装甲RNA在牡蛎中诺如病毒4种RNA提取方法比较研究中的应用*
2019-12-06江艳华李风铃逄凤娇王联珠翟毓秀
曲 梦 江艳华 李风铃 姚 琳 逄凤娇 王联珠 翟毓秀
装甲RNA在牡蛎中诺如病毒4种RNA提取方法比较研究中的应用*
曲 梦 江艳华 李风铃 姚 琳①逄凤娇 王联珠 翟毓秀
(中国水产科学研究院黄海水产研究所 农业农村部水产品质量安全检测与评价重点实验室 青岛 266071)
RNA提取是诺如病毒检测的关键步骤,而目前贝类中诺如病毒RNA提取、检测方法的比较与评价常囿于缺乏量值明确、无生物安全隐患的标准样品作为参考依据。本研究将前期制备的 GⅡ型诺如病毒装甲RNA (3.0×1010拷贝)作为标准样品,人工污染牡蛎()消化腺匀浆物,用4种常见RNA提取方法:TRIzol试剂、Viral RNA Kit、High Pure Viral Nucleic Acid Kit、柱式病毒RNAOUT试剂盒,分别提取RNA,经实时荧光RT-PCR检测后,利用标准曲线进行定量分析,分别计算4种方法对装甲RNA的回收率。结果显示,对于匀浆样本,TRIzol法对装甲RNA的回收率最高(6.80±0.89)%,显著高于Viral RNA Kit (4.51±2.28)%,二者的回收率又显著高于High Pure Viral Nucleic Acid Kit (0.24±0.05)%与柱式病毒RNAOUT试剂盒(0.11±0.02)% (˂0.05);对于冻干样本,Viral RNA Kit对装甲RNA的回收率最高(8.71±0.17)%,显著高于TRIzol试剂(7.12±0.64)%,二者的回收率又显著高于High Pure Viral Nucleic Acid Kit (0.33±0.12)%与柱式病毒RNAOUT试剂盒(0.06±0.01)% (˂0.05)。研究表明,TRIzol试剂与Viral RNA Kit对牡蛎消化腺样本中人工添加的装甲RNA均有良好的回收效果,同时也提示装甲RNA可作为一种良好的标准样品用于不同RNA提取试剂盒方法的评价与比较研究。
诺如病毒;装甲RNA;牡蛎;RNA提取方法;比较
人类各年龄段的非菌性胃肠炎中约有95%是由诺如病毒(Norovirus, NoVs)引起的(Ma, 2014)。我国是全球15个腹泻病高负担国家之一,NoVs在我国腹泻病例中的检出率在11.6%~59.52%之间(阎岩等, 2013; 余建兴等, 2015; 陈莉莉等, 2018),食物污染是NoVs传播的最主要途径,NoVs成为影响我国食品安全与公共卫生的重要病原。研究表明,多数NoVs疫情的暴发都与生食受污染牡蛎等贝类有关(Westrell, 2010)。牡蛎属于双壳滤食性生物,其内脏中存在NoVs受体及其合成酶(Le, 2006; 姜薇等, 2014; 姚琳等, 2016),牡蛎滤食海水中有机碎屑、微藻(任黎华等, 2013),极易将水体中污染的NoVs特异性富集于体内,因此被认为是引起NoVs食源性感染的高风险食品之一(Persson, 2018)。
实时荧光RT-PCR目前是NoVs检测的主要方法,而RNA提取是NoVs检测的基础,RNA的纯度、浓度及RT-PCR抑制物的多少对检测结果影响很大,如何提取高质量的RNA是检测的关键(Tan, 2009)。不同方法在RNA提取效率及对抑制物的去除效果方面有所差别,并最终影响检测结果(江涛等, 2017)。选择合适的提取方法,可从源头上最大程度避免NoVs检测结果的假阴性,确保检测结果的科学性和可靠性。由于NoVs无法按照传统方法进行体外培养,因此在比较或评价不同提取方法时,多数研究者用阳性NoVs腹泻样本或替代病毒—鼠诺如病毒添加到空白样本中(Uhrbrand, 2017; Kanwar, 2018)模拟实际样本,前者病毒含量不一且存在严重生物安全隐患,后者要经过繁琐的细胞培养与病毒含量测定。针对上述问题,近年来有研究开发出装甲RNA技术,即将检测靶标RNA包装在噬菌体病毒样颗粒内,一方面利用噬菌体衣壳蛋白保护易降解的RNA,另一方面病毒样颗粒高度模拟了病毒粒子结构,在提取、裂解等环节展示出与天然病毒的相似性(Pan, 2012),成为目前广泛应用的RNA病毒检测质控品。
本课题组前期建立了基于Qβ噬菌体的装甲RNA制备平台(张奇等, 2017),制备了多批内含NoVs检测靶标的装甲RNA。本研究将上述装甲RNA作为量值明确的标准样品添加到牡蛎内脏匀浆物中,以此模拟污染样本来比较TRIzol试剂等4种主要RNA提取方法对内脏匀浆物中装甲RNA的回收率,旨在筛选一种适合牡蛎中NoVs RNA提取的方法,同时探讨装甲RNA作为量值明确的标准样品用于方法比较研究的可行性。
1 材料与方法
1.1 试剂与仪器
RNA提取试剂TRIzol®购自Life公司;RNA 提取试剂盒Viral RNA Kit 购自OMEGA公司,High Pure Viral Nucleic Acid Kit购自Roche公司,柱式病毒RNAOUT购自北京TIANDZ基因科技有限公司;One Step PrimeScriptTMRT-PCR Kit购自宝生物工程(大连)有限公司;无水乙醇、异丙醇、氯仿等试剂均为分析纯。GⅡ型诺如病毒装甲RNA由本实验室制备、定量(3.0×108拷贝/μl)。参照GB 4789.42-2016《食品安全国家标准食品微生物学检验诺如病毒检验》,由生工生物工程(上海)股份有限公司合成检测GⅡ型NoVs所需的引物、探针,名称及序列见表1。实时荧光定量PCR仪为美国Roche公司LightCycler 2.0。
表1 引物、探针信息

Tab.1 Information of primers and probe
注:R=A/G;W=A/T;FAM=羧基荧光素;TAMARA=羧基四甲基罗丹明
Note: R=A/G; W=A/T; FAM=Carboxyfluorescein; TAMARA= Carboxytetramethylrhodamine
1.2 样品制备
将实验室收集的、经实时荧光RT-PCR检测GⅡ型诺如病毒呈阴性的牡蛎消化腺样品混合并匀浆,分装至48支15 ml无酶离心管中,每管2.0 g,分别添加3.0×1010拷贝的装甲RNA,充分混匀,室温放置1 h,制成牡蛎消化腺人工污染匀浆样品;从中随机取24管样品逐一称重后真空冷冻干燥,制成消化腺人工污染冻干样品,冻干后再次逐一称重,检测前根据每管失水的量加入相应重量的RNase-free H2O复水,涡旋混匀。
1.3 病毒RNA提取
按照说明书分别用TRIzol试剂、High Pure Viral Nucleic Acid Kit、柱式病毒RNAOUT、Viral RNA Kit提取1.2中制备的人工污染样品。每种样品提取5次。
1.4 实时荧光RT-PCR
用TaKaRa One Step PrimescriptTMRT-PCR kit 对提取的RNA进行实时荧光RT-PCR检测,反应体系和循环参数如下:2×one step RT-PCR Buffer Ⅲ 10 µl,TaKaRa ExHS(5 U/µl) 0.4 µl,PrimeScript RT EnzymeMix Ⅱ 0.4 µl, QNIF2(10 µmol/L)、COG2R(10 µmol/L)各0.3 µl, QNIFs(10 µmol/L)0.4 µl,RNA 2.0 µl,RNase- free H2O 6.2 µl;以RNase-free H2O为模板设置空白对照,以诺如病毒检测阴性且未添加装甲RNA的牡蛎内脏作为阴性对照。循环参数为42℃5 min,95℃ 10 sec;95℃ 5 sec,60℃ 20 sec,40个循环。每份样品的RNA平行测3次,记录t值。
1.5 回收率计算
利用本实验室前期建立的GⅡ型诺如病毒装甲RNA定量用标准曲线=-3.562+37.938 (张奇等, 2017),计算各样品中装甲RNA的拷贝数。根据不同的提取方法与样本类型,分别计算装甲RNA的回收率。计算公式:装甲RNA回收率=提取出的装甲RNA拷贝数/添加到样品中的装甲RNA拷贝数×100%。
1.6 统计学分析
将匀浆样品与冻干样品经4种不同提取方法提取RNA检测得到的t值和装甲RNA回收率采用SPSS 20.0进行单因素方差分析(One-way ANOVA),<0.05为差异显著。t值和回收率的分布用GraphPad Prism 6.0分析与绘图。
2 结果
2.1 4种提取方法所得RNA的Ct值分析
在牡蛎内脏匀浆物中添加3.0×1010拷贝GⅡ型诺如病毒装甲RNA,经4种方法提取后的RNA进行实时荧光RT-PCR检测,所得t值统计后见表2,t值分布经GraphPad Prism 6.0处理,见图1。对于匀浆样品,TRIzol法所提取的RNA得到的t值最小,其次是Viral RNA Kit,二者差异性显著(˂0.05),High Pure Viral Nucleic Acid Kit与柱式病毒RNAOUT试剂盒的t值较大,二者之间差异不显著,但与TRIzol法与Viral RNA Kit相比,差异显著(˂0.05)。对于冻干样品,TRIzol法与Viral RNA Kit的t值均很小,二者差异性不显著(˃0.05),High Pure Viral Nucleic Acid Kit与柱式病毒RNAOUT试剂盒的t值仍较大,二者之间差异不显著,但与TRIzol法与Viral RNA Kit相比,差异显著(˂0.05)。
表2 实时荧光RT-PCR分析4种提取方法所得RNA的t值(X±SD)

Tab.2 Ct values of RNA obtained from 4 extraction methods analyzed with realtime RT-PCR (X±SD)
注:同列数据中相同上标字母表示差异不显著(> 0.05),下同
Note: Values in the same column with same superscrips are not significantly different (>0.05), the same as below
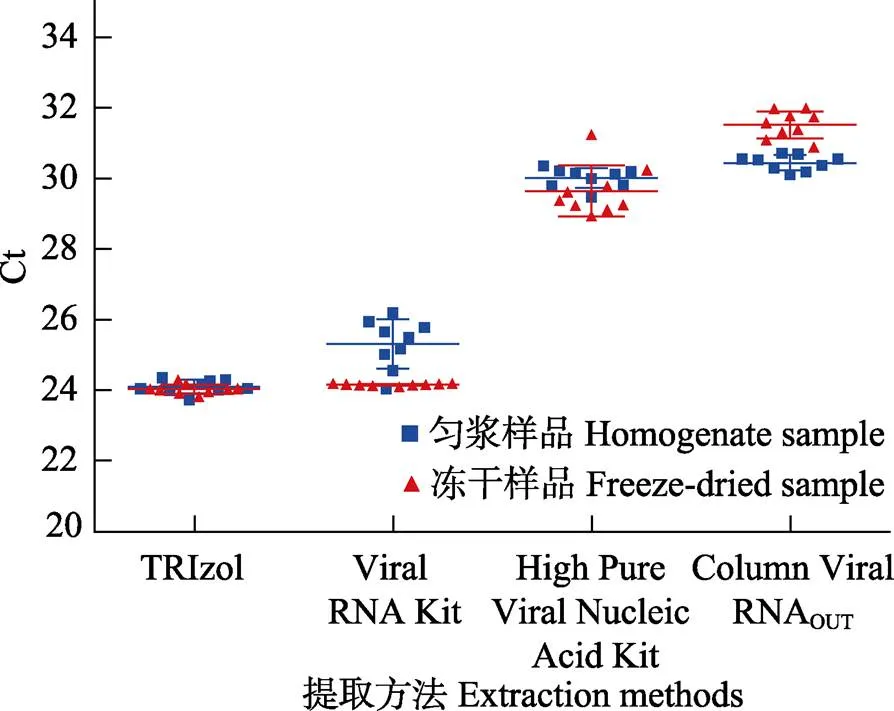
图1 4种RNA提取方法对应Ct值的分布
2.2 4种RNA提取方法对装甲RNA回收率分析
将4种RNA提取方法所得t值代入标准曲线计算出2.0 μl RNA所含的装甲RNA拷贝数,再根据不同试剂盒的提取流程,最终计算出4种提取方法对添加的装甲RNA的回收率,结果见表3。所得装甲RNA的回收率经GraphPad Prism 6.0分析后,结果见图2。对于匀浆样品,TRIzol法的回收率最高,其次是Viral RNA Kit,二者差异性显著(˂0.05),但均能达到GB 4789.42-2016中关于回收率应大于1%的要求,High Pure Viral Nucleic Acid Kit与柱式病毒RNAOUT试剂盒的回收率均很低,二者之间差异不显著,但与TRIzol法与Viral RNA Kit相比,差异显著(˂0.05)。对于冻干样品,Viral RNA Kit的回收率最高,其次是TRIzol法均超过2%,二者差异性不显著(˃0.05),High Pure Viral Nucleic Acid Kit与柱式病毒RNAOUT试剂盒的回收率很低,二者之间差异不显著,但与Viral RNA Kit与TRIzol法相比,差异显著(˂0.05)。
表3 4种RNA提取方法对装甲RNA的回收率(X±SD,=5, %)
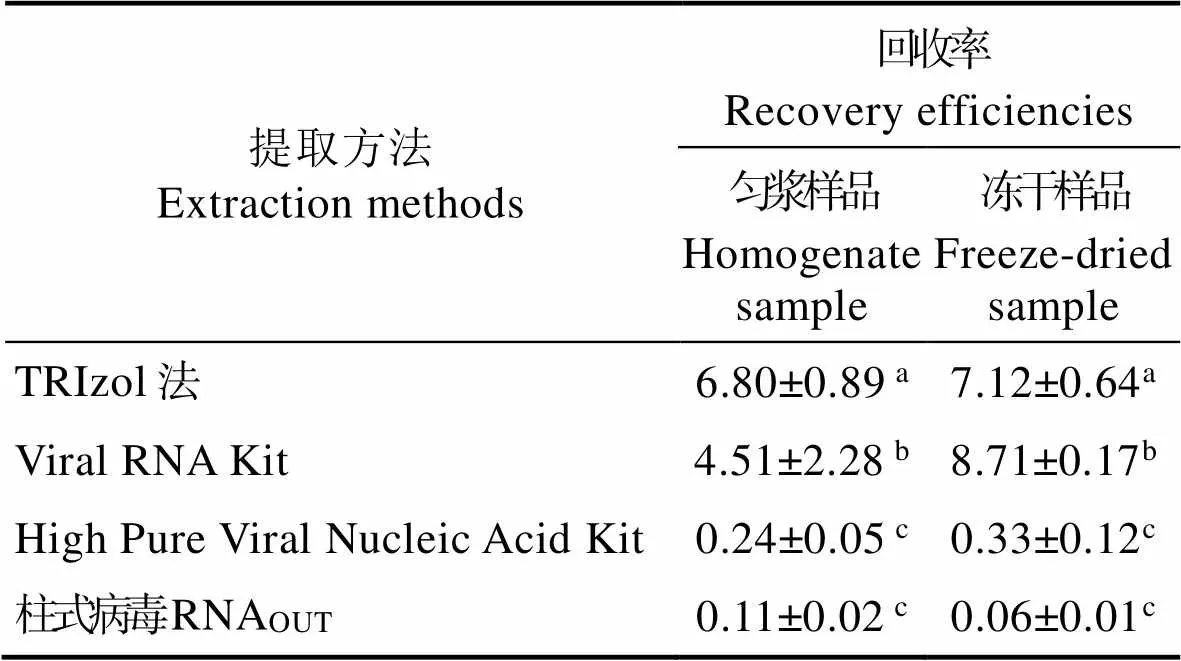
Tab.3 Recovery of 4 RNA extraction methods for armored RNA (X±SD, n=5, %)

图2 4种RNA提取方法对装甲RNA的回收率
3 讨论
NoVs是世界范围内的一种重要食源性病毒,是引起人类急性胃肠炎的主要病原。牡蛎因其自身生物学特性成为NoVs传播的重要载体之一,实时荧光RT- PCR目前是检测NoVs的“金标准”,然而受污染牡蛎自身存在大量RT-PCR抑制物、NoVs含量低、基因分型多等因素影响,其检测难度高于常见RNA病毒,提高检测方法的灵敏度和病毒回收率是各国学者一直关注的问题(Francoise, 2009)。部分提取方法对目标RNA的提取效率低、对样本中存在的RT-PCR抑制物去除效果不好、扩增前RNA出现降解等问题往往造成检测结果的假阴性(Das, 2011)。因此,比较病毒RNA提取方法成为优化、筛选NoVs检测方法的重点之一。虽然NoVs目前已可以基于干细胞转化出的肠上皮细胞进行人工培养(Ettayebi, 2016),但对实验条件与经验有严格的要求,短期内很难在大多从事应用性基础研究的实验室开展,因此在提取方法比较研究中,多数学者仍使用含病毒的临床腹泻样本和鼠诺如病毒等作为质控品添加到阴性样品中,但存在难以获得、量值不均一、生物安全隐患等不足。
本实验室前期基于Qβ噬菌体制备平台,成功制备了内含GⅡ型NoVs检测靶标的装甲RNA,证实其均匀性和稳定性良好,拷贝数高,可实现对NoVs核酸检测中的RNA提取、反转录、扩增等过程的定性、定量质量控制和评估(Villanova, 2007; 张奇, 2017)。由于牡蛎中的NoVs主要集中在消化腺,因此,本研究将牡蛎消化腺匀浆物与NoVs装甲RNA混匀后室温孵育,该人工污染的方法与用活牡蛎自然富集诺如病毒相比,简单有效,并且具有量值明确便于定量评估、可批量制备重复实验等多种优点。将人工污染后的样品分别以TRIzol法、High Pure Viral Nucleic Acid Kit、柱式病毒RNAOUT、Viral RNA Kit 4种提取方法提取病毒RNA,依据本实验室建立的GⅡ型诺如病毒标准曲线分析了不同提取方法对不同状态样品中装甲RNA的回收率,研究结果表明,无论是匀浆样品还是冻干样品,本研究涉及的4种RNA提取方法的回收效率相差较大。
有研究分别用磁珠法和TRIzol法提取血清样品中丙型肝炎病毒RNA,发现磁珠法提取的RNA更适合于血清样品中丙型肝炎病毒的检测(范公忍等, 2015)。Zhang等(2015)比较了TRIzol、异硫氰酸胍和二氧化硅纳米颗粒对丙型肝炎病毒RNA的提取效率,发现二氧化硅纳米颗粒的提取效率最高。上述研究结果中,TRIzol法并没有显示出明显的优势,可能是这些研究中的血清样品与本研究中的消化腺样品基质的差异,导致了本研究与上述研究结果的不同。
以二氧化硅或其衍生材料为基础的固相吸附技术是目前大多柱式提取试剂盒的核心技术,可以特异性地吸附RNA,样本中的蛋白质、脂质等其他组分可通过高盐缓冲液去除,然后用低盐缓冲液从二氧化硅材料中洗脱RNA,能较为有效地去除抑制物(Zhang, 2015; Xu, 2017),但在提取RNA的总量上与传统的TRIzol法、异硫氰酸胍方法相比,相差较大。不同试剂盒离心柱的二氧化硅材料及尺寸(小体积或大体积)等因素的差异或许是导致试剂盒提取效率不同的原因之一。此外,High Pure Viral Nucleic Acid Kit与柱式病毒RNAOUT试剂盒可以破坏较为分散的细胞并高效地暴露RNA,可能更适合血液、血浆、细胞培养物等样品;另外,High Pure Viral Nucleic Acid Kit使用过程中需要添加Poly(A),可特异性结合病毒基因组RNA,以增强后期纯化柱对病毒基因组的吸附作用,然而牡蛎样品盐分高,多糖等抑制物较多,很可能弱化了这一步骤的实际效果,从而最终导致该试剂盒对牡蛎消化腺样本中添加的装甲RNA回收率较低。与这2种试剂盒相比,Viral RNA Kit的裂解液等避免使用抑制RT-PCR的试剂,在操作上要求不高,处理步骤少,减少过度操作,RNA降解程度低,RT-PCR抑制物残留量少,该试剂盒较适用于牡蛎组织样品。
此外,考虑到保存期等因素,目前生物基质标准样品大多数为冻干样品,为了探讨样品冷冻干燥后,是否会影响RNA的提取,本研究还分析了4种方法对冻干样品的提取效果,TRIzol法与Viral RNA Kit的回收率与匀浆样品相比无明显差异,一方面表明这2种方法对样本状态适用性的多样化,另一方面,与新鲜样品相比,冻干样品脱水彻底,可在常温条件下长时间保存,已有研究表明,冻干可延长单纯装甲RNA稳定性(Stevenson, 2008),本研究进一步表明,在牡蛎内脏这样高盐、富含多糖的复杂基质样本中,冻干对装甲RNA的回收、检测等没有影响,从而为今后研制诺如病毒装甲RNA牡蛎内脏基质冻干标准样品提供了研究基础。
Chen LL, Chen J, Liao NB. Epidemiological characteristics of Norovirus infectious diarrhea in Zhejiang Province from 2014 to 2016. Chinese Journal of Health Laboratory Technology. 2018, 28(7): 863–866 [陈莉莉, 陈江, 廖宁波. 2014年-2016年浙江省诺如病毒感染性腹泻流行病学特征分析. 中国卫生检验杂志, 2018, 28(7): 863–866]
Das A, Beckham TR, McIntosh MT. Comparison of methods for improved RNA extraction from blood for early detection of Classical swine fever virus by real-time reverse transcription polymerase chain reaction. Journal of Veterinary Diagnostic Investigation, 2011, 23(4): 727–735
Ettayebi K, Crawford SE, Murakami K,. Replication of human noroviruses in stem cell-derived human enteroids. Science, 2016, 353(6306): 1387–1393
Francoise SG, Parnaudeau S, Schaeffer J,. Detection and quantitation of noroviruses in shellfish. Applied and Environmental Microbiology, 2009, 75(3): 618–624
Fan GR, Chen TB, Li B,. Comparison of effects between two kinds of nucleic acid extraction method for detecting HCV RNA and their application. Laboratory Medicine and Clinic, 2015, 12(1): 48–50 [范公忍, 陈天宝,李冰, 等. 两种核酸提取方法对丙型肝炎病毒RNA检测效果的比较及应用评价. 检验医学与临床, 2015, 12(1): 48–50]
Jiang W, Yao L, Jiang YH,. Molecular cloning and expressionof FUT2-like gene in the Oyster (). Progress in Fishery Sciences, 2014, 35(5): 70–75 [姜薇, 姚琳, 江艳华, 等. 太平洋牡蛎()类FUT2基因的克隆与组织表达. 渔业科学进展, 2014, 35(5): 70–75]
Jiang T, Han CH, Zhang HY,The detection of Noroviruses in oysters sold in Beijing by using Taqman-based one-step reverse transcription-polymerase chain reaction assays and quantitative analysis. Chinese Journal of Food Hygiene, 2017, 29(2): 126–130 [江涛, 韩春卉, 张宏元, 等. 北京市市售牡蛎中诺如病毒核酸检测及定量分析. 中国食品卫生杂志, 2017, 29(2): 126–130]
Ma Y, Duan Y, Wei Y,. Heat shock protein 70 enhances mucosal immunity against human norovirus when coexpressed from a vesicular stomatitis virus vector. Journal of Virology, 2014, 88(9): 5122–5137
Kanwar N, Hassan F, Barclay L,. Evaluation of RIDA®; GENE norovirus GI/GII real time RT-PCR using stool specimens collected from children and adults with acute gastroenteritis. Journal of Clinical Virology, 2018, 104:1–4
Le Guyader F, Loisy F, Atmar RL,. Norwalk virus-specific binding to oyster digestive tissues. Emerging Infectious Diseases, 2006, 12(6): 931–936
Pan Y, Zhang Y, Jia T,. Development of a microRNA delivery system based on bacteriophage MS2 virus-like particles. FEBS Journal, 2012, 279(7): 1198–1208
Persson S, Eriksson R, Lowther J,. Comparison between RT droplet digital PCR and RT real-time PCR for quantification of noroviruses in oysters. International Journal of Food Microbiology, 2018, 284: 73–83
Ren LH, Zhang JH, Fang JG,. A study on the daily rhythm of respiration, excretion and calcification in oysters. Progress in Fishery Sciences, 2013, 34 (1): 75–81 [任黎华, 张继红, 方建光, 等. 长牡蛎呼吸、排泄及钙化的日节律研究. 渔业科学进展, 2013, 34(1): 75–81]
Stevenson J, Hymas W, Hillyard D. The use of Armored RNA as a multi-purpose internal control for RT-PCR. Journal of Virological Methods, 2008, 150(1–2): 73–76
Tan SC, Chin YB. DNA, RNA, and protein extraction: The past and the present. Journal of Biomedicine and Biotechnology, 2009, 2009(3): 1–10
Uhrbrand K, Koponen IK, Schultz AC,. Evaluation of air samplers and filter materials for collection and recovery of airborne norovirus. Journal of Applied Microbiology, 2017, 124(4): 990–1000
Villanova GV, Gardiol D, Taborda MA,. Strategic approach to produce low-cost, efficient, and stable competitive internal controls for detection of RNA viruses by use of reverse transcription-PCR. Journal of Clinical Microbiology, 2007, 45(11): 3555
Westrell T, Dusch V, Ethelberg S,. Norovirus out breaks linked to oyster consumption in the United Kingdom, Norway, France, Sweden and Denmark, 2010. Euro Surveillance, 2010, 15(12): 1–4
Xu R, Shieh YC, Stewart DS. Comparison of RNA extraction kits for the purification and detection of an enteric virus surrogate on green onions via RT-PCR methods, Journal of Virological 2017, 239: 61–68
Yan Y, Wu Y, Guo J,. Sentinel surveillance and gene analysis of acute gastroenteritis norovirus in Guizhou province in 2011. Chinese Journal of Virology, 2013 (1): 51–55 [阎岩, 吴悦, 郭军,等. 贵州省2011年急性胃肠炎诺如病毒的哨点监测及其基因特征分析. 病毒学报, 2013(1): 51–55]
Yao L, Jiang YH, Li FL,. Codon Optimization and prokaryotic expression of α-1 and 2-fucosyltransferase in Oyster (). Progress in Fishery Sciences, 2016, 37(1): 74–79 [姚琳, 江艳华, 李风铃,等. 太平洋牡蛎()类α-1,2-岩藻糖基转移酶的密码子优化与原核表达. 渔业科学进展, 2016, 37(1): 74–79]
Yu JX, Lai SJ, Wang X,. Analysis of epidemic characteristics of Norovirus from 2009 to 2013 in 27 provinces (municipalities and autonomous regions) of China. Chinese Journal of Epidemiology, 2015, 36(3): 199–204 [余建兴, 赖圣杰, 王鑫, 等. 中国27省(市、自治区) 2009–2013年门诊腹泻病例诺如病毒流行特征分析. 中华流行病学杂志, 2015, 36(3): 199–204]
Zhang Q, Yao L, Jiang YH,. Development of Armored RNA reference material of Norovirus based on Qbeta bacteriophage. China Biotechnology, 2018, 38(1): 42–50 [张奇, 姚琳, 江艳华, 等. 基于Qbeta噬菌体装甲RNA技术的诺如病毒RNA标准参考样品的研制. 中国生物工程杂志, 2018, 38(1): 42–50]
Zhang B, Chen K, Ni E. Comparison of three methods for extraction of HCV RNA in sera collected from individuals with hyperlipidemia, hyperbilirubinemia and hyperglobulinemia. Journal of Virological Methods, 2015, 212: 44–46
Application of Armored RNA to Compare Four Norovirus RNA Extraction Methods in Oysters
QU Meng, JIANG Yanhua, LI Fengling, YAO Lin①, PANG Fengjiao, WANG Lianzhu, ZHAI Yuxiu
(Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Key Laboratory of Testing and Evaluation for Aquatic Product Safety and Quality, Ministry of Agriculture and Rural Affairs, Qingdao 266071)
Norovirus (NoVs) is the most prevalent worldwide foodborne pathogen and causes acute viral gastroenteritis. NoVs are transmitted mainly via the fecal–oral route and by person-to-person contact. It is thought that the majority of NoVs infections are caused by the consumption of contaminated food; the ingestion of contaminated oysters is the primary cause of foodborne NoVs infection since oyster digestive diverticula accumulate viral particles from seawater via filter feeding. Real time RT-PCR is commonly used to detect NoVs RNA in oysters; however, these assays are often hampered by the low viral titer in oysters and PCR inhibition due to matrix carryover during RNA extraction. Extraction is a critical step for obtaining sufficient high-quality viral RNA for amplification; however, studies comparing and evaluating NoVs RNA extraction and detection methods in shellfish are often limited by the lack of standard samples with clear quantitative values and a lack of biosafety hazards. In this study, four RNA extraction methods (TRIzol reagent, Viral RNA Kit, High Pure Viral Nucleic Acid Kit, and Column Virus RNAOUTKit) were used on oyster digestive gland homogenate samples and artificial freeze-dried samples contaminated with NoVs armored RNA (3.0´1010copies/sample) as a reference material. RNA extracted by the four methods was analyzed by real time RT-PCR and quantified using previously established standard curves. For the homogenized samples, the TRIzol method had the highest recovery rate (6.8± 0.89)% and was significantly higher than that by the Viral RNA Kit (4.51±2.28)%. The recovery rates of these two methods were both significantly higher than those by the High Pure Viral Nucleic Acid Kit (0.24±0.05)% and Column Virus RNAOUTkit [(0.11±0.02)%,˂0.05]. For the freeze-dried samples, the Viral RNA Kit had the highest recovery rate (8.71±0.17)% and was significantly higher than that by the TRIzol method (7.12±0.64)%. The recovery rates of these two methods were both significantly higher than those of the High Pure Viral Nucleic Acid Kit (0.33±0.12)% and Column Virus RNAOUTkit [(0.06± 0.01)%,˂0.05]. This study indicated that the TRIzol method and Viral RNA Kit could extract target RNA from oyster digestive gland homogenate samples with an ideal recovery rate; moreover, armored RNA could serve as a good reference material for comparing RNA extraction methods.
Norovirus; Armored RNA; Oyster; RNA extraction method; Comparison
Q33
A
2095-9869(2019)06-0180-06
10.19663/j.issn2095-9869.20180907002
http://www.yykxjz.cn/
曲梦, 江艳华, 李风铃, 姚琳, 逄凤娇, 王联珠, 翟毓秀. 装甲RNA在牡蛎中诺如病毒4种RNA提取方法比较研究中的应用. 渔业科学进展, 2019, 40(6): 180–185
Qu M, Jiang YH, Li FL, Yao L, Pang FJ, Wang LZ, Zhai YX. Application of armored RNA to compare four norovirus RNA extraction methods in oysters. Progress in Fishery Sciences, 2019, 40(6): 180–185
* 国家重点研发计划(2017YFC1600703)、科技部科技基础性工作专项(2013FY113300)和现代农业产业技术体系 (CARS-47)共同资助[This work was supported by National Key Research and Development Program of China (2017YFC1600703); Special Program for Science and Technology Basic Research of the Ministry of Science and Technology China (2013FY113300); Central Public-interest Scientific Institution Basal Research Found, CAFS (2016HY-ZD11);China Aquaculture Research System (CARS-47)]. 曲 梦, E-mail: 1874213448@qq.com
姚 琳,副研究员,E-mail: yaolin@ysfri.ac.cn
2018-09-07,
2018-10-31
YAO Lin, E-mail: yaolin@ysfri.ac.cn
(编辑 江润林)
