魁蚶Ets家族9个基因的克隆及其在病毒感染应答中的表达分析*
2019-12-06魏智薪辛鲁生白昌明李亚楠张淑敏李成华王崇明
魏智薪 辛鲁生 白昌明 李亚楠 张淑敏 李成华 王崇明
魁蚶Ets家族9个基因的克隆及其在病毒感染应答中的表达分析*
魏智薪1,3辛鲁生2,3白昌明2,3李亚楠3张淑敏3李成华1王崇明2,3①
(1. 宁波大学海洋学院 宁波 315211;2. 青岛海洋科学与技术试点国家实验室海洋渔业科学与食物产出过程功能实验室 青岛 266071;3. 中国水产科学研究院黄海水产研究所 农业农村部海水养殖病害防治重点实验室 青岛市海水养殖流行病学与生物安保重点实验室 青岛 266071)
Ets蛋白是宿主MAPK信号通路下游的一类可参与调控病毒基因转录复制的重要转录因子。本研究通过基因克隆成功获得魁蚶() Ets家族9条基因(分别命名为),开放阅读框(ORF)大小分别为1065、1290、1569、912、1344、1404、1521、1968和1191 bp,并分别编码354、429、522、303、447、468、506、655和396个氨基酸。系统进化树分类表明,本研究所获得的基因均属Ets家族。对ETS-1和ETS-3的氨基酸序列和三维结构分析表明,其均含有高度保守的ETS结构域。在不同水温条件下,通过人工注射牡蛎疱疹病毒(OsHV-1)对魁蚶进行感染,并对病毒拷贝数和的相对表达量进行定量分析。结果显示,和只在高温阳性组中相对表达量显著上调,与病毒拷贝数在高温条件下增长趋势呈正相关;和只在低温阳性组中相对表达量显著上调,但在高温阳性组中和的相对表达量与病毒拷贝数呈负相关;初步研究结果显示,从魁蚶基因中筛选出2条基因(和),在高温条件下(16±2)℃,其可能参与正向调控病毒OsHV-1的复制过程。本研究为进一步探索魁蚶在夏季因感染牡蛎疱疹病毒(OsHV-1)而导致的大量死亡提供了科学数据。
魁蚶;Ets家族;OsHV-1;克隆;温度
魁蚶()属瓣鳃纲(Lamelli- branchia)、翼形亚纲(Pteriomorphia)、蚶目(Arcoida)、蚶科(Arcidae)(刘寒苗等, 2017; 李瑶瑶等, 2018),是一种底栖冷水性大型贝类,主要分布于中国黄渤海、日本海、朝鲜半岛及俄罗斯东南部等沿海地区(毛雪英等, 2007; 张启刚等, 2007);在近海岸水深3 m到外海近60 m处的泥沙质或泥质海底均有分布(梁超等, 2010)。其营养价值极高、口感鲜美,深受消费者喜爱,成为中国对外出口创汇的重要的经济贝类之一(刘锡胤等, 2002)。
持续扩大的市场需求和有限的养殖环境使贝类养殖业向高密度养殖发展,随之而来的则是大规模的病害暴发,在高温季节,病害问题尤为严重,严重制约着海水贝类养殖业的可持续发展。贝类的病原(Elston, 1997; Hine, 2000)可分为细菌、病毒和寄生虫类等,其中,病毒类病原以致死率高、感染速度快等特征引起广泛关注,尤以牡蛎疱疹病毒OsHV-1 (Ostreid herpesvirus 1)的危害最为严重(胡宗福等, 2017; Roque, 2012; Webb, 2007; Barbosa- Solomieu, 2005)。自20世纪90年代以来,该疫病就在法国和美国牡蛎养殖海域连年暴发,随后蔓延至欧、美、亚等牡蛎养殖场,造成了巨大的经济损失(Renault, 2001; Segarra, 2010)。在中国,OsHV-1已造成了扇贝(Ren, 2013)和魁蚶的大规模死亡(Xia, 2015)。
病毒复制过程依赖宿主细胞,包括必需的能量和功能蛋白元件等均需要宿主细胞供给(Ruelas, 2013)。OsHV-1属于双链DNA病毒,需在宿主细胞核内进行转录复制(Jouaux, 2013; 胡宗福等, 2017)。Ets(E-Twenty-Six)转录因子家族是含有ETS结构域的一类转录因子,是宿主细胞核中一类可参与调控病毒基因转录复制的重要转录因子。Ets转录因子在MAP激酶、Ca2+依赖的信号通路以及TGF-β等信号通路的调节下,参与调控胚胎的发育、细胞的生长、分化和凋亡,进而调控许多生理和病理过程(曾红等, 2017; Suico, 2017)。Pleschka等(2001)研究表明,流感病毒的传播受到RAF/MEK/ERK信号级联的抑制。Bosselut等(1990)对HTLV-1的研究发现,人的Ets1和Ets2蛋白参与调控HTLV-1病毒的转录复制过程。本研究基于Ets的转录调控功能和OsHV-1中存在Ets结合位点,初步推断Ets转录因子可能参与OsHV-1的复制调控过程。本研究采用基因克隆和实时荧光定量PCR技术克隆魁蚶Ets转录因子,研究其在感染OsHV-1后的表达变化,以及进一步对Ets转录因子相对表达量变化与病毒拷贝数变化进行相关性分析。以期为OsHV-1感染魁蚶的致病机制提供科学依据。
1 材料与方法
1.1 实验材料
实验所用魁蚶捕捞自渤海海域。随机选取2龄魁蚶[壳长为(6.0±1.0) cm,壳高为(5.0±1.5) cm,体重为(90±10) g)],实验室室温暂养。实验用魁蚶暂养分为(16±2)℃的高温暂养和(10±2)℃的低温暂养,暂养时间为1周。暂养期间,每天更换1次海水,每次更换1/2体积;魁蚶日常投喂适量小球藻(),并始终充氧。魁蚶病料为自然感染OsHV-1个体,‒80℃保存(用于制备病毒悬液)。
1.2 实验方法
1.2.1 总RNA提取和cDNA合成 采用Trizol (TaKaRa)法进行样本总RNA提取。琼脂糖凝胶电泳检测RNA提取质量。使用NanoDrop ND-2000分光光度计测量其浓度。以提取的总RNA为模板,采用反转录试剂盒ReverTra Ace qPCR RT Master Mix with gDNA Remove(TOYOBO)合成第一链cDNA,实验方法按照说明书进行。‒80℃保存cDNA备用。
1.2.2 魁蚶Ets家族基因开放阅读框(ORF)的克隆及测序 根据本实验室的魁蚶转录组测序获得Ets家族的部分基因的全序列,使用Primer Premier 5.0软件设计ORF引物(表1),并利用KOD酶进行扩增。采用扩增体系(20 μl):cDNA模板1 μl、KOD酶0.4 μl、前引物0.5 μl、后引物0.5 μl、2×KOD酶缓冲液10 μl、超纯水3.6 μl、2 mmol/L dNTP 4 μl;反应程序:94℃预变性 4 min;94℃变性 30 s,58.0℃退火 30 s,68℃延伸2.0 min,35个循环。扩增产物经1.5%琼脂糖凝胶电泳检测后,利用琼脂糖凝胶回收试剂盒(TaKaRa)将目的条带进行切胶回收。随后,将回收的目的片段与pMD19-T载体(3∶1)在16℃连接过夜,取5 μl连接产物与50 μl大肠杆菌()DH5α感受态细胞在42℃热激30 s。利用载体多克隆位点两侧的引物RV-M(GAGCGGATAACAATTTCACACAG)和M13-47(CGCCAGGGTTTTCCCAGTCACGAC)对单克隆进行菌落PCR鉴定。阳性单克隆经验定后送生派生诺(青岛)股份有限公司进行测序。
1.2.3 魁蚶Ets家族基因的生物信息学分析 将测序结果正确的目的基因进行BLAST(https://www. ncbi.nlm.nih.gov/)同源性比对,利用在线网址(http:// www.expasy.org/)将基因的cDNA序列推导成氨基酸序列;使用MEGA7.0进行进化树分析;利用生物在线网站(https://zhanglab.ccmb.med.umich. edu/cgi-)bin/itasser_submit.cgi)对、编码的蛋白质进行3D结构、ETS结构域及酶催化位点进行预测。
1.2.4 OsHV-1病毒悬液的制备及拷贝数检测 将本课题组‒80℃保存的自然感染OsHV-1的魁蚶放置冰上解冻,随后取外套膜组织1~2 g,将样本润洗2~ 3次。无菌剪刀剪碎,按1∶9(g∶ml)加入无菌海水(0.22 μm过滤),50 ml无菌管组织匀浆(5~10 s/3次)。低速离心去除杂质:1000 g,4℃,5 min。取上清液 4倍稀释,过滤滤膜(5 μm–2 μm–0.45 μm–0.22 μm),4℃保存。按Roche High Pure Viral Nucleic Acid Kit试剂盒说明书提取病毒悬液DNA。将提取后产物的病毒拷贝数进行实时荧光定量检测。检测体系(20 μl):2× Mix 10 μl、BF引物0.8 μl、B4引物 0.8 μl、探针0.4 μl、超纯水0.4 μl;反应程序:95℃ 10 min;95℃ 10 s, 60℃ 15 s,72℃ 15 s,35个循环。使用的探针序列:BP6FAM-ATCGGGGGGGGGGGTTTTTTTTTTATBHQ-1;使用的引物序列为BF-GTCGCATCTTTGGA- TTTAACAA;B4-ACTGGGATCCGACTGACAAC。定量后的OsHV-病毒悬液用于后续实验。
表1 本实验用到的引物序列
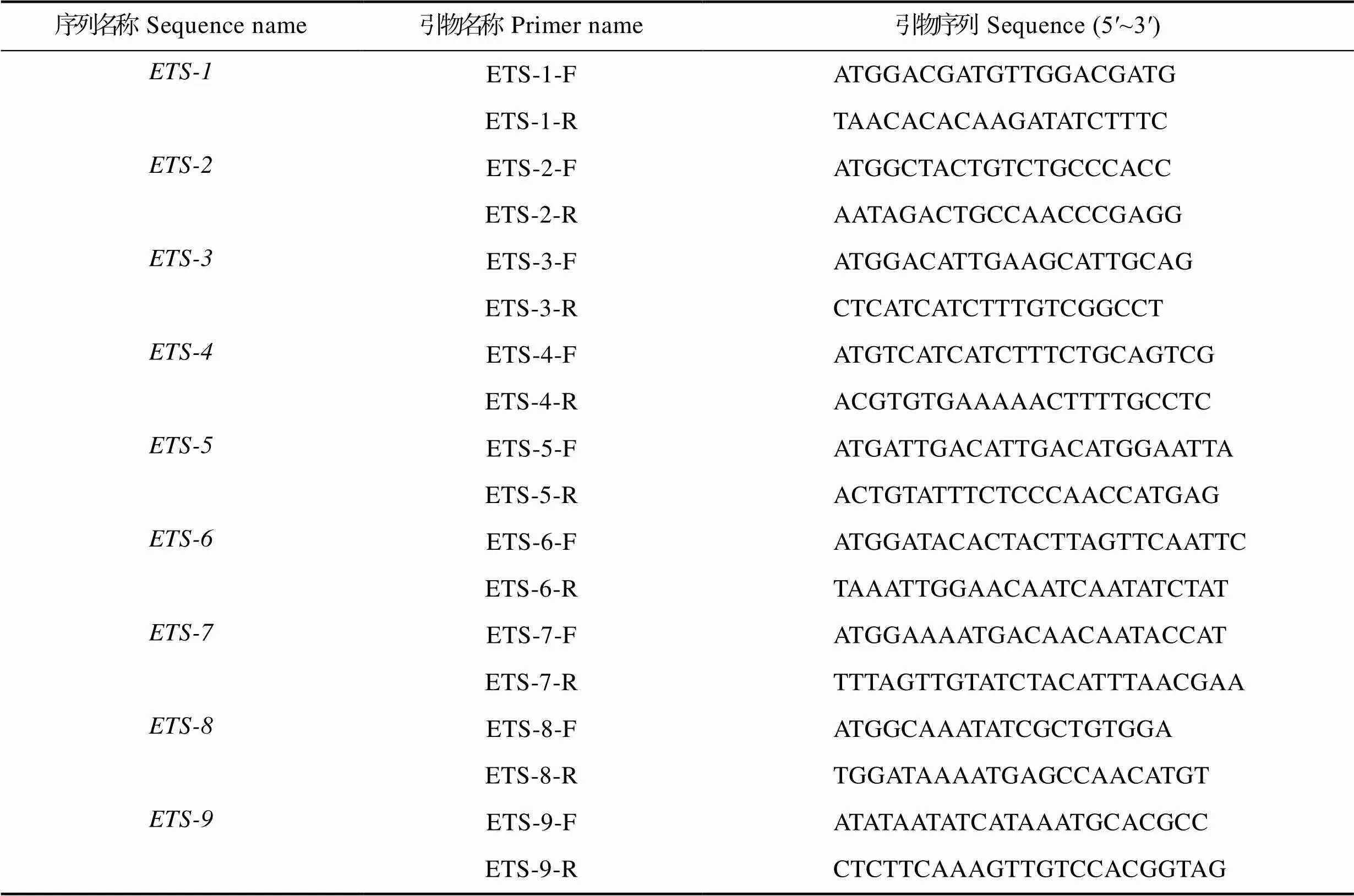
Tab.1 Sequences of primers used in this study
1.2.5 OsHV-1感染魁蚶实验 从实验室暂养的魁蚶中选取80只,随机分为4组:(16±2)℃高温阴性组与高温阳性组,(10±2)℃低温阴性组与低温阳性组,每组分别为20只。高温阳性组和低温阳性组通过腹足注射的方式每只魁蚶注射100 μl浓度约为105copies/μl的病毒悬液。高温阴性组和低温阴性组则注射100 μl健康魁蚶组织悬液。注射完成后,将魁蚶静置10 min,随后放入实验箱内。分别在注射后的0、6、12、24、48和72 h进行组织样品采集。随机从各组中选取3只魁蚶,采集血淋巴样品:用一次性注射器采集魁蚶血液,混匀后分装到1.5 ml EP管中,每管1 ml,4℃800 g离心5 min后,除去血清,并加入800 μl Trizol (TaKaRa),混匀后‒80℃保存。
1.2.6 魁蚶感染OsHV-1病毒后的表达研究 按照Trizol法提取血细胞总RNA,对提取的总RNA进行质量和浓度检测后,以总RNA为模板,用反转录试剂盒(Prime ScriptTMcDNA Synthesis Kit, TaKaRa)合成模板,操作方法按照说明书进行。以魁蚶RL15作为内参基因(Xin, 2018),使用Primer 5.0设计定量引物(表2),对进行qPCR检测。检测使用Universal qPCR Master Mix试剂盒(NEB),反应体系(20 μl):SYBR®Premix ExⅡ10 μl、前引物1 μl、后引物1 μl、模板2 μl、超纯水6 μl;反应程序:95℃ 5 min;95℃ 15 s,60℃ 20 s,72℃ 20 s,40个循环。每组实验均实验重复3次。2‒ΔΔCt方法对荧光定量PCR检测结果进行分析,利用SPSS 20.0对结果进行显著性分析。
表2基因定量引物

Tab.2 Quantitative primers of Ets genes
2 结果与分析
2.1 魁蚶Ets家族基因的cDNA开放阅读框(ORF)的克隆
从本课题组魁蚶转录组数据库中获得的9条序列片段,成功克隆获得9条基因的完整的ORF序列,从到对其进行分别命名。这9条基因的开放阅读框(ORF)大小分别为1065、1290、1569、912、1344、1404、1521、1968和1191 bp,并分别编码354、429、522、303、447、468、506、655和396个氨基酸(图1)。
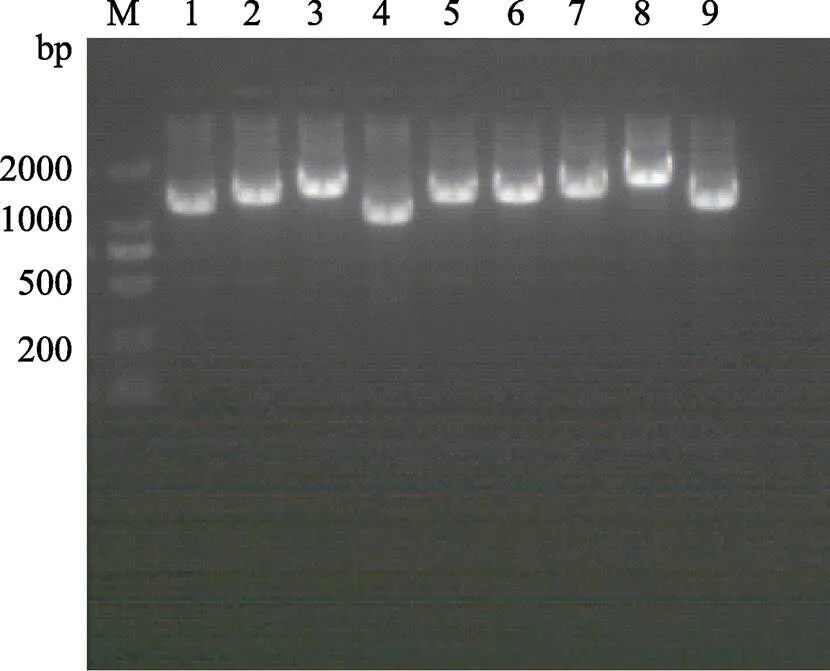
图1 魁蚶Ets家族基因克隆
M:Marker;1~9:~
2.2 Ets家族进化树分析
通过NCBI在线获取其他物种Ets蛋白序列并与获得的魁蚶Ets家族基因进行多序列比对,利用Mega 7.0软件建立系统进化树。结果显示,魁蚶Ets高度保守,与虾夷扇贝()的转录因子(OWF55447.1)进化关系较近,与丘疹旋毛虫的转录因子(KRZ74488.1)关系较远;与虾夷扇贝的转录因子(XP_021347939.1)进化关系较近;与虾夷扇贝的转录因子(XP_021366637.1)进化关系较近,与卡罗莱纳箱龟()的转录因子(XP_024065379.1)关系较远;与长牡蛎()转录因子(XP_ 019928533.1)进化关系较近;与长牡蛎的转录因子(XP_019917945.1)进化关系较近;与虾夷扇贝的转录因子(XP_021352365.1)进化关系较近;与虾夷扇贝的转录因子(XP_021360845.1)进化关系较近;与长牡蛎的转录因子(EKC21903.1)进化关系较近;与虾夷扇贝的(XP_ 021365446.1)进化关系较近(图2)。
2.3 OsHV-1感染后的病毒拷贝数检测
利用实时荧光定量PCR检测OsHV-1感染魁蚶后在不同时间段的病毒拷贝数的变化。结果显示,在低温阴性组和低温阳性组中,魁蚶均能够正常生长,OsHV-1拷贝数变化均在0~100 copies/ng范围内,属于本底水平,病毒没有增殖;在高温组中,高温阴性组正常生长,OsHV-1拷贝数变化在0~100 copies/ng范围内,属于本底水平,病毒没有增殖,但在高温阳性组中,病毒拷贝数随感染时间的延长,病毒拷贝逐步上升,在0~6 h,病毒拷贝数处于本底水平,在6~ 12 h,病毒拷贝数增长至1.0×103copies/ng,在12~24 h,拷贝数增长至约6.0×104copies/ng,在24~48 h,病毒拷贝数呈现爆炸式增长,约为3.4×106copies/ng,并持续增长,在72 h,病毒拷贝数达到最大值(7.4× 106copies/ng),此时魁蚶均已死亡(图6)。
2.4 魁蚶感染OsHV-1病毒后Ets基因的表达分析
利用实时荧光定量PCR技术对OsHV-1感染魁蚶后基因在不同时间点的表达情况分析。结果显示,在高温阳性组中,和的相对表达量显著上调(<0.05),其中,在12、24、48和72 h分别比高温阴性组上调3.9、2.8、7.4和15.6倍;低温阴性组与低温阳性组相对表达量无显著差异;低温阴性组与高温阴性组相对表达量无显著差异(图7A)。在高温阳性组中,在48 h后相对表达量显著上调,48和72 h分别比高温阴性组上调3.0和22.6倍;低温阴性组、低温阳性组和高温阴性组相对表达量无显著差异(图7C)。在低温阳性组中,和相对表达量显著上调(<0.05),其中,在24、48和72 h分别比低温阴性组上调4.3、7.0和8.2倍;高温阳性组、高温阴性组和低温阴性组相对表达量无显著差异 (图7D)。在低温阳性组24和48 h分别比低温阴性组上调2.4和1.8倍;高温阳性组、高温阴性组和低温阴性组相对表达量无显著差异(图7H)。其余5条转录因子(、、、、)相对表达量变化无明显规律(图7B、图7E、 图7F、图7G、图7I)。
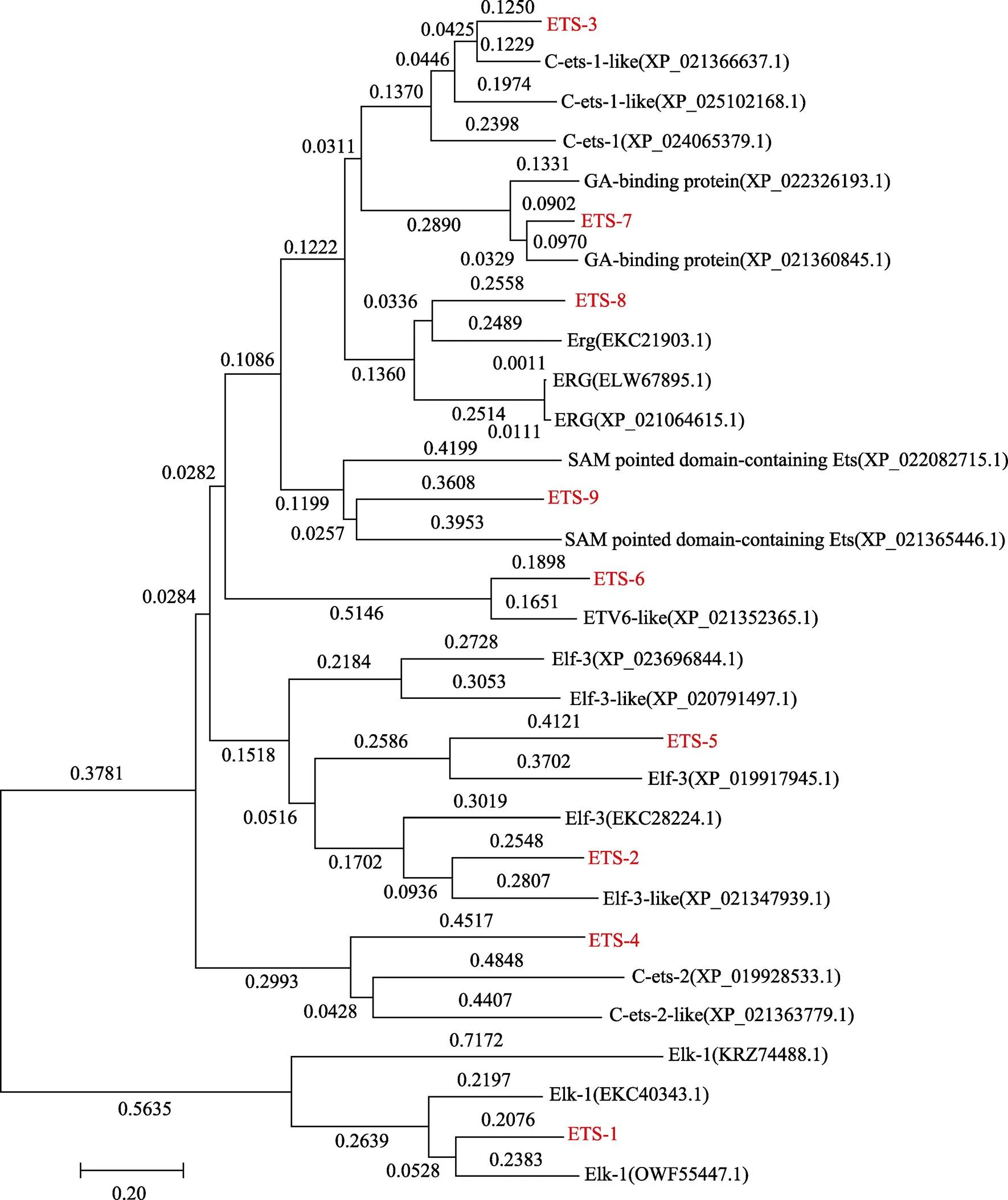
图2 基于NJ法构建的魁蚶Ets氨基酸序列的系统发育树

图3 ETS-1碱基和氨基酸序列
红色下划线表示ETS结构域氨基酸序列;黑色下划线表示BLLF1 superfamliy结构域氨基酸序列
The red underlined indicates the amino acid sequence of the ETS domain; the black underlined indicates the amino acid sequence of the BLLF1 superfamliy domain

图4 ETS-3碱基和氨基酸序列
红色下划线表示ETS结构域氨基酸序列;黑色下划线表示SAM superfamliy结构域氨基酸序列
The red underlined indicates the amino acid sequence of the ETS domain; the black underline indicates the SAM amino acid sequence of the superfamliy domain
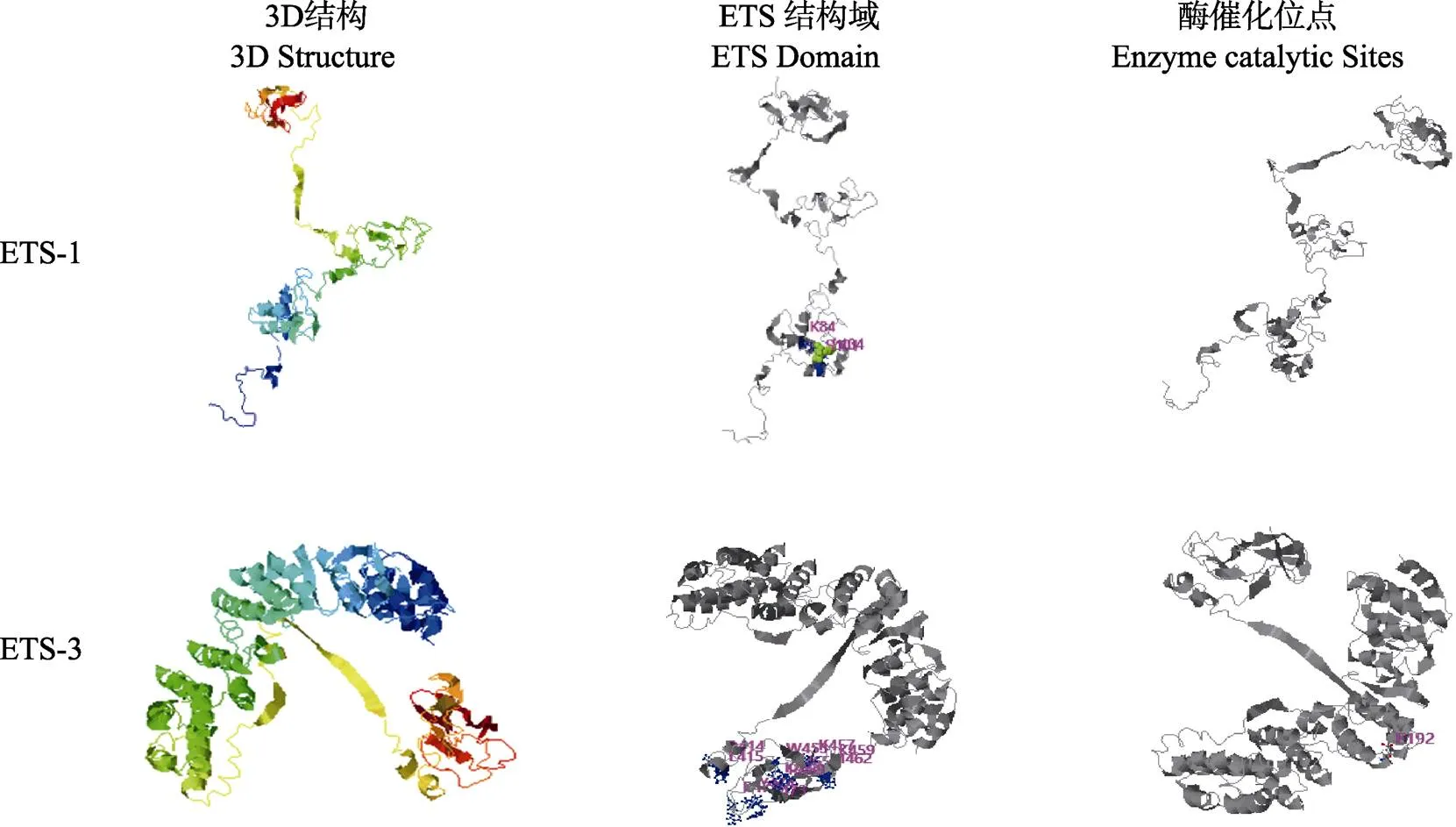
图5 Ets蛋白三维结构及功能位点
2.5 ETS-1和ETS-3生物信息学分析
对和进行生物信息学分析,结果显示,和除了含有ETS结构域外,还含有BLLF1 superfamliy结构域(图3),而还含有SAM superfamliy结构域氨基酸序列(图4),通过I-TASSER对、3D结构进行预测,的结构主要由4条α-螺旋环绕成的复杂结构,ETS结构域是保守的“螺旋-转角-螺旋”结构,无酶催化位点;的结构主要是由11个α-螺旋构成的复杂结构,ETS结构域是保守的“螺旋-转角-螺旋”结构,并且具有酶催化位点(图5)。
3 讨论
Ets转录因子的一个显著特征是均含有1个约85个氨基酸残基的进化保守的ETS结构域,该结构域能介导Ets蛋白与富含嘌呤的序列为GGAA/T和额外的侧翼核苷酸DNA序列发生特异性结合,进而参与靶基因的转录调控(Graves, 1998)。本研究对魁蚶家族基因进行了克隆鉴定,共获得了9个家族成员分子,生物信息学分析表明,其基因结构域高度保守。
Ets家族蛋白是MAPK信号通路下游级联信号转导的执行者(Oikawa, 2003; Wang, 2017; Wasylyk, 1998)。在高等生物中,Ets转录因子不仅参与正常细胞的生长、凋亡、分化等生理过程(Sharrocks, 2001),还可参与病毒的转录复制过程(Panagoulias,2017),例如,Ets家族蛋白可参与调控HIV-1病毒复制过程(Seth, 1993);人类免疫缺陷病毒1 (HIV-1)长末端重复(LTR)的远端增强子区域对HIV复制至关重要,其包含相邻的E-box和Ets 2个结合位点。Sieweke等(1998)发现,Ets-1与USF-1(Upstream stimulatory factor)蛋白相互作用,并分别与HIV-1病毒的E-box和Ets结合位点(‒130~ ‒166)结合,激活病毒复制。Ets在贝类中的功能有待研究(Ma, 2009)。OsHV-1是目前贝类中流行的主要病毒性病原,频繁造成全球范围内养殖贝类的大规模死亡。前期分析OsHV-1基因组序列时,鉴定出诸多Ets转录因子结合位点,初步推断魁蚶Ets转录因子可能参与病毒的复制调控。魁蚶在高温环境中感染OsHV-1后,和相对表达量显著上调,与病毒的拷贝数增长趋势呈正相关。魁蚶在低温环境中感染OsHV-1后,病毒拷贝数不增加,和的相对表达量处于本底水平状态。以上结果表明,和与病毒的复制密切相关,可能参与了病毒的复制调控过程。但它们是通过何种机制参与的、在病毒复制过程中是否起主导作用仍需要进一步研究。
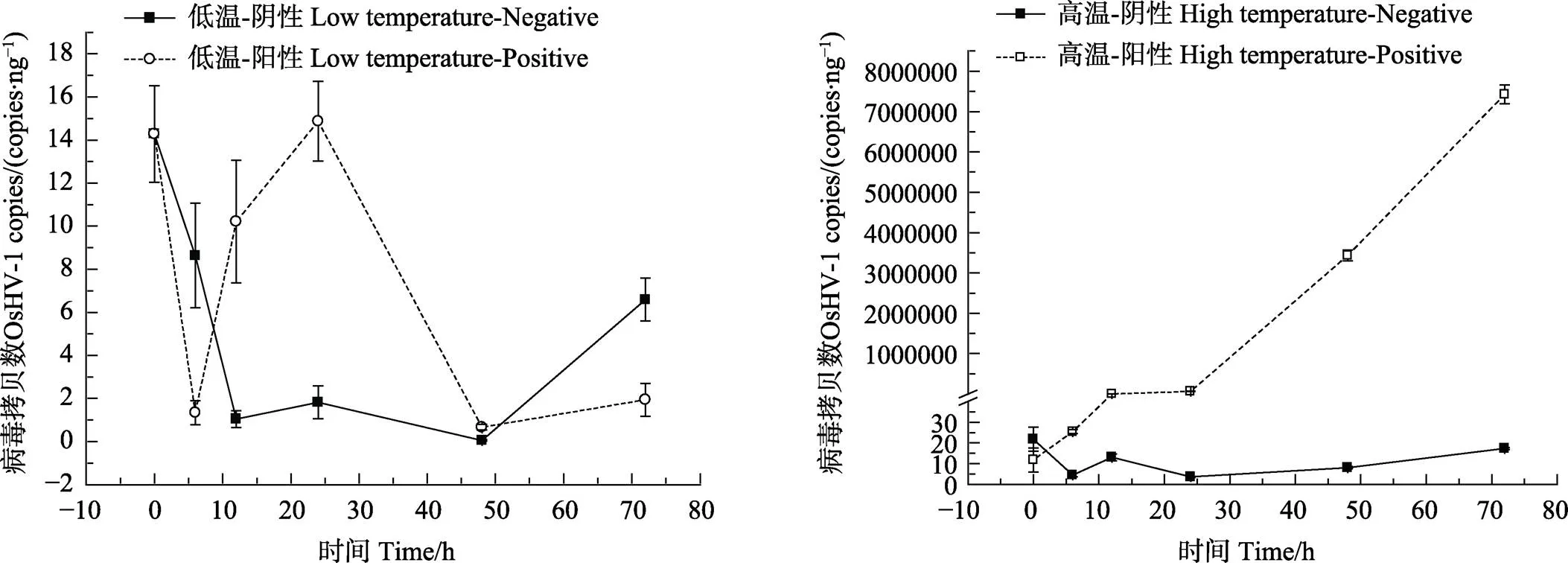
图6 OsHV-1感染后魁蚶血液中病毒拷贝数变化
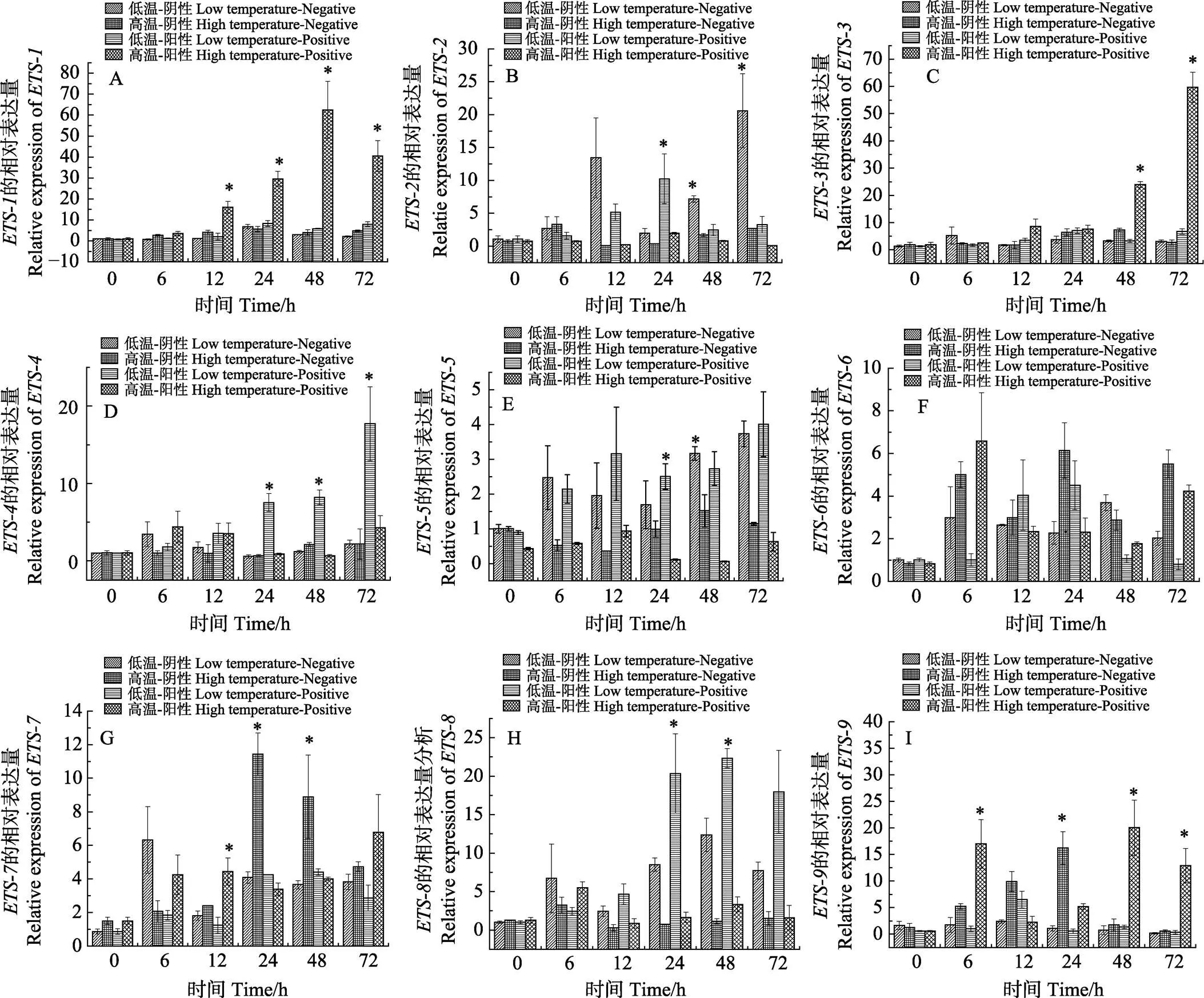
图7 OsHV-1感染后魁蚶血细胞Ets表达水平
*表示实验组和对照组之间差异显著(<0.05)
*indicated the difference between experiment and control group was statistically significant (<0.05)
本研究成功克隆出魁蚶9个Ets转录因子家族成员。通过OsHV-1人工感染魁蚶后对9个家族成员基因表达模式分析,初步确定了和参与病毒的复制调控。为深入研究魁蚶在夏季感染OsHV-1后暴发大规模死亡提供了一定的理论数据。
Barbosa-Solomieu V, Dégremont L, Vázquez-Juárez R,. Ostreid herpesvirus 1 (OsHV-1) detection among three successive generations of Pacific oysters (). Virus Research, 2005, 107(1): 47‒56
Bosselut R, Duvall JF, Gégonne A,. The product of the c-ets-1 proto-oncogene and the related Ets2 protein act as transcriptional activators of the long terminal repeat of human T cell leukemia virus HTLV-1. Embo Journal, 1990, 9(10): 3137‒3144
Elston R. Bivalve mollusc viruses. World Journal of Microbiology and Biotechnology, 1997, 13(4): 393‒403
Graves BJ, Petersen JM. Specificity within thefamily of transcription factors. Advances in Cancer Research, 1998, 75: 1‒57
Hine PM, Thorne T. A survey of some parasites and diseases of several species of bivalve mollusc in northern Western Australia. Diseases of Aquatic Organisms, 2000, 40(1): 67‒78
Hu ZF, Ren SJ, Li SG. Research progress of oyster herpes virus (OsHV-1). Fisheries Science and Technology Information, 2017, 44(5): 259‒264, 267 [胡宗福, 任绍杰, 李树国. 牡蛎疱疹病毒(OsHV-1)研究进展. 水产科技情报, 2017, 44(5): 259‒264, 267]
Jouaux A, Lafont M, Blin JL,. Physiological change under OsHV-1 contamination in Pacific oysterthrough massive mortality events on fields. BMC Genomics, 2013, 14(1): 590
Li YY, Liu YG, Liu XL,. The taxonomic status of Chinese population ofbased on sequences ofandgenes. Progress in Fishery Sciences, 2018, 39(5): 73‒80 [李瑶瑶, 刘云国, 刘晓玲, 等. 基于线粒体基因组和序列的魁蚶中国群体分类学地位研究. 渔业科学进展, 2018, 39(5): 73‒80]
Liang C, Yang AG, Liu ZH,. RAPD analysis of genetic variations in four geographical populations of. Progress in Fishery Sciences, 2010, 31(1): 59‒64 [梁超, 杨爱国, 刘志鸿, 等. 魁蚶4个地理群体遗传结构的RAPD分析. 渔业科学进展, 2010, 31(1): 59‒64]
Liu HM, Wu B, Liu ZL,. Genetic diversity and geographic population structures ofProgress in Fishery Sciences, 2017, 38(6): 92‒99 [刘寒苗, 吴彪, 刘志鸿, 等. 魁蚶()不同地理群体的遗传多样性及种群结构. 渔业科学进展, 2017, 38(6): 92‒99]
Liu XY, Xu HZ. Seedling technique of Ark clam () at room temperature. Sicentific Fish Faming, 2002(10): 14‒15 [刘锡胤, 徐惠章. 魁蚶常温育苗技术. 科学养鱼, 2002(10): 14‒15]
Ma H, Wang J, Wang B,. Characterization of an ETS transcription factor in the sea scallop. Developmental and Comparative Immunology, 2009, 33(9): 953‒958
Mao XY, Yan ZJ, Shao YQ. A new exploration of artificial seedling and seedling preservation in Ark clams (). Hebei Fisheries, 2007(4): 38‒39 [毛雪英, 阎子娟, 邵雁群. 魁蚶人工育苗及保苗的新探索. 河北渔业, 2007(4): 38‒39]
Oikawa T, Yamada T. Molecular biology of the Ets family of transcription factors. Gene, 2003, 303(1‒2): 11‒34
Panagoulias I, Karagiannis F, Aggeletopoulou I,. Ets-2 acts asa transcriptional repressor of the human immunodeficiencyvirus type 1 through binding to a repressor‒Activator target sequence of 5'-LTR. Frontiers Immunology, 2018, 8: 1924
Pleschka S, Wolff T, Ehrhardt C,. Influenza virus propagation is impaired by inhibition of the RAF/MEK/ERK signalling cascade. Nature Cell Biology, 2001, 3(3): 301‒305
Ren WC, Chen HX, Renault T,. Complete genome sequence of acute viral necrosis virus associated with massive mortality outbreaks in the Chinese scallop,. Virology Journal, 2013, 10: 110
Renault T, Lipart C, Arzul I. A herpes-like virus infectingandlarvae in France. Journal of Fish Diseases, 2001, 24(6): 369‒376
Roque A, Carrasco N, Andree KB,. First report of OsHV-1 microvar in Pacific oyster () cultured in Spain. Aquaculture, 2012, 324(3): 303‒306
Ruelas D, Greene W. An integrated overview of HIV-1 latency. Cell, 2013, 155(3): 519‒529
Segarra A, Pépin JF, Arzul I,. Detection and description of a particulargenotype associated with massive mortality outbreaks of Pacific oysters,, in France in 2008. Virus Research, 2010, 153(1): 92‒ 99
Seth A, Hodge DR, Thompson DM,. ETS family proteins activate transcription from HIV-1 long terminal repeat. Aids Research and Human Retroviruses, 1993, 9(19): 1017‒1023
Sharrocks AD, Brown AL, Ling Y,. The ETS-domain transcription factor family. International Journal of Biochemistry and Cell Biology, 1997, 29(12): 1371‒1387
Sieweke MH, Tekotte H, Jarosch U,. Cooperative interaction of Ets-1 with USF-1 required for HIV-1 enhancer activity in T cells. Embo Journal, 1998, 17(6): 1728‒1739
Suico MA, Shuto T, Kai H. Roles and regulations of the ETS transcription factor ELF4/MEF. Journal of Molecular Cell Biology, 2017, 9(3): 168‒177
Wang B, Krall EB, Aguirre AJ,. ATXN1L, CIC, and ETS transcription factors modulate sensitivity to MAPK pathway inhibition. Cell Reports, 2017, 18(6): 1543‒1557
Wasylyk B, Hagman J, Gutierrez-Hartmann A. Ets transcription factors: Nuclear effectors of the Ras-MAP-kinase signaling pathway. Trends in Biochemical Science, 1998, 23(6): 213‒ 216
Webb SC, Fidler A, Renault T,. Primers for PCR-based detection of ostreid herpes virus-1 (OsHV-1): Application in a survey of New Zealand molluscs. Aquaculture, 2007, 272(1‒4): 126‒139
Xia JY, Bai CM, Wang CM,. Complete genome sequence ofassociated with mortalities ofbroodstocks. Virology Journal, 2015, 12: 110
Xin LS, Huang BW, Bai CM,. Validation of housekeeping genes for quantitative mRNA expression analysis in OsHV-1 infected Ark clam,. Journal of Invertebrate Pathology, 2018, 155: 44‒51
Zeng H, Yang Y, An Y,. Physiogical functions of Ets family of transcription factors. China Pharmacological Bulletin, 2017, 33(12): 1645‒1650 [曾红, 杨洋, 安输, 等. Ets转录因子的生理作用研究进展. 中国药理学通报, 2017, 33(12): 1645‒1650]
Zhang QG, Wang RC, Wang XZ,. Selective breeding of strain of()reeve. Modern Fisheries Information, 2007, 22(7): 25‒26 [张启刚, 王如才, 王兴章, 等. 魁蚶(Reeve)品系选育. 现代渔业信息, 2007, 22(7): 25‒26]
Cloning of Nine Genes in the Ets Family of Ark Clam () and Their Expression in Response to Oyster Herpes Virus (OsHV-1) Infection
WEI Zhixin1,3, XIN Lusheng2,3, BAI Changming2,3, LI Yanan3, ZHANG Shumin3, LI Chenghua1, WANG Chongming2,3①
(1. School of Marine Sciences, Ningbo University, Ningbo 315211; 2. Laboratory for Marine Fisheries Science and Food Production Processes, Pilot National Laboratory for Marine Science and Technology (Qingdao), Qingdao 266071; 3. Key Laboratory of Maricultural Organism Disease Control, Ministry of Agriculture and Rural Affairs, Qingdao Key Laboratory of Mariculture Epidemiology and Biosecurity, Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Qingdao 266071)
Ets transcription-factor networks represent a model for how combinatorial gene expression is achieved. The characteristic feature of Ets factors is the conserved ETS domain (Helix-Turn-Helix). Thus, the Ets proteins bind to a core GGAA/T consensus sequence and regulate expression of several genes and play an important role in various cellular functions (mitosis, growth, development, differentiation, and apoptosis) and the regulation of immunity. In this experiment, 9genes (named, respectively) of the Ark clam () were successfully obtained by gene cloning technology, and the open reading frames (ORFs) were 1065 bp, 1290 bp, 1569 bp, 912 bp, 1344 bp, 1404 bp, 1521 bp, 1968 bp, and 1191 bp, respectively. Moreover, they encoded respectively 354, 429, 522, 303, 447, 468, 506, 655, and 396 amino acids. Evolutionary relationships of taxa showed that all genes in this chapter belonged to thefamily genes. The qPCR detection showed that the expression of twogenes (,) was significantly increased. Thus, the present study showed that the Ark clamandare involved in the replication process of the OsHV-1 under high temperature conditions (16±2)℃. In conclusion, the results of this study provide a scientific basis for further study concerning the large number of deaths of Ark clams due to infection with OsHV-1 in summer.
; Ets family; OsHV-1; Gene cloning; Temperature
S917.4
A
2095-9869(2019)06-0121-10
10.19663/j.issn2095-9869.20190304001
http://www.yykxjz.cn/
魏智薪, 辛鲁生, 白昌明, 李亚楠, 张淑敏, 李成华, 王崇明. 魁蚶Ets家族9个基因的克隆及其在病毒感染应答中的表达分析. 渔业科学进展, 2019, 40(6): 121–130
Wei ZX, Xin LS, Bai CM, Li YN, Zhang SM, Li CH, Wang CM. Cloning of nine genes in the Ets family of ark clam () and their expression in response to oyster herpes virus (OsHV-1) infection. Progress in Fishery Sciences, 2019, 40(6): 121–130
* 现代农业产业体系专项资金(CARS-49)、中央级公益性科研院所基本科研业务费(20603022017007; 20603022018014)共同资助[This work was supported by China Aquaculture Research System (CARS-49), and Special Scientific Research Funds for Central Non-Profit Institutes, Yellow Sea Fisheries Research Institute (20603022017007; 20603022018014)]. 魏智薪,E-mail: weizhixin315@yeah.net
王崇明,研究员,E-mail: wangcm@ysfri.ac.cn
2019-03-04,
2019-03-11
WANG Chongming, E-mail: wangcm@ysfri.ac.cn
(编辑 马璀艳)
