鼠伤寒沙门菌baeSR 和acrB 双基因缺失株相关耐药基因的RNA-seq 分析
2019-12-04徐军王瑞李锐高海侠张瑞良王文静赵霞李琳
徐军 王瑞 李锐 高海侠 张瑞良 王文静 赵霞 李琳
(安徽农业大学动物科技学院,合肥 230036)
鼠伤寒沙门菌(Salmonella typhimurium)是世界上分布最广泛的食源性病原菌之一,可广泛感染人和动物,引起食源性、感染性腹泻和败血症等,严重威胁养殖业发展和人类健康[1]。目前,抗生素是治疗鼠伤寒沙门菌感染的最有效手段。然而,长期不合理使用和滥用抗生素使得病原菌对抗生素产生耐药性,对兽医临床和动物性食品安全带来极大挑战[2]。多重耐药外排泵系统是引起细菌产生耐药性的最常见原因之一,同时也是细菌生物膜形成和毒力所必需的,因其作用底物广泛且与细菌的多种生理功能相关而被人们广泛关注[3]。其中AcrABTolC 是革兰阴性菌中最重要的多重耐药外排泵,也是目前研究最清楚的三联外排泵系统,其表达水平受多种调控基因的调节[4]。
双组份信号转导系统(Two-component system,TCS)普遍存在于细菌中,可调节包括运动性、毒力、新陈代谢、抗生素耐药性和应激反应等过程[5]。BaeSR 双组分系统首次在大肠杆菌中发现,随后相继在沙门菌和鲍曼不动杆菌中发现[6-8]。Lin 等[9]证明在沙门菌中BaeSR 可直接与STM3031 的启动子区域结合,从而调节其对头孢曲松的耐药性。Rosner 等[10]发现当某些有毒代谢物在细胞中积累时,BaeSR 和CpxAR 系统被激活,进而激活AcrD和MdtABC 多重耐药外排泵,调控大肠杆菌对多种抗生素的敏感性。本实验室前期发现,acrB的存在可能会掩盖其他外排泵的功能,从而阻碍baeSR对其他多重耐药外排泵的调控[11]。因此,BaeSR 和AcrB 对于研究抗生素耐药性产生的机制至关重要。
随着测序技术的不断进步,RNA-Seq 技术已经成为细菌耐药性研究中的一种常用实验手段。细菌在抗生素作用条件下,本身存在的多种耐药相关基因的表达量会发生一定的变化,利用转录组学研究方法,可以探究细菌耐药前后发生差异表达的基因,从而分析细菌产生耐药性的原因[12]。RNA-Seq技术不仅可以对已知的耐药机制进行确证,还可以从筛选得到的差异表达基因中寻找新的耐药机制,从而揭示其功能及其可能在代谢过程中发挥的作用。本研究通过对鼠伤寒沙门菌CR、CR△acrB及CR△baeSR△acrB基因缺失株进行转录组学测序分析,以期筛选与耐药相关差异表达基因并对其表达量变化、发挥的功能进行分析,为进一步探讨鼠伤寒沙门菌产生耐药性的机制,寻找潜在或新的药物靶标,遏制细菌耐药性的产生和传播提供理论依据。
1 材料与方法
1.1 材料
鼠伤寒沙门菌ATCC13311 体外诱导环丙沙星耐药株CR 由本实验室保存,CR△acrB、CR△baeSR△acrB基因缺失株均由本实验室构建。LB 肉汤购自上海生工生物有限公司,RNA 提取试剂盒、RNA纯化试剂盒均购自QIAGEN 公司,TransStart Green qPCR SuperMix 购自北京全式金生物技术有限公司。
1.2 方法
1.2.1 样品采集 将鼠伤寒沙门菌CR、CR△acrB和CR△baeSR△acrB分别划线接种于LB 固体平板,置于37℃培养16 h 后,分别挑取单菌落接种于LB 液体培养基中,37℃ 180 r/min 过夜培养;次日,1∶100 转接于新鲜LB 液体培养基中,生长至对数生长期时收集菌液,于4℃ 5 000 r/min 离心10 min 后弃上清,并用PBS 洗3 次,收集菌体,液氮速冻,-80℃保存备用。
1.2.2 RNA 的提取与检测 由北京诺禾致源科技股份有限公司对所有样品进行RNA 抽提,用琼脂糖凝胶电泳对RNA 降解程度以及是否有污染进行检测,用Nanodrop(OD260/OD280)对RNA 的纯度进行检测,用Qubit 对RNA 的浓度进行精确定量检测,最后使用Agilent 2100 对RNA 的完整性进行精确检测。
1.2.3 文库构建与测序 检测合格后的样品通过Ribo-zero 试剂盒去除rRNA,加入fragmentation buffer 对样品进行片段化处理以及合成双链cDNA,并用AMPure XP beads 对其纯化。对纯化后的双链cDNA 进行末端修复、加A 尾并连接测序接头,再用AMPure XP beads 进行片段大小选择。最后进行PCR 扩增,并用AMPure XP beads 纯化PCR 产物,得到最终的cDNA 文库,将构建好的文库使用HiSeq测序平台进行测序。
1.2.4 差异基因表达分析 采用HTSeq 软件对各样品进行基因表达水平分析,比对结果使用 FPKM(Fragments per kilobase of exon per million fragments mapped)法进行计算,差异表达基因的筛选标准为|log2(Fold Change)| > 1 且q value < 0.005。
1.2.5 差异基因GO 和KEGG 富集分析 采用GOseq 方法对所有筛选到的差异表达基因进行GO富集分析,明确差异表达基因在 Gene Ontology 中的分布状况。以KEGG 数据库中Pathway 为单位,应用超几何检验,找出在差异表达基因中显著性富集的Pathway,进一步确定差异表达基因所参与的代谢通路及发挥的生物学意义。
1.2.6 差异表达基因的验证 随机选取7 个耐药相关差异表达基因,根据NCBI 中公布的鼠伤寒沙门菌的基因组序列(CP009102.1),以沙门菌GAPDH为内参基因,利用Oligo 6.0 设计所用引物,引物名称及序列见表1。采用SYBR Green I 法进行mRNA表达量分析,数据采用2-△△Ct处理。

表1 引物序列
2 结果
2.1 测序数据质量评估及基因组比对结果
检测样品中CR、CR△acrB和CR△baeSR△acrB的 质 量 浓 度 分 别 为1 188,372 和688 ng/μL;OD260/280分 别 为2.077、2.11 和2.02,OD260/230分 别为2.055、1.01 和0.76,满足建库测序的要求。从转录组测序数据分析报告中得知,CR、CR△acrB和CR△baeSR△acrB分别获得 11 693 470、13 342 112和108 24 816 条原始序列,对原始数据进行过滤后,剔除低质量数据的Clean reads 分别为11 311 842、12 959 780 和10 500 966,其错误率分别为0.03%、0.02%和0.02%。通过Bowtie2 将过滤后的测序序列进行基因组定位分析,结果显示鼠伤寒沙门菌CR、CR△acrB和CR△baeSR△acrB的比对结果分别是95.74%、97.60%和97.19%(表2)。该结果证明本次测序准确性较高,后续可直接用该部分数据进行分析。
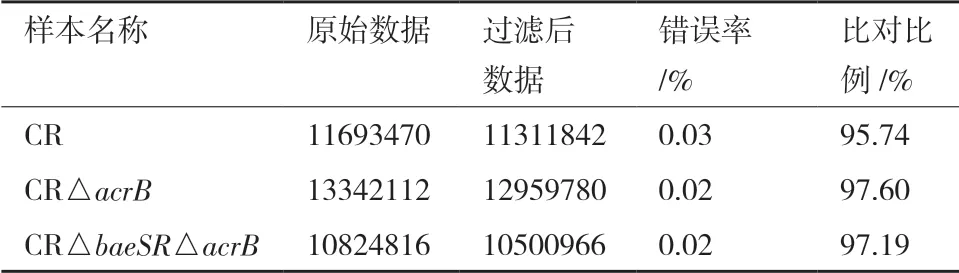
表2 数据产出质量统计
2.2 基因差异表达分析
采用HTSeq 软件对各样品进行基因表达水平分 析,以 同 时 满 足|log2(Fold Change)| > 1 且q value < 0.005 为标准对差异表达基因进行筛选。以CR△acrB、CR 为比较组,共筛选到1 320 个差异表达基因,其中894 个基因表达下调,426 个基因表达上调(图1-A);以CR△baeSR△acrB、CR 为比较组,共筛选到1 377 个差异表达基因,其中972 个基因表达下调,405 个基因表达上调(图1-B),从上述结果中筛选部分耐药相关差异表达基因(表3)。
2.3 差异表达基因的GO富集分析
采用GOseq 将差异表达基因注释在Gene Ontology 中,GO 分为分子功能(Molecular function)、生物过程(Biological process)和细胞组分功能(Cellular component)3 个部分。根据试验目的筛选差异基因后,富集分析差异基因在 Gene Ontology 中的分布状况。以CR△acrB、CR 为比较组得到30 个GO 功能注释(图2-A),其中分子功能类4 个,无显著富集;生物过程16 个,主要集中在跨膜运输、细胞定位、细胞黏附等功能;细胞组分功能10 个,主要集中在细胞膜及其相关组成成分。以CR△baeSR△acrB、CR 为比较组得到30 个GO 功能注释(图2-B),其中分子功能类5 个,无显著富集;生物过程16 个,主要集中在细胞运动、跨膜运输、细胞黏附、纤毛或鞭毛运动、细胞定位等功能;细胞组分功能9 个,主要集中在细胞膜及其膜的组成部分。

图1 差异基因分布火山图

表3 耐药相关差异表达基因
2.4 差异表达基因的KEGG Pathway富集分析
在生物体内,不同基因相互协调行使其生物学功能,通过Pathway 显著性富集能确定差异表达基因参与的最主要生化代谢和信号转导途径。以CR△acrB、CR 为比较组(图3-A),结果显示1 071个差异表达基因在数据库中注释到88 个信号通路,其中581 个差异基因表达下调,490 个差异基因表达上调;以CR△baeSR△acrB、CR 为比较组(图3-B),结果显示1 086 个差异表达基因在数据库中注释到86 个信号通路,其中605 个差异基因表达下调,481 个差异基因表达上调。差异基因主要富集在代谢途径、次生代谢产物合成、ABC 转运系统、双组分信号转导系统、鞭毛组装、β-内酰胺抗性等通路。
2.5 差异表达基因的qRT-PCR验证
随机筛选7 个差异表达基因,以GADPH 为内参基因,与RNA-Seq 分析结果相比,qRT-PCR 的检测结果与其基本一致(图4)。随机选取的7 个差异表达基因在qRT-PCR 和RNA-Seq 中的诱导表达变化趋势相同,从而证实了RNA-Seq 分析结果的可靠性。
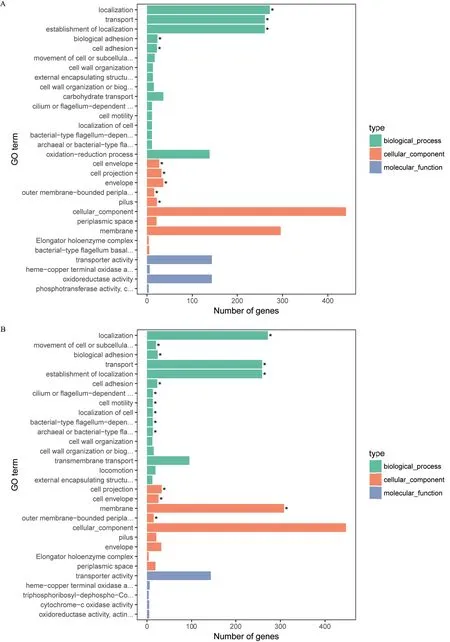
图2 GO 功能富集
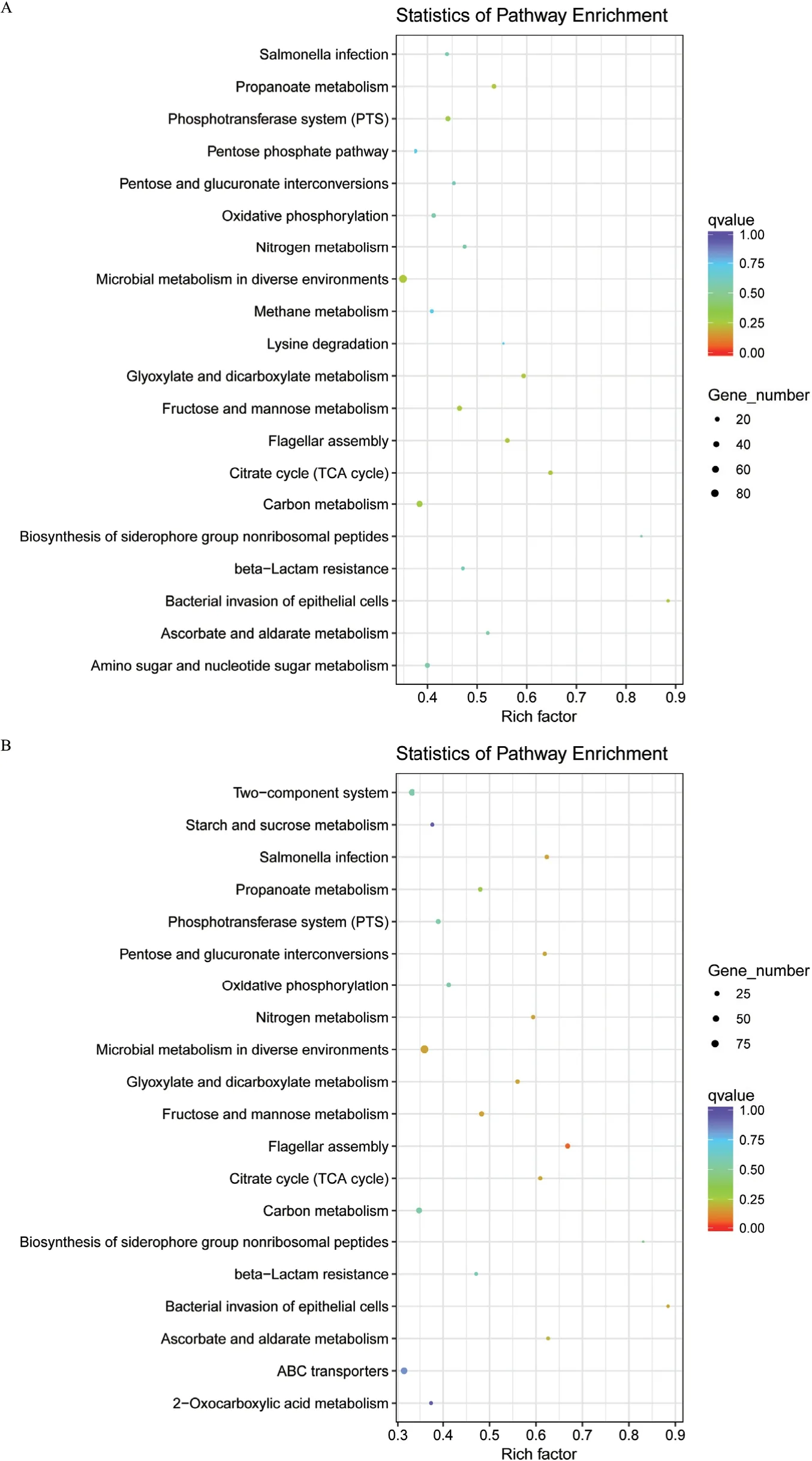
图3 KEGG 通路富集
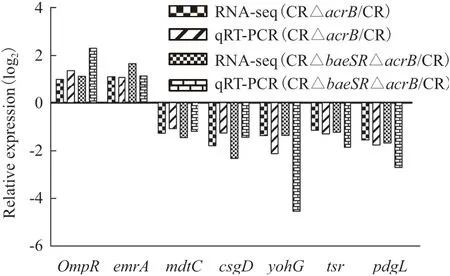
图4 部分差异表达基因qRT-PCR 验证结果
3 讨论
近年来,从分子水平上揭示沙门菌的耐药机制,已成为沙门菌最重要的研究方向之一[13]。鼠伤寒沙门菌的耐药性主要依赖于外排泵系统、双组分信号转导系统、生物膜等因素。抗菌药物耐药相关外排泵可分为5 大家族,其中RND 家族主要以三聚体形式转运底物,通常由染色体基因编码,其结构由膜融合蛋白、RND 转运蛋白和外膜因子组成。在革兰阴性菌中,RND 家族外排泵作为最主要的多重耐药外排泵通过从细菌细胞中泵出多种抗微生物化合物来赋予其多重耐药性,在沙门菌耐药性方面发挥着不可替代的作用[14]。AcrB因其作用底物广泛且在多种革兰阴性菌中均存在其同源物,被认为是对人类健康最重要的RND 系统[15]。双组分信号转导机制可以调节细菌对环境的适应性从而影响其对抗生素的敏感性,如BaeSR 可通过激活RND 型外排泵AcrD 和MdtABC 的转录改变沙门菌的多要耐药性[10,16-17]。本实验室致力于鼠伤寒沙门菌耐药机制的研究,通过自杀质粒介导的同源重组系统构建acrB和baeSR的基因缺失株,并对其进行运动性、生物膜的形成、耐药性等方面的研究,结果显示,acrB和baeSR基因缺失导致生物膜形成能力下降,耐药性减弱,表明基因acrB和baeSR可调控鼠伤寒沙门菌的耐药性。为进一步探究其耐药机理,对CR、CR△acrB以及CR△baeSR△acrB进行转录组测序。
转录组测序结果显示CR△acrB/CR、CR△baeSR△acrB/CR 分别筛选到1 320、1 377 个差异表达基因。CR△acrB/CR 中894 个基因表达下调,426 个基因表达上调;CR△baeSR△acrB/CR 中972个基因表达下调,405 个基因表达上调。通过对差异表达基因进行GO 功能分类发现,差异表达基因主要参与跨膜运输、细胞黏附、纤毛或鞭毛运动以及细胞定位等生物学功能,生物膜以及膜的组成部分等细胞组分功能,这些生物过程均与细菌耐药性有着密切联系。先前得到的acrB和baeSR基因缺失可以导致生物膜形成能力下降、运动性降低、耐药性减弱等试验结果与其基本一致。通过KEGG 富集分析和以往文献报道对转录组学结果进行深入分析,筛选出CR△acrB和CR△baeSR△acrB共同变化的耐药相关差异表达基因,包括STM3031、pmrF、fliA、OmpR、csgD、emrA、mdtC和yejE等。
STM3031 已被证实可赋予沙门菌头孢曲松抗性,本团队对acrB基因缺失后可造成STM3031 表达量显著下调,acrB和baeSR基因缺失后表达量进一步下降,与Lin 等[9]实验结果一致。KEGG Pathway 富集分析显示pmrF属于氨基糖和核苷酸糖代谢途径,可参与调节细菌对多黏菌素的抗性[18]。细菌生物膜的形成与临床感染密切相关,鞭毛在生物膜的形成初期起着重要的介导黏附作用,并为生物膜的形成提供动力[19]。因此,生物膜和鞭毛运动都是引起细菌产生耐药性的重要原因。本研究中OmpR基因表达量显示上调,csgD、fliA、fliC、fliG等相关基因均显示不同程度的下调。其中OmpR属于双组份信号转导系统,可激活外膜孔蛋白相关基因、脂多糖修饰相关基因等的转录[20]。CSG 区由csgBA和csgDEFG组成,可编码菌毛等相关基因,csgD是重要的生物膜转录调节因子,在细胞黏附、聚集和生物膜形成方面发挥至关重要的作用[21]。朱春红等[22]通过构建的不同肠炎沙门菌的fliC基因缺失株,证明其在生物膜形成过程中发挥着重要作用。本研究结果证实acrB和baeSR可通过调控沙门菌的鞭毛运动和生物膜形成能力改变其对抗生素的敏感性。
外排泵在细菌多重耐药性方面发挥至关重要的作用,本研究筛选到多个外排泵相关基因均与以往报道相符。其中emrA属于主要易化因子家族(MFS),可与emrB、tolC结合形成三联体系;mdtC属于RND 家族外排泵,BaeSR 可激活mdtC的表达;yejE属于ABC 转运系统,均可介导菌株对黏菌素、新生霉素的耐药性[23]。因此,多种外排泵相关基因的表达量发生变化,可能是acrB和baeSR调控鼠伤寒沙门菌产生耐药性的主要原因。在后续研究中,我们将对STM1545、fliA、csgD和pmrf进行EMSA 验证,进一步明确鼠伤寒沙门菌双组分系统BaeSR 和AcrB的耐药调控机制。
4 结论
本研究初步探讨了鼠伤寒沙门菌acrB和baeSR基因缺失后的表达变化,发现菌株对抗生素的敏感性可能通过AcrB 和BaeSR 介导的多重耐药外排泵基因的表达、细胞膜的通透性、核苷酸代谢以及鞭毛运动等多种耐药机制调控。
