单粒花生主要脂肪酸含量近红外预测模型的建立及其应用
2019-11-13李建国薛晓梦张照华王志慧晏立英陈玉宁万丽云康彦平淮东欣姜慧芳廖伯寿
李建国 薛晓梦 张照华 王志慧 晏立英 陈玉宁 万丽云 康彦平 淮东欣 姜慧芳 雷 永 廖伯寿
单粒花生主要脂肪酸含量近红外预测模型的建立及其应用
李建国 薛晓梦 张照华 王志慧 晏立英 陈玉宁 万丽云 康彦平 淮东欣*姜慧芳 雷 永*廖伯寿
中国农业科学院油料作物研究所/ 农业部油料作物生物学与遗传育种重点实验室, 湖北武汉 430062
脂肪酸组成是影响花生营养价值和货架寿命的主要因素, 高油酸花生以其营养保健价值高、化学稳定性好、耐储藏等特点, 深受广大消费者和花生加工企业的喜爱。因此, 培育高油酸品种是花生育种的重要目标, 建立快速、高效、准确检测花生中主要脂肪酸含量的无损方法是加快花生脂肪酸改良和高油酸品种选育进程的重要技术保障。本研究利用近红外光谱技术建立了可以非破坏性地快速检测单粒花生中油酸、亚油酸、棕榈酸含量的数学模型, 其中油酸模型的决定系数(2)为0.907, 均方差为3.463; 亚油酸模型的决定系数为0.918, 均方差为2.824; 棕榈酸模型的决定系数为0.824, 均方差为0.782。使用100粒花生验证该模型的准确性, 结果油酸、亚油酸和棕榈酸的近红外预测值与化学值的相关系数分别为0.88、0.90和0.71, 表明此模型可以准确地预测单粒花生中这3种脂肪酸的含量。本研究借助该模型建立了一种不依赖分子标记的快速、高效选育高油酸花生的方法, 并成功应用于高油酸花生育种, 选育出高油酸花生品种中花215。
单粒花生; 脂肪酸; 近红外模型; 高油酸; 育种方法
花生(L)是我国重要的油料与经济作物[1]。在国内大宗油料作物中, 花生总产、单位面积产油量、种植业产值均居首位, 在花生总产仅一半用于榨油的情况下, 花生油年产量约270万吨, 占国产植物油产量的四分之一, 是国产植物油的第二大来源, 仅次于菜籽油[2]。脂肪酸组成是影响花生及其加工产品的营养保健价值、加工特性与效率、耐储性以及市场竞争力的关键品质性状。花生种子的脂肪酸以油酸(C18:1)、亚油酸(C18:2)、棕榈酸(C16:0)为主,三者之和占脂肪酸总量的90%以上, 这3种脂肪酸的含量是花生品质的重要影响因素[3]。油酸是单不饱和脂肪酸, 它能选择性地降低人体血液中有害的低密度胆固醇, 而保持有益的高密度胆固醇, 有效预防心血管疾病的发生。亚油酸是多不饱和脂肪酸, 具有2个双键, 在加工和储存过程中易被氧化变质, 含量过多不利于储存和加工[4]。棕榈酸是饱和脂肪酸, 摄入过多棕榈酸会引起人体血清中总胆固醇的升高, 增加心血管疾病的发病率, 不利于人体健康[5-6]。因此, 培育油酸含量高、亚油酸和棕榈酸含量低的优质品种是花生育种的重要目标, 而建立花生中这3种脂肪酸含量的快速、高效、准确的检测技术是加快育种进程的重要技术保障。
目前, 利用气相色谱法(gas chromatography, GC)检测花生中的脂肪酸组成是最准确的方法, 但是该方法对花生种子具有破坏性[7-8], 且费时费力、操作繁琐, 还需要专业人员和昂贵设备, 检测成本高。Chamberlin等[9]开发利用毛细管电泳检测花生油亚比, 该方法测定结果与GC测定值的决定系数(2)高达0.96, 但是该方法从制备到分析出实验结果仍需5~6 h。近年来, 鉴于近红外光谱检测技术具有成本低、无损、快速等优点, 花生中也建立了相应的近红外检测脂肪酸的方法。禹山林等[10]利用近红外光谱技术建立检测花生中油酸、亚油酸、棕榈酸和硬脂酸含量的方法。之后, 花生中又相继开发了一系列用于分析花生中主要脂肪酸含量的近红外模型[11-13]。但是, 目前建立的这些方法都需要10~100 g (20~ 150粒)花生种子作为样品, 难以应用于早期杂交分离世代(F2~F4)基因型高度复杂群体中少量纯合单粒的筛选, 不能有效缩减育种规模, 因此, 建立更加高效的检测单粒花生中主要脂肪酸的技术非常必要[14-15]。Tillman等[16]建立了检测单粒花生中油酸和亚油酸含量的近红外模型。Kavera等[17]也建立了单粒花生脂肪酸含量的近红外模型, 但建模样品油酸含量均低于70%, 因此该模型用于花生高油酸育种中的选择效果不佳。国内尚未开发出可用于检测单粒花生种子中脂肪酸含量的近红外模型。
分子标记辅助选择是当前高油酸花生育种主要采用的选择鉴定方法。FAD2 (D12脂肪酸脱氢酶)是脂肪酸生物合成途径中催化油酸在第12碳位引入双键生成亚油酸的关键酶[18-19], 花生中已针对基因的突变位点开发了AS-PCR、CAPS、KASP等分子标记[20-21], 但是使用这些分子标记费时费力、操作繁琐, 还需要专业人员和昂贵设备, 检测成本高。本团队通过研究发现单粒花生种子中突变的基因个数与其油酸含量存在累加效应: 当4个基因都发生突变(基因型为aabb)时, 其油酸含量平均为80% (变异范围79.0%~82.5%), 均在75%以上; 当3个基因发生突变(基因型为Aabb或aaBb)时, 其油酸含量在64.5%~74.4%之间; 当突变的基因数目少于3个(基因型为AaBb、AAbb、aaBB、AABb、AaBB或AABB)时, 其油酸含量不超过70% (45.3%~70.0%)[22]。根据此规律, 利用近红外光谱检测单粒花生种子中油酸的含量, 根据油酸含量就可以快速选择出基因型为aabb的高油酸花生种子。如果将单粒花生中脂肪酸含量的近红外检测模型应用于高油酸花生育种, 可实现在杂交的早期世代对单粒花生中油酸含量定向快速选择, 大大降低育种的人工和时间成本, 提高选择效率。
本研究针对上述问题, 选用1202粒有代表性的花生种子, 分别建立单粒花生中油酸、亚油酸和棕榈酸含量的近红外分析模型, 利用上述模型开发了一种不依赖分子标记的快速、高效选育高油酸花生的方法, 并成功选育出高油酸花生新品种。
1 材料与方法
1.1 材料
第一部分是建立油酸、亚油酸和棕榈酸预测数学模型的标准样品集, 共来源于520份材料的1202粒饱满花生种子, 其中包含中国花生微微核心种质99份(297粒)、ICRISAT花生微核心种质184份(368粒)、栽培品种及地方品种237份(537粒); 第二部分是近红外模型的外部验证样品集, 包含100粒饱满的花生种子; 第三部分是杂交亲本及其后代分离材料, 以普通油酸含量花生品种冀花4号(油酸含量51.9%)为母本, 高油酸花生品种开农58(油酸含量为80.5%)为父本。以上所有材料均由中国农业科学院油料研究所花生研究室提供。
1.2 方法
1.2.1 标准样品集近红外光谱的测定 近红外光谱仪预热后, 将干燥除杂后的花生种子置近红外光谱扫描操作台。近红外光谱扫描谱区范围为4000~12,500 cm-1, 取每个样品1粒种子, 重复扫描3次, 并且第2次和第3次扫描时要将花生换个角度重新放置在扫描孔上, 以得到同一样品的多个近红外光谱[23]。
1.2.2 标准样品集化学值的测定 参照GB/T17377-2008, 由中国农业科学院油料所花生研究室测定标准样品集化学值。
1.2.3 NIRS模型的建立 对单粒花生中油酸、亚油酸和棕榈酸的化学值和近红外光谱预测值分别进行拟合光谱处理, 采用偏最小二乘法(PLS)的化学计量学方法建立数学模型, 反复采用内部交叉验证剔除奇异值, 通过比较模型的决定系数(2)和标准差(RMSECV)衡量模型质量, 筛选最佳模型, 并通过比较样品预测值与化学值的决定系数(2)和均方差(RMSECV)来衡量模型的质量[24-25]。
1.2.4 模型的外部验证 选取100粒饱满的花生种子, 利用本方法检测其油酸、亚油酸和棕榈酸的含量, 记录近红外光谱(NIR)的预测值, 再利用气相色谱法(GC)分析每粒种子的脂肪酸组成, 比较每粒种子NIR预测值与GC化学值的相关性和准确性。
1.2.5 品种选育过程 2013年春季, 以普通油酸含量花生品种冀花4号为母本, 高油酸花生品种开农58为父本配置杂交组合, 并收获F1。2013年冬季, 将F1种植于三亚, 并收获F2。2014年春季, 使用近红外检测单粒F2种子中油酸含量, 选择油酸含量在70%以上的种子, 种植于武汉, 并收获F3。2014年冬季, 使用近红外检测单粒F3种子中油酸含量, 选择油酸含量在75%以上的种子, 种植于三亚, 并收获F4。2014年春季, 将收获的F4按株系种植于武汉, 调查其农艺性状, 并收获F5。利用气相色谱法分析各株系的脂肪酸组成, 淘汰油酸含量低于75%的株系。2014年冬季, 三亚扩繁筛选出的株系。2015— 2016年间持续进行株系选择和扩繁, 筛选出优异株系, 编号15-2015。通过参加2017—2018年的多点试验(品系代号中花215), 收集相关数据, 预计于2019年登记高油酸花生品种中花215。
2 结果与分析
2.1 花生籽粒的近红外反射光谱分析
共使用1202粒饱满的花生种子用于构建近红外分析模型。花生标准样品集的近红光谱曲线趋势大致相同(图2), 但不同样品的吸收峰强度不同, 即脂肪酸含量不同。结果表明, 花生籽粒的近红外光谱图可以用于花生籽粒脂肪酸组成的定量分析。
2.2 花生籽粒的化学值测定
1202粒用于脂肪酸含量建模的花生种子经过气相色谱分析, 其油酸含量变异范围为32.8%~ 80.8%; 亚油酸含量变异范围为0.7%~43.9%; 棕榈酸含量变异范围为5.9%~16.6% (表1)。本研究选取的花生种子脂肪酸含量变幅较宽, 满足建立近红外模型的要求。

图1 本试验中使用的近红外检测仪Unity-SpectrastarXL

图2 样品集的光谱分析

表1 建立数学模型使用的单粒花生种子的油酸、亚油酸和棕榈酸含量
2.3 模型构建
对单粒花生中油酸、亚油酸和棕榈酸的化学值和采集的近红外光谱数据分别进行拟合光谱处理, 采用偏最小二乘法(PLS)的化学计量学方法建立数学模型, 反复采用内部交叉验证剔除奇异值, 通过比较模型的决定系数(2)和标准差(RMSECV)衡量模型质量, 筛选最佳模型。油酸模型的决定系数为0.907, 均方差为3.463 (图3-A); 亚油酸模型的决定系数为0.918, 均方差为2.824 (图3-B); 棕榈酸模型的决定系数为0.824, 均方差为0.782 (图3-C)。油酸和亚油酸模型的决定系数均大于0.9, 可以进行有效预测, 由于花生中棕榈酸含量相对油酸、亚油酸较低, 其决定系数低于油酸、亚油酸, 但大于0.8, 均方差为0.782, 也可以进行有效预测。
2.4 模型的外部验证
另取100粒饱满的花生种子作为外部验证样品, 检验模型的预测效果(附表1)。油酸的NIR预测值与GC化学值的相关系数达到0.88 (图4-A), 亚油酸的NIR预测值与GC化学值的相关系数达到0.90 (图4-B), 棕榈酸的NIR预测值与GC化学值的相关系数为0.71 (图4-C), 表明利用本方法得到的NIR预测值准确。
2.5 模型在高油酸花生育种中的应用
2.5.1 F1种子的获取 以冀花4号(油酸含量51.9%)为母本, 开农58 (油酸含量为80.5%)为父本, 进行杂交, 收获12粒F1种子。
2.5.2 F2~F3种子油酸含量测定与筛选 将F1代自交, 分单株收获, 共得174粒F2种子。利用近红外仪配合本研究建立的脂肪酸检测模型, 检测单粒F2花生种子中油酸的含量(附表2), 共筛选到38粒油酸含量在70%以上的花生种子; 将F2代按照油酸含量由高到低种植, 自交, 分单株收获, 共得563粒F3种子; 利用近红外光谱检测单粒F3花生种子中油酸的含量(附表3), 共筛选到196粒油酸含量在75%以上的花生种子; 将F3代按照油酸含量由高到低种植, 自交, 根据其农艺性状严格选择, 收获农艺性状优异的10个F4单株。
2.5.3 F4优异单株、株系选择与油酸测定及基因型确证 利用近红外光谱检测同一株收获的10粒F4种子中油酸含量, 发现其油酸含量全部高于75%。再将上述10粒种子混合取样并利用气相色谱分析发现, 其油酸含量均在80%及以上(表2)。将来源于同一单株F4种子种植于1~3行内, 按行收集该行内每一单株的一片新叶, 提取混合样的DNA, 利用KASP标记检测AhFAD2的基因型, 发现这10个F4株系基因型全部为aabb (表3)。

图3 本方法中油酸模型、亚油酸模型和棕榈酸模型的决定系数

图4 检验使用的单粒花生种子的油酸(A)、亚油酸(B)、棕榈酸(C)近红外预测值与化学值相关性
C18:1: 油酸; C18:2: 亚油酸; C16:0-棕榈酸。
C18:1: oleic acid; C18:2: linoleic acid; C16:0: palmitic acid.
2.5.4 优良品系多点试验 根据农艺性状精选, 通过扩大繁殖、参加株系和产量比较试验, 最终选育出高油酸花生品种中花215。中花215属普通型高油酸花生品种, 全生育期123 d, 株高37.4 cm, 平均单产荚果370.98 kg 667 m–2, 籽仁252.93 kg 667 m–2, 百果重194.8 g 667 m–2, 百仁重84.16 g 667 m–2, 出仁率67.45%, 油酸含量80.2%, 亚油酸含量1.6%, 含油量53.3%, 准备于2019年登记。
3 讨论
3.1 本研究建立的单粒花生脂肪酸近红外模型适用性广
本研究成功建立了利用近红外技术检测单粒花生种子中油酸、亚油酸和棕榈酸含量的模型。虽然之前国际上已建立了一些利用近红外检测单粒花生脂肪酸含量的模型, 但是Tillman等[16]建立的模型仅使用了132粒花生种子, 涉及89个花生品种和品系, Kavera等[17]则使用的是1000份突变体材料遗传背景单一。本研究在建立模型时使用了1200多粒花生种子, 而且来源于遗传背景非常丰富的花生种质资源, 其中包括中国微微核心种质、ICRISAT微核心种质以及地方品种和育成品种, 选择的材料遗传背景丰富且复杂, 涉及到的材料也十分广泛, 非常具有代表性。因此, 本研究建立的模型适用性更广。

表2 单粒近红外光谱检测花生F4种子中的油酸含量(%)及其混样气相色谱检测结果
a: 品系名称; b: 单粒近红外检测结果; c: 混样气相检测结果。
a: the name of the strain; b: oleic acid content of single seed detected by NIR; c: GC for mixed seeds.
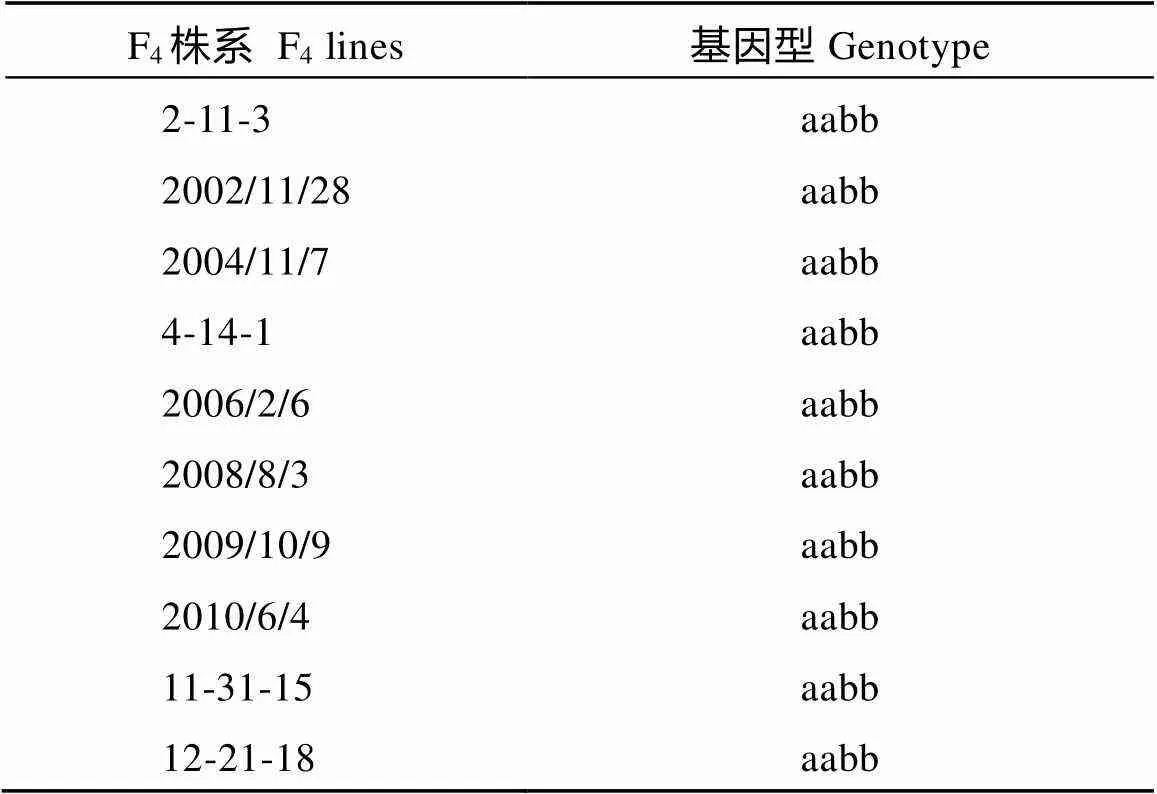
表3 各F4株系AhFAD2的基因型
3.2 单粒花生脂肪酸近红外模型在高油酸花生育种中应用的关键点
将单粒花生脂肪酸近红外模型应用于高油酸花生育种的关键在于如何在杂种的早期世代确定保留种子的油酸含量标准, 如果选择的条件过于严苛(油酸含量标准定的过高), 例如只选油酸含量75%以上的种子, 可能造成保留的F2代种子太少, 难以在其后代中筛选到农艺性状优良的单株; 如果选择的条件过于宽松, 例如选择油酸含量高于65%的种子, 可能造成引入携带AaBb、AAbb或aaBB这些基因型的种子, 当这些种子再自交一代后, AaBb基因型材料自交后代分离复杂, AAbb或aaBB基因型的材料不可能再分离产生高油酸花生, 都为后续的田间种植和选择工作增加了负担。本研究所建立的育种方法选择在F2代时保留油酸含量在70%以上的花生种子, 是依据“当突变的基因数目少于3个(基因型为AaBb、AAbb、aaBB、AABb、AaBB或AABB)时, 其油酸含量不超过70%”[26-27]。这一规律确定的, 并不是单纯的折中选择, 选用70%油酸含量为标准, 确保所选种子的基因型为aabb、Aabb和aaBb之一, 其后代分离简单, 易于选择, 并且为后续的农艺性状筛选保留了足够多的F2种子。而在F3代时选择保留油酸含量在75%以上的花生种子, 此时提高选择条件可以确保所选择的花生种子基因型都为aabb, 不再混入其他基因型的种子, 在后续选择中可以不再针对油酸性状, 有效减少工作量。
3.3 单粒花生脂肪酸近红外模型应用于高油酸花生育种的优势
本研究建立了利用近红外检测单粒花生主要脂肪酸含量的模型, 使用该模型配合高油酸性状的遗传特性, 可高效地用于高油酸花生品种培育, 该方法不依赖分子标记和基因型鉴定, 依然可以实现在杂交早期后代对油酸含量进行快速、高效选择。该方法快速、高效、简便、成本低廉、易操作, 可以显著加快高油酸花生品种的育种进程。
4 结论
使用遗传背景广泛的1202粒花生种子, 建立了基于近红外光谱技术检测单粒花生中油酸、亚油酸和棕榈酸含量的模型。同时, 利用该模型开发了育种方法, 该方法基于单粒花生种子中基因突变个数与其油酸含量的规律, 不依赖分子标记, 可以在杂交的早期世代对油酸含量进行选择, 有效加快育种进程, 并成功选育出高油酸花生品种中花215。
附表 请见网络版: 1) 本刊网站http://zwxb.chinacrops.org/; 2) 中国知网http://www.cnki.net/; 3) 万方数据http://c.wanfangdata.com.cn/Periodical-zuowxb. aspx。
[1] 万书波. 我国花生产业面临的机遇与科技发展战略. 中国农业科技导报, 2009, 11(1): 7–12. Wan S B. Opportunities facing peanut industry in China and strategies for its science and technology development., 2009, 11(1): 7–12 (in Chinese with English abstract).
[2] 刘娟, 汤丰收, 张俊, 臧秀旺, 董文召, 易明林, 郝西. 国内花生生产技术现状及发展趋势研究. 中国农学通报, 2017, 33(22): 13–18. Liu J, Tang F S, Zhang J, Zang X W, Dong W Z, Yi M L, Hao X. Current status and development trends of peanut production technology in China., 2017, 33(22): 13–18 (in Chinese with English abstract).
[3] 郭建斌. 花生含油量及脂肪酸组成的QTL分析. 华中农业大学硕士学位论文, 湖北武汉, 2016. Guo J B. QTL Analysis for Oil Content and Fatty Acid Traits in Peanut (L.).MS Thesis of Huazhong Agricultural University, Wuhan, Hubei, China, 2016 (in Chinese with English abstract).
[4] Pandey M K, Wang M L, Qiao L X, Feng S P, Khera P, Wang H, Tonnis B, Barkley N A, Wang J P, Holbrook C C, Culbreath A K, Varshney R K, Guo B Z. Identification of QTLs associated with oil content and mappinggenes and their relative contribution to oil quality in peanut (L.)., 2014, 15(1): 133.
[5] Yin D , Deng S , Zhan K, Cui D. High-oleic peanut oils produced by hpRNA-mediated gene silencing of oleate desaturase., 2007, 25: 154–163.
[6] 张晓科, 王珊珊, 曲伟华. 近红外定量分析自然风干花生油酸和亚油酸及棕榈酸含量的探索.农业开发与装备, 2017, (7): 76–77. Zhang X K, Wang S S, Qu W H. Quantitative analysis of natural air–dried arachidonic acid, linoleic acid and palmitic acid by near infrared spectroscopy., 2017, (7): 76–77 (in Chinese with English abstract).
[7] 王传堂. 高油酸花生. 上海:上海科学出版社, 2017. pp 113–114. Wang C T. High Oleic Acid Peanuts. Shanghai: Shanghai Scientific and Technical Publishers, 2017. pp 113–114 (in Chinese).
[8] Idriss M R.基于高光谱成像技术和近红外光谱技术测定花生种子及花生油中油酸和亚油酸含量的方法研究. 中国农业科学院博士学位论文, 北京, 2016. Idriss M R. Determination of Oleic Acid and Linoleic Acid in Peanut Seeds and Peanut Oil by High Spectral Imaging and Near Infrared Spectroscopy. PhD Dissertation of Chinese Academy of Agricultural Sciences, Beijing, China, 2016 (in Chinese with English abstract).
[9] Chamberlin K D, Melouk H A, Madden R, Dillwith J W, Bannore Y, Rassi Z E, Payton M. Determining the oleic/linoleic acid ratio in a single peanut seed: a comparison of two methods., 2011, 38: 78–84.
[10] 禹山林, 杨庆利, 潘丽娟, 曹玉良, 闵平, 朱雨杰. 一种非破坏性测定花生种子主要脂肪酸含量的方法. 中国专利, 2009, CN101887018B.Yu S L, Yang Q L, Pan L J, Cao Y L, Min P, Zhu Y J. A nondestructive method for the determination of main fatty acids in peanut seeds. Chinese patent, 2009, CN101887018B (in Chinese).
[11] 张严, 谢岩黎, 孙淑敏, 马明扬, 李琳琳. 近红外分析花生籽粒脂肪酸含量的研究. 河南工业大学学报(自然科学版), 2014, 35(2): 54–58. Zhang Y, Xie Y L, Sun S M, Ma M Y, Li L L. Analysis of fatty acids in peanut seeds by near infrared spectroscopy.(Nat Sci Edn), 2014, 35(2): 54–58 (in Chinese with English abstract).
[12] Chamberlin K D, Barkley N A, Tillman B L, Dillwith J W, Madden R, Payton M E, Bennett R S. A comparison of methods used to determine the oleic/linoleic acid ratio in cultivated peanut (L.)., 2014, 5: 227–237.
[13] Lee J M, Pae S B, Choung M G, Lee M H, Kim S U, Oh E Y, Oh K W, Jung C S, Oh I S. Determination of fatty acid composition in peanut seed by near infrared reflectance spectroscopy., 2016, 61: 64–69.
[14] 王传堂. 生物技术和近红外技术在花生育种中的应用. 中国海洋大学博士学位论文, 山东青岛, 2010. Wang C T. Application of Biotechnology and NIRS in Peanut Breeding. PhD Dissertation of Ocean University of China, Qingdao, Shandong, China, 2010 (in Chinese with English abstract).
[15] 张丽君, 刘龙龙, 马名川, 崔林, 周建萍. 燕麦蛋白质近红外定量模型的创建及其在育种中的应用. 安徽农业科学, 2017, 45(8): 10–13. Zhang L J, Liu L L, Ma M C, Cui L, Zhou J P. Establishment of near infrared quantitative model of oat protein and its application in breeding., 2017, 45(8): 10–13 (in Chinese with English abstract).
[16] Tillman B L, Gorbet D W, Person G. Predicting oleic and linoleic acid content of single peanut seeds using near-infrared reflectance spectroscopy., 2006, 46: 2121. doi: 10.2135/cropsci2006. 01.0031.
[17] Kavera, Nadaf H L, Hanchinal R R. Near infrared reflectance spectroscopy (NIRS) for large scale screening of fatty acid profile in peanut (L.)., 2014, 37: 272. doi: 10.5958/j.0976-0571.37.3.041.
[18] 迟晓元, 郝翠翠, 陈明娜, 潘丽娟, 陈娜, 王通, 王冕, 杨珍, 梁成伟, 禹山林. 花生基因与油酸/亚油酸比值的关系. 花生学报, 2016, 45(4): 20–24. Chi X Y, Hao C C, Chen M N, Pan L J, Chen N, Wang T, Wang M, Yang Z, Liang C W, Yu S L. Relationship betweengene and oleic acid/linoleic acid ratio in Peanut., 2016, 45(4): 20–24 (in Chinese with English abstract).
[19] 禹山林, 朱雨杰, 闵平, 杨庆利, 曹玉良, 王传堂, 刘旭, 周学秋. 傅立叶近红外漫反射非破坏性测定花生种子主要脂肪酸含量. 花生学报, 2010, 39(1): 11–14. Yu S L, Zhu Y J, Min P, Yang Q L, Cao Y L, Wang C T, Liu X, Zhou X Q. Nondestructive determination of main fatty acids in peanut seeds with near infrared reflectance spectroscopy., 2010, 39(1): 11–14 (in Chinese with English abstract).
[20] 徐平丽, 唐桂英, 付春. 高通量检测花生油酸含量相关基因等位变异的方法. 农业生物技术学报, 2016, 24: 1364–1373. Xu P L, Tang G Y, Fu C. Methods for high-throughput detecting the allelic variation ofgene related with oleic acid content in peanut (L.)., 2016, 24: 1364–1373 (in Chinese with English abstract).
[21] 只升华, 苏同兵, 于拴仓, 张凤兰, 余阳俊, 张德双, 赵岫云, 汪维红, 卢桂香, 朱月林. 利用全基因组关联分析获得白菜A01染色体定位的霜霉病抗病位点和相关分子标记开发. 植物生理学报, 2016, 52: 693–702. Zhi S H, Su T B, Yu S C, Zhang F L, Yu Y J, Zhang D S, Zhao X Y, Wang W H, Lu G X, Zhu Y L. Genetic characteristics of A01-located resistant loci to downy mildew in Chinese cabbage by genome-wide association studies., 2016, 52: 693–702 (in Chinese with English abstract).
[22] 张照华, 王志慧, 淮东欣, 谭家壮, 陈剑洪, 晏立英, 王晓军, 万丽云, 陈傲, 康彦平, 姜慧芳, 雷永, 廖伯寿. 利用回交和标记辅助选择快速培育高油酸花生品种及其评价. 中国农业科学, 2018, 51: 1641–1652. Zhang Z H, Wang Z H, Huai D X, Tan J Z, Chen J H, Yan L Y, Wang X J, Wan L Y, Chen A, Kang Y P, Jiang H F, Lei Y, Liao B S. Fast development of high oleate peanut cultivars by using marker-assisted backcrossing and their evaluation., 2018, 51: 1641–1652 (in Chinese with English abstract).
[23] 吴静珠, 刘翠玲, 李慧, 孙晓荣. 近红外光谱技术在食用油种类鉴别及脂肪酸含量检测中的应用. 食品科学技术学报, 2010, 28(5): 56–59. Wu J Z, Liu C L, Li H, Sun X R. Application of near infrared spectroscopy in identification of edible oils and detection of fatty acids content., 2010, 28(5): 56–59 (in Chinese with English abstract).
[24] 周小华, 张玫, 相秉仁. 近红外光谱结合区间偏最小二乘法应用于花生油酸价的测定. 粮油食品科技, 2017, 25(2): 62–64. Zhou X H, Zhang M, Xiang B R. Detetmination of fatty acid value of peanut oil by near-infrared spectrometry combined with interval partial least squares., 2017, 25(2): 62–64 (in Chinese with English abstract).
[25] 张建成, 王传堂, 王秀贞, 唐月异, 张树伟, 李桂杰. 花生自然风干种子油酸、亚油酸和棕榈酸含量的近红外分析模型构建. 中国农学通报, 2011, 27(3): 90–93. Zhang J C, Wang C T, Wang X Z, Tang Y Y, Zhang S W, Li G J. NIRS calibration models predictive of oleic, linoleic and palmitic acid content in sun-dried bulk peanut seed samples., 2011, 27(3): 90–93 (in Chinese with English abstract).
[26] 张照华. 利用MAS回交技术创制花生高油酸新种质及基因遗传效应分析. 中国农业科学院硕士学位论文, 北京, 2018. Zhang Z H. Creation of New Peanut Germplasm with High Oleic Acid Using MAS Backcrossing Technology and the Genetic Effect ofGene. MS Thesis of Chinese Academy of Agricultural Sciences, Beijing, China, 2018 (in Chinese with English abstract).
[27] Barkley N A, Isleib T G, Wang M L, Pittman R N. Genotypic effect ofon fatty acid profiles in six segregating peanut (L.) populations., 2013, 14: 62. doi: 10.1186/1471-2156-14-62.
Establishment and applicant of near-infrared reflectance spectroscopy models for predicting main fatty acid contents of single seed in peanut
LI Jian-Guo, XUE Xiao-Meng, ZHANG Zhao-Hua, WANG Zhi-Hui, YAN Li-Ying, CHEN Yu-Ning, WAN Li-Yun, KANG Yan-Ping, HUAI Dong-Xin*, JIANG Hui-Fang, LEI Yong*, and LIAO Bo-Shou
Oil Crops Research Institute, Chinese Academy of Agricultural Sciences / Key Laboratory of Biology and Genetic Improvement of Oil Crops, Ministry of Agriculture, Wuhan 430062, Hubei, China
Profile of fatty acid is the main factor determining the nutritional value and shelf life of peanut. High oleate peanut has been increasingly favored by customers and peanut processing enterprises, as it provides a prolonged shelf-life and a beneficial effect on human health. Therefore, creating high oleate peanut cultivar is an important objective for peanut breeding. Developing a nondestructive method which could detect the fatty acid composition in peanut rapidly, efficiently and accurately is an important technical support for accelerating the processing. In this study, models for predicting the contents of main fatty acids (oleic acid, linoleic acid and palmitic acid) in a single peanut seed were built up using near-infrared reflectance spectroscopy (NIRS). The coefficient of multiple determination (2) and the root mean squared error of external calibration (RMSECV) of prediction models were 0.907 and 3.463 for oleic acid, 0.918 and 2.824 for linoleic acid, 0.824 and 0.782 for palmitic acid, respectively. One hundred peanut seeds were analyzed by both NIR and gas chromatography (GC) to validate the accuracy of the prediction models. The correlation coefficients of oleic acid, linoleic acid and palmitic acid between NIR values and GC values were 0.88, 0.90, and 0.71, respectively, suggesting that these models could accurately predict the contents of the three main fatty acids in a single peanut seed. Furthermore, based on these prediction models, a breeding method of high oleate peanut without assistance of molecular marker was developed, and a high oleate peanut cultivar Zhonghua 215 was successfully bred.
single peanut seed; fatty acid; near infrared model; high oleic acid; breeding method
本研究由玛氏-中国高油酸花生育种计划项目(MARS-China HOAP 2013-2018), 国家自然科学基金项目(31671734, 31461143022, 31770250, 31371662), 中央级科研院所基本科研业务费专项(Y2018PT52)和国家现代农业产业技术体系建设专项(CARS-13)资助。
This study was supported by the Mars-China High Oleic Acid Peanut Breeding Project (MARS-China HOAP 2013-2018), the National Natural Science Foundation of China (31671734, 31461143022, 31770250, 31371662), the Fundamental Research Funds for Central Non-profit Scientific Institution (Y2018PT52), and the China Agriculture Research System (CARS-13).
淮东欣, E-mail: dxhuai@caas.cn; 雷永, E-mail: leiyong@caas.cn
E-mail: 15611821765@163.com
2019-05-15;
2019-06-20;
2019-07-16.
URL: http://kns.cnki.net/kcms/detail/11.1809.S.20190715.1610.006.html
10.3724/SP.J.1006.2019.94016
