大豆抗SC3候选基因的克隆及分析
2019-11-13向文扬杨永庆任秋燕晋彤彤王丽群王大刚智海剑
向文扬 杨永庆 任秋燕 晋彤彤 王丽群 王大刚 智海剑
大豆抗SC3候选基因的克隆及分析
向文扬 杨永庆 任秋燕 晋彤彤 王丽群 王大刚 智海剑*
南京农业大学大豆研究所/ 农业部大豆生物学与遗传育种重点实验室/ 国家大豆改良中心/ 作物遗传与种质创新国家重点实验室, 江苏南京 210095
大豆花叶病毒(, SMV)病是大豆主要的病害之一, 给我国大豆生产带来了巨大的损失。大豆抗病育种是目前防治大豆花叶病毒病最为经济有效的措施, 发掘抗病基因是抗病育种的基础。本文在前期对大豆抗SMV株系SC3基因精细定位的基础上, 克隆了2个具有TIR-NBS-LRR典型抗病结构域的基因(和)。生物信息学分析表明,和基因均在抗感品种中存在氨基酸位点的突变, 而且突变位点都位于保守结构域内, 这2个基因编码的蛋白质预测为烟草花叶病毒(TMV)抗性N蛋白; 物种间同源比对结果显示,和基因与野生大豆亲缘较近。qRT-PCR结果表明,和能够响应SMV的侵染增加表达量, 且在抗病品种中的表达量高于感病品种。2个基因存在IN1、IN2和IN3不同的剪接体, 所有的剪接体都能够响应病毒的诱导增加表达量, 且在抗病品种中的表达量高于感病品种, IN1和IN2的表达量随时间的变化较为明显, IN3的表达量则相对稳定, 说明这些剪接体可能参与大豆对SMV的抗病过程。本研究为后续基因功能的研究奠定了基础。
大豆花叶病毒; 抗病基因; 诱导表达; 可变剪接
大豆花叶病毒病是由大豆花叶病毒(SMV)引发的一种大豆病害, 在我国主要大豆产区都有分布, 给我国大豆的产量和品质带来严重影响[1]。目前大豆花叶病毒的防治主要从3个方面入手, 第一是控制SMV初侵染源, 带毒大豆种子是SMV初侵染来源[2], 优选大豆种子, 特别是不用前一年发病植株上收获的种子可以一定程度上防治SMV的危害; 第二是控制SMV的田间传播, 蚜虫是SMV田间传播的主要媒介[1,3], 及时发现并杀灭蚜虫能减少SMV在田间的传播; 第三是选育优良的大豆抗SMV品种, 这是最经济有效的防治方法。
国内外已经有许多关于抗SMV基因研究的报道。美国学者从不同的抗SMV种质中分别发现了4个抗病位点、、和[4-7], 并将其定位在D1b (第2染色体)、F (第13染色体)和B2 (第14染色体) 3个连锁群上, 并在随后的研究中发现这些抗病位点的多个等位位点; 我国针对不同的SMV株系从抗性种质中定位出了多个抗病基因, 分别命名为R抗病位点等, 这些基因分布在D1b、F、C2 (第6染色体)和B2四个连锁群上。从物理和遗传图谱位置上来看, 美国和我国定位出来的抗病基因可能存在重叠或者处于相近区域。
目前关于基因(resistance gene)的研究在国内外有很多的报道, 在不同的物种中已经有几十个抗性基因被成功克隆[8-11]。这些基因介导对不同病原的抗性, 尽管其来源不同, 但是绝大多数的基因具有高度保守的蛋白结构域, 其中典型的保守结构域包括核苷酸结合位点(nucleotide binding site, NBS)、亮氨酸拉链结构(leucine zipper, LZ)、丝氨酸/苏氨酸蛋白激酶结构域(serine-threonine kinase, STK)、toll蛋白/白细胞介素-1受体类似结构域(toll- interleukin-1 receptor, TIR)、亮氨酸富集重复结构(leucine-rich repeat, LRR)、跨膜结构域(transmembrane domain, TM)等, 其中TIR-NBS-LRR类结构基因是目前鉴定的基因中最多的一类。
大豆抗SMV基因的分离和鉴定已经取得了一定的研究进展。Davis[12]在大豆品种L29中发现了抗SMV候选基因, 并通过烟草瞬时表达系统在烟草中共表达该基因与大豆花叶病毒蛋白CI, 结果表明该基因与SMV蛋白CI相互作用导致烟草植株出现了坏死症状, 说明可能参与对SMV的抗性。Tran等[13]在不含的大豆品种瞬时表达使其获得了对SMV株系G5H的系统抗性; 同时使用病毒诱导的基因沉默技术(virus-induced gene silencing, VIGS)沉默含有大豆品种中的使其丧失了对G5H的抗性。G533和实际上为同一个基因, 该结果初步确认基因与大豆对SMV的抗性有关。
大部分的植物基因含有内含子, 这些基因转录形成的前体mRNA需要经过剪接后才能形成成熟的mRNA, 前体mRNA的剪接方式可以分为组成性剪接(constitutive splicing)和选择性剪接(alternative splicing)[14-15], 其中选择性剪接是高等生物普遍存在的一种基因转录后调控机制, 能够增加基因产物的多样性, 提高植物对环境变化的适应能力, 已有研究表明, 可变剪接在植物抗病反应、抗胁迫逆境反应及生长发育过程中发挥着十分重要的作用[16-18]。按照剪接方式的不同, 可变剪接主要分为内含子保留(intron retention)、外显子丢失(exon skip)、可变供体(alternative donor site)、可变受体(alternative donor site)及外显子互斥(mutually exclusive exons)等[19], 内含子保留是最为常见的可变剪接形式[20]。研究植物的可变剪接可以提高对植物基因功能多样性的理解, 目前关于大豆抗SMV基因定位的研究已有很多,但鉴定分离成功的抗病基因还很少, 候选基因可变剪接体的分析可以为大豆抗SMV基因的分离鉴定提供全新的视角。
杨永庆等[21]在PI96983品种中将大豆抗SC3基因R定位在了大豆第13染色体为345 kb的物理区间内, 本文在该研究的基础上对这个区间内的可能的抗病基因进行筛选, 搜索到了2个高度同源(99%同源性)的具有TIR-NBS-LRR典型抗病结构域的基因和, 克隆这2个基因并对其进行生物信息学分析, 同时检测了这2个基因及其剪接体在病毒诱导条件下的表达, 初步探讨了和的结构及其可能的功能, 为后续基因功能的研究奠定了基础。
1 材料与方法
1.1 材料
大豆品种南农1138-2、齐黄1号、Williams 82及PI96983均由国家大豆改良中心保存提供, SMV株系SC3由本课题组保存提供。其中齐黄1号和PI96983为抗SMV株系SC3的品种, Wiliams 82和南农1138-2是 SMV株系SC3的感病品种。
1.2 材料种植及处理
本实验所使用的大豆材料均种植于温度为25℃、湿度为65℃、光照10 h/黑夜8 h的恒温培养室中; 在大豆第1对真叶展开时利用摩擦接种法[22]接种, 实验组处理为接种SMV株系SC3, 对照组为接种磷酸缓冲液(0.01 mol L-1磷酸钠, 0.01 mol L-1磷酸钾或0.05 mol L-1柠檬酸钠, pH 7)。
1.3 植物总RNA的提取及第1链cDNA的反转录
提取植物总RNA的严格按照RNAprep pure Plant Kit (购于TIANGEN)说明书。利用琼脂糖凝胶电泳法对提取的RNA进行质量检测, 选取质量合格的RNA进行第1链cDNA的反转录, 按照PrimeScript 1st Strand cDNA Synthesis Kit (购于TAKARA)说明书进行反转录。
1.4 基因克隆
由于及具有高度同源性, 故采用特异性较高的巢式PCR (Nested-PCR)进行扩增。以NCBI上公布的Williams 82基因组Wm82.a2.v1数据库为参考, 利用Primer premier 5软件设计引物(表1)。首先使用引物对GmR47-1和GmR51-1进行第1步PCR, 反应体系为50 μL, 包含酶25 μL 2×PrimerStar Master Mix (Takara)、上下游引物(浓度为10 μmol L-1)各1 μL、模板cDNA 1 μL、灭菌ddH2O 22 μL。PCR程序为95℃ 3 min; 98℃ 10 s, 58℃ 15 s, 72℃ 45 s, 30个循环; 72℃ 5 min。然后使用引物对GmR47-2和GmR51-2进行第2步PCR, 反应体系为50 μL, 包含酶25 μL 2×PrimerStar Master Mix (Takara)、上下游引物(浓度为10 μmol L-1)各1 μL、模板为第1步PCR产物稀释100倍后取1 μL、灭菌ddH2O 22 μL。反应程序同上。利用琼脂糖凝胶电泳法检测第2步PCR反应产物, 回收条带大小正确的产物。并将回收产物使用T4连接酶连接至T-easy载体pMD20-T, 将连接产物转化大肠杆菌感受态DH5α中过夜培养, 挑取单克隆, 将检测为阳性的单克隆送至通用生物公司测序。
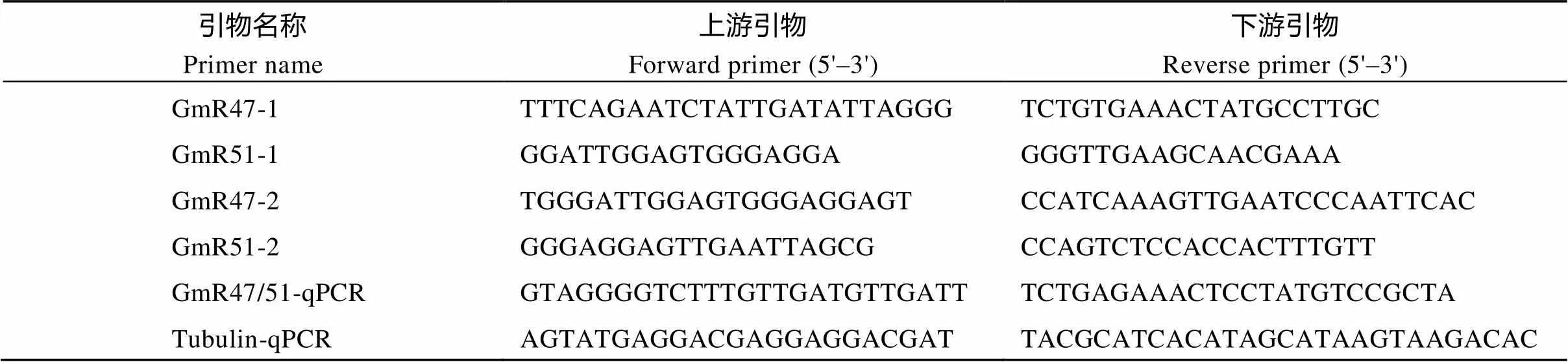
表1 基因克隆引物及荧光定量引物
引物名称中含有qPCR的为实时荧光定量引物。
The primer’s name contained ‘qPCR’ means its Quantitative Real-time PCR (qRT-PCR) primer.
1.5 序列生物信息学分析
比对碱基及氨基酸序列使用BioXM软件; 通过NCBI BLASTP (https://blast.ncbi.nlm.nih.gov/Blast. cgi)搜索下载烟草、拟南芥、野生大豆等10个不同植物中候选基因的氨基酸同源序列, 使用MEGA 7软件及邻近法(Neighbor-Joining, NJ)构建氨基酸序列系统进化树; 使用Interpro (http://www.ebi.ac.uk/ interpro)软件分析蛋白质保守结构域及预测功能; 使用SOPMA软件(https://npsa-prabi.ibcp.fr/cgi-bin/)预测蛋白质的二级结构, 使用Phyre2 (http://www. sbg.bio.ic.ac.uk/phyre2)软件预测蛋白质的三级结构。
1.6 荧光定量
按照荧光定量试剂盒ChamQ Universal SYBR qPCR Master Mix (购于VEZYME)说明书进行荧光定量反应, 内参基因选择(登录号为AY907703), 每个样品和内参均设置3个平行实验, qPCR条件为95℃ 1 min; 98℃ 10 s, 60℃ 15 s, 72℃ 20 s, 40个循环。和相似度高达99%, 无法利用1对引物将其区分, 故将这2个基因的表达量统一讨论,及荧光定量引物见表1。分析荧光定量结果采用2-DDCt法[23], 以接种磷酸缓冲液的对照组候选基因的表达量为基准品, 计算接种SC3实验组候选基因的相对表达量。
1.7 可变剪接体的检测及诱导表达分析
和均存在3个含子, 分别在这3个内含子处设计了2对引物, 1对是跨内含子, 设计在2个相邻外显子上的常规引物, 另外1对是引物在内含子内的特异性引物, 内含子检测引物见表2。以PI96983、南农1138-2和齐黄1号3个大豆品种的cDNA为模板进行PCR, 检测可变剪接是否在这2个基因中存在, 此处使用的cDNA模板经过去基因组DNA处理, 排除cDNA模板中的基因组DNA的污染。按照HiScript III RT SuperMix for qPCR (+gDNA wiper)荧光反转录试剂盒(购于VEZYME)说明书进行去基因组DNA污染的反转录。可变剪接体的诱导表达分析以每个内含子处的总剪接体数为单位进行, 每个剪接体处的荧光定量引物见表2。

表2 可变剪接检测引物及荧光定量引物
引物名中含有Uni的为不含特异性的常规引物; 含Spe的为内含子上的特异性引物; 含qPCR的为荧光定量引物。
The primer’s name contained ‘Uni’ means the conventional primers without specificity; those contained ‘Spe’ means intron specific primer; those contained ‘qPCR’ means qRT-PCR primer.
1.8 亚细胞定位
使用在引物5'端含有GateWay元件的引物扩增和完整的开放阅读框, 其中不包含终止子。利用GateWay系统构建载体, 经过BP反应及LR反应, 将目的基因构入pGWB5载体获得目的基因与融合的载体pGWB5--和pGWB5--。使用农杆菌注射的方法侵染烟草[24], 侵染烟草48 h后, 利用Leica TCS SP2激光共聚焦荧光显微镜观察注射烟草细胞中荧光信号的分布。
2 结果与分析
2.1 候选基因的克隆
通过已公布的Williams 82基因组数据, 分别获得和的参考序列, 比对参考序列可知2个基因具有高度同源性。克隆大豆品种PI96983中的抗病候选基因和, 设计2对特异性引物分别对这2个基因进行巢式PCR扩增, 以大豆品种PI96983的cDNA为模板扩增获得大小为3500 bp左右的目标片段。测序结果显示, 大豆品种PI96983中和基因均含有一个大小为3288 bp的开放阅读框(ORF), 均编码1095个氨基酸残基。编码合成的蛋白质分子量为125.34 kD, 等电点为6.69;编码合成的蛋白质分子量为125.4 kD, 等电点为6.64。
2.2 候选基因生物信息学分析
和编码的蛋白产物含有相同的蛋白结构域, 从N端到C端分别为TIR、NBS及LRR结构域, 是典型的TIR-NBS-LRR家族基因(图1), 预测的蛋白功能为烟草花叶病毒(TMV)抗性N蛋白。、在抗病品种PI96983及感病品种南农1138-2之间的氨基酸序列差异(图2)显示,在抗感品种之间存在8个SNP的差异, 导致5个氨基酸突变, 突变位点在TIR、NBS和LRR结构域中均有分布;在抗感品种之间存在8个SNP差异, 导致3个氨基酸突变, 突变位点分布在NBS和LRR结构域中。和在不同物种间的同源比对结果(图3)显示,和基因在野生大豆中的亲缘性最近, 其次是苜蓿。
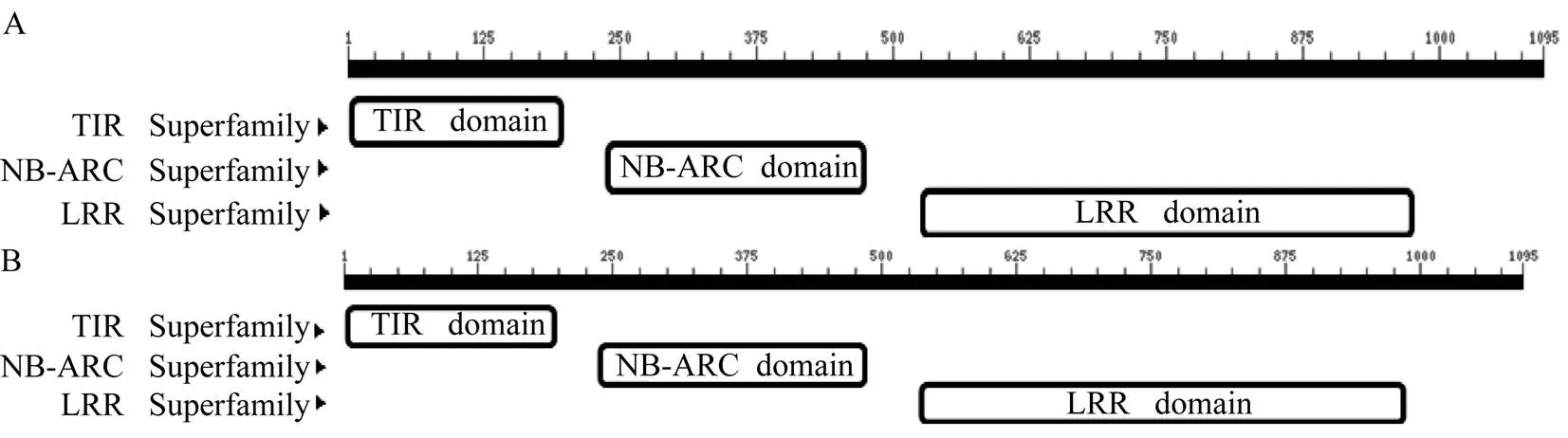
图1 GmR47和GmR51编码蛋白质的保守结构域预测
A:编码蛋白质的保守结构域预测。B:编码蛋白质的保守结构域预测。
A: conservative domain prediction of-encoded proteins. B: conservative domain prediction of-encoded proteins.

图2 GmR47和GmR51编码的核苷酸序列在抗病品种(PI96983)及感病品种(南农1138-2)之间的比对结果
A: GmR47编码氨基酸序列的比对结果。B: GmR51编码氨基酸序列比对的结果。
A: alignment result of amino acid sequence encoded by GmR47; B: alignment result of amino acid sequence encoded by GmR51.
2.3 GmR47和GmR51基因编码蛋白的二级结构和三级结构预测
图4-A表明, 在二级结构中α螺旋结构占45.84%, 延伸链占15.07%, β转角结构占5.57%, 不规则卷曲占33.52%;编码的蛋白产物二级结构(图4-B)中α螺旋结构占45.02%, 延伸链占16.07%, β转角结构占5.57%, 不规则卷曲占33.33%。抗感病品种中和编码蛋白产物预测三级结构见图5, 显示和基因编码蛋白产物三级结构在抗感品种中存在差异。
2.4 候选基因在抗感品种之间的表达量差异
利用SC3接种抗感品种诱导候选基因的表达, 检测抗感品种之间表达量的差异。实验组采用SMV株系SC3接种, 对照组用磷酸缓冲液处理。分别在处理后的0、1、2、4、8、12、24、48、72 h以及7 d采集对照组和实验组的处理叶片。荧光定量的结果显示(图6), 候选基因在抗病品种中的表达量变化趋势是先升后降再升, 分别在2 h、7 d达到峰值, 最高值约为未接种时表达量的2倍; 在24 h达到最低值, 此时表达量与未接种时持平。候选基因在感病品种表达量的变化不大, 在12 h时取得最低值, 约为未接种时表达量的0.5倍, 在24 h达到峰值, 约为未接种时表达量的1.6倍; 由此看出, 感病品种中候选基因的表达量相对平稳, 而在抗病品种中表达量则有较大的起伏变化, 在抗病品种中的表达量要高于感病品种中。说明候选基因和能够积极的响应SMV的诱导提高表达量。
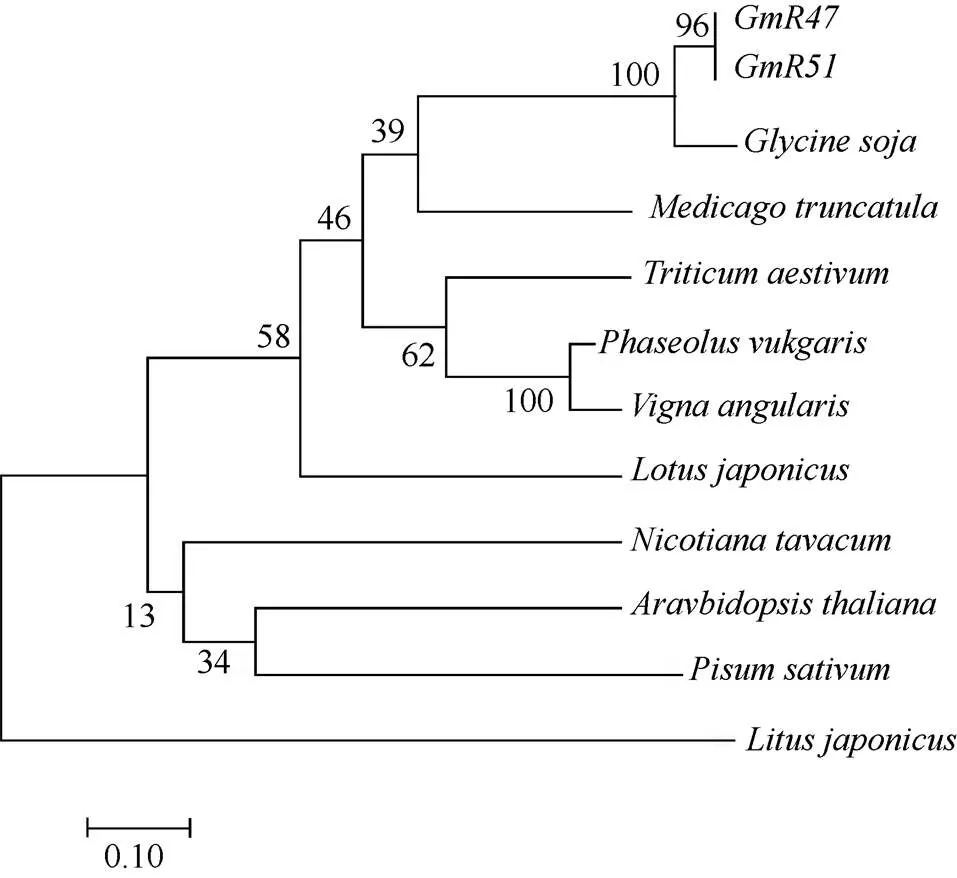
图3 GmR47和GmR51基因在不同物种间的同源进化树
图4 GmR47 (A)和GmR51 (B)基因编码的氨基酸序列二级结构预测结果
2.5 可变剪接体的分析
和基因均存在3个内含子, 为检测其内含子处的可变剪接现象, 在每个内含子处设计了1对常规引物和1对特异性引物, 以齐黄1号、PI96983、南农1138-2和Williams 82的去基因组污染的cDNA为模板进行PCR扩增。扩增的结果显示(图7), 在这3个内含子处, 跨内含子设计在相邻外显子上的常规引物均扩增出2条或2条以上的条带, 且设计在内含子上的特异性引物同样扩增出了条带。说明和基因在这3个内含子处均存在可变剪接的现象, 并且可变剪接现象在4个大豆品种中均存在。将常规引物PCR扩增产物回收后测序显示(图8), 3个内含子处均存在内含子保留可变剪接体, 由于和基因的高度同源性, 未能检测出每个基因的不同可变剪接体。
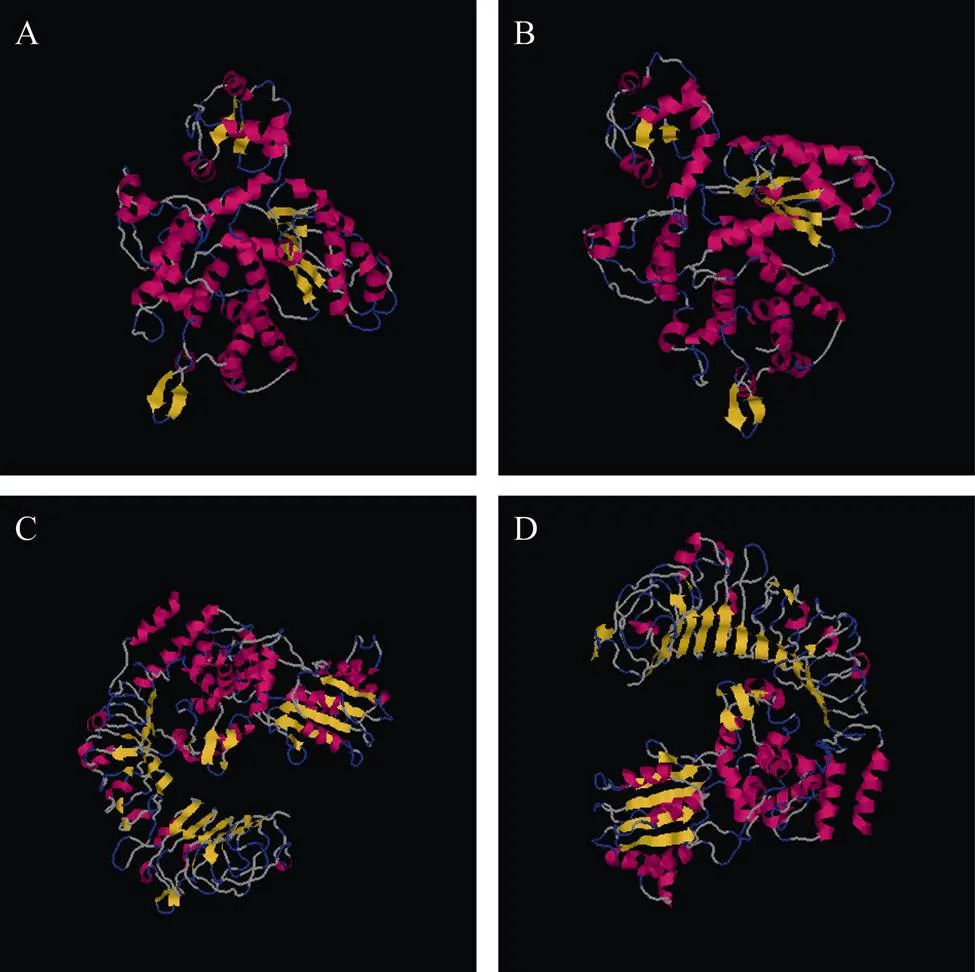
图5 抗感品种中GmR47和GmR51基因编码合成的蛋白质三维结构预测结果
A: PI96983; B: PI96983; C: 南农1138-2; D: 南农1138-2。
A: PI96983; B: PI96983; C: Nannong 1138-2; D: Nannong 1138-2.
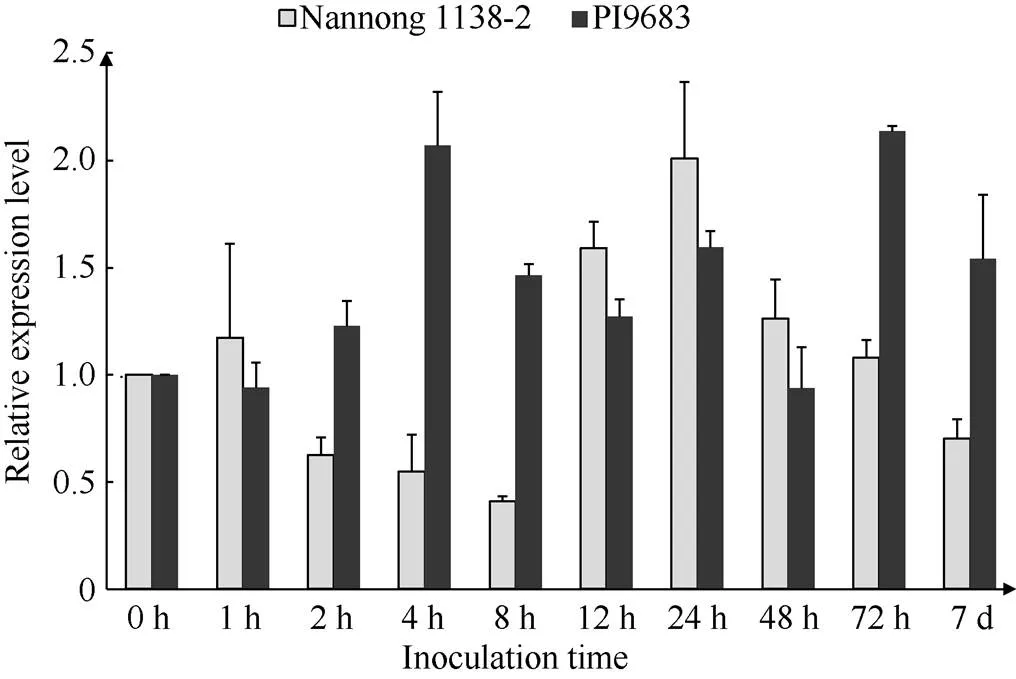
图6 PI96983和南农1138-2接种SMV后GmR47及GmR51基因的总表达量随时间的变化
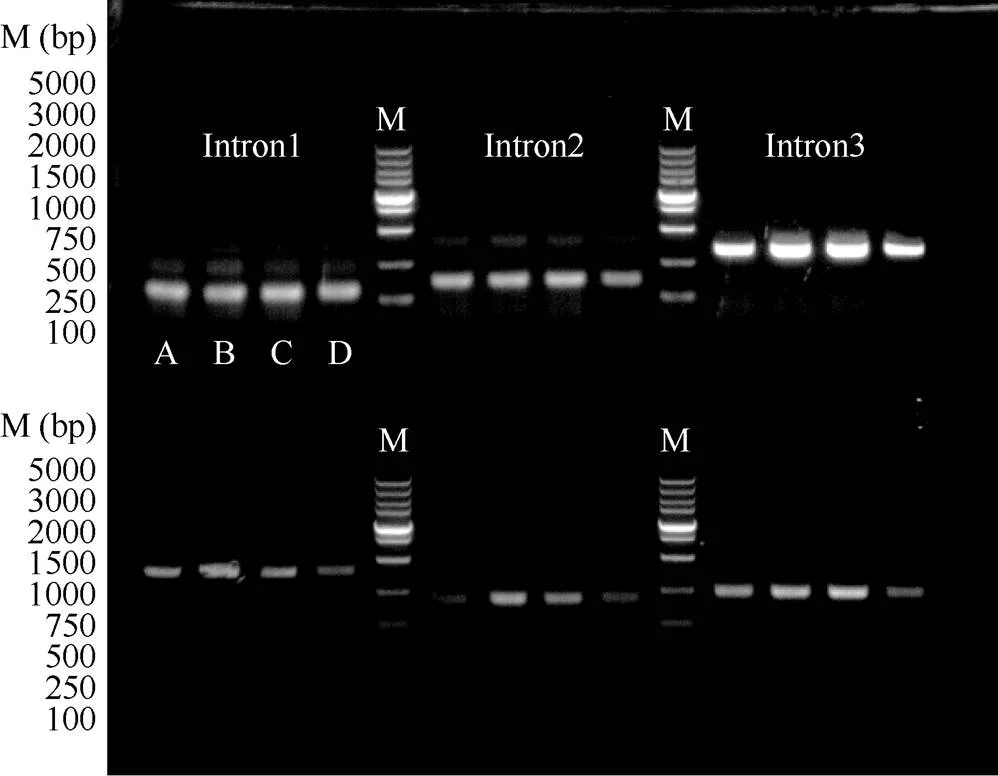
图7 可变剪接体的凝胶电泳检测结果
上排条带为常规引物扩增的结果, 下排条带为内含子特异性引物的扩增结果, 相邻的4个条带为4个品种间的扩增结果。A: 齐黄1号; B: PI96983; C: 南农1138-2; D: Williams 82; M: marker。
The upper bands are the result of amplification of theconventional primer, the lower bands are the amplification result of the intron-specific primer, adjacent four bands are amplification from four varieties. A: Qihuang 1; B: PI96983; C: Nannong 1138-2; D: Williams 82; M: marker.
由于和基因具有高度同源性, 且基因组较大, 无法单独对某个剪接体进行分析, 为研究不同剪接体对SMV的响应情况, 本文以每个内含子处存在的总剪接体为单位进行表达量分析, 即在每个内含子内设计特异性引物进行荧光定量, 将3个内含子处存在的总剪接体分别命名为IN1、IN2和IN3。可变剪接诱导表达所采用的实验材料为PI9683和南农1138-2, 实验组处理为接种SC3病毒, 对照组为接种磷酸缓冲液, 抗病品种及感病品种均设置实验组和对照组。在处理后的0、1、2、4、8、12、24、48、72 h以及7 d内采集处理叶片, 并进行荧光定量分析。荧光定量结果(图9)显示, IN1的表达量(图9-A)在抗病品种中的趋势是先升再降再升, 在感病品种中的趋势是先降再升再降, 其中2、4、8、72 h在抗感品种之间存在显著差异; IN2的表达量(图9-B)在抗感病品种中的变化趋势是先升再降, 抗病品种中候选基因在24 h表达量达到峰值, 在感病品种中在12 h表达量达到峰值; IN3的表达量(图9-C)在抗病品种中的变化趋势是先升后维持较高表达水平, 在感病品种中表达量变化不大。以上结果表明, IN1、IN2和IN3的的表达量都能响应病毒的诱导, 并且抗病品种中的表达量要高于感病品种。不同内含子间可变剪接体的表达量存在差异, IN1表达量随病毒侵染时间的变化较为明显, 而IN2和IN3的表达量则相对平稳。总的来说,和在3个内含子处均含有可变剪接现象, 而且能够响应病毒的诱导表达, 说明这些可变剪接体可能参与大豆对SMV的抗性。
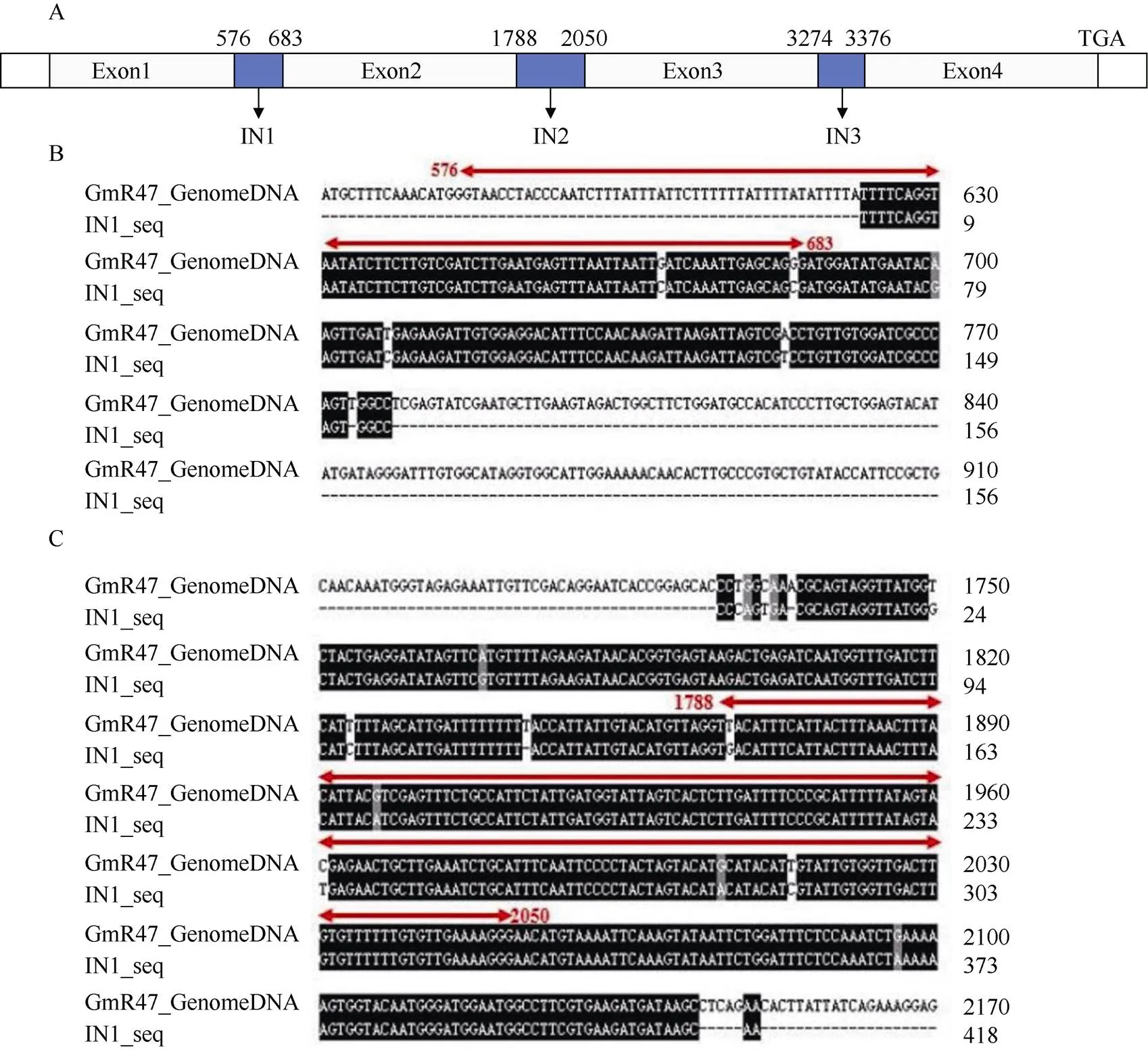
(图8)
A:基因可变剪接体示意图; B: IN1序列; C: IN2序列; D: IN3序列。
A: schematic diagram of alternative splicing ofgene; B: IN1 sequence; C: IN2 sequence; D: IN3 sequence.
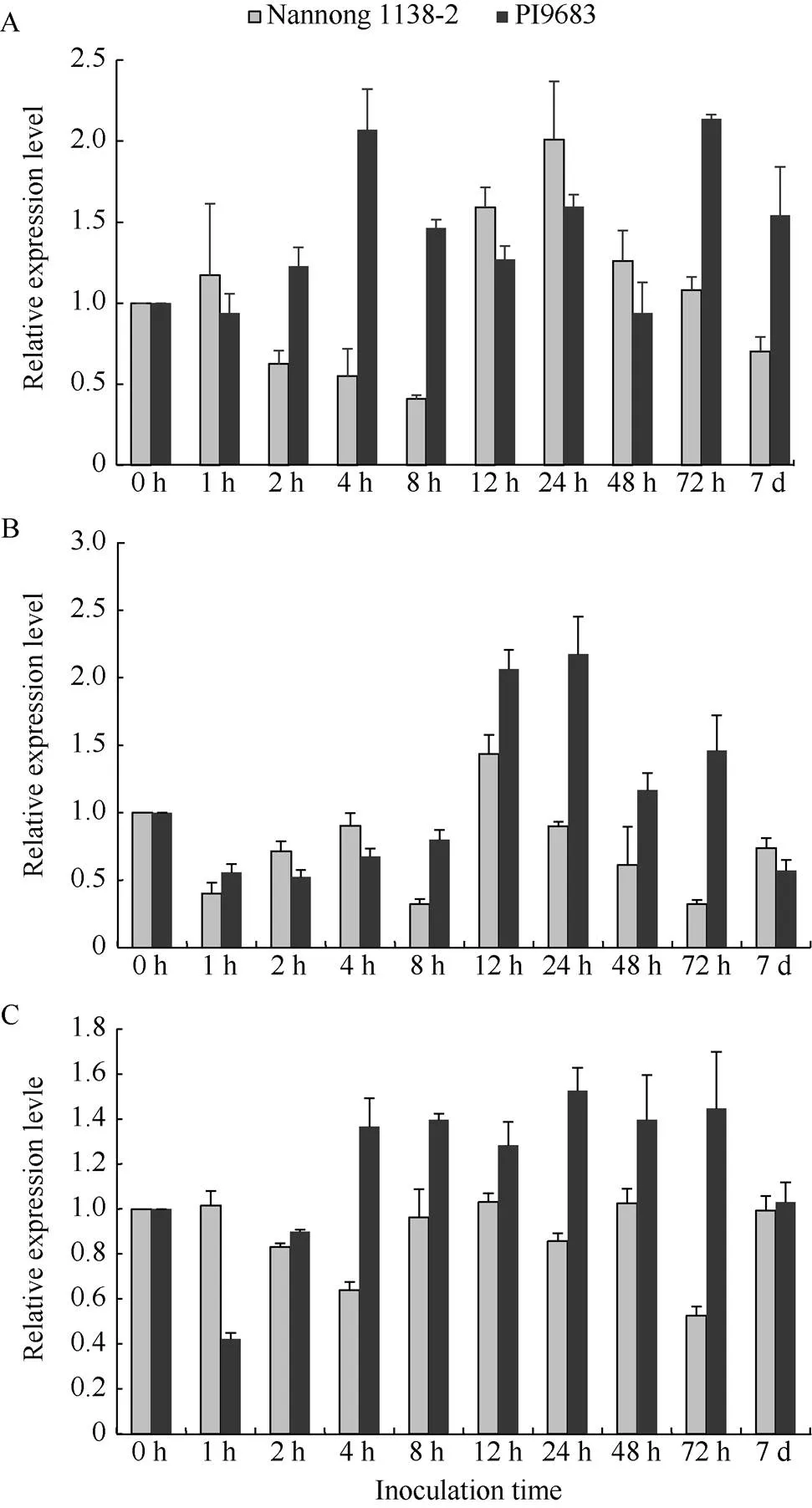
图9 PI96983和南农1138-2接种SMV后GmR47及GmR51基因在不同内含子处的剪切体的总表达量随时间的变化
A: 内含子1处的剪接体的总表达量; B: 内含子2处的剪接体的总表达量; C: 内含子3处的剪接体的总表达量。
A: total expression of alternative splicing in Intron 1; B: total expression of alternative splicing in Intron 2; C: total expression of alternative splicing in Intron 3.
2.6 候选基因的亚细胞定位
利用Cell-PLoc (http://www.csbio.sjtu.edu.cn/ bioinf/Cell-PLoc-2/)对和编码的蛋白质作用的位置预测显示,编码的蛋白产物分布在细胞膜上,编码的蛋白产物分布在细胞膜和细胞质中。构建植物表达载体pGWB5--及pGWB5--并利用PCR法, 酶切法和测序法对其验证, 选择构建正确的载体进行烟草侵染。在侵染烟草后的48 h观察烟草细胞中的荧光分布, 结果显示(图10), pGWB5--和pGWB5--融合蛋白在在细胞膜和核膜上有荧光信号, 证实了和编码的蛋白产物均分布在细胞膜和核膜上, 以上结果说明,和编码的蛋白产物为膜蛋白。
3 讨论
本文发现候选基因能响应SMV的侵染而增加表达量, 说明候选基因能够参与对SMV的抗性反应, 但其表达量与CP含量的变化趋势并不一致, 推测其原因可能是植物抗病过程由多个基因共同作用, 单个基因的高表达并不一定能使抗性增强而直接降低病毒量。
美国以不同抗性的种质定位出、等抗病基因, 目前已经从PI96983中初步鉴定了具有NBS-LRR结构的抗SMV基因[25], 该基因有可能为, 从遗传距离和物理距离上来看,和我国定位的距离很近, 可能互为等位基因或处于相近区域; 韩国学者鉴定L29中的为抗病基因, 证明其有可能为[8], 该基因与我国大白麻含有的基因位点相同, 初步说明L29与大白麻可能含有相同抗病基因; 在我国, 目前确定为抗SMV基因的候选基因还较少,黄赛花等[26]利用VIGS沉默齐黄1号中R候选基因使抗SMV植株的抗病性发生改变, 初步证明为抗病基因参与齐黄1号对SMV的抗性。本文根据生物信息学和表达量分析的结果, 初步推测和基因为抗SMV基因, 至于候选基因是否为SMV抗性基因, 还需要VIGS、转基因以及CRIPSR-CAS9基因靶向编辑等实验后续证明。
可变剪接可以使一个基因生成多种转录产物, 极大地提高基因产物的多样性, 是一种重要的转录后调控机制, 在植物的抗病、抗逆、生长发育中发挥重要作用。目前关于大豆抗病基因的研究主要是以基因转录生成的前体RNA组成型切割体为基础, 而较少基于可变剪接体, 但在其他作物上已有关于可变剪接体参与植物抗性反应的报道, 如水稻抗稻瘟病基因转录后存在2种可变剪接体, 其中介导对稻瘟病的抗性, 而另一种剪接形式则没有表现出对稻瘟病的抗性[27], 结果说明基因转录产生的不同剪接体可能具有截然不同的功能。本文在大豆抗病候选基因及中检测到可变剪接现象的存在, 发现这些可变剪接体能够响应SMV的侵染增加表达量, 并且在抗病品种和感病品种之间的表达量存在较为明显的差异, 说明这2个基因的可变剪接体可能参与抗病品种对SMV的抗性。和基因剪接体的发现, 为其基因功能的鉴定提供了新的视角。但由于和基因具有较高同源性, 且基因各个内含子处均存在可变剪接, 候选基因每个可变剪接体的存在形式还需要进一步的实验证明。
4 结论
从大豆品种PI96983中扩增了2个TIR-NBS- LRR结构基因, 分别命名为和。它们高度同源, 均含有一个3288 bp的开放阅读框, 编码1095个氨基酸。在抗病品种(PI96983)和感病品种(南农1138-2)中存在8个SNP差异, 最终导致5个氨基酸发生突变;在抗病品种和感病品种中也存在8个SNP差异, 最终导致3个氨基酸突变。和亲缘最近的物种是野生大豆。和基因编码的蛋白产物主要分布在细胞膜和核膜上。和基因在抗感病品种中均存在可变剪接现象, 2个基因及其可变剪接体均能够响应病毒的诱导提高表达量, 且在抗病品种中的表达量高于感病品种, 其中可变剪接体IN1和IN2的表达量变化较为明显, IN3的表达量则相对平稳。初步推测候选基因和为抗SMV基因, 其可变剪接体可能参与对SMV的抗性。
[1] Hill J H, Whitham S A. Control of virus diseases in soybeans., 2014, 90: 355–390.
[2] Kendrick J B, Gardner M W. Soybean mosaic: seed transmission and effect on yield., 1924, 27: 91–98.
[3] Heinze K, Köhler E. The mosaic disease of the soybean and its transmission by insects., 1940, 13: 207–242.
[4] Yu Y G, Maroof M A S, Buss G R. Divergence and allelomorphic relationship of a soybean virus resistance gene based on tightly linked DNA microsatellite and RFLP markers., 1996, 92: 64–69.
[5] Hayes A J, Ma G, Buss G R, Maroof S. Molecular marker mapping of RSV4, a gene conferring resistance to all known strains of., 2000, 40: 1434–1437.
[6] Jeong S C, Hayes A J, Biyashev R M, Maroof S. Diversity and evolution of a non-TIR-NBS sequence family that clusters to a chromosomal “hotspot” for disease resistance genes in soybean., 2001, 103: 406–414.
[7] Klepadlo M, Chen P, Shi A, Mason R E, Korth K L, Srivastava V. Single nucleotide polymorphism markers for rapid detection of thelocus forresistance in diverse germplasm., 2017, 37: 10.
[8] Bent A F, Kunkel B N, Dahlbeck D, Brown K L, Schmidt R L, Giraudat J, Leung J L, Staskawicz B J. RPS2 of: a leucine-rich repeat class of plant disease resistance genes., 1994, 265: 1856–1860.
[9] Grant M R, Godiard L, Straube E, Ashfield T, Lewald J, Sattler A, Innes R W, Dangl J L. Structure of thegene enabling dual specificity disease resistance., 1995, 269: 843–846.
[10] Parker J E, Coleman M J, Szab V, Frost L N, Schmidt R, Biezen E A V D, Moores T, Dean C, Daniels M J, Jones J D. The arabidopsis downy mildew resistance gene RPP5 shares similarity to the toll and interleukin-1 receptors with N and L6., 1997, 9: 879–894.
[11] Yoshimura S, Yamanouchi U, Katayose Y, Toki S, Wang Z X, Kono I, Kurata N, Yano M, Iwata N, Sasaki T. Expression of Xa1, a bacterial blight-resistance gene in rice, is induced by bacterial inoculation., 1998, 95: 1663–1668.
[12] Davis C L. Identification, Validation, and Mapping ofandResistance Genes in Soybean. PhD Dissertation of Virginia Tech, Blacksburg, USA, 2017.
[13] Tran P T, Widyasari K, Seo J K, Kim K H. Isolation and validation of a candidategene from a soybean genotype that confers strain-specific resistance to., 2018, 513: 153–159.
[14] 郭小勤, 李德葆. 植物前体mRNA的选择性剪接. 农业生物技术学报, 2006, 14: 809–815. Guo X Q, Li D B. Pre-mRNA alternative splicing in plants., 2006, 14: 809–815 (in Chinese with English abstract).
[15] Brack C, Hirama M, Lenhardschuller R, Tonegawa S. A complete immunoglobulin gene is created by somatic recombination., 1978, 15: 1–14.
[16] Ali G S, Reddy A S N. Regulation of Alternative Splicing of Pre-mRNAs by Stresses. Heidelberg: Springer, 2008. pp 257– 275.
[17] Gassmann W. Alternative splicing in plant defense., 2008, 326: 219–233.
[18] Jang Y H, Lee J H, Park H Y, Kim S K, Lee B Y, Suh M C, Kim J K. OsFCA transcripts show more complex alternative processing patterns than itscounterparts., 2009, 52: 161–166.
[19] Modrek B, Lee C. A genomic view of alternative splicing., 2001, 30: 13–19.
[20] Lal S, Choi J H, Shaw J R, Hannah L C. A splice site mutant of maize activates cryptic splice sites, elicits intron inclusion and exon exclusion, and permits branch point elucidation., 1999, 121: 411–418.
[21] Yang Y, Zheng G, Han L, Wang D G, Yang X F, Yuan Y, Huang S H, Zhi H J. Genetic analysis and mapping of genes for resistance to multiple strains ofin a single resistant soybean accession PI96983., 2013, 126: 1783–1791.
[22] 刘玉芝, 廖林, 孙大敏. 对大豆花叶病毒(SMV)病抗源的筛选. 吉林农业科学, 1997, 1: 30–34. Liu Y Z, Liao L, Sun D M. Screening for resistant sources of soybean germplasm to SMV.. 1997, 1: 30–34 (in Chinese with English abstract).
[23] 纪冬, 辛绍杰. 实时荧光定量PCR的发展和数据分析. 生物技术通讯, 2009, 20: 598–600. Ji D, Xin S J. Development and data analysis of real-time fluorescent quantitative PCR., 2009, 20: 598–600 (in Chinese with English abstract).
[24] 李晓君, 王绍梅, 谢艳兰, 和敏. 农杆菌渗透法转化烟草条件的优化. 江苏农业科学, 2014, 42(9): 45–47. Li X J, Wang S M, Xie Y L, He M. Optimization of agrobacterium-infiltration method for transformation of tobacco., 2014, 42(9): 45–47 (in Chinese).
[25] Hayes A J, Jeong S C, Gore M A, Yu Y G, Buss G R, Tolin S, Maroof S. Recombination within a Nucleotide-Binding-Site/ Leucine-Rich-Repeat gene vluster produces new variants conditioning resistance toin soybeans., 2004, 166: 493–503.
[26] 黄赛花, 郑桂杰, 杨永庆, 智海剑. 利用VIGS技术对抗SMV候选基因的功能分析. 大豆科学, 2015, 34: 582–587.Huang S H, Zheng G J, Yang Y Q, Zhi H J. Analysis on the candidate resistance geneto soybean mosaic virus by VIGS., 2015, 34: 582–587 (in Chinese with English abstract).
[27] Cesari S, Thilliez G, Ribot C, Chalvon V, Michel C, Jauneau A, Rivas S, Alaux L, Kanzaki H, Okuyama Y, Morel J B, Fournier E, Tharreau D, Terauchi R, Kroj T. The rice resistance protein pair RGA4/RGA5 recognizes theeffectors AVR-Pia and AVR1-CO39 by direct binding., 2013, 25: 1463–1481.
Cloning and analysis of candidate gene resistant to SC3 in soybean
XIANG Wen-Yang, YANG Yong-Qing, REN Qiu-Yan, JIN Tong-Tong, WANG Li-Qun, WANG Da-Gang, and ZHI Hai-Jian*
Institute of Soybean, Nanjing Agricultural University / Key Laboratory of Soybean Biology and Genetic Breeding, Ministry of Agriculture / National Center for Soybean Improvement / State Key Laboratory of Crop Genetics & Germplasm Innovation, Nanjing 210095, Jiangsu, China
(SMV) is one of the prevalent pathogens of soybean, causing great reduction of soybean yield worldwide. Soybean disease resistance-breeding is currently the most cost-effective measure to control SMV, and identification of resistance genes is the basis of disease resistance breeding. According to the previous mapping result of resistance gene to SMV strain SC3, two genes (,) with TIR-NBS-LRR domain were cloned. Bioinformatics analysis showed that bothandgenes have SNP mutations in the susceptible varieties and resistant varieties, and the mutation sites are located in the conserved domain. These two proteins encoded byandgenes are predicted to be(TMV) resistant N proteins. The results of homologous alignment between species indicated thatandgenes were close to those of wild soybean. The expression ofandwas analyzed after inoculation with soybean mosaic virus in soybean, demonstrating thatandcould increase the expression level in response to SMV infection, with the higher level in resistant varieties than in susceptible varieties. Analysis of the alternative splicing ofandrevealed that the two genes have different splice variants IN1, IN2, and IN3. The response analysis of splices to SMV showed that all splices were able to increase the expression in response to virus induction, with the higher level in resistant varieties than in susceptible varieties. It indicated that these alternative splicing may be involved in the disease resistance process of soybean to SMV. The result of this study lay a foundation for the study of subsequent gene function.
(SMV); resistance gene; inducing expression; alternative splicing
本研究由国家转基因生物新品种培育科技重大专项(2016ZX08004-004), 国家自然科学基金项目(31571690, 31571687), 中央高校基本科研业务费专项(KYT201801), 长江学者和创新团队发展计划项目(PCSIRT_17R55), 国家现代农业产业技术体系建设专项(CARS-04), 江苏省现代作物生产协同创新项目(JCIC-MCP)和国家重点研发计划项目(2017YFD0101501)资助。
This study was supported by the Fund of Transgenic Breeding for Soybean Resistance to Soybean mosaic virus (2016ZX08004-004 ), the National Natural Science Foundation of China (31571690, 31571687), the Fundamental Research Funds for the Central Universities (KYT201801), the Program for Changjiang Scholars and Innovative Research Team in University (PCSIRT_17R55), the National Soybean Industrial Technology System of China (CARS-004), Jiangsu Collaborative Innovation Center for Modern Crop Production (JCIC-MCP), and the National Key R&D Program of China (2017YFD0101501).
智海剑, E-mail: zhj@njau.edu.cn
E-mail: a15295572307@163.com
2019-04-02;
2019-06-22;
2019-07-15.
URL: http://kns.cnki.net/kcms/detail/11.1809.S.20190712.1641.010.html
10.3724/SP.J.1006.2019.94054
