陆地棉漆酶基因家族鉴定及在黄萎病菌胁迫下的表达分析*
2019-11-13李旭彤梁学忠王志城吴立强王省芬张桂寅马峙英
赵 晶 李旭彤 梁学忠 王志城 崔 静 陈 斌 吴立强 王省芬 张桂寅 马峙英 张 艳
陆地棉漆酶基因家族鉴定及在黄萎病菌胁迫下的表达分析*
赵 晶**李旭彤**梁学忠 王志城 崔 静 陈 斌 吴立强 王省芬 张桂寅 马峙英 张 艳*
河北农业大学 / 华北作物种质资源研究与利用重点实验室 / 河北省棉花产业协同创新中心, 河北保定 071001
黄萎病是降低棉花产量和品质较为严重的维管束病害。棉花在抵抗病原菌的过程中, 抗病基因起着尤为重要的作用。漆酶是一种多功能氧化酶, 在植物木质素合成和提高植株抗逆性等方面发挥着重要作用。高质量版本的基因组是提升基因家族分析准确性的必要条件。本研究以目前最新的陆地棉TM-1高质量版本基因组为参考, 通过生物信息学分析鉴定了陆地棉基因组中的()基因家族, 并对其进行了理化性质、基因结构、染色体定位以及在黄萎病胁迫下的表达模式分析。结果表明, 陆地棉TM-1基因组中共包含83个家族成员, 分布在24条染色体上, 所有GhirLAC蛋白均定位在胞外, 且具有相同/相似的保守基序。系统发育树分析显示,基因家族成员可分为7个亚组。结合黄萎病胁迫下棉花转录组数据, 将基因家族各成员的表达分为3种模式, 其中第1类和第2类基因在黄萎病菌胁迫下分别呈现有规律的表达量升高和降低, 推测这些基因在棉花抗黄萎病反应中发挥重要功能。荧光定量PCR结果表明,()、()和() 3个候选基因均受黄萎病菌诱导表达, 其表达趋势与转录组数据相吻合。本研究为以后深入解析棉花基因的抗病功能及分子机制奠定基础。
陆地棉; 漆酶; 基因家族; 黄萎病; 基因表达
棉花(spp)是世界上第一大天然纤维作物, 具有重要的经济价值, 也是食品、饲料和生物燃料的重要来源[1]。在生长过程中, 棉花的品质和产量会受到多种生物和非生物胁迫, 其中危害最大的是由大丽轮枝菌(Kleb.)引起的黄萎病[2]。黄萎病菌寄主范围极其广泛, 其产生的菌核对极端温度变化适应能力强, 可在土壤中存活数十年, 因此防治难度很大, 每年造成巨大经济损失[3-4]。研究表明利用品种的遗传抗性可以有效降低黄萎病带来的危害, 实现棉花的高产、稳产[5]。
细胞壁是植物抵御病原体侵袭的天然屏障, 当病原菌侵染时, 寄主植物细胞在细胞壁、胞间层、细胞质等不同部位产生一系列抗性反应, 可以有效地阻止病原菌的再度侵染和蔓延[6-8]。已有研究表明, 在细胞壁的木质化过程中, 从薄壁组织细胞内合成的木质醇单体分泌至胞外经脱氢聚合反应形成木质素[9]。植物细胞壁的木质化能够抵抗病原菌侵入的机械压力、抵抗真菌酶类对细胞壁的降解和阻断病原菌与寄主植物间的物质交流[10]。木质素是构成植物细胞壁的成分之一, 而漆酶又是木质素合成途经中的关键酶之一, 因此漆酶在植物细胞壁形成和抗病过程中的作用值得深入研究。
漆酶属于铜蓝蛋白家族, 是一种结合多个铜离子的糖蛋白, 广泛存在于细菌、真菌和动植物中。目前高等植物中已有部分漆酶基因成员的研究报道, 如短柄草对其细胞壁木质化具有重要作用[11]; 亚洲棉漆酶基因导入新疆杨后能够导致转基因植株中总木质素含量的增加, 表明参与了植物木质素的合成[12]; 赵先炎等[13]研究表明, 增强番茄中漆酶基因的活性, 可以提高植株对酚类物质的抗性; 田奇琳等[14]克隆了龙眼基因, 并发现其可能参与茉莉酸、水杨酸和脱落酸的逆境胁迫信号转导途径, 调控龙眼多种非生物胁迫应答过程; 黄晨等[15]发现龙井茶树在茉莉酸处理、机械损伤和茶尺蠖取食后, 漆酶基因和的表达量有明显的变化。此外, 在水稻[16]、甜高粱[17]和亚麻[18]中, 漆酶基因家族成员也被相继鉴定出来, 由此可见, 不同植物漆酶基因在生物和非生物胁迫中具有重要功能。
已有关于漆酶基因家族的研究多集中在二倍体植物[16-18], 棉花属于异源四倍体, 基因组庞大而复杂, 目前漆酶基因在栽培棉花中的研究报道相对较少, 课题组前期基于SSH文库筛选到, 研究发现其通过防御诱导的木质素的合成增强黄萎病抗性[19]; Hu等[20]超表达棉花基因同时提高了植株对黄萎病菌及棉铃虫的抗性。Balasubramanian等[21-22]以2015年公布的陆地棉TM-1基因组为参考, 通过全基因组鉴定家族成员并明确了不同成员在陆地棉纤维发育中的表达模式。随着测序技术的不断提升和基因组质量的进一步完善, Wang等[23]通过三代基因组获得的最新的陆地棉TM-1基因组较之前版本基因组质量有很大提升, 为全基因组基因家族分析提供更为可靠的参考序列; 另外棉花中漆酶基因家族除目前已报道的两个成员外[19-20], 其他成员是否参与抗黄萎病反应尚不清楚。因此, 本研究参考最新的陆地棉TM-1基因组, 利用生物信息学方法, 提取并鉴定得基因家族成员, 并对各成员的理化性质、基因结构、系统进化和染色体定位等方面进行系统分析, 结合黄萎病胁迫处理后的转录组数据, 明确基因家族成员的表达规律, 为深入解析棉花基因的抗病功能及分子机制奠定基础。
1 材料与方法
1.1 基于陆地棉TM-1三代基因组的漆酶基因家族的鉴定及理化性质分析
本研究利用最新公布的陆地棉TM-1基因组序列为参考, 相关基因组和蛋白质组数据从COTTONGEN (http://www.cottongen.org/)数据库中下载[24]。借助基因结构域的隐马可夫模型(PF00394、PF07731和PF07732), 搜索棉花蛋白质组中的LAC蛋白序列。去除冗余后, 将所有的候选基因在Pfam (http:// pfam.xfam.org/)数据库中进行结构域验证, 对搜索出来的棉花基因家族成员进一步确认。利用ExPASy软件(http://cn.expasy.org/tools)计算棉花LAC蛋白序列的长度; 采用EuLoc在线软件(http://euloc.mbc.nctu.edu.tw/)对棉花LAC蛋白进行亚细胞定位预测。
1.2 陆地棉漆酶基因家族的系统进化分析
利用MEGA 7软件中的Clustal W功能对83个陆地棉LAC蛋白序列和17个拟南芥LAC蛋白序列进行序列比对, 然后基于序列比对的结果, 采用邻近法构建系统进化树, 其中Bootstrap值设定为1000。
1.3 陆地棉漆酶基因家族的染色体定位、基因结构和保守结构域分析
从陆地棉基因组注释文件中提取基因家族成员的染色体位置信息, 利用MapChart软件2.32绘制基因的染色体定位图, GSDS在线软件(http://gsds.cbi.pku.edu.cn/)绘制基因结构图, MEME (http://meme-suite.org/tools/meme)分析LAC蛋白的保守结构域。
1.4 陆地棉漆酶基因家族在黄萎病胁迫下的表达模式分析
本课题组前期以抗病陆地棉品种农大601为试验材料, 获得在处理组(黄萎病菌胁迫处理后) 2、6、12、24和48 h和对照组(各对应时间点接种水)的根部转录组数据。将陆地棉基因家族各成员的表达量数据进行log2(1+RPKM)处理后, 利用MEV软件, 绘制基因家族各成员的表达热图。
1.5 陆地棉漆酶基因家族在黄萎病胁迫下的表达模式分析
根据陆地棉家族基因的cDNA信息, 采用Primer 5.0软件在基因序列5′端、ORF或3′端的特异区域设计引物(表1), 扩增片段约为200 bp。分别以陆地棉农大601根、茎、叶不同组织和接菌后6、12、24、36和48 h的根组织cDNA为模板, 利用qRT-PCR方法检测候选基因的表达情况, 设置每个样本3次重复, 内参基因为[25]。qRT-PCR体系为10.0 μL, 其中cDNA 1 μL、2×SYBR预混液5.0 μL、基因特异正向引物(10 μmol L–1) 0.5 μL、反向引物(10 μmol L–1) 0.5 μL、RNase-Free H2O 2.8 μL、ROX校正液0.2 μL。qRT-PCR在ABI7500仪器反应, 反应程序为94℃预变性30 s; 95℃变性5 s, 57℃退火5 s, 72℃延伸34 s, 40个循环。采用2–ΔΔCt方法计算基因的相对表达量[26]。
2 结果与分析
2.1 陆地棉LAC基因家族成员的鉴定
通过Hmmsearch搜索和数据库验证, 在陆地棉基因组中共鉴定出83个LAC基因家族成员, 根据其在染色体上的位置信息, 命名为(表1), 其中和没有定位到染色体上。除A亚组的第7染色体和D亚组的第7染色外, 其他染色体都不均匀地分布着LAC基因(图1)。所有LAC基因所编码蛋白的氨基酸数量在420~583个之间, 差异较小, 且所有基因的亚细胞定位结果都在胞外, 属于分泌蛋白。
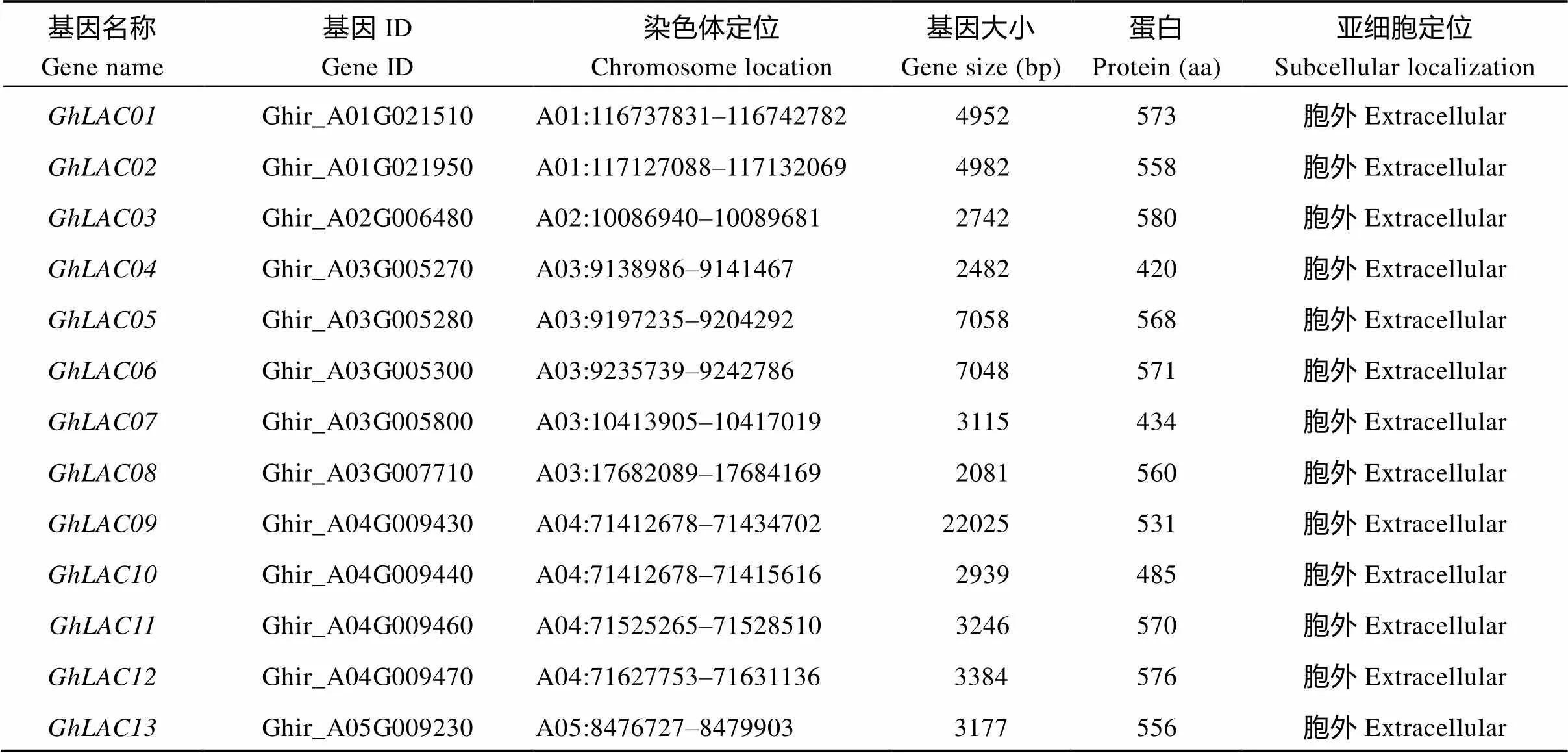
表1 陆地棉LAC基因家族信息
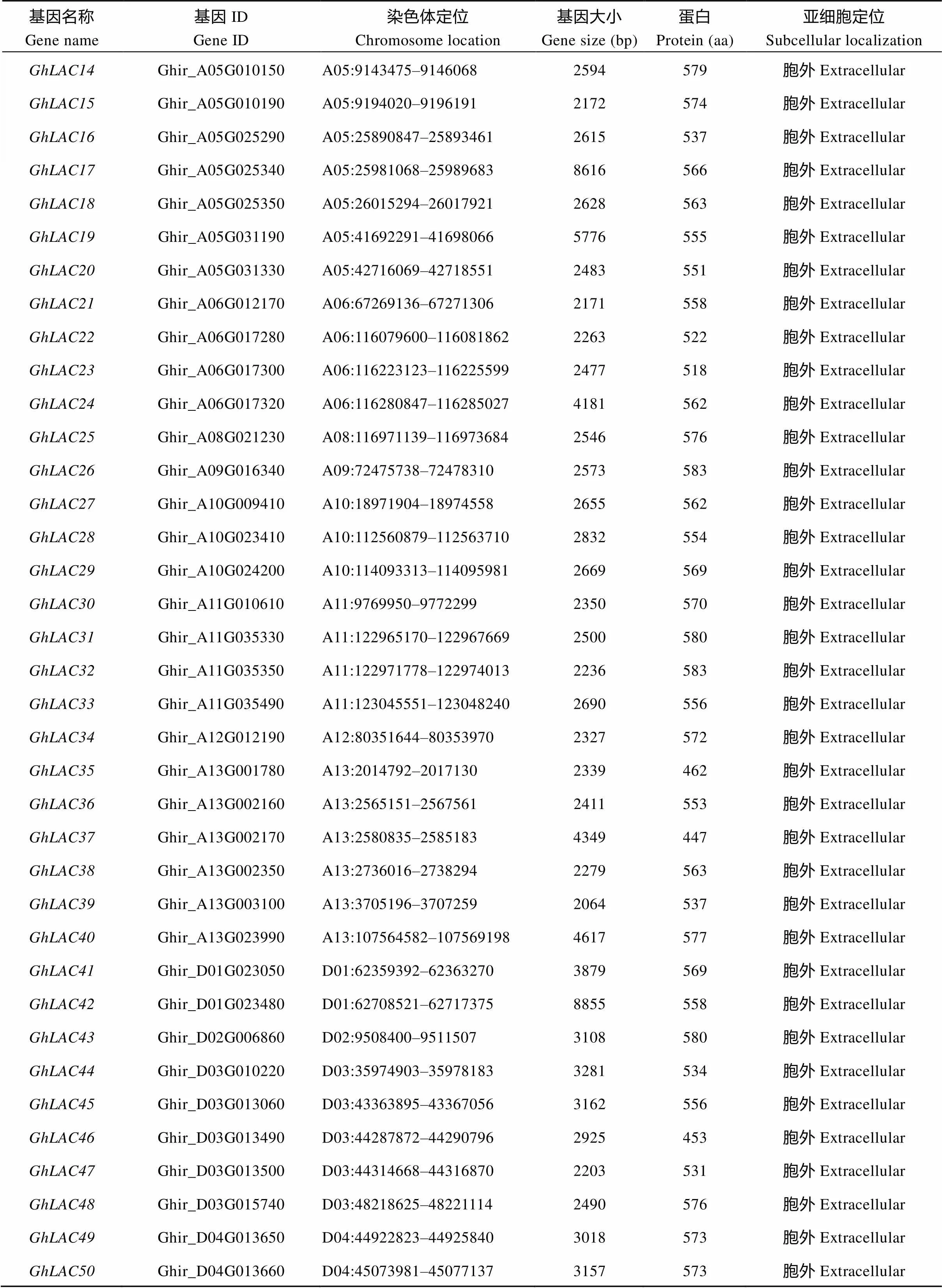
(续表1)
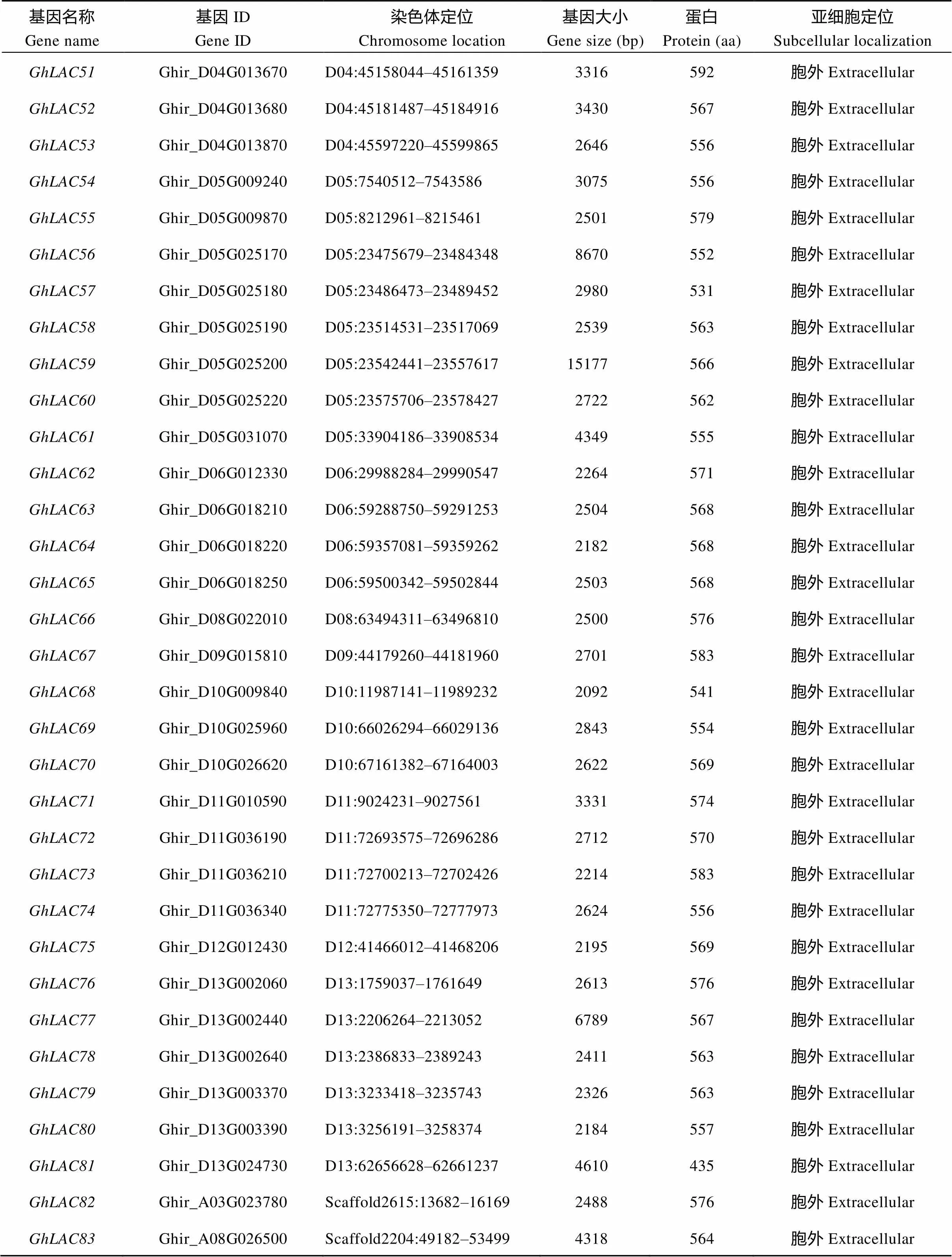
(续表1)
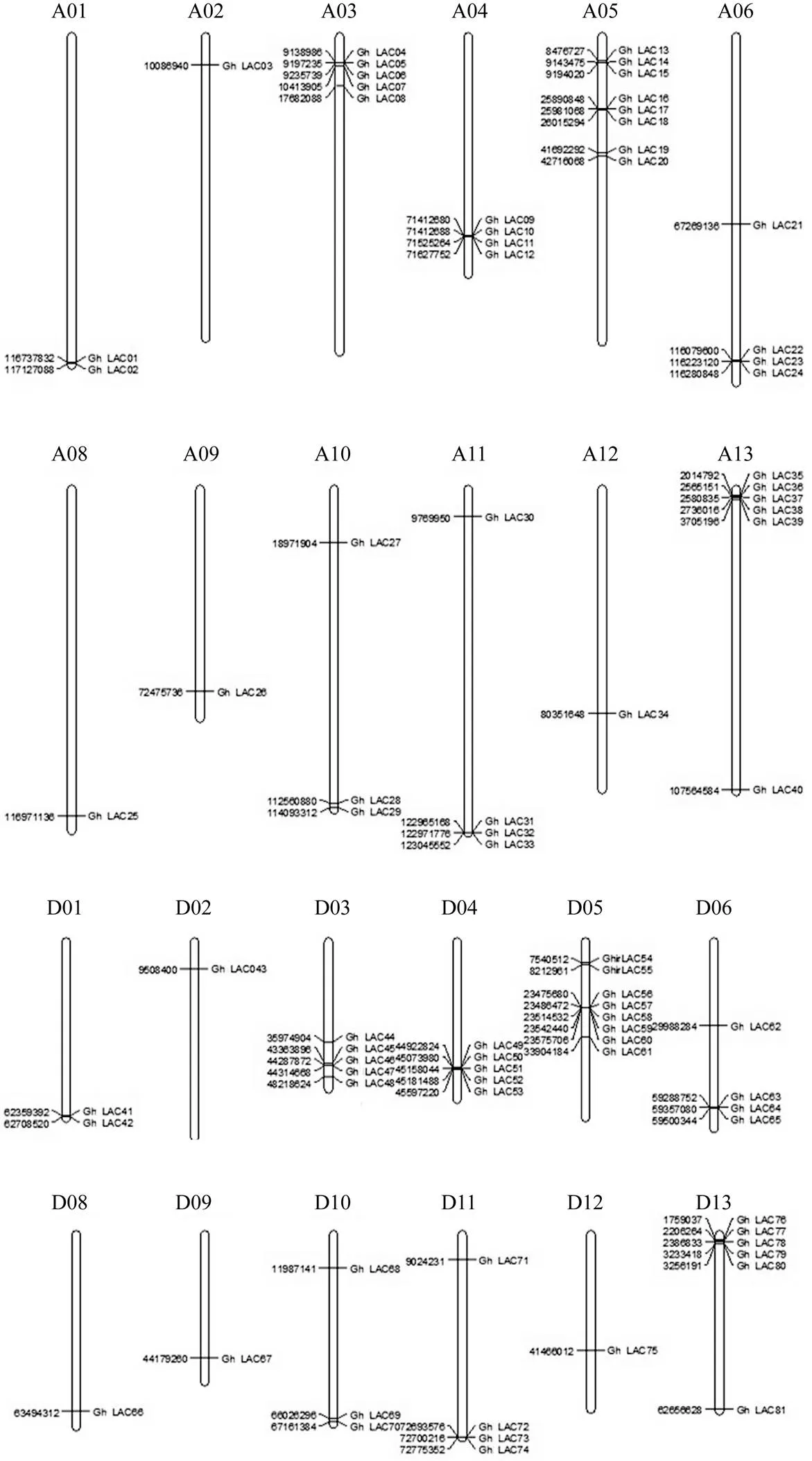
图1 陆地棉LAC基因家族成员的染色体定位分析
2.2 陆地棉漆酶家族基因系统发育分析
用83个陆地棉GhirLACs蛋白序列和17个拟南芥AtLACs蛋白序列构建系统发育树, 并根据拟南芥漆酶基因的功能[27], 将棉花漆酶分为7组(图2)。第1组包括23个陆地棉漆酶成员和2个拟南芥漆酶成员(和), 第2组包括22个陆地棉漆酶成员和4个拟南芥漆酶成员(、、和), 拟南芥、和在调控木质素单体和细胞壁木质化方面具有重要作用, 属于参与木质素生物合成的单木质素漆酶, 基于该进化树结果, 初步认为棉花第1组与第2组漆酶成员具有类似的功能[27-29]。第3组包括13个陆地棉漆酶成员和4个拟南芥漆酶基因(、、和), 其中、和对ABA信号有响应[29];第4组包括12个陆地棉漆酶成员和2个拟南芥漆酶基因(和), 可能与干旱、低温和盐胁迫有关[29]; 第5组包括9个陆地棉漆酶成员和3个拟南芥漆酶成员(、和); 第6组只有1个拟南芥漆酶成员第7组包括4个陆地棉漆酶成员和1个拟南芥漆酶成员(), 其在生物胁迫下表达会下调[30]。
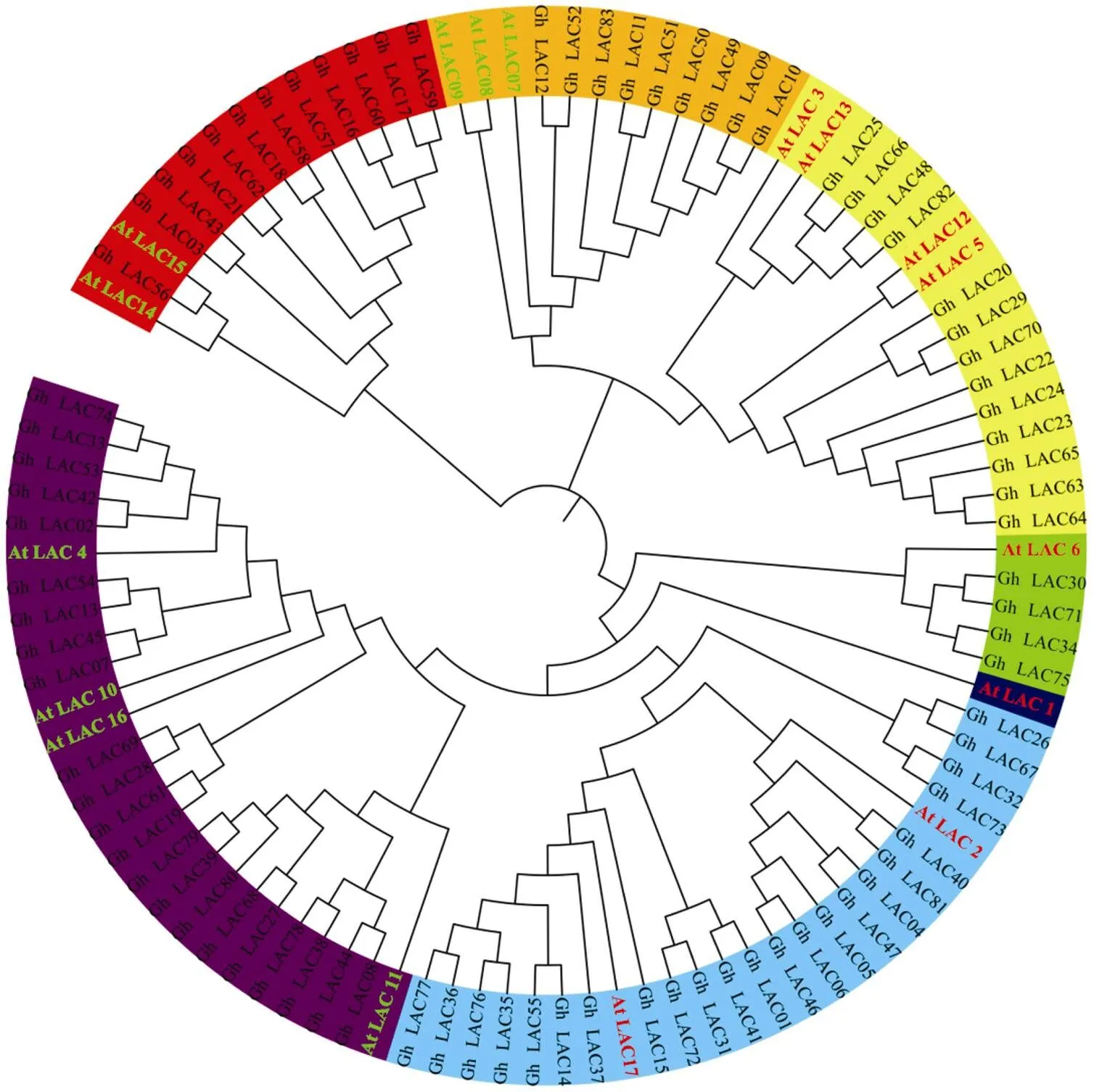
图2 陆地棉和拟南芥LACs基因家族进化树分析
2.3 陆地棉漆酶基因家族的结构和保守域分析
对陆地棉LAC基因家族83个成员的基因结构和保守域分析结果如图3所示, 所有LAC基因的外显子个数在4~7个之间变化, 且基本都含有相同的保守结构域(Motif 1、Motif 2、Motif 3、Motif4、Motif5、Motif 9和Motif 10; 图4)。经Pfam数据库验证, Motif 1和Motif 9完全对应于Cu_oxidase_3结构域; Motif 3与Cu_oxidase结构域相对应; Motif2和Motif4则构成了Cu_oxidase_2结构域。某些保守基序只存在于某一亚组中, 如Motif 8只存在于第7亚组中, motif是蛋白质分子中具有特定空间构象和特定功能的结构成分, 是结构域的亚单位, 与特定的功能联系在一起, 推测具有不同motif的漆酶成员在进化和功能上可能存在差异。
2.4 陆地棉漆酶基因家族在黄萎病胁迫下的表达分析
基于本课题组前期已有的抗病农大601黄萎病菌胁迫下的根转录组数据[31], 将不同处理时间点下的RPKM值进行log2(1+RPKM) 处理后, 构建LAC基因家族成员的表达热图。结果显示, 在83个陆地棉LAC基因中, 有80个基因的表达量在黄萎病胁迫下都发生了显著的变化。80个差异基因在根中的表达模式分为3种类型, 第1类基因呈现出病原菌诱导后表达上调, 包括、等24个成员。根据该类基因对黄萎病菌的响应速度和程度, 进一步划分为3个亚组, 即快速上调表达基因(I), 包括和, 这些成员在接菌后2 h基因表达达到高峰; 较快上调表达基因(II),包括77、等9个成员, 该亚组基因在接菌后6 h达到表达高峰; 以及第III亚组, 包括01等10个成员, 受黄萎病菌诱导后这些基因在12~24 h之间达到表达高峰。第2类基因受病原菌胁迫后呈现不同程度下调表达, 包括、、、等56个成员, 接菌后这些基因的表达明显受到抑制, 暗示这些基因在棉花抗黄萎病反应中起负调控作用; 第3类基因不响应黄萎病菌处理, 推测其不参与棉花抗黄萎病过程, 包括、和(图5)。
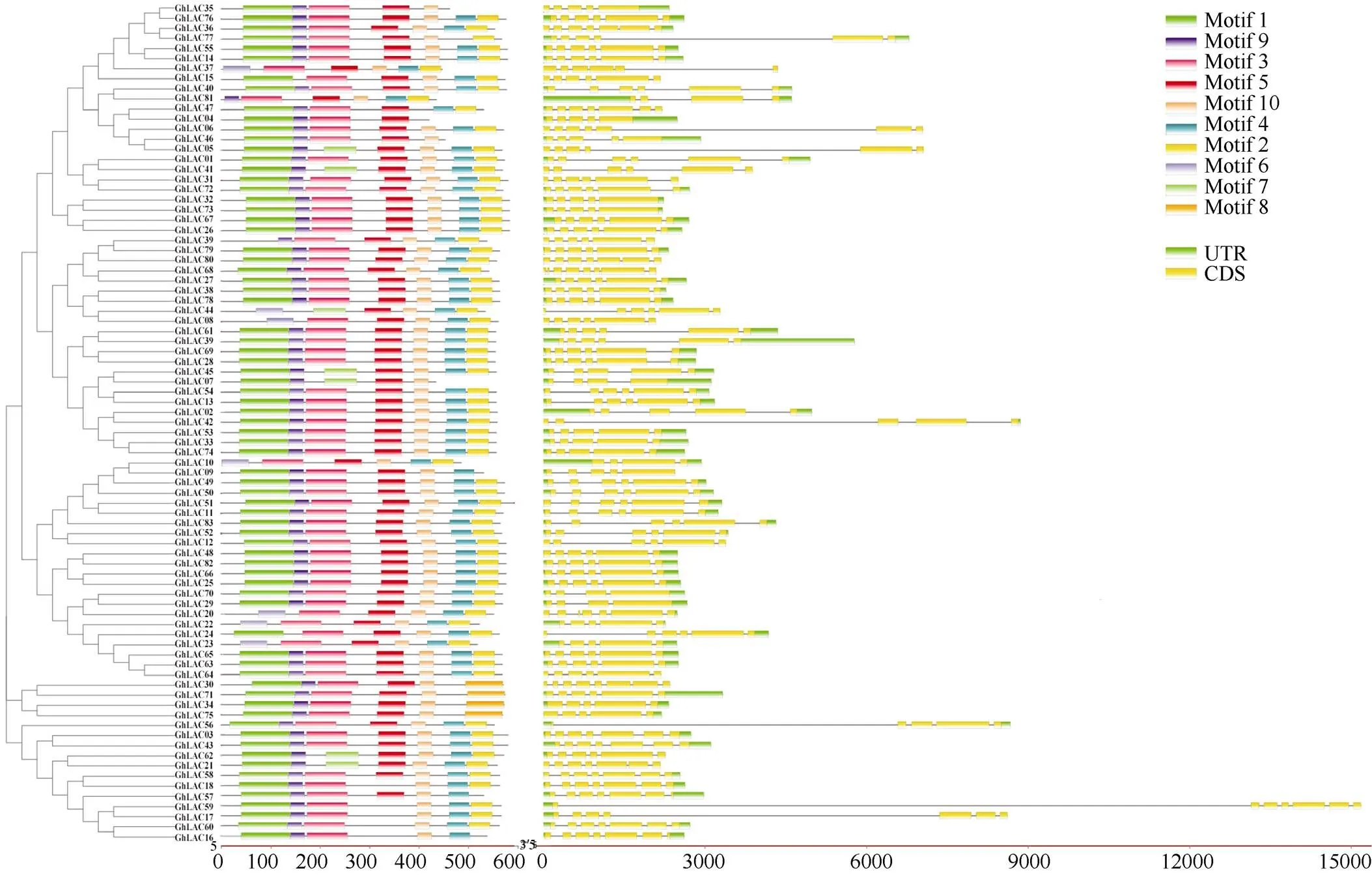
图3 陆地棉LAC家族成员基因结构分析

图4 陆地棉LAC基因家族保守基序
2.5 陆地棉漆酶基因家族在黄萎病胁迫下的表达分析
结合抗病转录组数据、全长cDNA文库筛选以及功能尚未报道的前提, 本研究进一步挑选出3个分别与拟南芥、和直系同源的新基因, 分别命名为()、()和()。荧光定量PCR结果表明,在接黄萎病后6 h表达量显著提高;受黄萎病菌诱导明显, 接菌后6 h快速响应, 24 h达到表达高峰;的表达在接菌后各时间点均显著上调。这3个基因的荧光定量结果与转录组数据的趋势相吻合, 该结果在进一步明确三个基因的表达规律的同时也反映了转录组数据的可靠性(图6)。组织特异性表达结果显示3个基因在棉花根、茎、叶中均有表达;在茎中表达量最高,和在根中表达量最高(图6)。
3 讨论
基因组质量的完整性是全基因组水平分析基因家族的基础。随着基因组测序技术的不断深入, 棉花基因组从二倍体棉到四倍体陆地棉不断更新[22,32-33]。基于二代基因组测序组装获得的四倍体陆地棉TM-1的基因组草图为开展全基因组水平分析提供了可能, 但由于棉花属于异源多倍体, 基因组庞大而复杂, 高质量版本的基因组成为提升基因家族分析准确性的必要条件。最新的研究释放了基于超高深度测序、10×Genomics、BioNano光学图谱及Hi-C数据等多技术联合组装的陆地棉TM-1基因组[23], 较以前发表的基因组草图在连续性和完整性上提高了10~20倍, 并对具有高度重复的区域(如着丝粒)进行了成功组装。此外, 新版本基因组预测出72,761。个高度可信的蛋白编码基因, 较旧版本预测的70,478个蛋白编码基因多出了2283个新基因。因此, 利用更新的陆地棉参考基因组分析复杂的基因家族, 将为深入研究该基因家族的功能提供更为准确的信息
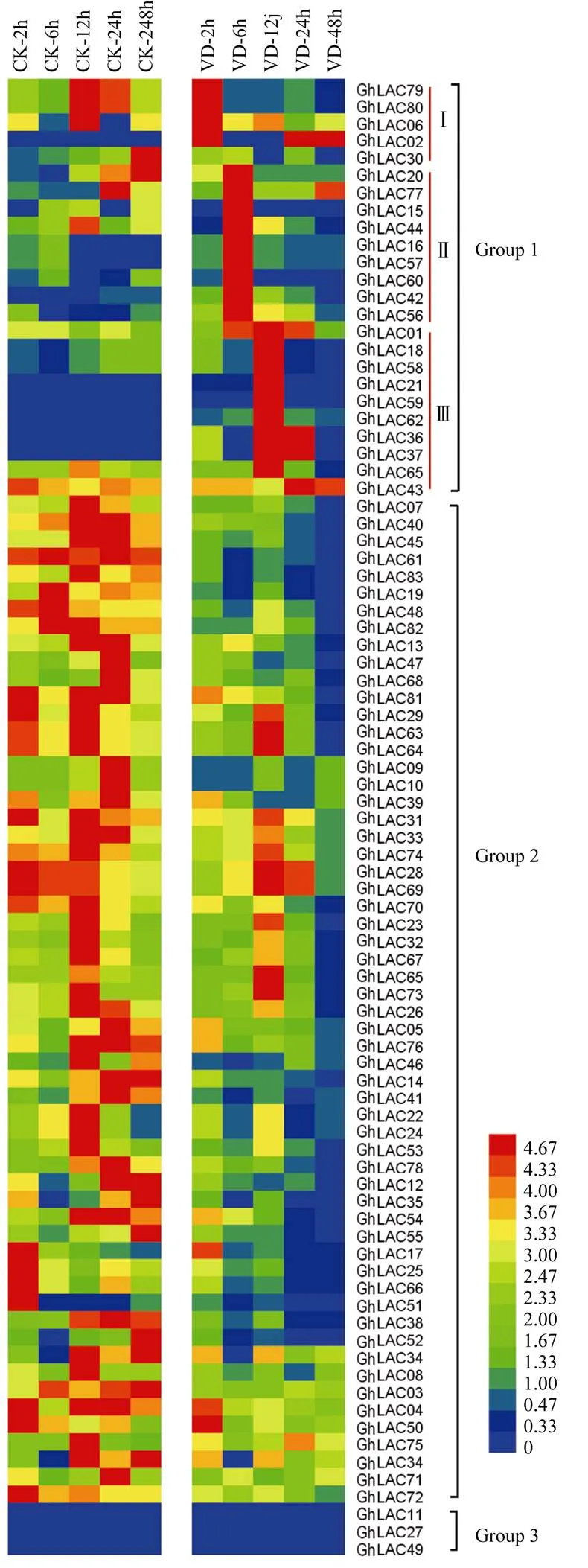
图5 陆地棉LAC基因在黄萎病胁迫下的表达热图
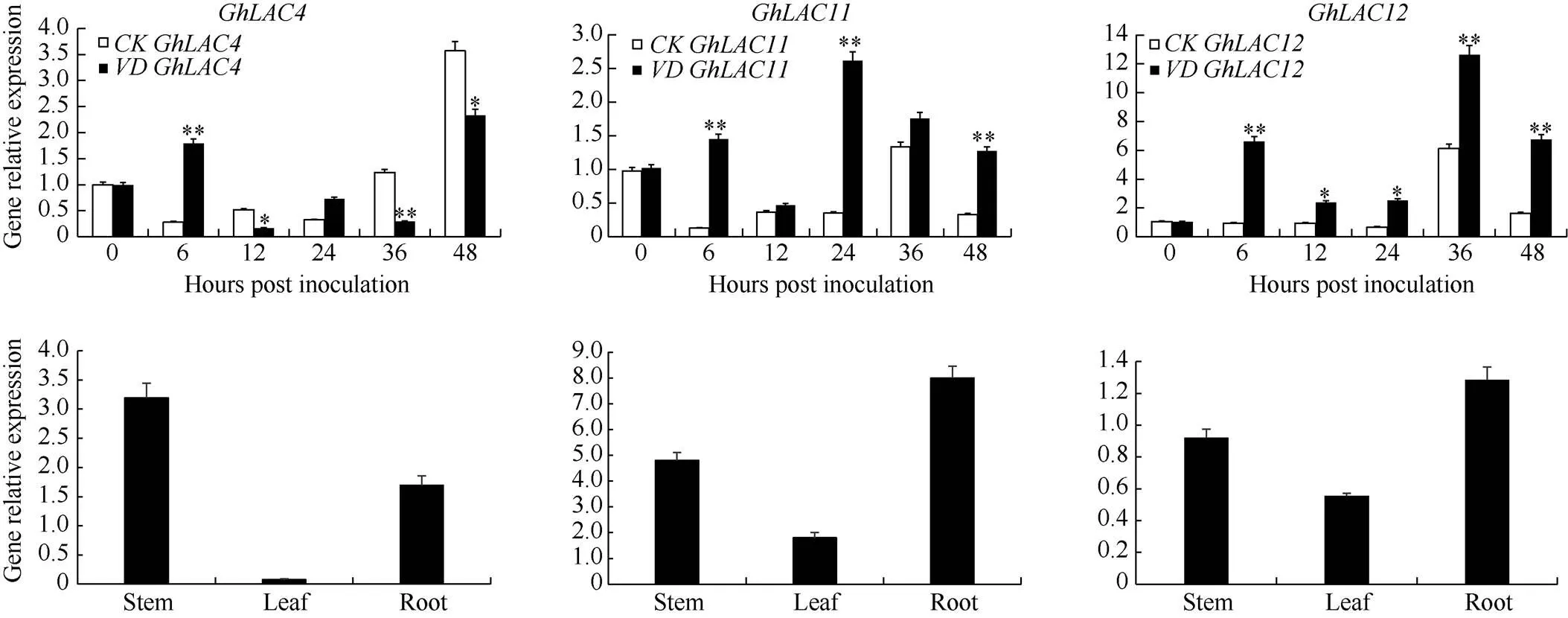
图6 黄萎病菌胁迫下GhLAC4、GhLAC11和GhLAC12基因的表达
本研究通过生物信息学方法从高质量的陆地棉TM-1全基因组中鉴定出83个漆酶基因成员, 经过分析发现, 所有基因的外显子个数在4~7之间, 所编码蛋白的氨基酸数量在500个左右, 且基本含有相同的保守基序, 表明基因家族不同成员之间具有较强的保守性。已有研究报道拟南芥、和属于参与木质素生物合成的单木质素漆酶, 在木质素合成方面具有重要功能, 基于本研究棉花漆酶与拟南芥直系同源基因进化树分析, 初步推测分别与拟南芥、和基因聚为一组的棉花漆酶成员可能具有类似的功能[27-29]。本研究结果与2016年报道的一篇关于棉花漆酶基因家族的分析结果存在较大差异。本研究基于高质量的TM-1基因组鉴定到83个LAC成员, 除At07和Dt07染色体外, 其他染色体都不均匀地分布着LAC基因, 其中At05和Dt05上LAC基因分布最多, 分别为8个; Balasubramanian等[21]基于旧版本的TM-1基因组草图鉴定到84个LAC成员, 其结果显示在3条染色体At07、Dt07、At08中均没有LAC的分布, LAC成员分布最多的染色体位于At05, 分布着11个LAC基因。这些信息的不匹配性为我们深入研究LAC基因家族带来很大困扰甚至是误导, 因此本研究以目前最高质量的基因组版本系统分析LAC基因家族, 为深入研究LAC重要成员的功能奠定基础。
细胞壁是抵御病原菌入侵的天然屏障, 木质素是细胞壁的重要组成部分, 在植物抗病防御反应中发挥重要功能。漆酶位于木质素合成途径的最后一步, 负责将G、S、H不同木质素单体交联聚合形成多聚物-木质素。自然条件下, 细胞壁作为植物固有的基本组成部分保证植株生长发育, 在此过程中漆酶参与的木质素合成被定义为specific-lignification, 如拟南芥、和[27-29]; Chezem等[34]研究表明拟南芥可以激活防御诱导的木质素的合成, 参与该过程的木质素合成基因受丁香加单包杆菌的诱导表达, 课题组前期的研究也发现()对黄萎病菌迅速响应, 其表达趋势与本研究在转录组中的表达趋势类似, 超表达基因通过增加植株防御诱导的木质素的合成而提高抗病性[19]。Hu等[20]研究了棉花基因, 发现超表达的植株木质素增加, 从而提高了对黄萎病和棉铃虫的抗性; 抑制基因植株苯丙烷代谢流发生改变, 木质素合成降低而黄酮代谢增强, 同时还激活了植物激素茉莉酸的合成, 从而富集了更多对病虫有害的次生代谢物质。自然条件下, 植物生长面临生物和非生物胁迫等复杂的外界环境, 植物通过能量与物质的重新分配来调控生长与防御的平衡[35]。由此推测, 病菌胁迫条件下, 植物暂停或暂缓生长发育进度, 能量物质向防御反应方向倾斜。从本研究的家族的表达结果来看, 其中属于第二类的各成员受病菌胁迫后, 基因的表达量或快速、或大幅度下调, 推测这些基因在调控生长与防御的平衡方面发挥重要作用。此外, 从第一类基因的表达来看, 该类基因的大部分成员在自然生长状态下处于低或较低表达水平, 受病菌诱导后这些基因在某个时间点达到表达峰值, 随后表达水平逐渐回落, 表明这些基因属于防御诱导的一类抗病漆酶基因, 对病原菌迅速响应, 推测植株一旦防御体系建立, 为了协调防御和植株生长发育, 该类基因的表达随后降低。综上, 本研究基于表达, 认为80个响应黄萎病菌的成员在棉花抗黄萎病反应中具有功能, 但第一类基因可能侧重于增加木质素的合成提高抗病性; 第二类基因可能侧重于改变苯丙烷代谢流来提高抗病性; 二者也可能存在部分交叉。该研究结果为进一步研究的功能提供一定指导。
4 结论
本研究鉴定了陆地棉基因组中的Laccase基因家族, 83个家族成员分布在24条染色体上, 所有GhirLAC蛋白均定位在胞外, GhirLAC家族成员可分为7个亚组。黄萎病菌胁迫下,家族各成员的表达分为3种模式, 其中第1类和第2类基因在棉花抗黄萎病反应中发挥重要功能。()、()和() 3个候选基因均受黄萎病菌诱导表达。本研究为以后深入解析棉花基因的抗病功能及分子机制奠定基础。
[1] Gao X Q, Wheeler T, Li Z H, Kenerley C M, He P, Shan L B. Silencingandcompromises cotton resistance to Verticillium wilt., 2011, 66: 293–305.
[2] Bolek Y, Elzik K M, Pepper A E,Bell A A, Magill C W, Thaxton P M, Reddy O U K. Mapping of Verticillium wilt resistance genes in cotton., 2005, 168: 1581–1590.
[3] Zhang Y, Wang X F, Rong W, Yang J, Li Z K, Wu L Q, Zhang G Y, Ma Z Y. Histochemical analyses reveal that stronger intrinsic defenses inthan inare associated with resistance to., 2017, 30: 984–996.
[4] Cai Y F, He X H, Mo J C, Sun Q, Yang J P, Liu J G. Molecular research and genetic engineering of resistance toin cotton: a review., 2009, 8: 7363–7372.
[5] 张天真, 周兆华, 闵留芳, 郭旺珍, 潘家驹, 何金龙, 纵瑞收, 汤杰珍, 郭小平, 蒯本科, 王谧, 朱协飞, 陈兆夏, 唐灿明, 刘康, 孙敬, 惠书勤, 黄在进. 棉花对黄萎病的抗性遗传模式及抗(耐)病品种的选育技术. 作物学报, 2000, 26: 673–680. Zhang T Z, Zhou Z H, Min L F, Guo W Z, Pan J J, He J L, Zong R S, Tang J Z, Guo X P, Kuai B K, Wang M, Zhu X F, Chen Z X, Tang C M, Liu K, Sun J, Hui S Q, Huang Z J. Inheritance of cotton resistance toand strategies to develop resistant or tolerant cultivars.,2000, 26: 673–680 (in Chinese with English abstract).
[6] Malinovsky F G, Fangel J U, Willats W G T. The role of the cell wall in plant immunity., 2014, 5: 178.
[7] Bowers J H, Nameth S T, Riedel R M,Rowe R C. Infection and colonization of potato roots byas affected byand., 1996, 86: 614–621.
[8] Fradin E F, Thomma B P H J. Physiology and molecular aspects of Verticillium wilt caused byand., 2006, 7: 71–86.
[9] 赵蕾, 张天宇. 植物病原菌产生的降解酶及其作用. 微生物学通报, 2002, 29: 89–93. Zhao L, Zhang T Y. Production and roles of the degrading enzymes prodused by phytopathogen., 2002, 29: 89–93 (in Chinese with English abstract).
[10] Smit F, Dubery I A. Cell wall reinforcement in cotton hypocotyls in response to aelicitor., 1997, 44: 811–815.
[11] Wang Y, Coussa O B, Lebris P, Antelme S, Soulhat C, Gineau E, Dalmais M, Bendahmane A, Morin H, Mouille G, Legée F, Cézard L, Lapierre C, Sibout R. LACCASE 5 is required for lignification of theculm., 2017, 168: 192–204.
[12] 王骥, 朱木兰, 卫志明. 棉花漆酶基因在转基因新疆杨中的表达及其对木质素合成的影响. 分子细胞生物学报, 2008, (1): 11–18.W J, Zhu M L, Wei Z M. Cotton Laccase gene overexpression in transgenicvar.and its effects on the lignin biosynthesis in transgenic plants.2008, (1): 11–18 (in Chinese with English abstract).
[13] 赵先炎, 庞明利, 赵强, 任怡然, 郝玉金, 由春香. 番茄漆酶基因的克隆与表达分析. 园艺学报, 2015, 42: 1285–1298.Zhao X Y, Pang M L, Zhao Q, Ren Y R, Hao Y J, You C X. Cloning and expression analysis of tomatogene., 2015, 42: 1285–1298 (in Chinese with English abstract).
[14] 田奇琳, 林玉玲, 郑庆游, 苏荣峰, 赖钟雄. 龙眼的克隆及其表达调控分析. 果树学报, 2016, 33: 1185–1193.Tian Q L, Lin Y L, Zheng Q Y, Su R F, Lai Z X. Cloning and expression analyses ofin., 2016, 33: 1185–1193 (in Chinese with English abstract).
[15] 黄晨, 陈帅, 程小芳, 张新, 黎星辉, 孙晓玲. 茶树漆酶基因和的克隆与表达分析. 植物保护学报, 2018, 45: 1069–1077.Huang C, Chen S, Cheng X F, Zhang X, Li X H, Sun X L. Cloning and expression analysis of the laccase genesandfrom the tea plant.2018, 45: 1069–1077 (in Chinese with English abstract).
[16] Liu Q Q, Luo L, Wang X X,Shen Z G, Zheng L QComprehensive analysis of rice laccasegene () family and ectopic expression ofenhances tolerance to copperstress in Arabidopsis., 2017, 18: 1–16.
[17] Wang J H, Feng J J, Jia W T, Fan P X, Bao H X G D L, Li S Z, Li Y X. Genome-wide dentification of sorghum bicolor laccases reveals potential targets for lignin modification., 2017, 8: 714.
[18] Roy J L, Blervacq A S, Créach A, Huss B, Hawkins S, Neutelings G. Spatial regulation of monolignol biosynthesis and laccase genes control developmental and stress-related lignin in flax., 2017, 17: 124.
[19] Zhang Y, Wu L Z, Wang X F, Chen B, Zhao J, Cui J, Li Z K, Yang J, Wu L Q, Wu J H, Zhang G Y, Ma Z Y. The cotton laccase geneenhancedresistance via increasing defense-induced lignification and lignin components in the cell wall of plants., 2019, 20: 309–322.
[20] Hu Q, Min L, Yang X Y, Jin S X, Zhang L, Li Y Y, Ma Y Z, Qi X W, Li D Q, Liu H B, Lindsey K, Zhu L F, Zhang X L. Laccasemodulates broad-spectrum biotic stress tolerance via DAMP-triggered immunity., 2017, 176: 1–34.
[21] Balasubramanian V K, Rai K M, Thu S W, Hii M M, Mendu V. Genome-wide identification of multifunctional laccase gene family in cotton (spp.); expression and biochemical analysis during fiber development., 2016, 29: 6.
[22] Li F G, Fan G Y, Lu C R, Xiao G H, Zou C S, Kohel R J, Ma Z Y, Shang H H, Ma X F, Wu J Y, Liang X M, Huang G, Percy R G, Liu K, Yang W H, Chen W B, Du X M, Shi C C, Yuan Y L, Ye W W, Liu X, Zhang X Y, Liu W Q, Wei H L, Wei S J, Huang G D, Zhang X L, Zhu S J, Zhang H, Sun F M, Wang X F, Liang J, Wang J H, He Q, Huang L H, Wang J, Cui J J, Song G L, Wang K B, Xu X, Yu J Z, Zhu Y X, Yu S X. Genome sequence of cultivated Upland cotton (TM-1) provides insights into genome evolution., 2015, 33: 524–530.
[23] Wang M J, Tu L L, Yuan D J, Zhu D, Shen C, Li J Y, Liu F Y, Pei L L, Wang P C, Zhao G N, Ye Z X, Huang H, Yan F L, Ma Y Z, Zhang L, Liu M, You J Q, Yang Y C, Liu Z P, Huang F, Li B Q, Qiu P, Zhang Q H, Zhu L F, Jin S X, Yang X Y, Min L, Li G L, Chen L L, Zheng H K, Lindsey K, Lin Z X, Udall J A, Zhang X L. Reference genome sequences of two cultivated allotetraploid cottons,and., 2019, 51: 224–229.
[24] Yu J, Jung S, Cheng C H, Ficklin1 S P, Lee T, Zheng P, Jones D, Percy R G, Main D. CottonGen: a genomics, genetics and breeding database for cotton research., 2014, 42: 1229–1236.
[25] Zhang Y, Wang X F, Rong W, Yang J, Ma Z Y. Island cotton enhanced disease susceptibility 1 gene encoding a lipase-like protein plays a crucial role in response toby regulating the SA level and H2O2accumulation., 2016, 7: 1830.
[26] Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) method.,2001, 25: 402–408.
[27] Berthet S, Demont-Caulet N, Pollet B, Bidzinski P, Cézard L, Bris P L, Borrega N, Hervé J, Blondet E, Balzergue S, Lapierre C, Jouanin L. Disruption ofandresults in tissue-specific alterations to lignification ofstems., 2011, 23: 1124–1137.
[28] Zhao Q, Nakashima J, Chen F, Yin Y B, Fu C X, Yun J F, Shao H, Wang X Q, Wang Z Y, Dixon R A.is necessary and nonredundant withfor lignin polymerization during vascular development in., 2013, 25: 3976–3987.
[29] Turlapati P V, Kim K W, Davin L B, Lewis N G. The laccase multigene family in: towards addressing the mystery of their gene function(s)., 2011, 233: 439–470.
[30] Szakasits D, Heinen P, Wieczorek K, Hofmann J, Wagner F, Kreil D P, Sykacek P, Grundler F M W, Bohlmann H. The transcriptome of syncytia induced by the cyst nematodei in Arabidopsis roots., 2010, 57: 771–784.
[31] Yang J, Zhang Y, Wang X F, Wang W Q, Li Z K, Wu J H, Wang G N, Wu L Q, Zhang G Y, Ma Z Y. HyPRP1 performs a role in negatively regulating cotton resistance tovia the thickening of cell walls and ROS accumulation., 2018, 18: 339.
[32] Li F G, Fan G Y, Wang K B, Sun F M, Yuan Y L, Song G L, Li Q, Ma Z Y, Lu C R, Zou C S, Chen W B , Liang X M , Shang H H , Liu W Q, Shi C C , Xiao G H, Gou C Y , Ye W W, Xu X, Zhang X Y, Wei H L, Li Z F, Zhang G Y, Wang G Y, Liu K, Kohel R J, Percy R G, Yu J Z, Zhu Y X, Wang J, Yu S X. Genome sequence of the cultivated cotton., 2014, 46: 567–572.
[33] Zhang T Z, Hu Y, Jiang W K, Fang L, Guan X Y, Chen J D, Zhang J B, Saski C A, Scheffler B E, Stelly D M, Hulse-Kemp A M, Wan Q, Liu B L, Liu C X, Wang S, Pan M Q, Wang Y K, Wang D, Ye W X, Chang L J, Zhang W P, Song Q X, Kirkbride R C, Chen X Y, Dennis E, Llewellyn D J, Peterson D G, Thaxton P, Jones D C, Wang Q, Xu X Y, Zhang H, Wu H T, Zhou L, Mei G F, Chen S Q, Tian Y, Xiang D, Li X H, Ding J, Zuo Q Y, Tao L N, Liu Y C, Li J, Lin Y, Hui Y Y, Cao Z S, Cai C P, Zhu X F, Jiang Z, Zhou B L, Guo W Z, Li R Q, Chen Z J. Sequencing of allotetraploid cotton (L. accTM-1) provides a resource for fiber improvement., 2015, 33: 531–537.
[34] Chezem W R, Memon A, Li F S, Weng J K, Clay N. SG2-Type R2R3-MYB Transcription factor MYB15 controls defense- induced lignification and basal immunity in Arabidopsis., 2017, 29: 1907–1926.
[35] Ye J, Zhong T, Zhang D, Ma C, Wang L, Yao L, Zhang Q, Zhu M, Xu M. The auxin-regulated protein ZmAuxRP1 coordinates the balance between root growth and stalk rot disease resistance in maize., 2019, 12: 360–373.
Genome-wide identification of Laccase gene family in updateL. genome and expression analysis understress
ZHAO Jing**, LI Xu-Tong**, LIANG Xue-Zhong, WANG Zhi-Cheng, CUI Jing, CHEN Bin, WU Li-Qiang, WANG Xing-Fen, ZHANG Gui-Yin, MA Zhi-Ying, and ZHANG Yan*
Hebei Agricultural University / North China Key Laboratory for Crop Germplasm Resources of Education Ministry / Co-Innovation Center for Cotton Industry of Hebei Province, Baoding 071001, Hebei, China
stress causes a disease in vascular bundle that decreases cotton yield and fiber quality. During cotton defense against pathogen infection, disease resistance genes play important roles. Laccase is a multifunctional oxidase that plays an important role in lignin synthesis and plant resistance. High-quality cotton reference genome is necessary to improve the accuracy of gene family analysis. In this study, the laccase () family genes in the update genome ofL. cv. TM-1 were identified by bioinformatics, and its physical and chemical properties, gene structure, chromosome location and expression pattern understress were analyzed. There were 83 members offamily in the genome ofL., which distributed on 24 chromosomes. All GhirLAC proteins predicted were located in extracellular and had the same conserved motif. Phylogenetic analysis showed that the members of thegenes family were divided into seven subgroups. According to the analysis results of cotton transcription understress, it was clear that the expression pattern ofgenes could be divided into three groups, of which, group 1 and group 2genes displayed down-regulation and up-regulation expression patterns, respectively, suggesting that these genes should play important roles in cottonresistance. Furthermore, we identified three candidate genes expression patterns induced by, including(),(), and(), the qPCR results were consistent with the expression trend based on transcriptome data. This study lays a foundation for further analysis of disease resistance function and molecular mechanism ofgene in cotton.
L.; laccase; gene family; Verticillium wilt; gene expression
本研究由河北省优秀青年科学基金项目(C2017204011), 河北省高等学校科学技术研究重点项目(ZD2014019)和河北省青年拔尖人才支持计划资助。
This study was supported by the Natural Science Foundation of Hebei Province (C2017204011), Key Scientific and Technological Research Projects of University in Hebei Province (ZD2014019), and Talents Support Program of Hebei Province.
张艳, E-mail: zhangyan7235@126.com
**同等贡献(Contributed equally to this work)
赵晶, E-mail: 824802835@qq.com
2019-04-02;
2019-06-22;
2019-07-13.
URL:http://kns.cnki.net/kcms/detail/11.1809.S.20190711.1619.006.html
10.3724/SP.J.1006.2019.94053
