蝉拟青霉类枯草杆菌蛋白酶基因的克隆及其序列和蛋白质分析
2019-10-28陈官菊柴一秋厉晓腊刘又高金轶伟
陈官菊,柴一秋,厉晓腊,方 鸣,刘又高,金轶伟
(浙江省亚热带作物研究所,浙江 温州 325005)
蝉拟青霉(IsariacicadaeMique)既是我国传统中药蝉花,又是一种昆虫的寄生真菌,其寄主范围包括鳞翅目、膜翅目和同翅目[1-3]。因此,蝉拟青霉被广泛应用于生物防治。虫生真菌通过主动寄生于昆虫,经过体表附着、体壁穿透、体内定殖和致死3个阶段[4],最终杀死寄主。大量的研究表明,虫生真菌在入侵昆虫体壁的过程中,能够分泌蛋白酶、几丁质酶、脂酶、淀粉酶等体壁降解酶以及分解纤维素和酚类等化合物的酶[5],这些酶与入侵过程中表皮的降解、侵染结构的形成以及菌株的毒力等有关,表皮降解酶一旦作用于目标靶位,便逐渐将体壁结构物质降解而形成病菌进入寄主血腔的通道。上述所列酶系中,研究较多的是胞外蛋白酶系。因为蛋白质是昆虫表皮的主要结构成分,因此,蛋白酶是决定病菌杀虫毒力的关键酶。
随着分子生物学技术水平的不断提高,从基因水平研究蝉拟青霉对昆虫的致死能力与蛋白酶活力之间的关系,对于了解蝉拟青霉侵染致病的分子机理有一定的指导意义。同时,其研究成果可用于将蝉拟青霉蛋白酶基因转入杀虫真菌受体菌,构建蝉拟青霉杀虫超级菌株,以提高蝉拟青霉的杀虫效率,这为解决蝉拟青霉致死慢、防效不稳定等问题提供了一套行之有效的方法[6]。
本研究将以蝉拟青霉为材料,根据GenBank中已注册的虫生真菌蛋白酶基因的序列,设计一对引物,用RT-PCR和3′/5′-RACE相结合的方法力图从蝉拟青霉中克隆蛋白酶基因的cDNA全序列,为蝉拟青霉作为高效稳定的杀虫物质资源的开发应用提供基础。
1 材料与方法
1.1 菌株来源
蝉拟青霉I.cicadaeAPC20为浙江省亚热带作物研究所虫生真菌研究室实验室分离、保存。其寄主为山蝉(CicadaflammataDist.),采自浙江省温州市雁荡山风景区。
1.2 主要试剂
TaqDNA聚合酶、DNA Marker DL2000、PMD18-T载体、及胶回收试剂盒等购自TaKaRa公司;E.Z.N.A.TM Total RNA Kit Ⅰ购自OMEGA公司;引物合成及测序、pUCm-T载体、两步法qRT-PCR试剂盒、GeneRacer TM Kit购自Invitrogen公司;感受态细胞E.coliTrans1-T1及JM109由南京农业大学馈赠。
1.3 总RNA的提取
按照E.Z.N.A.TMTotal RNA Kit Ⅰ提供的方法进行:培养蝉拟青霉菌丝体3 d,取30 mg组织经液氮和石英砂研磨成粉末,转移粉末至TRK裂解液裂解,20 ℃,20 000 ×g,离心10 min;取上清,加入等体积的70%乙醇,充分混匀,将微型柱置于2 mL离心管之上, 并将所有的混合液加入到微型柱中,20 ℃,10 000 ×g,离心60 s,弃去滤液;加入300 μL RNA洗脱液Ⅰ,20 ℃,10 000 ×g,离心60 s,弃滤液;加入DNA酶Ⅰ消化,静置15 min,加入500 μL RNA洗脱液Ⅰ,20 ℃,10 000 ×g,离心60 s,弃滤液;将微型柱置于新的2 mL离心管上,加入500 μL乙醇稀释过的RNA洗脱液Ⅱ,20 ℃,10 000 ×g,离心60 s,弃滤液;加入500 μL RNA洗脱液Ⅱ再次洗涤微型柱,20 ℃,20 000×g,离心60 s,弃滤液;将离心柱置于空的2 mL离心管上,20 ℃,10 000 ×g,离心2 min。转移微型柱至新的1.5 mL的离心管上,加入30 μL的DEPC处理的水洗脱柱子,20 ℃,20 000×g,离心3 min。-70 ℃保存备用。
1.4 cDNA特异片段的RT-PCR扩增
取总RNA(约1 μg),加入1 μL oligo (dT)12~18(500 μg·mL-1),1 μL dNTP(10 mmol·L-1),加双蒸水至12 μL,65 ℃水浴5 min,稍微离心甩一下,冰上放置5 min;加入4 μL 5×第一链合成缓冲液,2 μL DDT(0.1 mmol·L-1),1 μL RNaseOUTTM核酸酶抑制剂(40 U·μL-1),37 ℃温浴2 min;加入1 μL M-MLV逆转录酶,混匀,37 ℃温浴50 min,70 ℃加热15 min终止反应。-20 ℃保存备用。
根据GenBank中已登录的淡紫拟青霉、粉拟青霉、球孢白僵菌和金龟子绿僵菌的类枯草杆菌蛋白酶基因序列,用DNAStar软件中的MegAlign比较其同源性,发现一对较为保守的DNA区域序列,据其设计的一对引物为:PCPr1F,5′-CTGGCTTCCGTGGTTATGCTGG-3′;PCPr1R,5′-CACCACCAAGAGACATGTTGGCGAC-3′。
PCR反应体系(25 μL)含有:2.5 μL 10×Taq酶缓冲液,dNTP各0.2 mmol·L-1,MgCl22.0mmol·L-1,引物各0.01 nmol·L-1,rTaq0.5 μL,模板DNA 1.0 μL。扩增反应在PCR仪上进行:95 ℃ 4 min,1个循环;95 ℃ 45 s,60 ℃ 40 s,72 ℃ 1 min,30个循环;最后72 ℃ 10 min。扩增产物在1.2%琼脂糖凝胶(含EB)中电泳1 h,凝胶成像仪检测结果。
使用TaKaRa Agarose Gel DNA Purification kit Ver 2.0电泳片段回收试剂盒进行特异条带的回收,回收片段连接PMD18-T载体,转化感受态细胞Trans1-T1,获得转化质粒,经菌落PCR验证后委托上海Invitrogen测序公司进行测序。所有扩增反应均在美国Bio-Rad公司生产的PTC-200 PCR仪上进行。
1.5 3′/5′端RACE-PCR扩增
按GeneRacer TM Kit提供的方法进行,其中,GeneRacerTMRNA Oligo 序列为:5′-CGACUGGAGCACGAGGACACUGACAUGGACUGAAGGAGUAGAAA-3′;GeneRa-cerTMOligo dT 引物序列为: 5′-GCTGTCAACGATACGCTACGTAACGGCATGACAGTG(T)24-3′ (60 bases)
5′模板合成:加入7 μL总RNA,1 μL 10×CIP Buffer,1 μL RNase OUT(10 U·μL-1),1 μL CIP(10 U·μL-1),用枪头轻轻混匀,瞬时离心,50 ℃水浴1 h,瞬时离心,置冰上;依次加入90 μL DEPC水和100 μL酚/氯仿混合液,上下混匀,最大转速离心5 min,转移上层至新的离心管;加入2 μL 10 mg·mL-1糖原,10 μL 3 mol·L-1醋酸钠(pH 5.2),混匀,再加入220 μL 95%乙醇,上下混匀,-20 ℃过夜保存,4 ℃,最大转速离心20 min,弃去上清,加入500 μL 70%乙醇,颠倒几次,上下混匀,4 ℃,最大转速离心2 min,去乙醇,再次离心去除残余乙醇,室温干燥1~2 min;加入7 μL DEPC水重悬RNA;依次加入1 μL 10×TAP缓冲液,1 μL RNase OUT(10 U·μL-1),1 μL TAP(0.5 U·μL-1),用枪头轻轻吸打混匀,瞬时离心,37 ℃温浴1 h,瞬时离心,置冰上,沉淀RNA沉淀(方法同上);加入7 μL DEPC水重悬RNA;将6 μL去磷酸化,去帽子结构的RNA加入分装好RNA Oligo(0.25 μg)的试剂管(先离心),吸打混匀,瞬时离心,65 ℃温浴5 min,冰上放置约2 min,瞬时离心,加入1 μL 10×Ligase Buffer,1 μL ATP(10 mmol·L-1),1 μL RNase Out(10 U·μL-1),1 μL T4 RNA ligase(5 U·μL-1),混匀,且瞬时离心;37 ℃温浴1 h,瞬时离心,置冰上;依次加入10 μL RNA连接液,1 μL GeneRace oligodT,1 μL dNTP Mix,1 μL DEPC水,65 ℃温浴5 min,冰上放置至2 min,瞬时离心,加入4 μL 5×First Strand Buffer,1 μL DTT(0.1 mol·L-1),1 μL RNase Out(10 U·μL-1),1 μL SuperSeript Ⅲ RT,枪头吸打混匀,瞬时离心,50 ℃温浴50 min,然后70 ℃温浴15 min,冰上放置2 min,最大离心力瞬时离心,加入1 μL RNase H(2 U),37 ℃温浴20 min,瞬时离心,-70 ℃保存。
根据已获得的蝉拟青霉Pr1基因片段序列,设计5′RACE引物序列:其中,SLP 5′RACE引物:rSLP-R1,5′-CGACATGGTGCCCGAGCCGTTAGA-3′(465-488);rSLP-R2,5′-CCTCAACGTCAGGGTGGTTGCGAAT-3′(51-75)。5′GeneRacer outer primer,5′-CGACTGGAGCACGAGGACACTGA-3′;5′GeneRacer inner primer,5′-GGACACTGACATGGACTGAAGGAGTA-3′。
5′RACE扩增反应:第一轮反应体系(25 μL):0.5 μL 5′RACE模板,1.0 μL 5′GeneRacer outer primer(10 μmol·L-1),1.0 μL rSLP-R1(10 μmol·L-1),22.5 μL Platinum PCR Supermix High Fidelity。Touch-down PCR循环条件为:94 ℃ 2 min;94 ℃ 30 s,72 ℃ 1 min,5个循环; 94 ℃ 30 s,70 ℃ 1 min,5个循环;94 ℃ 30 s,66 ℃ 30 s,68 ℃ 1 min,25个循环。进行第二轮反应,PCR体系(25 μL):0.5 μL第一轮PCR产物,1.0 μL 5′GeneRacer inner primer(10 μmol·L-1),1.0 μL rSLP-R2(10 μmol·L-1),22.5 μL Platinum PCR Supermix High Fidelity。循环条件为:94 ℃ 2 min;94 ℃ 30 s,66 ℃ 30 s,68 ℃ 1 min,30个循环。二次反应结束后,1.2%琼脂糖电泳检测结果。切胶回收,连接pUCm-T载体,转化感受态细胞JM109,送往上海Invitrogen公司进行测序。
3′模板合成:加入10 μL总RNA,1 μL GeneRacerTMOligo dT Primer,1 μL dNTP Mix,1 μL DEPC水,65 ℃温浴5 min,冰上放置2 min,瞬时离心;依次加入4 μL 5×First Strand Buffer,1 μL DDT(0.1 mol·L-1),1 μL RNaseOut(10 U·μL-1),1 μL SuperSeript Ⅲ RT,枪头吸打混匀,瞬时离心;50 ℃温浴50 min,70 ℃温浴15 min,冰上放置2 min,瞬时离心;加入1 μL RNase H(2 U),37 ℃温浴20 min,瞬时离心,-70 ℃保存。
根据已获得的蝉拟青霉Pr1基因片段序列,设计3′RACE引物序列。其中,SLP 3′RACE引物:rSLP-F1,5′-CACCACCGTCCCCGTCACCACCA-3′;rSLP-F2,5′-GACACTCTCGGTTTCGGCACCTTCAA-3′。3′GeneRacer outer primer,5′-GCTGTCAACGATACGCTACGTAACG-3′;3′GeneRacer inner primer,5′-CGCTACGTAACGGCATGACAGTG-3′。
3′RACE扩增反应。第一轮反应体系(25 μL):0.5 μL 3′RACE模板,1.0 μL 3′GeneRacer outer primer(10 μmol·L-1),1.0 μL rSLP-F1(10 μmol·L-1),22.5 μL Platinum PCR Supermix High Fidelity。Touch-down PCR循环条件为:94 ℃ 2 min;94 ℃ 30 s,72 ℃ 2 min,5个循环;94 ℃ 30 s,70 ℃ 2 min,5个循环;94 ℃ 30 s,65 ℃ 30 s,68 ℃ 2 min,25个循环。进行第二轮反应,PCR体系(25 μL):0.5 μL第一轮PCR产物,1.0 μL 3′GeneRacer inner primer(10 μmol·L-1),1.0 μL rSLP-F2(10 μmol·L-1),22.5 μL Platinum PCR Supermix High Fidelity。循环条件为:94 ℃ 2 min;94 ℃ 30 s,65 ℃ 30 s,68 ℃ 1.5 min,30个循环。二次反应结束后,1.2%琼脂糖电泳检测结果。切胶回收,连接pUCm-T载体,转化感受态细胞JM109,送往上海Invitrogen公司进行测序。
1.6 全长基因的PCR检验
利用最终序列,设计引物rSLP-F: 5′-GCGTCAGCAACTCTCTACACT-3′;rSLP-R: 5′-CGACGCCACAACGGACAAGTT-3′,模板为5′、3′RACE扩增产物。PCR反应体系(25 μL)含有:模板DNA各0.5 μL,引物(10 μmol·L-1)各1.0 μL,Platinum PCR Supermix High Fidelity(Invitrogen)22 μL。扩增反应在PCR仪上进行:94 ℃ 2 min,1个循环;94 ℃ 30 s,60 ℃ 30 s,68 ℃ 1.5 min,30个循环;最后72 ℃ 10 min。扩增产物在1.2%琼脂糖凝胶(含 EB)中电泳1 h,凝胶成像仪检测结果。
1.7 蛋白结构分析和预测
蛋白质结构分析主要用DNAStar软件分析;信号肽预测、等电点和分子量用网上服务器http://www.expasy.org分析工具SignalP、compute PI/MW。
2 结果与分析
2.1 蝉拟青霉类枯草杆菌蛋白酶cDNA特异片段的RT-PCR扩增
用引物PCPr1F和PCPr1R进行特异性扩增,经1.2%的琼脂糖凝胶电泳检测(图1),得到一条特异性很好的条带,估计长度600 bp左右,测序实得的序列长度为605 bp,与设计引物时所预测的序列长度(656 bp)有51 bp的差距。但用Blast在GenBank中进行比较的结果表明,该片段的核苷酸序列与虫草菌(Cordycepscicadae)的PR1H(AGK07304.1)同源性最高,达到100%。
2.2 蝉拟青霉类枯草杆菌蛋白酶cDNA的3′/5′-RACE扩增
在第一轮RACE-PCR反应中,无论是3′-RACE反应,还是5′-RACE反应均为弥散的DNA条带。但在第二轮的RACE-PCR反应中,分别得到一条很亮的特异条带,其长度约为1 300 bp和500 bp(图2)。对3′-RACE和5′-RACE的第二轮扩增产物进行测序,其结果分别为1 354 bp和549 bp,与上述特异片段分别有521个核苷酸和28个核苷酸重叠,所以第二轮扩增产物分别为同一cDNA的3′端和5′端。

M,Marker DL2000;1,扩增片段转化DH5α后菌落PCR产物;2,扩增片段转化DH5α后提取质粒PCR产物。M, Ladder marker of DL2000; 1, Cloned special fragment of DH5α colony; 2, Cloned special fragment of DH5α plasmid.
2.3 蝉拟青霉类枯草杆菌蛋白酶cDNA全序列分析
通过RT-PCR和3′/5′-RACE扩增片段的测序结果,除去重叠区域,最后确定蝉拟青霉类枯草杆菌蛋白酶cDNA全序列为2 031 bp(KC508612.1,GenBank),与PCR方法检查3′/5′-RACE扩增片段的电泳结果相吻合(图3)。此蛋白起始密码子(ATG)和终止子(TAA)分别在171碱基和1 770碱基处,编码阅读框由1 599个核苷酸组成,5′-非翻译区(5’-UTR)与3′-非翻译区(3′-UTR)分别为170个核苷酸和262个核苷酸(图4)。
在GenBank中通过Blast进行比对,该基因编码的蛋白序列与虫草棒束孢(Isariafarinosa)、球孢白僵菌(Beauveriabassiana)、蛹虫草(Cordycepsmilitaris)和哈茨木霉(Trichodermaharzianum)类枯草杆菌蛋白酶(subtilisin-like protease)有较高的同源性,同源性分别为88%、88%、89%和71%,说明该基因确为蝉拟青霉类枯草杆菌蛋白酶基因。

M,Marker DL5000;5′,5′-RACE DNA扩增片段;3′,3′-RACE DNA扩增片段。M, Ladder marker of DL5000; 5′, 5′-RACE with rSLP-F1 and rSLP-R2; 3′, 3′-RACE with rSLP-R1 and rSLP-F2.
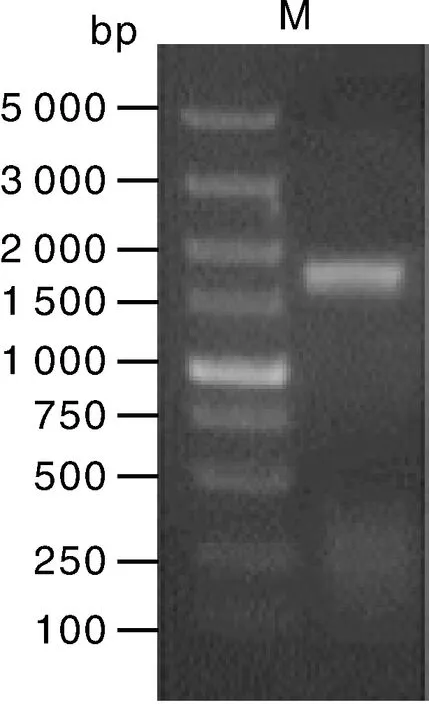
M,Marker DL5000;1,目的基因。M, Ladder marker of DL5000; 1, Target gene.
2.4 蝉拟青霉类枯草杆菌蛋白酶的生物信息学分析及其同源性分析
蝉拟青霉类枯草杆菌蛋白酶前体蛋白理论分子量为56.9 ku,理论等电点为6.329,为水溶性蛋白,共有532个氨基酸组成,其中含酸性氨基酸68个(D、E),碱性氨基酸56个(K、R),极性氨基酸129个(Y、S、T、C、M、N、Q),疏水氨基酸187个(W、A、V、L、I、F),分别占总氨基酸数量的12.8%,10.5%,24.2%,35.2%。
N端的18个氨基酸被Signal P在线预测为信号肽,这段信号肽碱性氨基酸1个(K),极性氨基酸5个(S、Q),疏水氨基酸12个(M、V、I、A、L)。
根据同源性比较,成熟的枯草杆菌蛋白酶应当从152的Lys开始,有5个半胱氨酸(Cys232、Cys335、Cys366、Cys401、Cys459),有4个潜在的N-糖基化位点(258NGS、298NMS、336NYS和474NRS)(图4)。
根据软件预测结果显示,蝉拟青霉枯草杆菌蛋白酶(PR1)位于细胞的液泡中与分泌途径相关。通过SOPM方法和DNAMAN 4.0软件预测蛋蛋白二级结构,表明蝉拟青霉枯草杆菌蛋白酶(PR1)二级结构中以α-螺旋和无规则卷曲为主,分别占32.52%和33.46%,β-片层占22.56%,β-转角占11.47%。依据同源建模原理,运用SWISS-MODEL工具和X-RAY DIFFRACTION 2.10A方法对PR1蛋白的三级结构分析结果显示,选取与蝉拟青霉枯草杆菌蛋白酶(PR1)相似率为31.14%的3whi.1.A作为模型,构建了蝉拟青霉枯草杆菌蛋白酶(PR1)的三级结构(图5)。

图4 蝉拟青霉类枯草杆菌蛋白酶cDNA和推导氨基酸序列

蓝色部分表示α-螺旋;橙黄色部分表示无规则卷曲;红色部分表示β-片层;绿色部分表示β-转角。The blue part represented α-helixes; The orange part represented random colis; The red part represented β-sheets; The green part represented β-turns.
3 讨论
目前已知昆虫病原真菌的胞外酶系主要有蛋白酶、几丁质酶、酯酶、脂肪酶、DNA酶及其他分解纤维素和酚类化合物的酶或酶系。上述所列酶系中,研究较为清楚的是蛋白酶,其次是几丁质酶。因为蛋白质是昆虫表皮的主要结构成分,因此,蛋白酶是决定病菌杀虫毒力的关键酶[7-8]。在昆虫病原真菌中与毒力有关的蛋白酶目前主要分为两类:一类为丝氨酸弹性凝乳蛋白酶(类枯草杆菌蛋白酶,简称为Pr1),另一类为丝氨酸类胰蛋白酶(简称为Pr2)。丝氨酸弹性凝乳蛋白酶是昆虫病原真菌的重要毒力因子,并且是由多个同源蛋白酶组成的家族。已报道的与杀虫活性有关蛋白降解酶20多种,包括Pr1A、Pr1B、Pr1C、Pr1D、Pr1E、Pr1F、Pr1H、Pr1I、Pr2和Pr3等类型。在昆虫病原真菌中,第一个被克隆出来的表皮降解酶基因就是金龟子绿僵菌的Pr1A(当时被称为Pr1)基因[9],在蝉拟青霉中,本研究首次克隆了与侵染过程相关的类枯草杆菌蛋白酶基因。序列显示它合成一个前体肽,包含信号肽和前肽。对于信号肽区域来讲,通常认为疏水核心区含9个疏水氨基酸就可以满足跨膜的需要[10]。这段信号肽符合上述特点,因此我们认定该蛋白为胞外分泌蛋白,可以分泌到细胞膜外来分解昆虫体壁上的蛋白质,以协助真菌完成侵染过程。Leger等[9]克隆的Pr1A基因的信号肽也有18个氨基酸,不同的是其核心区仅含8个疏水氨基酸。前肽的作用可能是在转运过程中屏蔽自身分解蛋白的活性,这是蛋白酶具有前肽的特点。但该区域和其他虫生真菌PR1H的前肽同源性不高,Leger等[9]的研究曾指出该区域序列不保守,因而进化较快。
蝉拟青霉作为一类潜在的农业害虫生防菌,在对其开发利用方面也存在虫生真菌的通病。但是有关蝉拟青霉中降解昆虫体壁蛋白酶基因的报道尚处于空白。尽管不同虫生真菌的Pr1酶的功能是相同或相似的,但由于其编码基因的部分核苷酸的变化可能使氨基酸序列发生改变,会造成二级结构差异,导致这些Pr1酶在亲疏水性能和抗原性上表现出一定的差异[6],因此,从蝉拟青霉中克隆具有降解昆虫体壁功能的蛋白酶基因并对其功能进行鉴定分析,对了解蝉拟青霉的致病机理有着重要意义。对蝉拟青霉中具有降解昆虫体壁功能的蛋白酶基因进行克隆,将为从分子水平探索病原菌-昆虫的互作机制奠定基础,并为新型高效安全的杀虫剂的研发提供新思路,开辟新途径。
本研究报道了蝉拟青霉类枯草杆菌蛋白酶基因片段的克隆,通过序列分析发现其与其他病原菌的类枯草杆菌蛋白酶基因之间存在较高的同源性,这为进一步获取蝉拟青霉蛋白酶基因全序列奠定了基础,并对今后研究蝉拟青霉蛋白酶结构和功能提供了依据。
