基于高通量测序技术的鱼类环境DNA研究中通用引物的筛选验证
2019-10-28张爱菊郝雅宾郭爱环刘金殿练青平周志明
张爱菊, 郝雅宾, 郭爱环, 刘金殿, 练青平, 周志明
(浙江省淡水水产研究所,中国水产科学研究院东海水产研究所浙江研究中心,浙江省淡水水产遗传育种重点实验室,农业农村部淡水渔业健康养殖重点实验室,浙江 湖州 313001)
2008年,法国学者Ficetola等[1]利用从水中提取的环境DNA(environmental DNA,eDNA)检测水域中是否有入侵物种美国牛蛙(Ranacatesbeiana),开启了eDNA技术在水生生物实时监测中的研究与应用。eDNA是指可以从环境样品(如水、土壤、空气等)中直接提取到的DNA片段总和,既包含生物体经由皮肤、尿液、粪便、黏液等释放到环境中表皮细胞中的胞内DNA,也包括细胞死亡后裂解释放到环境中的胞外DNA[2]。eDNA技术的原理是在确定调查物种或种群的特异性基因识别片段的基础上,利用各种分子手段检测从环境介质中所提取eDNA包含识别片段的情况,进而确定取样环境中生物的分布状况[3-4]。
eDNA被提取后,需要采用高通量测序或克隆测序等技术进行后续实验分析。目前欧美或日本等国将eDNA技术应用于目标物种(如入侵物种、濒危物种及其他稀有物种)的检测、资源量的估测、水体生物多样性的调查等方面[5-7]。国内eDNA技术的利用研究尚处于起步阶段,多采用克隆测序技术检测水中的目标物种和物种多样性[8-11]。然而,不论哪种测序技术,利用eDNA进行群落结构分析都需要有合适的通用性引物。由于环境中DNA易于受到紫外线和微生物分解作用的影响,物种的长DNA片段十分容易降解,故短DNA片段普遍被用于eDNA研究[2,12-13]。然而,不同的引物获得的扩增结果往往差异很大[14]。因此,筛选出一个通用性和适用性都比较理想的通用引物是利用eDNA技术研究鱼类群落结构的关键技术之一。
高通量测序技术的核心思想是边合成边测序,相比于克隆测序具有耗时短、获得信息全等优点。Illumina Miseq测序平台是高通量测序的一款小型测序平台,可以高通量、并行对核酸片段进行深度测序,目前可以支持500~600 bp的读长。由于Miseq测序读长的限制,同时也为了保证测序质量,最佳测序的插入片段范围是200~450 bp,故选取的引物扩增产物片段大小也应在此范围。由于鱼类区系的差异,我国水域生态系统中的鱼类群落结构与国外不同,因此国外研究中提供的鱼类通用引物的通用性和适用性也有待进一步验证。
钱塘江是浙江省第一大河,流域面积约5.5万km2,历来是杭州市的主要饮用水源地,除此之外,钱塘江还兼具发电、防洪、灌溉、游览等多种功能。近年来,随着钱塘江流域经济的快速发展和城市化进程的加快,以及水利工程建设、采砂作业、渔业资源的过度捕捞等人为因素的影响,水域环境发生变迁,其渔业资源群体结构也随之发生变化。据统计,20世纪70年代末80年代初,钱塘江可以采集到的鱼类标本有136种;1986—2001年钱塘江杭州段可采集到的鱼类标本为127种;2014—2015年,钱塘江杭州段采集到的鱼类标本为70余种。这些结果均通过传统的捕捞调查方法获得,但由于方法的限制,可能导致一些常规种类未采集到的情况。本研究从国内外文献中筛选出5对线粒体基因组部分片段的通用引物,采用Illumina Miseq法对这些引物在钱塘江富阳段环境样品中的通用性和适用性进行了研究,以期为钱塘江水系鱼类多样性及水生生态系统保护提供参考。
1 材料与方法
1.1 水样采集和DNA提取
2017年6月在钱塘江富阳段中下游选取9个点采集表层水样,每份样品5 L,立即带回实验室后放入4 ℃冷藏箱,24 h内对水样进行抽滤处理,使用滤膜孔径为0.45 μm[8,15]。使用Omega公司生产的试剂盒EZNA water DNA kit提取滤膜中的eDNA。提取好的DNA溶于TE缓冲液中,-20 ℃保存备用。
1.2 引物选择
与核DNA相比,鱼类线粒体DNA具有分子小、结构简单、易于提取且是母系遗传的特点,因此线粒体DNA片段常作为分析鱼类种群结构、物种分类鉴定的分子标记[16]。迄今为止,国内外学者常选用Cytb、COⅠ基因或16S rDNA等的片段作为鱼类的识别片段[6-8]。参照国内外文献[11,17-19],找到5对线粒体基因组部分序列的鱼类通用引物,其扩增产物分别属于线粒体Cytb基因、Cytb基因、16S rDNA、16S rDNA和COⅠ基因片段。引物均由杭州联川生物技术股份有限公司合成,详见表1。
1.3 PCR扩增及质控
PCR初筛:用选取的5对引物分别对2个水样eDNA进行扩增。25 μL反应体系包括:2.5 μL 10×buffer,2 μL dNTP(2.5 mmol·L-1),各1 μL正反引物(10 μmol·L-1),0.15 μLTaq酶(5 U·μL-1),1 μL DNA模板(约50 ng),17.35 μL ddH2O。反应条件为94 ℃预变性2 min;94 ℃变性30 s,退火40 s(各引物退火温度见表1),72 ℃延伸1 min,35个循环;71 ℃延伸10 min。扩增后各取5 μL的PCR产物于1%的琼脂糖凝胶中进行电泳检测,电压100 V,电泳60 min,用溴化乙锭染色8 min,最后在凝胶成像系统上拍照,并统计每对引物的扩增效果。
PCR验证:依据初筛结果,选取扩增效果良好的引物,分别再对剩余7个eDNA样品进行扩增,反应条件和体系、凝胶成像条件不变,以此验证引物的实际通用效果,每对引物设置1个阴性对照。
1.4 目的片段文库构建、测序及数据分析
使用PCR扩增产物建库,文库构建步骤遵循Illumina测序仪文库构建方法,其中引物序列采用筛选出的能够对环境样品良好扩增的引物。通过2%琼脂糖凝胶电泳检测PCR扩增产物,并采用AxyPrep PCR Cleanup Kit回收试剂盒对目标片段进行回收。
对纯化后的PCR产物采用Quant-iT PicoGreen dsDNA Assay Kit在Promega QuantiFluor 荧光定量系统上对文库进行定量。将合格的上机测序文库(文库浓度≥2 nmol·L-1)梯度稀释后,依据所需测序量按相应比例混合,并经NaOH变性为单链上机测序。测序采用Illumina Miseq平台。测序获得的数据经拆分、拼接和过滤等分析后,得到高质量的序列。应用软件Vsearch 2.3.4对高质量序列在97%的相似性水平上进行聚类,产生可操作性分类单元(operational taxonomic units,OTUs)。以每个OTUs中最长的序列作为代表性序列,通过BLAST在NCBI数据库中查找同源序列,将最相近且可信度达到90%以上序列的种属信息,作为该序列的物种注释信息。由于本研究只关注于鱼类多样性的研究,故只标注注释上的鱼类物种信息。
2 结果与分析
2.1 DNA提取结果
各样本的核酸浓度为321.10~523.3 ng·μL-1,核酸总量为14.09~20.93 μg,符合后续扩增和建库要求。
表1PCR所用的5对通用引物
Table1Five pairs of universal primers for PCR
2.2 初筛扩增结果
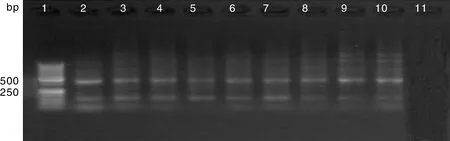
分别用5对引物对提取的2个水样eDNA进行PCR扩增,扩增产物的凝胶电泳如图1。引物CY45M在2~3泳道的相似位置出现了相对清晰的条带,该扩增条带长度符合预期长度。引物1634M在4~5泳道的相似位置出现了明亮清晰的条带,且该条带的长度符合预期长度,显示出引物1634M对eDNA具有良好的扩增效果。泳道6~9均未见明显的扩增条带,说明引物16ACM和COⅠM在水样eDNA中无法扩增出目的条带。引物CYFM在泳道10~11上分别有一条暗淡的条带,该条带长度超过1 000 bp,不符合目的条带的预期长度,显示该引物对eDNA的扩增效果不佳。
2.3 验证扩增结果
选取具有良好扩增效果的引物CY45M和1634M,对剩余7个水样eDNA进行PCR扩增。以9个水样eDNA为模板的扩增产物的凝胶电泳如图2和图3。结果发现,引物CY45M扩增出了所有环境样品中的Cytb基因片段,目的条带清晰,但是在目的条带下方出现了一条杂带。引物1634M的扩增条带干净清晰,无非特异性条带,无拖带现象。总体来看,2对引物对环境样品的扩增效果均能满足后续的实验要求。
2.4 Illumina测序结果
为进一步验证测序效果,采用Illumina MiSeq测序平台对9个水样eDNA的扩增产物进行高通量测序。CY45M扩增产物测序共获得50 510~136 715条原始序列,优化后获得32 070~76 930条高质量序列,有效数据率为50.69%~71.42%;1634M扩增产物测序共获得36 811~77 604条原始序列,优化后获得35 916~74 557条高质量序列,有效数据率为95.17%~97.75%。所有高质量序列在97%的相似性水平上对其进行聚类,结果引物CY45M共获得3 738个OTUs,而1634M共获得2 305个。

泳道1,GeneRuler 50 bp DNA ladder;泳道2—3,引物CY45M的扩增产物;泳道4—5,引物1634M的扩增产物;泳道6—7,引物16ACM的扩增产物;泳道8—9,引物COⅠM的扩增产物;泳道10—11,引物CYFM的扩增产物。Lane 1, GeneRuler 50 bp DNA ladder; Lane 2-3, PCR products of CY45M; Lane 4-5, PCR products of 1634M; Lane 6-7, PCR products of 16ACM; Lane 8-9, PCR products of COⅠM; Lane 10-11, PCR products of CYFM.
泳道1,GeneRuler 50 bp DNA ladder;泳道2—10,引物CY45M依次以1至9号水样eDNA为模板的扩增产物;泳道11,阴性对照。Lane 1, GeneRuler 50 bp DNA ladder; Lane 2-10, PCR products of nine water eDNA samples amplified by the primer CY45M; Lane 11, Negative control.
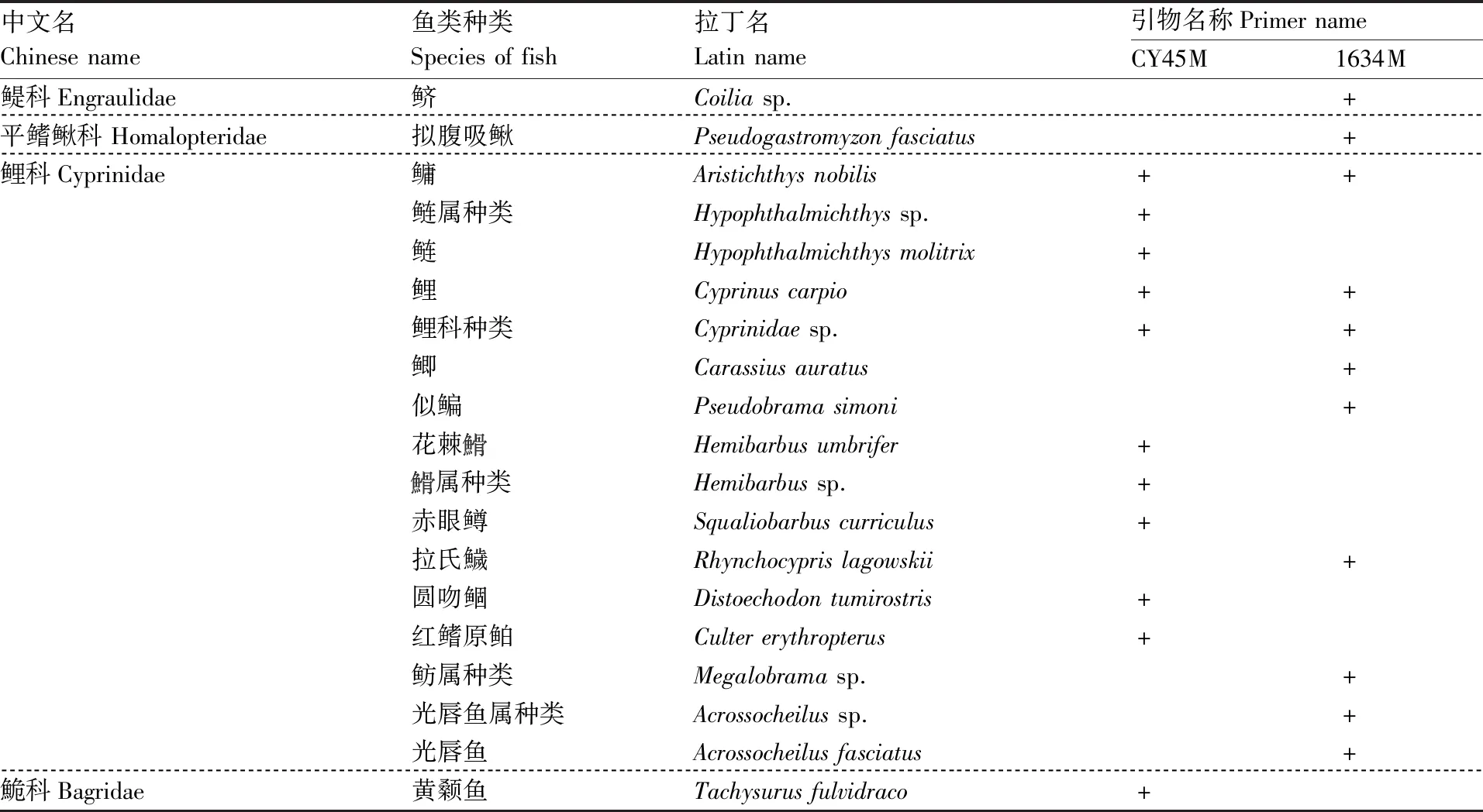
通过BLAST对OTUs的代表性序列进行物种注释,引物CY45M扩增产物注释到物种信息的OTUs数目占所有OTUs数目的0.02%~0.16%,共注释到鱼类物种2目2科8属11种,其中鲤科10种,鮠科1种(表2);1634M扩增产物注释到物种信息的OTUs数目占所有OTUs数目的0.00%~0.71%,注释到2目3科10属11种鱼类物种,其中鳀科1种,平鳍鳅科1种,鲤科9种(表2)。总体上看,两对引物共检出鱼类种类19种,其中鲤科种类数最多,为16种,占检出种类总数的84.21%。同时2对引物注释上的鱼类种类既存在差异也存在重复。在19种注释上的鱼类中,引物CY45M特异注释上的有鲢(Hypophthalmichthysmolitrix)、花棘(Hemibarbusumbrifer)、赤眼鳟(Squaliobarbuscurriculus)、圆吻鲴(Distoechodontumirostris)、红鳍原鲌(Cultererythropterus)、黄颡鱼(Tachysurusfulvidraco)等8种,而引物1634M特异注释上的鱼类有拟腹吸鳅(Pseudogastromyzonfasciatus)、鲫(Carassiusauratus)、似鳊(Pseudobramasimoni)、拉氏鱥(Rhynchocyprislagowskii)、光唇鱼(Acrossocheilusfasciatus)等8种,2对引物的特异性扩增率均达到72.73%,而共同注释上的鱼类有鳙(Hypophthalmichthysnobilis)、鲤(Cyprinuscarpio)、鲤科种类(Cyprinidaesp.)3种。
表2 使用引物CY45M和1634M扩增环境样品的产物比对结果
Table2The results of BLAST of PCR production amplified by primer CY45M and 1634M from environmental samples
中文名Chinesename鱼类种类Speciesoffish拉丁名Latinname引物名称PrimernameCY45M1634M鳀科Engraulidae鲚Coiliasp.+平鳍鳅科Homalopteridae拟腹吸鳅Pseudogastromyzonfasciatus+ 鲤科Cyprinidae鳙Aristichthysnobilis++ 鲢属种类Hypophthalmichthyssp.+鲢Hypophthalmichthysmolitrix+鲤Cyprinuscarpio++鲤科种类Cyprinidaesp.++鲫Carassiusauratus+似鳊Pseudobramasimoni+花棘Hemibarbusumbrifer+属种类Hemibarbussp.+赤眼鳟Squaliobarbuscurriculus+拉氏鱥Rhynchocyprislagowskii+圆吻鲴Distoechodontumirostris+红鳍原鲌Cultererythropterus+鲂属种类Megalobramasp.+光唇鱼属种类Acrossocheilussp.+光唇鱼Acrossocheilusfasciatus+鮠科Bagridae黄颡鱼Tachysurusfulvidraco+
“+”代表该种类可被检测到。
The ‘+’ indicated the corresponding species could be detected.
本研究发现,不同水样使用同种引物注释上的鱼类种类存在差异,如同样使用CY45M,9个水样扩增产物注释上的鱼类为2~7种,最高种类数出现在4号水样,最低出现在3号水样,而使用1634M注释上的鱼类为0~8种。虽然各注释上物种的相对丰度在不同水样间存在差异,鲤科鱼类的相对丰度始终占据优势地位。此外,在相同的水样中,2对引物扩增产物的注释结果也不同,一般来说,CY45M注释上的种类数多于1634M。然而,所有水样中不能被注释的OTUs仍占绝大多数(图4、图5)。

图4 引物CY45M扩增钱塘江调查水域各水样eDNA鱼类物种注释结果
图5 引物1634M扩增钱塘江调查水域各水样eDNA鱼类物种注释结果
3 讨论
3.1 水样采集和DNA提取方法
国内外研究中,采用eDNA方法研究鱼类物种多样性的水样采样量从15 mL到10 L不等,其中采用最多的采样量为1~2 L[2],国内采用的水样采集量多为2 L[8-9]。研究表明,不同采样量所对应结果的准确度与水体中目标物种的密度有关。实验室条件下,非洲宝石鱼(Hemichromisletourneuxi)密度每增加2.53尾·m-3,每升水样的检测成功率随之增加4.86倍[24]。考虑到本次调查水域为开放性水域且水体流动性强等特点,建议采用的采集量为5 L。同时,本研究使用的DNA提取试剂盒采用裂解法和硅胶柱纯化方式提取水样中的总DNA,结果显示,该试剂盒可以有效且稳定地提取出水样中的eDNA。
3.2 鱼类物种多样性引物筛选及验证
在水体中,为了调查鱼类多样性,需要设计通用引物,该引物应能够扩增尽可能多的鱼类物种的识别片段。刘军等[14]、Kitano等[21]认为,Cytb区域、16S rDNA中有适合设计鱼类通用引物的基因片段。本研究的引物CY45M和1634M分别在Cytb基因和16S rDNA的保守区域设计,其扩增产物片段大小在Miseq测序读长的限制范围内,在脊椎动物(包括哺乳动物、鸟类、鱼类、两栖类、爬行类)中均能获得良好扩增[17,21]。如引物CY45M在琵琶湖的61种鱼类中发现仅有3.8个碱基不匹配[17],对千岛湖45种鱼类DNA样品进行扩增后成功获得42种鱼类的目的基因片段[14]。引物1634M在鱼类DNA样品中也存在良好的扩增效果[21]。
本研究中,CY45M扩增出了所有9个环境样品中的Cytb基因片段,而刘军等[14]利用此引物无法扩增出10个环境样品中的目的基因。分析原因可能是该引物的扩增条件差异造成的,本研究使用的扩增条件更有利于环境样品eDNA的扩增。
大量研究发现,即使是对水样eDNA具有良好的扩增效果的通用引物,在对水样eDNA进行扩增测序后可检测到的鱼类物种普遍较少[14,17]。刘军等[14]采用16s rDNA基因引物对千岛湖水样eDNA进行扩增,其产物克隆测序后仅检出黄尾鲴(Xenocyprisdavidi)、蓝鳃太阳鱼(Lepomismacrochirus)、细鳞鲴(Plagiognathopsmicrolepis)、蒙古鲌(Chanodichthysmongolicus)、鲢和鳙这6种鱼类的序列;采用COⅠ基因引物获得的水样eDNA扩增产物经比对发现均为细菌的细胞色素氧化酶第一亚基。Minamoto等[17]利用引物CY45M对日本Yura江中的水样eDNA进行扩增测序后比对上了6种鱼类的物种信息。本研究测序结果表明,引物CY45M和1634M扩增产物注释上的鱼类均为11种,特异注释上的鱼类也均为8种,而造成这一结果的原因仍需进一步研究。虽然本研究仅检出了钱塘江富阳段中分布鱼类的一小部分,但仍很有借鉴意义。
同时,本次研究发现,不同引物获得的扩增结果往往具有较大差异,且即使选用同一个基因片段,获得较小的扩增产物片段的引物更有利于获得较高的扩增成功率[20]。本研究结果进一步证实了这一观点,即使用2种在不同保守区域设计的引物注释上的鱼类物种存在较大的差异。分析后发现,绝大部分水样使用CY45M注释上的物种数高于1634M。推测可能是1634M引物的特异性较强、2对引物扩增的目的基因片段降解率不同、比对数据库中的目的基因登录信息差异等原因造成的。同时,不同水样使用同种引物注释上的物种也存在差异。如使用CY45M注释上的鱼类物种为2~7种,使用1634M注释上的鱼类物种为0~8种,采样点差异可能是造成这一结果的主要原因。鉴于该两对引物分别在线粒体不同Cytb基因和16S rDNA的保守区域设计,扩增出的片段存在互补性,故认为该两对引物均可作为鱼类群落结构eDNA研究的通用引物。
3.3 鱼类多样性的检测
与传统调查方法相比,eDNA技术具有灵敏度高、省时省力、对调查对象无损伤等优点,不要求调查者具有传统的生物识别及鉴定经验。此次利用eDNA技术共检出鱼类种类19种,为钱塘江的主要常见鱼类科属,其中以鲤科种类最多,占检出总种类数的84.21%以上。同时,检出的鱼类种类中不仅包括钱塘江富阳段原有的土著类群,如敞水带的鲫、似鳊等,沿岸带的光唇鱼等,底栖的鮠类等,还包括了主要的放养种类,如鲢、鳙等。因此,此次检出的鱼类无论从来源、生态类型还是分类地位,钱塘江都具有代表性。
在高通量测序比对上的种类中,大部分为传统捕捞方法可以采集到的种类,如鲫、鲤、鲢、鳙、似鳊等。此外,也检测到了常规调查方法未在钱塘江富阳段记录到的种类,如光唇鱼、拟腹吸鳅、拉氏鱥,然而这些种类多为钱塘江上游溪流性鱼类或下游河口型鱼类。分析原因可能是水体流动性所致。在流动水体中,生物释放的eDNA会随着水流迁移一段时间,且迁移距离与种类有关。相同实验条件下,长刺水蚤(Daphnialongispina)的迁移距离大于12.3 km,而白乳玉螺(Uniotumidus)的迁移距离则小于9.1 km[22],表明在流动水体中,在某处检测出目标种类的eDNA,其存在范围可至上游约10 km。本次研究的采样水域为钱塘江富阳段,属于富春江的一部分,而富春江是钱塘江的中游段,富春江电站为潮区界,以下均为感潮河段。富春江的这种水文特征更有利于水体中eDNA存在范围的上下游扩展。
同时,此次的检出种类中也存在一些未曾在钱塘江发现的鱼类,如花棘,究其原因可能与BLAST比对时设置的可信度参数(90%)等因素有关,需要进一步探究。
3.4 eDNA技术在钱塘江鱼类多样性保护中的应用
鱼类是水域生态系统中的重要组成部分,了解其资源状况,有利于保护和开发鱼类资源,有利于渔业增殖放流工作,也有利于维持生态系统的稳定。在浙江内陆渔业水域调查中,捕捞鱼类的渔具可分为刺网、掩网、抄网、敷网、张网、钓具、刺耙等7类,还有违禁的地笼网、电捕等。而在钱塘江富阳段,刺网、钓具、地笼网是使用最多的渔具。由于这种捕捞方法的限制,可能导致一些常规种类有时未采集到、濒危或稀有种类不能采集到的情况。采用eDNA技术作为一种补充手段,对钱塘江水域的渔业资源进行调查,弥补了单一采用传统调查方法的缺陷,能更加方便地检测目标物种是否存在,更加全面地了解水域的鱼类多样性和分布情况。在野外调查取样的工作量上,eDNA调查方法也比较简单快速。本文尝试采用Illumina MiSeq法来研究钱塘江富阳段鱼类种类组成,结果表明这种方法是可行的。
然而,采用eDNA调查方法时,水样eDNA的采集方法、提取方法、引物可靠性和稳定性都会影响最终的鱼类多样性判断。比如,影响每个种类检出序列数的因素包括群体生物量、eDNA来源、DNA序列与引物的同源度、随机抽样误差等方面。本次研究中发现测序获得的序列大多仍存在无法判别的情况,在后续研究中需要进一步通过改善eDNA的获取、筛选开发更有效的分子标记等方法用于分析和检测。
