雷蒙德棉第Ⅲ类过氧化物酶全基因组鉴定和表达分析
2019-10-14高正银孙文杰宋晓云左开井
高正银, 孙文杰, 宋晓云, 胡 轼, 左开井
上海交通大学农业与生物学院, 上海 200240
棉花纤维是重要的天然纺织材料。棉花纤维是由棉花胚珠种皮细胞发育而来的单细胞结构[1]。成熟的棉花纤维主要由纤维素(大于90%)组成,此外还有少量的非纤维素成分,例如:果胶、木聚糖、木质素和木质素类酚醛物质[2,3],其中,木质素的合成过程影响了成熟纤维的品质包括纤维的长度和强度等,其分子机理有待进一步的研究。
过氧化物酶是一个重要的酶家族,广泛存在于植物、动物和微生物中,催化氧化还原反应[4]。过氧化物酶分为三类:①第Ⅰ类过氧化物酶(Class Ⅲ Peroxidases,Class Ⅰ Prxs)为细胞内过氧化物酶,包含抗坏血酸过氧化物酶、细胞色素C过氧化物酶以及过氧化氢酶。抗坏血酸过氧化物酶存在于植物和酵母的叶绿体或细胞质中,可以控制过氧化氢分子的浓度。细胞色素C过氧化物酶能够与细胞色素C形成复合物,催化过氧化氢还原为水[5]。②第Ⅱ类过氧化物酶(Class Ⅱ Peroxidases,Class Ⅲ Prxs)包括木质素酶和其他细胞外真菌过氧化物酶[6]。③第Ⅲ类过氧化物酶(Class Ⅲ Peroxidases,Class Ⅲ Prxs,secreted plant peroxidases)是植物特有的一个巨大的过氧化物酶家族,存在于植物细胞外空间或液泡中[7]。该类蛋白的三维结构由大部分的α螺旋和小部分的β折叠组成,血红素分子结合于近C端结构域和N端结构域之间的α螺旋上(图1B)[8]。其蛋白序列存在高度保守的序列和不保守的可变序列,蛋白一级结构中有4个高度保守的结构域:血红素结合位点、活性位点、底物结合位点和2个保守的钙结合位点[9~12]。家族的所有成员共享一个血红素假体基团,以过氧化氢作为电子受体,催化一个多步骤氧化反应,与过氧化氢解毒、生长素分解代谢、木质素生物合成和应激反应有关。
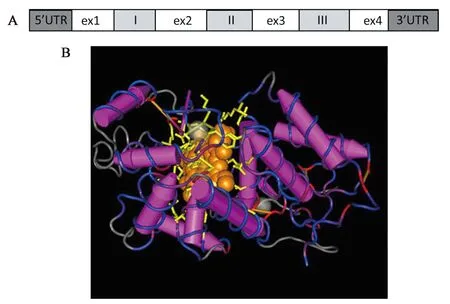
图1 第Ⅲ类过氧化物酶基因和三维蛋白结构示意图Fig.1 Gene and 3D protein structure of Class Ⅲ Peroxidases.A:第Ⅲ类过氧化物酶基因编码序列经典模式结构示意图;Ⅰ、Ⅱ和Ⅲ 为3个高度保守的内含子区域。B: 第Ⅲ类过氧化物酶基蛋白三维结构示意图;α螺旋和β折叠分别用蓝色和红色表示,血红素分子(粉色)结合于N端结构域和C端结构域之间的α螺旋上,钙原子用灰色球形表示。
第Ⅲ类过氧化物酶(Class Ⅲ Prxs)又称为分泌性植物过氧化物酶,广泛存在于植物各组织的细胞外空间或液泡中,参与过氧化氢解毒、生长素分解代谢、木质素生物合成和应激反应等生理过程[13], 然而参与这些生理过程的机理仍然未被解释。在拟南芥基因组中有73个第Ⅲ类过氧化物酶被鉴定[7, 14],大多数第Ⅲ类过氧化物酶基因广泛在植物各组织中表达,目前拟南芥中只有3个基因被证实在根部特异性表达[15]。第Ⅲ类过氧化物酶木质素是维管束植物细胞壁的主要成分,是一种复杂的芳香杂聚物,木质素的聚合是通过第Ⅲ类过氧化物酶或漆酶催化的氧化偶联过程,是一种新的单体与细胞壁中生长的聚合物(或低聚体)发生重复自由基偶联而进行的[16~18]。在烟草、拟南芥、白杨等植物中都有研究表明过氧化物酶参与催化木质素的合成过程,不同的过氧化物酶在特定的器官、细胞和发育阶段参与木质素合成[19~21]。第Ⅲ类过氧化物酶能够通过消耗或产生活性氧ROS参与多种抗逆过程,如重金属胁迫、病原体入侵、臭氧过多、温度胁迫、缺氧、磷酸盐缺乏、硫缺乏和缺钾等[22]。因此,过氧化物酶基因的表达依赖于多种不良的环境因素。研究表明,还有一些第Ⅲ类过氧化物酶通过氧化生长素以及产生·OH参与了植物生长过程。拟南芥中大多数第Ⅲ类过氧化物酶的编码序列具有经典模式结构:包含有4个外显子和3个完全保守的内含子,目前在拟南芥基因组中有73个第Ⅲ类过氧化物酶被鉴定[7,14],而只有3个基因被证实在根部特异性表达[15],研究发现,拟南芥AtPrx33和AtPrx34除了参与抗逆过程还参与了根的伸长[23]。在葡萄和西葫芦中也发现第Ⅲ类过氧化物酶能够调控IAA的含量进而影响细胞的伸长[24,25]。
在棉花植株生长的过程中,第Ⅲ类过氧化物酶起到重要的功能。例如,抵抗病原体攻击、耐盐、参与植物激素和生物碱的代谢[26~28]。此外,在棉花纤维的伸长发育过程中,有多种细胞活动需要第Ⅲ类过氧化物酶参与催化氧化还原反应,例如细胞壁化合物的聚合[4,26,29,30]。在棉花纤维发育过程中特异高表达的第Ⅲ类过氧化物酶可能参与调控棉花纤维的起始、伸长以及次生壁合成等过程,尤为值得关注。有研究表明GhPOX1在快速伸长的纤维细胞中大量表达,qRT-PCR分析发现该基因在伸长的纤维细胞中的表达量是胚珠、花、根、茎和叶片中的400倍以上,进一步实验也证明该基因确实参与调控棉花纤维的伸长过程[31]。
为了进一步系统分析第Ⅲ类过氧化物酶在纤维伸长过程中的功能,鉴定与纤维发育有关的过氧化物酶基因,本研究拟通过生物信息学手段和转录组数据分析对雷蒙德第Ⅲ类过氧化物酶家族基因进行系统分类和表达分析,鉴定可能参与调控棉花纤维的起始、伸长以及次生壁合成等过程的关键基因,为进一步探究第Ⅲ类过氧化物酶在棉花纤维发育过程中的功能提供参考依据。
1 材料方法
1.1 雷蒙德棉第Ⅲ类过氧化物酶基因确定和序列获取
在Cottongen(https://www.cottongen.org/find/genes)棉花基因组数据库,以peroxidase为关键词搜索Gossypiumraimondii(D5) genome JGI assembly v2.0 (annot v2.1)数据库,获得候选序列301条。进行序列比对、进化树构建,蛋白亚细胞定位预测(http://cello.life.nctu.edu.tw/)[32,33],以及保守域结构分析(http://www.ncbi.nlm.nih.gov/Struc-ture/cdd/wrpsb.cgi)[34],进一步对第Ⅲ类过氧化物酶候选基因进行确定。利用ExPASy (http://www.expasy.org/) 计算候选蛋白的等电点和分子量。
1.2 雷蒙德棉第Ⅲ类过氧化物酶基因结构分析以及进化树构建
利用 ClustalX 2.0进行多重序列比对[35],然后用MEGA 6.0 构建Neighbor-joining (NJ) 进化树[36]。用Gene Structure Display (http://gsds.cbi.pku.edu.cn/)[37],对候选蛋白的CDS和基因组序列进行分析,获得其基因结构。
1.3 雷蒙德棉第Ⅲ类过氧化物酶家族基因表达分析
从NCBI的SRA (https://www.ncbi.nlm.nih.gov)转录组数据库中筛选陆地棉TM-1不同组织(根、茎、叶和不同发育时期纤维0~25DPA)转录组数据并下载SRP044705。对转录组数据进行分析,具体如下:①数据解压:将数据从.sra格式转化为.fastq格式;②数据剪切:剪切数据中低质量部分,数据会压缩;③建立棉花参考基因组的bowtie2的索引文件;④利用TopHat2 将RNA-Seq 数据进行快速剪接映射到棉花基因组上;⑤利用HTSeq-count计算比对数;⑥计算所有基因组各个位点的rpkm值[38]。从所有rpkm值中提取候选基因的对应值,绘制heatmap进行分析。
1.4 第Ⅲ类过氧化物酶家族基因qRT-PCR分析
本研究所用棉花材料陆地棉品种冀棉14。引用Hasenfratz挂牌法,取开花期间不同时期(0、5 DPA、10 DPA、20 DPA、25 DPA) 的纤维材料迅速放于液氮中冷冻存储。使用多糖多酚植物总RNA提取试剂盒(购自天根公司)提取棉花总RNA,将所得RNA于-70℃ 条件下冻存备用。进一步反转录获得棉花纤维组织cDNA。
根据要定量分析的基因序列设计基因特异性引物(表1)。利用ubiquitin基因作为内参,以△CT法对棉花纤维不同发育时期基因的表达水平进行qRT-PCR分析。
2 结果与分析
2.1 雷蒙德棉第Ⅲ类过氧化物酶家族蛋白的确定和聚类分析
在Cottongen(https://www.cottongen.org/find/genes)棉花基因组数据库,以“peroxidase”为关键词搜索Gossypiumraimondii(D5) genome JGI assembly v2.0 (annot v2.1)数据库,获得了301个棉花过氧化物酶家族候选蛋白序列,下载并进行分析。利用NCBI conserved domain 工具对下载的蛋白序列进行比对分析,选择具有第Ⅲ类过氧化物酶保守结构、血红素结合位点、活性位点、底物结合位点和2个保守的钙结合位点的蛋白,排除其他201个第Ⅰ类、第Ⅱ类过氧化物酶家族的蛋白,最终确认雷蒙德棉花基因组中有90个第Ⅲ类过氧化物酶蛋白。所有的这90个蛋白具有第Ⅲ类过氧化物酶的典型特征,包含4个保守结构域。用Neighbour-joining法对90个第Ⅲ类过氧化物酶蛋白进行了进化树分析,根据聚类分析的结果将第Ⅲ类过氧化物酶蛋白分为8个亚类,每个亚类用不同的颜色加以区分如图2,其中第Ⅳ亚类包含的蛋白数量最多。对90个蛋白进行亚细胞定位预测,发现这些蛋白分别定位于细胞质、细胞膜、胞外和周质空间等(表2)。

表1 本研究所用的引物序列Table 1 Primers used in this investigation.
2.2 雷蒙德棉第Ⅲ类过氧化物酶家族蛋白的结构和分类特征
本文对雷蒙德棉第Ⅲ类过氧化物酶家族基因的结构分类和进化关系进行了分析。该家族总共有90个成员,分布于棉花的13条染色体上。参考拟南芥和水稻第Ⅲ类过氧化物酶基因结构的分类方法,对雷蒙德棉第Ⅲ类过氧化物酶家族基因的结构进行分析,结果显示该家族基因存在高度保守的序列和不保守的可变序列。其序列保守性主要表现在外显子序列的高度保守和内含子位点高度的保守(图3)。
此外,在经典的4个外显子-3个内含子模型的基础上,内含子的数量发生了缺失和增加。图4为雷蒙德棉第Ⅲ类过氧化物酶基因结构分类示意图,黄色区域表示外显子,对应的数字表示外显子序列长度范围,黑色线段表示内含子插入位点。在90个成员中有55个基因(占整个家族成员数量的61%)具有过氧化物酶基因典型的4个外显子-3个内含子的结构(图4H,I),这种结构也广泛存在于拟南芥和水稻中[7,14],还有35个基因在4个外显子-3个内含子的模型上缺失或增加1~3个内含子(图4A~G,J,K)。
表2 雷蒙德棉第Ⅲ类过氧化物酶蛋白八个亚群保守域和亚细胞定位分析结果Table 2 The conserved domain and protein subcellular localization analysis of eight subgroup of Gossypium raimondii Class Ⅲ peroxidases.
续表 Continued亚群基因 ID基因名称CDD Accession蛋白质名称可能的定位IVGorai.008G007700GrPrx45cd00693secretory_peroxidase周质Gorai.008G007500GrPrx43cd00693secretory_peroxidase细胞质Gorai.008G007600GrPrx44cd00693secretory_peroxidase细胞质Gorai.006G074900GrPrx28cd00693secretory_peroxidase细胞质Gorai.011G184600GrPrx73cd00693secretory_peroxidase周质Gorai.011G184500GrPrx72cd00693secretory_peroxidase周质Gorai.008G172200GrPrx49cd00693secretory_peroxidase周质Gorai.005G150500GrPrx26cd00693secretory_peroxidase周质Gorai.005G150600GrPrx27cd00693secretory_peroxidase周质Gorai.013G099400GrPrx85cd00693secretory_peroxidase外膜Gorai.013G099600GrPrx87cd00693secretory_peroxidase外膜Gorai.013G099500GrPrx86cd00693secretory_peroxidase外膜Gorai.009G017700GrPrx55cd00693secretory_peroxidase周质Gorai.002G208800GrPrx9cd00693secretory_peroxidase细胞质Gorai.012G046400GrPrx79cd00693secretory_peroxidase周质Gorai.008G264300GrPrx52cd00693secretory_peroxidase周质Gorai.004G049000GrPrx13cd00693secretory_peroxidase周质Gorai.004G049200GrPrx15cd00693secretory_peroxidase周质Gorai.004G049100GrPrx14cd00693secretory_peroxidase周质Gorai.009G064700GrPrx56cd00693secretory_peroxidase周质Gorai.001G039200GrPrx2cd00693secretory_peroxidase外膜Gorai.008G281600GrPrx53cd00693secretory_peroxidase周质Gorai.003G006600GrPrx10cd00693secretory_peroxidase周质Gorai.010G001900GrPrx65cd00693secretory_peroxidase周质Gorai.009G192200GrPrx60cd00693secretory_peroxidase周质VGorai.010G131800GrPrx67cd00693secretory_peroxidase周质Gorai.005G077600GrPrx23cd00693secretory_peroxidase外膜Gorai.008G090200GrPrx48cd00693secretory_peroxidase周质Gorai.007G234600GrPrx40cd00693secretory_peroxidase细胞质VIGorai.007G065200GrPrx37cd00693secretory_peroxidase细胞质Gorai.007G049900GrPrx36cd00693secretory_peroxidase细胞质Gorai.007G070800GrPrx38cd00693secretory_peroxidase细胞质Gorai.007G199700GrPrx39cd00693secretory_peroxidase细胞质Gorai.009G198700GrPrx61cd00693secretory_peroxidase周质Gorai.008G228500GrPrx51cd00693secretory_peroxidase周质Gorai.005G068200GrPrx22cd00693secretory_peroxidase外膜Gorai.008G174200GrPrx50cd00693secretory_peroxidase周质Gorai.005G149000GrPrx25cd00693secretory_peroxidase周质Gorai.012G150400GrPrx83cd00693secretory_peroxidase外膜Gorai.008G073800GrPrx46cd00693secretory_peroxidase周质Gorai.010G127400GrPrx66cd00693secretory_peroxidase周质Gorai.004G093200GrPrx17cd00693secretory_peroxidase周质Gorai.006G143400GrPrx29cd00693secretory_peroxidase细胞质Gorai.007G238800GrPrx41cd00693secretory_peroxidase细胞质Gorai.004G265900GrPrx21cd00693secretory_peroxidase周质Gorai.004G093000GrPrx16cd00693secretory_peroxidase周质Gorai.002G168400GrPrx8cd00693secretory_peroxidase外膜
续表 Continued亚群基因 ID基因名称CDD Accession蛋白质名称可能的定位VIIGorai.N001200GrPrx88cd00693secretory_peroxidase外膜Gorai.006G234200GrPrx35cd00693secretory_peroxidase细胞质Gorai.006G234100GrPrx34cd00693secretory_peroxidase细胞质Gorai.N001400GrPrx90cd00693secretory_peroxidase外膜Gorai.N001300GrPrx89cd00693secretory_peroxidase外膜Gorai.006G233900GrPrx33cd00693secretory_peroxidase细胞质Gorai.006G233800GrPrx32cd00693secretory_peroxidase细胞质Gorai.011G129800GrPrx70cd00693secretory_peroxidase周质ⅧGorai.011G211300GrPrx76cd00693secretory_peroxidase周质Gorai.011G211100GrPrx75cd00693secretory_peroxidase周质Gorai.011G211400GrPrx77cd00693secretory_peroxidase周质Gorai.011G129900GrPrx71cd00693secretory_peroxidase周质Gorai.004G234900GrPrx19cd00693secretory_peroxidase周质Gorai.009G361800GrPrx63cd00693secretory_peroxidase周质Gorai.003G151700GrPrx12cd00693secretory_peroxidase周质Gorai.013G005800GrPrx84cd00693secretory_peroxidase外膜Gorai.012G016200GrPrx78cd00693secretory_peroxidase周质Gorai.009G295200GrPrx62cd00693secretory_peroxidase周质
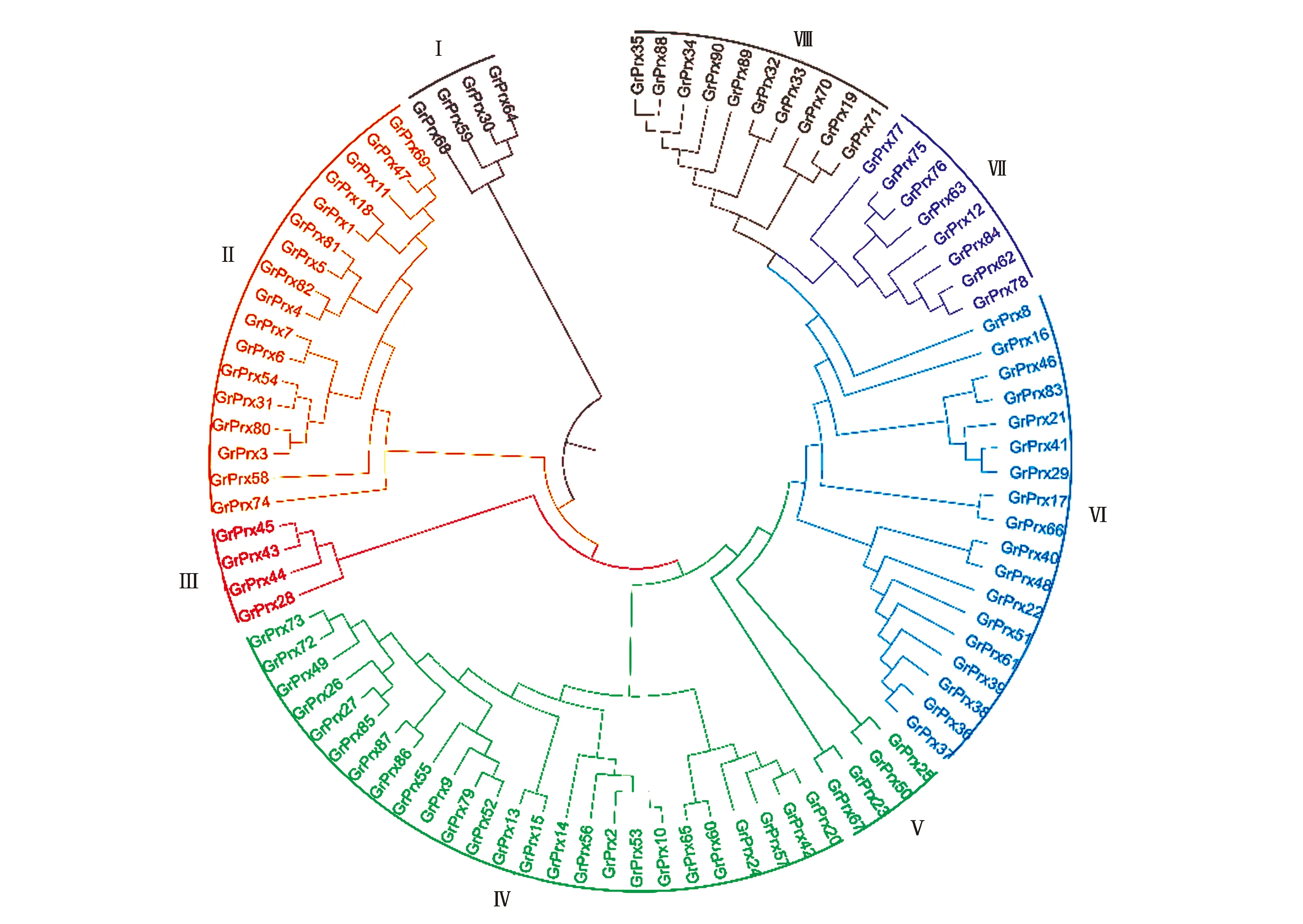
图2 雷蒙德棉第Ⅲ类过氧化物酶蛋白聚类分析Fig.2 Phylogenetic analysis of Gossypium raimondii Class Ⅲ peroxidases.
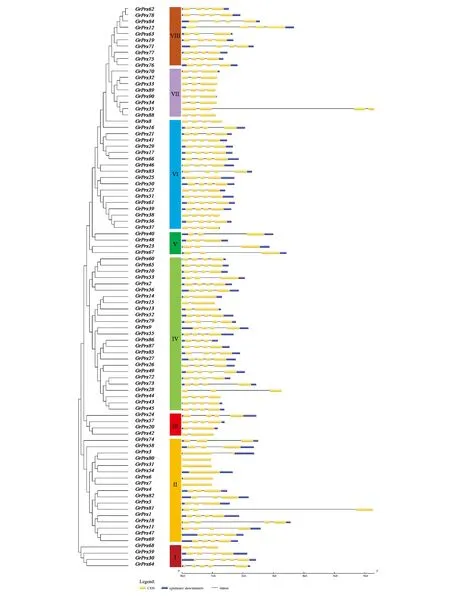
图3 雷蒙德棉第Ⅲ类过氧化物酶基因结构分析图Fig.3 Gene structure analysis of Gossypium raimondii Class Ⅲ peroxidases.注:每个亚家族用不同的颜色加以区分,蓝色区域表示5’和3’非编码区,黄色区域表示外显子,黑色线段表示内含子。
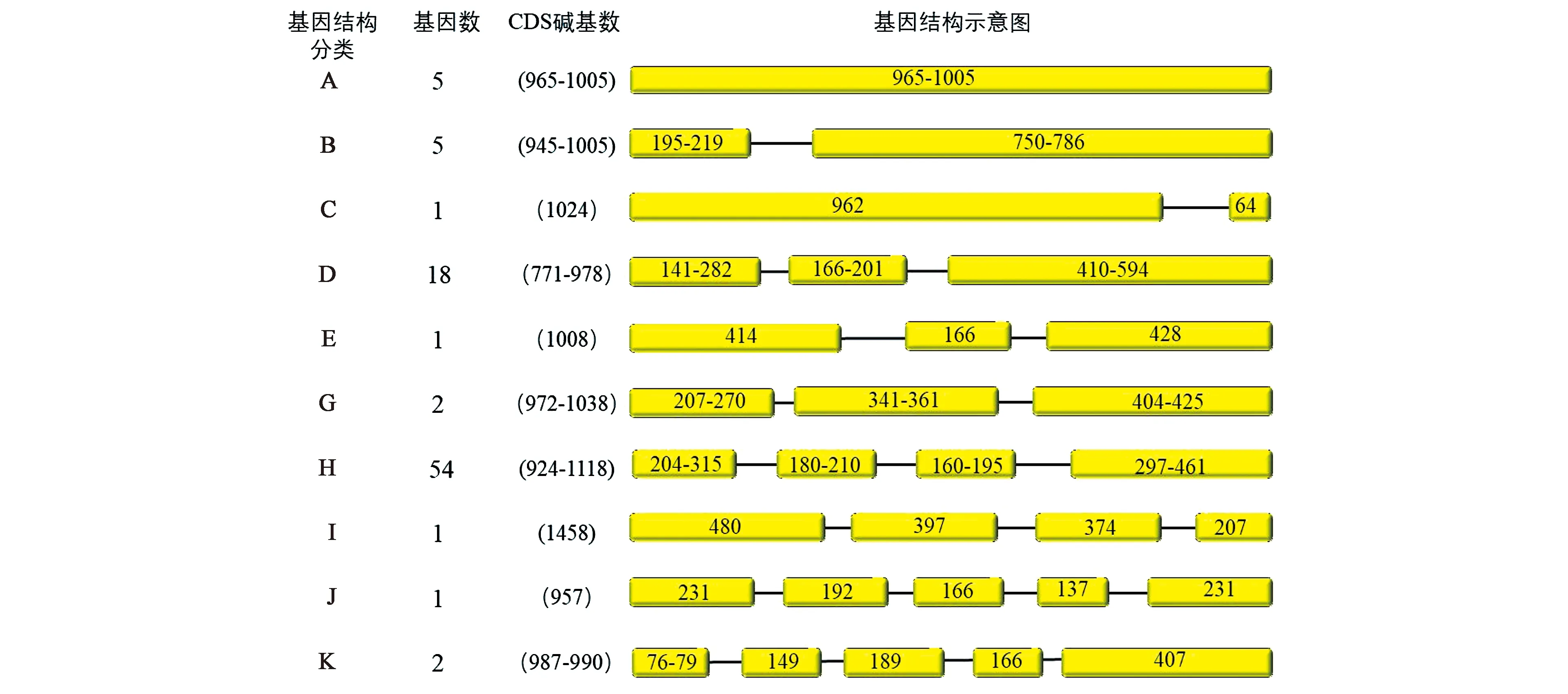
图4 雷蒙德棉第Ⅲ类过氧化物酶基因结构分类示意图Fig.4 Structure classification of Gossypium raimondii Class Ⅲ peroxidase genes.
2.3 第Ⅲ类过氧化物酶家族基因在棉花纤维发育过程中的表达模式分析
目前生产应用的棉花均为AADD的四倍体棉花,为了分析第Ⅲ类过氧化物酶基因家族在纤维起始发育及伸长发育过程中的可能功能,因此本研究利用陆地棉TM-1转录组数据对所有的第Ⅲ类过氧化物酶家族基因在纤维发育过程中的表达模式进行了分析。结果显示第Ⅲ类过氧化物酶基因在纤维发育的不同阶段表达的基因有所不同,具有3种不同的表达模式(图5),而它们的表达趋势变化暗示它们参与不同的生化途径,行使不同的功能。
进一步对3种表达模式的代表性基因进行组织时空表达模式分析,结果如图6所示,表达模式一:该类过氧化物酶基因在棉纤维发育的起始阶段表达量上调,如GrPrx21、GrPrx36、GrPrx57和GrPrx59等。这类基因的表达量在纤维起始阶段0 DPA达到最高,进入纤维伸长期后立即快速下降。表达模式二:该类过氧化物酶基因在棉纤维发育的伸长阶段表达量持续显著上升,然后随着进入次生壁合成期后表达量显著降低,如GrPrx3、GrPrx32、GrPrx56和GrPrx61等基因,暗示其可能在棉花纤维的快速伸长期起到重要的作用。表达模式三:过氧化物酶基因在纤维快速伸长期结束进入次生壁合成期后表达量迅速上升,如GrPrx11、GrPrx16、GrPrx66和GrPrx77等,这类基因可能在棉花纤维次生壁合成过程中起到重要的作用。
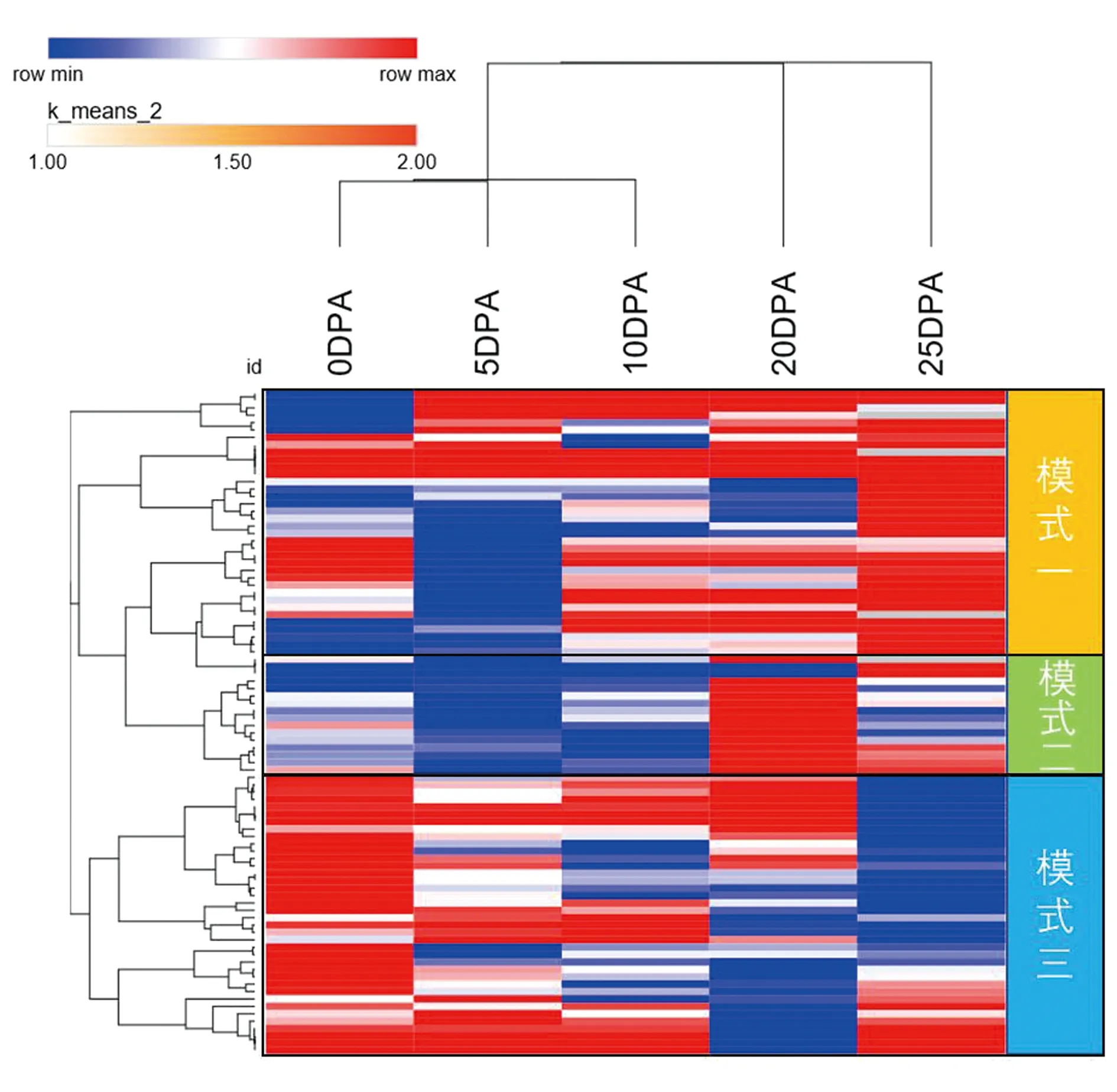
图5 90个雷蒙德棉第Ⅲ类过氧化物酶家族基因在纤维(0 DPA、5 DPA、10 DPA、20 DPA、25DPA)中的表达模式Fig.5 The expression pattern of 90 Gossypium raimondii Class Ⅲ peroxidase genes in fiber(0 DPA、5 DPA、10 DPA、20 DPA、25DPA).注:红色表示表达量较高,蓝色表示表达量较低。
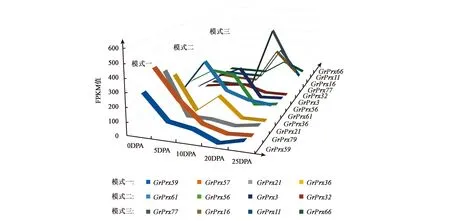
图6 纤维发育过程中三种不同表达模式的雷蒙德棉第Ⅲ类过氧化物酶基因表达示例Fig.6 The illustration of three different expression pattern of Gossypium raimondii Class Ⅲ peroxidases genes during cotton fiber development.
为了进一步验证转录组数据分析结果的可靠性,选取具有代表性的三种不同表达模式的基因分别进行qRT-PCR分析。转录组分析结果表明,GrPrx21、GrPrx3和GrPrx11分别属于三种不同表达模式。如图7所示,对三个不同表达模式的基因在纤维发育的0 DPA、5 DPA、10 DPA、20 DPA和25 DPA时期的基因表达水平进行qRT-PCR分析,发现:GrPrx21在纤维发育0 DPA时基因的表达量最高,进入纤维伸长期后表达水平持续降低;GrPrx3基因的表达水平从0 DPA到25 DPA呈现先上升后下降的模式,在10 DPA表达水平达到最高;GrPrx11在0 DPA、5 DPA、10DPA基因的表达量很低,直到进入次生壁合成阶段(20~25 DPA),其表达水平急速上升;上述qRT-PCR结果均与转录组分析结果相一致。
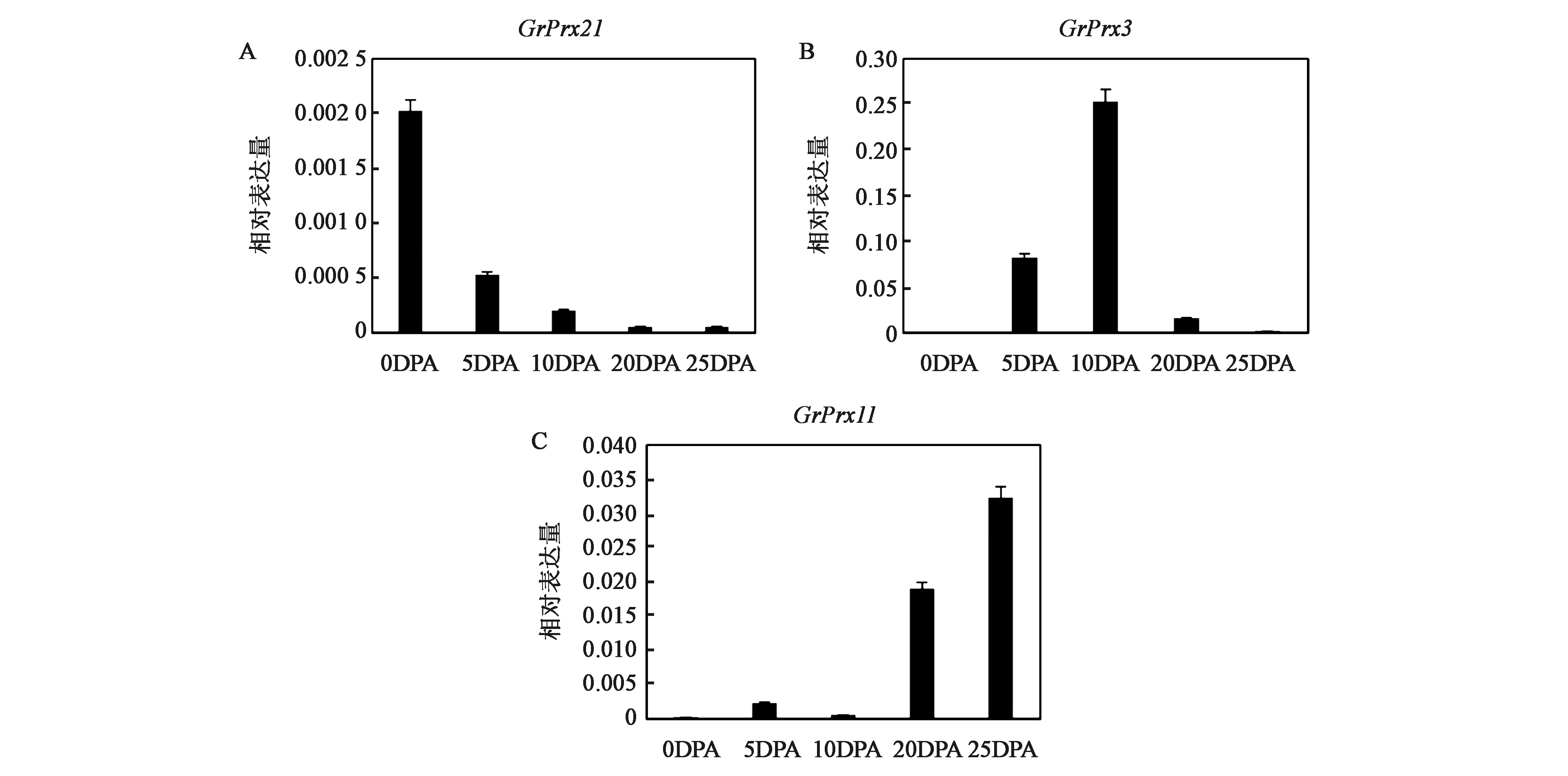
图7 纤维发育过程中三种不同表达模式的第Ⅲ类过氧化物酶基因定量分析Fig.7 qRT-PCR analysis of three different expression pattern Gossypium raimondii Class Ⅲ peroxidases genes during cotton fiber development.A.GrPrx21基因; B.GrPrx3基因; C:GrPrx11基因。
3 讨论
第Ⅲ类过氧化物酶作为重要的催化植物体内各种氧化还原反应的保护酶类家族参与调控植株生长、发育和抗逆等多种生化途径。相关研究表明,过氧化物酶在棉花纤维的伸长过程中起到重要的调控作用,可能参与棉花雄性生殖过程以及响应抗病等过程。例如:GhPOX1在快速伸长的纤维细胞中大量表达,参与了棉花纤维的发育过程,qRT-PCR分析发现该基因在伸长的纤维细胞中的表达量是胚珠、花、根、茎和叶片中的400倍以上[31]。Ghpod(cDNA GenBank Accession Number: EU196676)在棉花的花器官中特异表达,可能参与棉花雄性生殖过程[39]。在大丽轮枝菌侵染棉花植株后,一些第Ⅲ类过氧化物酶基因响应并且表达量发生了变化[40]。
拟南芥、水稻、玉米、杨树、梨和二穗短柄草全基因组的第Ⅲ类过氧化物酶已经被鉴定[7,14,41~44]。在棉花中关于第Ⅲ类过氧化物酶的研究比较少,因此系统全面的研究棉花中的第Ⅲ类过氧化物酶具有重要的意义。本文通过对已经注释的雷蒙德棉基因组进行搜索,鉴定得到了90个雷蒙德棉第Ⅲ类过氧化物酶基因。对所有的第Ⅲ类过氧化物酶蛋白及其CDS序列进行同源比对、构建进化树,整合已有的拟南芥过氧化物酶功能研究结果,将90个过氧化物酶蛋白分为8个亚家族。进化树分析结果表明第Ⅲ类过氧化物酶蛋白序列是相对保守的,暗示同一亚家族的基因可能在植株生长、发育和抗逆过程中承担相同或相似的功能。进一步的基因结构分析显示雷蒙德棉第Ⅲ类过氧化物酶基因结构、位点都相对保守,雷蒙德棉大多数(61%)的第Ⅲ类过氧化物酶保留了经典的第Ⅲ类过氧化物酶的基因结构。此外,蒙德棉第Ⅲ类过氧化物酶基因的结构存在一定的多样性,90个雷蒙德棉第Ⅲ类过氧化物酶成员基因的内含子数目有0~4个不等,大多数的基因包含2个及以上的内含子,说明雷蒙德棉第Ⅲ类过氧化物酶基因家族存在一定的多样性。雷蒙德棉第Ⅲ类过氧化物酶在不同亚家族之间也存在这些特征的差异表现,说明雷蒙德棉第Ⅲ类过氧化物酶亚家族成员在功能上是多样化的。在每个亚家族中存在其特定的保守序列,各成员在此保守序列的基础上插入或缺失内含子增加了亚家族成员的序列和功能多样性。
基因的表达模式可以为有关基因的功能研究提供重要的线索。本研究通过结合转录组数据和qRT-PCR分析来研究第Ⅲ类过氧化物酶基因的表达模式。分析发现,许多第Ⅲ类过氧化物酶基因在不同组织和纤维发育的过程中呈现特异性的沉默或特异性高表达,暗示这些基因在棉花植株和纤维发育过程中承担相应的功能。进一步对在纤维发育过程中棉花第Ⅲ类过氧化物酶的表达水平进行分析,发现了3种不同表达模式的基因,在纤维发育过程中不同阶段有特异的基因表达,表明在纤维发育的起始期、伸长期以及次生壁合成阶段分别有不同的第Ⅲ类过氧化物酶行使相应的功能,为进一步探究第Ⅲ类过氧化物酶在纤维发育过程中的功能提供依据。
本研究对雷蒙德棉第Ⅲ类过氧化物酶家族成员的进化起源、系统发育关系和外显子/内含子结构进行了分析,并对纤维发育不同阶段第Ⅲ类过氧化物酶基因的时空表达模式进行了检测。研究结果为进一步了解第Ⅲ类过氧化物酶基因家族的进化和每个第Ⅲ类过氧化物酶基因在纤维发育过程中的潜在功能提供了一个有用的参考。由于迄今为止,棉花中只有少数第Ⅲ类过氧化物酶基因明确了功能特征,本研究结果有助于进一步研究其生物学功能,并应用于纤维品质改良和生产。
