一种分布式爬虫系统的设计与应用
2019-10-11陈远平
周 逸,李 新,陈远平
1.中国科学院计算机网络信息中心,北京 100190
2.中国科学院大学, 北京 100049
目前,要了解一个学科的发展情况,主要通过查找文献,阅读专家撰写的综述或直接看大量有关这一学科的文献,从科学家个人角度看待这一学科,缺乏定量分析,有一定的局限性。[1]而且随着时间推移、学术发展、科技进步,文献数量呈爆发式增长,不可能做到逐一阅读。因此,要了解一个学科的发展态势单纯凭借人工力量有一定的难度,而文献计量学从定量分析的角度很好地解决了这一问题。
1969年,“文献计量学”这一术语的出现标志着文献计量学的正式诞生[2]。它是一门定量性、实用性很强的学科,集数学、统计学、文献学为一体,注重量化的综合性知识体系,无论是理论研究还是实际应用都必须要有一定规模的资料数据支持。因此 ,我们必须建立系统化 、规范化的资料来源工具和原始资料的获取渠道。[3]
Web of Science 是是全球高质量核心期刊论文及全球会议文献的检索工具,是获取高质量文献的权威渠道。本文正是基于此,设计并实现了一种基于 scrapy-redis 的分布式爬虫系统用于自动获取web of Science 中文献相关信息用以支持文献计量学的分析,同时设计了 Web 服务以便于向用户展示分析结果。
1 学科发展态势分析研究现状
学科发展态势分析已被应用于多学科领域的分析。例如刘昊[4]等对信息学学科发展态势做的文献计量分析,揭示了信息学发展经历了封闭萌芽期、交叉发展期、开放增长期,且在医学、生物学、计算机科学这些具有较大的数据体量或信息技术优势的领域发展快,图情领域信息学相关研究偏少的特点。陆颖、杨志萍、王春明[5]等利用 Web of Science 数据库、ESI数据库开展文献计量分析,获得我国天文台 SCI 论文产出发展态势和国际竞争力,寻找与国外类似研究机构相比的优势与差距。
这些研究的数据大多来源于美国汤森路透公司开发的 Web of Science 数据库,是全球高质量核心期刊论文及全球会议文献的检索工具。对 Web of Science数据进行解析、分析的工具也很成熟,比如数据清洗方面的工具 Bibexcel,Hiscite,ISI.exe;可视化方面工具有 VOSviewer、Citespace、SPSS 等 3 种分析可视化软件[4]。
以往的这些研究,一般需要手动先将文献相关信息从 web of science 下载到本地,然后通过上述数据清洗工具进行预处理,再将预处理结果输入可视化软件进行展示。而且以往对于交叉学科的研究分析,其中用到的期刊名称-所属学科映射表也是依靠人工查询获取。这一系列步骤都是手动的,较为繁琐。所以本文提出一种分布式自动化的爬虫系统,用户只需要输入初始待爬取页面的 URL (Uniform Resource Locator),爬虫会自动爬取所有文献并解析为结构化数据存入 MongoDB 数据库,同时利用 Django 搭建的web 服务读取 MongoDB 中的数据进行可视化展示。
系统总体分为两部分,第一部分是负责完成数据预处理的基于 scrapy-redis 的分布式爬虫,爬虫解析 web of science 页面中的论文条目,存为 MongoDB中的结构化数据,完成数据积累。第二部分是利用Django 搭建的负责可视化展示的 Web 服务。
2 基于 scrapy-redis 的分布式爬虫设计思路及实现
2.1 scrapy-redis框架介绍
scrapy 是一款基于 Python 开发的开源 web 爬虫框架,可快速抓取 Web 站点并提取页面中的结构化数据,具有高度的扩展性[6]。redis 是一个高性能的可基于内存亦可持久化的日志型 key-value 数据库。
scrapy-redis 是一款开源的 python 库,是以scrapy 为基础针对分布式进行的改进。把 scrapy 爬虫框架与 redis 数据库相结合,将 redis 作为缓存,利用redis 进行 URL 去重,实现了爬虫的分布式。
scrapy-reids 库中的主要组件及作用如下[7,8]{李代祎,2017 #16;李代祎,2017 #16}:
· Connections.py:负责根据 settings 中的相关配置建立 redis connection 的实例。涉及到 redis 存取的操作都要用到此模块,它被 dupefilter.py 和 scheduler.py 调用。
· dupefilter.py:负责利用 redis 的 set 结构执行request 的去重。是对 scarpy 库本身去重方法的重写。scrapy 库中的去重只需读取本地内存中的 request 队列或者持久化的 request 队列就能判断这次要发出的request URL 是否已经请求过或者正在调度。而分布式爬虫,需要各个主机上爬虫的 scheduler 调度器都连接同一个数据库的同一个 request 池来判断一次请求是否重复,所以在scrapy-redis 框架中,redis 实现了记录 request 池的功能,记录所有访问过的 URL 进行去重。
· queue.py:负责实现队列,当 request 不重复时,将其存入到队列中,调度时将其弹出。
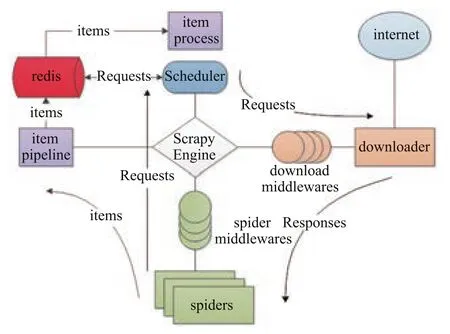
图1 scrapy-redis原理图Fig.1 scrapy-redis principle
· scheduler.py:是对 scrapy 自带的 scheduler 的替代,利用此模块实现调度器以支持爬虫的分布式调度,其利用的数据结构正是 queue.py 中实现的队列。有三种可供选择的参数:先进先出的 SpiderQueue,可以设置优先级的 SpiderPriorityQueue,先进后出的SpiderStack,本系统选择先进先出的 SpiderQueue 实现队列。
· pipelines.py:将处理过的 item 持久化到 redis中,以便后期进行数据分析。
· spider.py:从 redis 中读取待爬取的 URL,然后执行爬取和解析网页。
2.2 分布式爬虫系统设计方案
用户需要给爬虫系统输入一个 URL 以启动爬取。本文选取的数据样本设定如下:在Web of Science 上选择数据库为“Web of Science 核心合集”,基本检索中按主题“data mining”检索,时间跨度选择“自定义年份范围”,选择 2008年–2018年内的文献数据,机构扩展选择“CHINESE ACADEMY OF SCIENCES”,这些限定条件设置完毕后即可获得待爬取的初始 URL。数据样本包含了中科院在过去10年内在data mining 领域发表的 Web of Science 核心合集论文。
分布式爬虫分为 master 端和 slave 端,整体架构图如图2。
用户向 master 端的爬虫输入初始 URL,master的爬只虫负责翻页,将每页的 URL 存入 redis 数据库中的 wos_slave_spider:slave_urls KEY 中。各个 slave端的爬虫从 wos_slave_spider:slave_urls 中读取 URL并解析为结构化数据,持久化到 MongoDB 中。
MongoDB 是一个基于分布式文件存储的数据库,是一个介于关系数据库和非关系数据库之间的产品,是非关系数据库当中功能最丰富,最像关系数据库的一款产品。MongoDB 的优点在于不必提前定义 schema,是一款 schemaless 的数据库,格式灵活,不用为了格式不一样的信息专门设计统一的格式,极大的减少开发工作。所以,本系统选择 MongoDB 存储文献相关信息。
由于上文提到的 redis 数据库是一款基于内存的高读写速度的 key-value 型数据库,所以选择redis存储待爬取的 URL 和期刊名称-所属学科映射关系表。
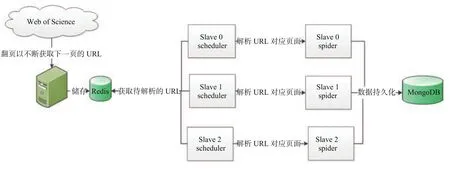
图2 分布式爬虫架构图Fig.2 Distributed crawlers Architecture
2.2.1 master端爬虫设计与实现
首先配置好 Master 端环境,见表1。
web of science 每页有 10 篇论文,如果单机爬虫进行爬取,需对一个页面进行解析,解析完毕后才能提取下一页的 URL,递归调用上述过程。所以单机爬虫是串行化的:一页一页地爬取,爬取并解析完一页才能翻下一页。而分布式爬虫中,master 端只负责翻页,将每页的 UR L存储在redis 数据库中的 wos_master_spider:slave_URLs 的 key 里,可根据任务扩展slave 端的爬虫数量,大大提高了扩展性、并发星 ,缩短了运行时间。
2.2.2 slave 端爬虫设计与实现
首先配置好 slave 端开发环境:Ubuntu+anaconda3.7+selenium
slave 端从 wos_slave_spider:slave_urls 取 URL(见图3),访问并解析之,将生成的 item 发送到itempipeline,通过 itempipeline 将数据持久化到master 服务器的 MongoDB 中。
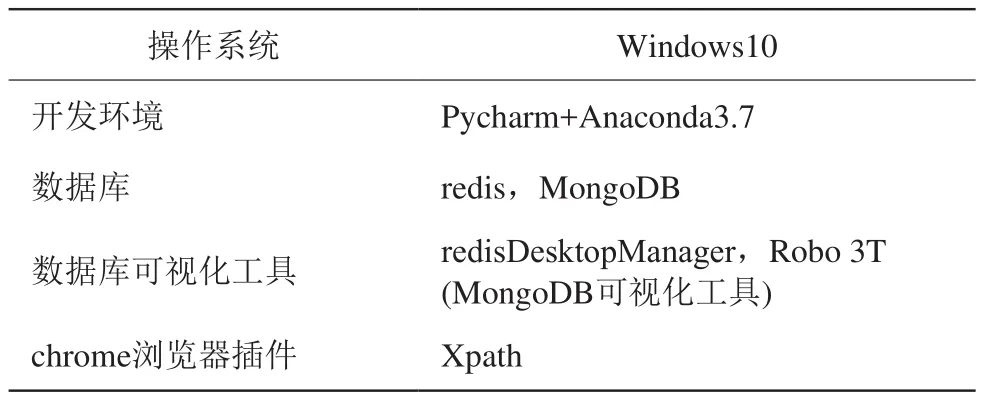
表1 master 服务器环境配置Table 1 Configuration of master server
在slave 端的 items.py 定义解析网页所需获得的字段,包括论文序列号、论文名称、作者名单、期刊名称、出版时间、论文链接、学科分布、参考文献列表(见表2)。因为作者名单、参考文献所属期刊列表两项不知其具体数目,故以列表形式存储。
图3 wos_slave_spider:slave_urls存储urlFig.3 wos_slave_spider:slave_urls stores url

表2 document字段定义及含义Table 2 Document field definition and meaning
MongoDB 中定义名为 wos 的 database,在其中定义名为 wos_paper 的 collection 用于存放文献相关信息,一篇文献相关信息存为一个 document (见图5)。
利用 xpath 浏览器插件帮助提取页面上对应字段的 xpath 表达式,比如提取文献序列号的关键代码如下,其他字段xpath表达式同理可得。

代码中的 yield paper_item 代表把数据持久化到MongoDB 中,这需要在爬虫工程的 pipelines.py 文件中做如下设置:

同时需要在工程的 settings.py 里设置 MongoDB相关参数并启用 MongoPipeline。配置完毕后,运行项目时我们可以看到一篇文献在MongoDB 中的存储形式如图4。
学科分布字段有助于展开基于引文分析的研究,该字段是以 python 字典形式存储的,key 为学科名称,value 为属于该学科的参考文献数量,形式如:{学科 1:15,学科 2:7,学科 3:4}。该字段可用于挖掘一篇文献所属的交叉学科情况。
该字段由参考文献列表字段计算得出,这就需要维护一个保存期刊名称--所属学科类别映射关系的哈希表(见图5),这可以利用 redis 的hash结构实现。在之前的研究中获取期刊名称-所属学科映射关系的方法是静态的,例如韩正琪等将从 JCR 数据库中获得的期刊全称-学科类别对照表与 ESI Journal List 的期刊全称-缩写对照表进行关联,得到期刊全称-期刊缩写-所属学科类别对照表[9]。这种方法需要提前准备好映射关系表,且对于非在校学生、非科研人员而言,没有途径进入 JCR 数据库,给研究造成阻碍。为解决这些问题,本系统提出一种动态获取期刊名称 (全称或缩写均可)-所属学科类别对照表的方法,算法思路如下:
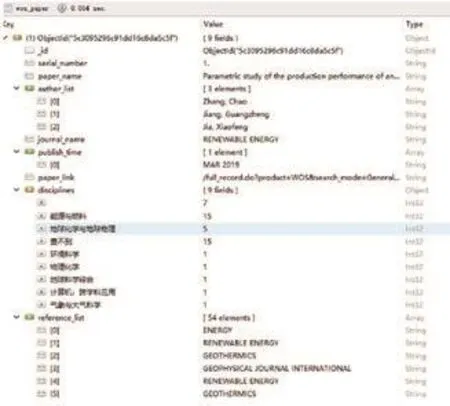
图4 文献在MongoDB 中的存储形式Fig.4 Storage format of literature in MongoDB
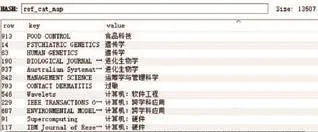
图5 期刊名称—所属学科类别映射Fig.5 Journal name-discipline catagory mapping

算法1 求参一篇文献的参考文献的学科分布输入:所有参考文献所属期刊列表ref_journal_list输出:以python字典形式存储的学科分布distuibiton 1 function get_Distributin(ref_journal_list):2 用selenium库模拟打开第三方查询期刊分区网站“letpub”;3 初始化期刊-所属学科映射表ref_cat_map 4 distribution←{}5 for j in ref_journal_list:6 if j_name in ref_journal_list.keys:7 categoryy←ref_cat_map[j_name]8 else:9 category←在letpub中输入期刊名称提取分区10 end if 11 if category in distribution:12 distribution[category]++13 else:14 distribution[category]←1 15 end if 16 end for 17 return distribution 18 end function
selenium 库是 python 的一款开源的自动化 web应用程序测试工具,引入 selenium 库来实现模拟输入期刊名称,输入回车等操作。本系统选用火狐浏览器配合 geckodriver 驱动实现。
leptub 是一个第三方的期刊分区查询系统,在该网站中输入期刊全称或缩写,均可得到该期刊所属的大类学科和小类学科,本系统记录小类学科。
2.2.3 slave 端配置优化
(1) 设置 USER-AGENT,减少被识别为爬虫的风险
在middlewares 设置 USER_AGENT,模拟request headers。设定 USER_AGENT 列表,每次发起request 请求时随机在列表中选择一个 user_agent 作为请求的 headers 参数。
(2) 防止出现爬虫丢失网页现象,保障数据完整性
在实际爬取过程中,个别情况会发生 TimeoutError。在settings.py 中配置 LOG_LEVEL = "DEBUG",将控制台信息存入日志,LOG_FILE 配置为 LOG 文件路径。可以在scrapy日志中查看错误信息提示为[scrapy.downloadermiddlewares.retry]DEBUG: Gave up retrying
为了解决此问题,在settings.py 里做如下设置:设置 RETRY_ENABLE=True 开启下载重试功能,增加重试次数,本系统将 RETRY_TIMES 设为10。DOWNLOAD_TIMEOUT 参数代表下载器在超时前等待的时间量,默认为 180 秒,增长等待时间有助于等待网速恢复正常,本系统设置为 480 秒。CONCURRENT_REQUESTS 参数规定了最大并发REQUEST 数量,默认为 16,本系统设置为8。尽管如此,还是可能由于短暂性的网速过慢造成丢失 URL现象,所以本系统重写了 RETRY 中间件作为最后保障。当出现 TimeoutError 时将此时访问的 URL 再放入 redis 中的 wos_slave_spider:slave_urls 队列中的末尾,以便后续再次被爬虫请求到。关键代码如下,其中r为 redis 连接的对象。

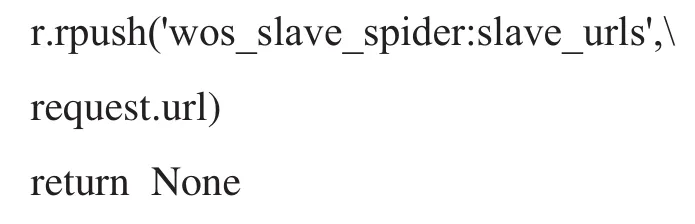
3 Django Web 可视化服务
当 slave 爬虫将数据存储到 MongoDB 完成后,就已经完成数据积累了。为了让用户便捷直观地感受到学科发展态势,利用 Django Web MVC 框架搭建网站对数据进行分析、展示。Django 是一款基于 Python的 Web 开发框架,符合 MVC 架构,以其简单易学、组件的可重用性和可插拔性、开发周期短而著称[10]。Django 框架原理如图6所示。
Model 层是 MongoDB 中 collections 的逻辑映射,必须把 MongoDB 中的所有字段都一一对应定义(字段名称必须相同) 。
Controller 层在URLs.py 里规定 URL 映射规则,如下。

这代表当用户在URL 栏中输 127.0.0.1:8000/paperCount/ 时,会调用 views.paperCountShow 函数。
而在paper_analysis_web 中 views.py 规定业务处理逻辑,并将数据以json格式返回给 templates 中的HTML 网页。
View 层在整个项目的 templates 里面存放 html页面。前端页面用 Echart 组件展示,Echart 是百度出品的一款强大的,交互友好的可视化图表js 库。我们只需引用Echart中的图表,传入数据即可。比如views.py 传来的 jso 数据中有两个关键字:val1,val2,那么 paperCountShow.html 中接收数据的方式为{{val1|safe}},{{val2|safe}}。
用户可以通过在浏览器输入网址查看分析结果,效果如图7所示。
用户可以对图表进行个性化的解读,比如从图中我们可以看出,2010–2015年中科院在data mining领域发表文献数量稳步递增,2015–2017年数量平稳没有较大变化,2017–2018年数量减少,这可能是由于 data mining 领域文献数量愈多质量愈高竞争愈加激烈、录用难度提升所致。
同理,更多基于文献计量学的学科发展态势分析模块可根据用户需求进行设定,比如:可根据author_list 字段观察合著者人数规律,根据 disciplines字段分析某一领域的文献其引文中较常见的学科有哪些,在此不做展开。
4 系统运行结果分析
本系统查询 web of science 核心合集,以 data minging 为主题,选取 2008-2018年时间范围内扩展机构为 Chinese Acadecemy of Sciecne 的所有文献,获取初始 initialURL。符合条件的文献共 1561 篇,分为157 页,每页最多 10 篇文献。
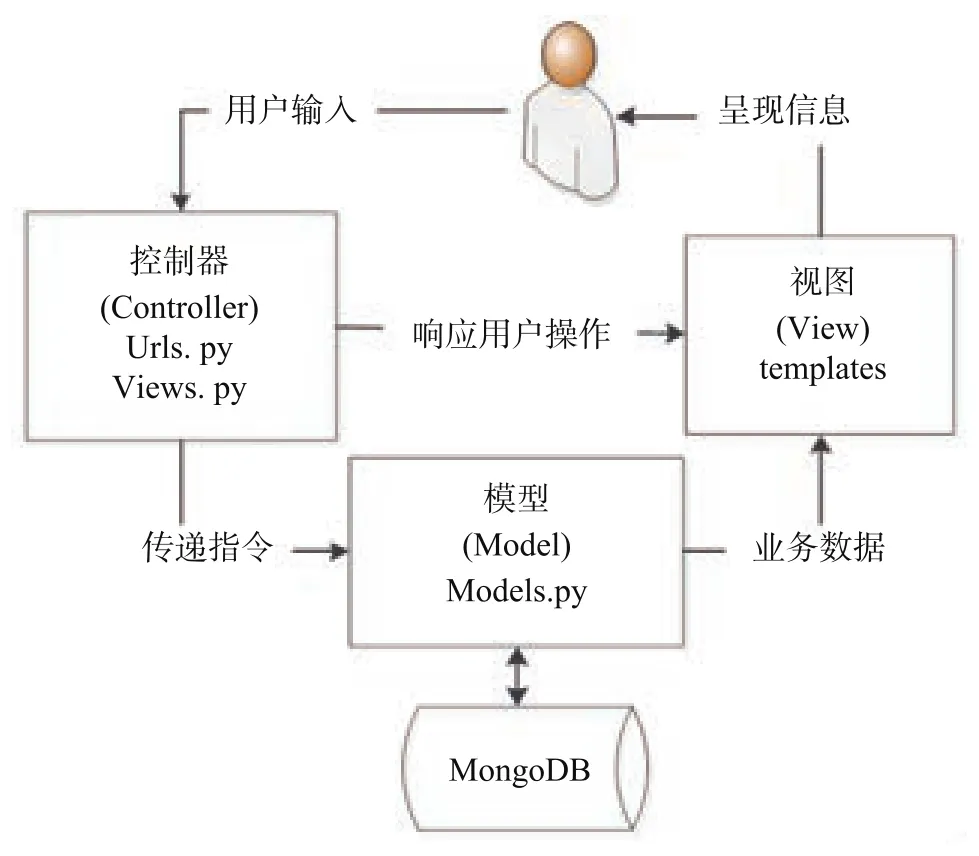
图6 Django Web 原理图Fig.6 Principle of Django Web
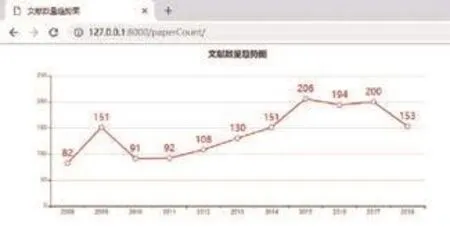
图7 文献数量趋势图Fig.7 Trend of number of literature
我们同时启动 master,slave0,slave1,slave2 爬虫,此时各爬虫处于待命状态。在redis client 中输入lpush wos_master_spider:start_urls initalURL 命令使各爬虫开始工作。
master 端爬虫在网速 100Mbps 的情况下,用11min 爬取了 157 个页面链接。3 个 slave 爬虫在平均网速 148Mbps 的情况下,解析这 157 个链接共耗时220min,平均每分钟系统共解析 5 篇文献。
我们可以在MongoDB 中通过如下语句查询每个爬虫解析了多少篇文献,结果见表2。
db.wos_paper.aggregate([{$group:{_id: '$spider_number', paperCount: {$sum: 1}}}])
因为每篇文献的参考数量不同,所以每篇文献需要的解析时间不同,且由于不同主机间网速存在差异,所以每个爬虫分担的工作量不尽相同,但大致相当。
同时我们可以看到,redis 里的 ref_cat_map 期刊名称及其所属学科映射表共积累了 13507 条映射关系。
5 总结与展望
本文设计了一种基于 scrapy-redis 分布式爬虫的学科发展态势分析系统,slave 爬虫从 redis 队列中读取 master 存储的 url 并进行解析,将结果持久化到MongoDB。DjangoWeb 从 MongoDB 读取数据进行可视化展示。分布式爬虫相较于单机爬虫而言大大提高了爬取效率,为文献计量学提供了一种有效的分析手段。
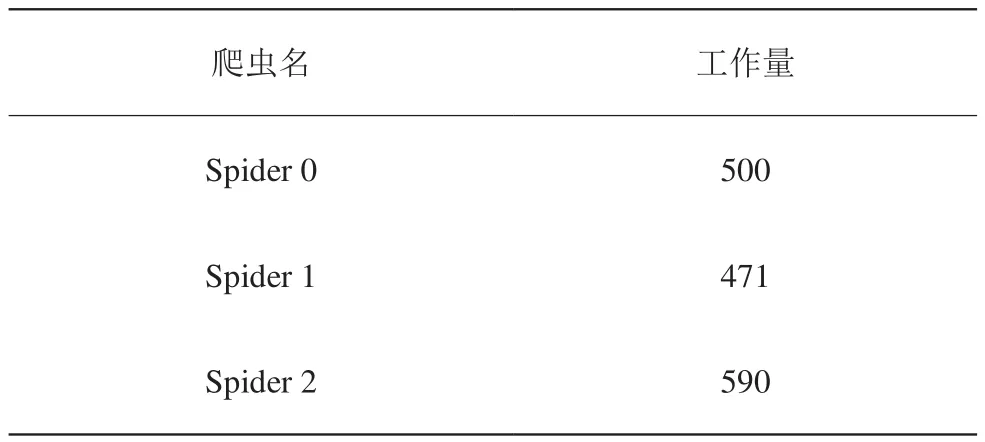
表3 爬虫工作量分布表Table 3 Crawlers workload distribution
但是目前 slave 爬虫数量还需人工根据经验预估提前设定,未来可以向根据任务量自动设定爬虫数量方向进行改进。同时目前多个爬虫分担的工作量还不够均衡,可以从负载均衡角度出发进行改进,充分发挥每个爬虫的资源。
