病例研究:未知病因的凡纳滨对虾溞状幼体的病原和微生物组分析*
2019-09-27万晓媛谢国驷
孙 静 万晓媛 杨 倩 谢国驷 董 宣 黄 倢
病例研究:未知病因的凡纳滨对虾溞状幼体的病原和微生物组分析*
孙 静 万晓媛 杨 倩 谢国驷 董 宣 黄 倢①
(中国水产科学研究院黄海水产研究所 农业农村部海水养殖病害防治重点实验室 青岛海洋科学与技术试点国家实验室海洋渔业科学与食物产出过程功能实验室 青岛市海水养殖流行病学与生物安保重点实验室 青岛 266071)
从某对虾育苗场2个育苗池中分别采集了发生摄食下降、活力降低及死亡率增高等症状的未知疾病的凡纳滨对虾()溞状幼体3期(Z3)和1期(Z1)的样品。用PCR检测白斑综合征病毒(WSSV)、传染性皮下及造血组织坏死病病毒(IHHNV)、急性肝胰腺坏死病副溶血弧菌(AHPND)、桃拉综合征病毒(TSV)、传染性肌坏死病毒(IMNV)、黄头病毒(YHV)、虾肝肠胞虫(EHP)、偷死野田村病毒(CMNV)和虾血细胞虹彩病毒(SHIV)等9种已知病原为阴性。组织病理学诊断观察到肝胰腺小管上皮细胞内存在不明褐色团块。使用2216E培养基对致病菌分离鉴定,得到2株溶藻弧菌(),浸浴感染悉生卤虫()幼体后的平均存活率分别为58%和83%。采用Illumina HiSeq高通量测序方法对这2个溞状幼体样品中细菌16S rRNA基因的2个高变区(V3~V4)进行总细菌菌群的测序,分析揭示了未知病因病虾中细菌菌群的多样性和相对丰度,门水平上2个样品的优势菌群均包括变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)和厚壁菌门(Firmicutes),但相对丰度有显著差异;在属水平上相对丰度最高的均为弧菌属(),在2个样品中的丰度分别为74.3%和60.5%,此外,Z1样品相对高丰度(21.9%)的属为黏着杆菌属()。育苗管理信息及上述病例分析结果提示,该疾病可能是由于在过期存放的幼体饲料质量下降所致的营养障碍情况下,有一定致病性的溶藻弧菌条件性感染所引起的疾病。
病例研究;凡纳滨对虾;溞状幼体;未知病因;菌群多样性
对虾养殖产业产生了巨大的经济效益,然而,急性肝胰腺坏死病等细菌性病害暴发,严重威胁对虾养殖产业(张宝存等, 2012; FAO, 2013)。目前,对致病微生物的研究还主要依赖于可培养的微生物(阎冰等, 2004),然而,迄今为止,在实验室条件下超过99%的微生物还无法被分离及纯培养(Kellenberger, 2001),因此采用传统培养技术调查养殖系统中的微生物群落会造成片面的认识,许多未知疾病的致病原也尚未被发现。1998年,宏基因组(Metagenomics)的概念首次被提出(Handelsman, 1998),它是将微生物生态群落中所有微生物的基因组看作一个整体,通过研究这个整体中物种的组成、丰度和功能来探索微生物群落及其生境之间的相互关系(Handelsman, 1998; 叶雷等, 2016)。与传统研究方法相比,基于宏基因组的高通量测序技术具有一系列优点:首先,不需要依赖培养技术对微生物进行分离;其次,可利用较少的样本量检测到低丰度的微生物;而且可以同时对多个样本进行分析,从而更加快速和完整地反映样品中微生物的群落特征(Handelsman, 1998; 孙欣等, 2013; Williams, 2014)。因此,近几年来,越来越多的研究者采用高通量测序技术来进行未知及复杂疾病的病原检测(Alavandi, 2012; 浦绍艳等, 2014)。采用Ion Torrent PGM平台,研究者对未知病因足部溃烂病人样品的16S rDNA基因V1~V2高变区进行测序,发现了一部分与足部慢性溃疡有关的细菌(刘文丽等, 2016)。Johnston等(2017)研究牛呼吸道疾病(BRD)的肺部组织样品,通过Illumina Miseq测序16S rDNA基因的V3~V4区,揭示了肺和淋巴结中的不同优势菌组成,并发现导致牛呼吸道疾病的病原菌可能从属于纤毛菌科(Leptotrichiaceae)中一个新的菌属。尽管高通量测序在人体及陆生动物疾病诊断中已经得到应用,但在水生动物疾病研究中还鲜有报道。
2017年2月,在浙江某对虾育苗场了解到有2个苗池的凡纳滨对虾()溞状幼体发生未知病因的疾病,养殖池水体的理化指标均正常,但幼体出现摄食少、活动力差和死亡等情况。在2个苗池分别采集病虾样品进行多种病原检测,并用组织病理学、微生物分离鉴定和微生物组学方法分析了各样品的潜在病原、病理和细菌菌群结构,为该病例的发病原因分析提供线索。
1 材料与方法
1.1 样品采集
凡纳滨对虾的溞状幼体样品于2017年2月取自浙江某育苗场,现场了解育苗情况及病害情况后,取2个患病苗池的幼体各0.1,分别标记为20170222002 (溞状幼体3期, Z3)和20170222003(溞状幼体1期, Z1),使用Davidson’s AFA (DAFA) (Bell, 1988)和3倍体积95%乙醇保存(陈大菾等, 2015),另取一部分样品匀浆后接种于2216E液体培养基中。样品运回实验室后,95%乙醇保存样品,于–80℃冰箱备用。
1.2 组织病理观察
固定24 h后,将DAFA固定的对虾组织更换于70%酒精中保存,并进行梯度脱水、浸蜡、透明、石蜡包埋和切片。切片经苏木精-伊红(HE)染色制片(Bell, 1988),于光学显微镜(Nikon E800)下观察。
1.3 细菌分离鉴定及其致病力检测
将2216E液体培养基保存的菌液,划线接种在新鲜的2216E固体培养基平板上,28℃过夜培养。之后根据平板上菌落形态加以区分,从形态一致、数量较多的菌落中随机选取进行保种,并采用16S rRNA基因对优势菌株进行PCR扩增并测序。进一步采用弧菌基因的特异引物(5¢-GAGAACCCGACAGAA GCGAAG-3¢和5¢-CCTAGTGCGGTGATCAGTGTTG- 3¢) (Wei, 2014)对待鉴定菌株进行进一步比对。MEGA6软件用于基因的系统进化树构建,并进行1000次的Bootstraps重复检验。
分离并鉴定出的细菌采用悉生卤虫()幼体进行致病力检测。分别将各受试菌于28℃下震荡培养至OD600 nm=1备用。取50 ml无菌玻璃瓶,加入10 ml灭菌海水及20只悉生卤虫幼体,并加入已制备的菌液至终浓度为107CFU/ml进行攻毒。空白对照组未加入细菌,阴性对照组加入相同浓度的徐氏弧菌()。每组实验设置3个重复。28℃下光照培养48 h后,统计并计算不同实验组卤虫的存活率。
1.4 总基因组DNA和RNA提取
用研磨珠将2管溞状幼体组织匀浆,取60 µl样品,8000 r/min离心5 min,弃上清液,在室温下放置10 min,使管中的酒精充分挥发。使用TIANamp海洋动物组织基因组试剂盒(天根, 北京)提取DNA,RNAiso Plus (TaKaRa, 大连)提取总RNA。每管溞状幼体3个重复。
1.5 病原携带或感染背景筛查
参照OIE《水生动物疾病诊断手册》(OIE, 2017)的标准检测方法分别筛查白斑综合征病毒(White spot syndrome virus, WSSV)、传染性皮下及造血组织坏死病病毒(Infectious hypodermal and hematopoietic necrosis virus, IHHNV)、急性肝胰腺坏死病副溶血弧菌(Acute hepatopancreatic necrosis disease caused by,AHPND)、桃拉综合征病毒(Taura syndrome virus, TSV)、传染性肌坏死病毒(Infectious myonecrosis virus, IMNV)及黄头病毒(Yellow head virus, YHV);并参考相关文献检测虾肝肠胞虫(, EHP) (Jaroenlak, 2016)、偷死野田村病毒(Covert mortality nodavirus, CMNV) (Zhang, 2014)及虾血细胞虹彩病毒(Shrimp hemocyte iridescent virus, SHIV) (Qiu, 2017)。引物序列详见表1。
表1 常见已知对虾病原PCR检测的引物
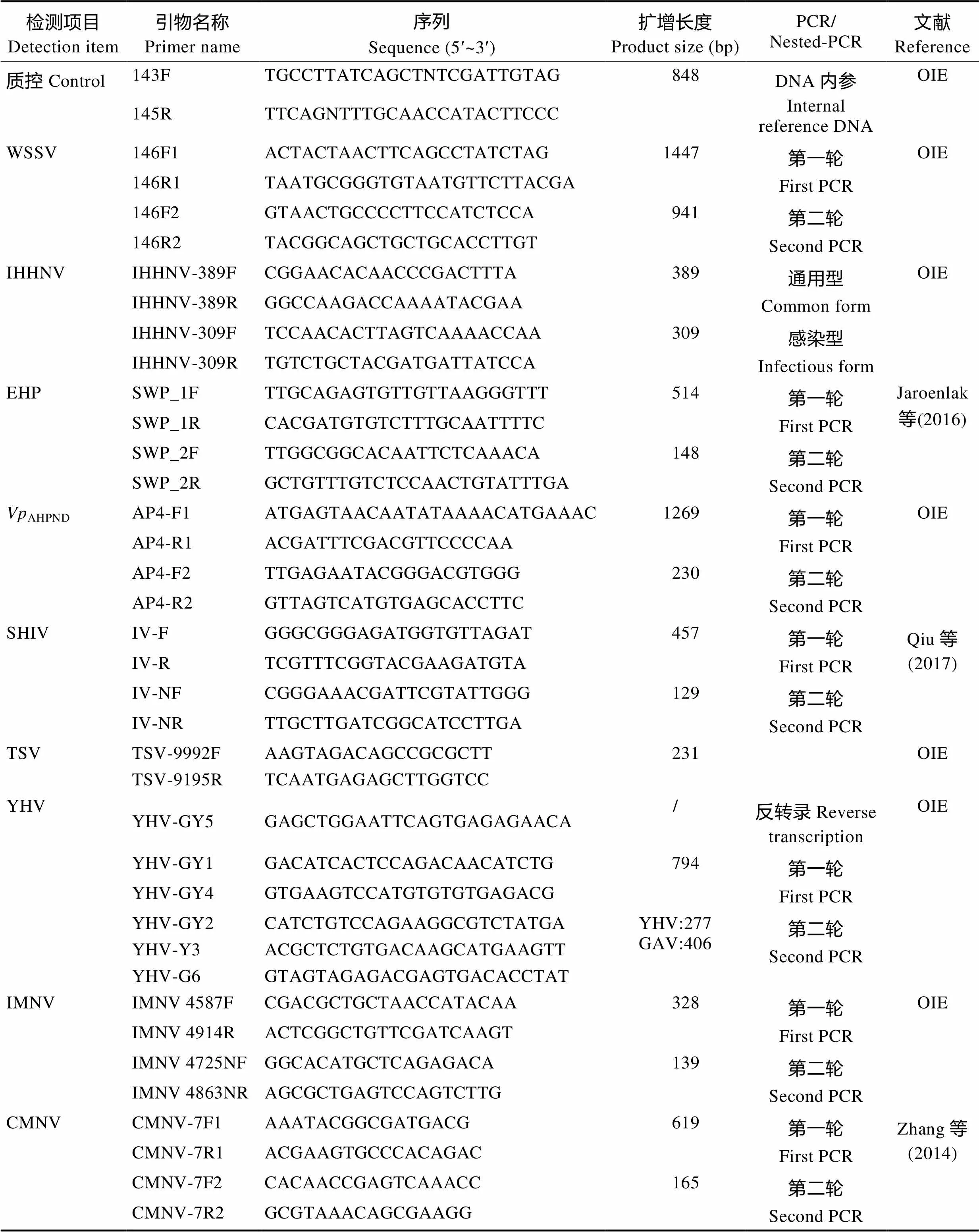
Tab.1 Primers of PCR detection for usual known pathogens of penaeid shrimp
1.6 PCR扩增及Illumina HiSeq高通量测序
将提取的基因组DNA作为模板,扩增16S rDNA序列的V3~V4可变区,引物序列为常规引物341F(5¢- CCTACGGGNGGCWGCAG-3¢)和805R(5¢-GACTAC HVGGGTATCTAATCC-3¢)。PCR反应体系为30 μl,含2×Phusion Master Mix (Thermo Scientific, 美国) 15 μl、2 μmol/L引物混合物3 μl和1 ng/μl gDNA 10 μl。经98℃预变性1 min;98℃ 10 s、50℃ 30 s和72℃ 30 s,30个循环;72℃延伸5 min。每个样品取等量PCR产物混合,用2%琼脂糖凝胶电泳回收目的条带,用geneJET胶回收试剂盒(Thermo Fisher Scientific, 美国)回收产物。使用TruSeq®DNA PCR-Free Sample Preparation Kit (Illumina, 美国)进行文库构建,合格的文库使用HiSeq2500 PE250进行上机测序(诺禾致源公司, 北京)。
1.7 测序数据处理和统计分析
利用Quantitative Insights Into Microbial Ecology (QIIME, v1.7.0) (Caporaso, 2010)对拼接得到的Raw Tags进行严格的质控和去除嵌合体,从而得到高质量的有效数据(Effective tags)。使用Uparse (v7.0.1001) (Edgar, 2013)在97%的相似性水平对有效序列聚类到分类操作单元(Operational taxonomic units, OTU),选取每个OTU中丰度最高的序列作为代表序列,对OTUs代表序列进行物种注释,用Mothur方法(Schloss, 2009)与SILVA (Wang, 2007)的SSU rRNA数据库(Quast, 2013)进行物种注释分析(设定阈值为0.8~1),获得分类学信息并分别在界、门、纲、目、科、属和种等各个分类水平统计各样本的群落组成。
使用QIIME (v1.7.0)计算alpha多样性指数(Observed-species, Chao1, Shannon, Simpson, ACE, Goods-coverage),选用-test检验比较2组样品的差异。LEfSe分析使用LEfSe软件展示了2组样品之间具有统计学差异的物种[默认设置Linear discriminant analysis (LDA) Score的筛选值为4]。
2 结果与分析
2.1 常规分析
2.1.1 虾苗症状和病理分析 2批虾苗均来自进口某品牌的无特定病原(SPF)亲虾产卵所孵化的幼体,亲虾引进时经检疫部门检测合格。虾卵经孵化和无节幼体培育,进入溞状幼体期后均只使用某品牌虾片,该批虾片在4℃~10℃的冷库已存放接近6个月,辅以螺旋藻粉、黑粒、车元等添加剂配制的饲料喂养,未投喂单细胞藻类。采样时,虾苗分别处于溞状幼体3期(Z3)和溞状幼体1期(Z1),其中,Z3出现饲料摄食减少、虾苗活动能力差、虾苗批量死亡的现象,取样后因为死亡率高而全池销毁;Z1出现虾苗活动力下降、饲料摄食量降低、拖便呈细丝状或偏少的情况,后期在变态中损失较大。
对2批虾苗样品进行组织切片,结果显示,均没有可辨认的常见已知病原感染的组织病理症状,但在Z3肝胰腺小管细胞内的脂滴中观察到大量棕黄色的不明团块,Z1肝胰腺小管和部分细胞内也出现棕色颗粒(图1)。
2.1.2 常见已知病原检测 样品分别取自同一个养殖场的2个发病池塘,实验室分子检测结果见表2。如表2所示,2份样品均未检出常见的已知对虾病原,对近几年导致重大对虾疾病的EHP、AHPND和SHIV等进行检测,结果也均为阴性。

图1 病理组织观察
A:溞状幼体3期(Z3)样品;B:溞状幼体1期(Z1)样品
A: zoea stage Ⅲ (Z3); B: zoea stage Ⅰ (Z1)

表2 对样品进行已知病原的检测结果
注:N表示阴性
Note: N means negative result
2.1.3 常规方法分离的细菌及其致病力检测 根据16S rDNA的序列比对结果,进一步采用弧菌属特异引物对待鉴定菌株进行测序比对,最终确定该菌株为溶藻弧菌() (图2)。使用悉生卤虫分别对2个池塘分离的溶藻弧菌致病力进行评估,结果显示,2份样品中分离出的溶藻弧菌均对卤虫具有致病性,且Z3期样品分离株致病性较强(<0.05) (表3)。

图2 基于Neighbor-Joining方法的gyrB基因系统进化树
表3 不同溶藻弧菌分离株对卤虫的致病力检测

Tab.3 Survival of brine shrimp larvae challenged with different V. alginolyticus isolates
注:1:为阴性对照;2:从Z3期样品中分离出的;3:从Z1期样品中分离出的
Note: 1:is negative control; 2:was isolated from Z3 sample; 3:was isolated from Z1 sample
2.2 宏基因组分析
2.2.1 基因组DNA提取及PCR扩增 以提取的总菌群基因组DNA为模板,经PCR扩增所得16S rDNA的V3~V4区目的片段条带清晰(图3),可以满足后续测序实验的要求。
2.2.2 样品菌群的多样性统计分析 上述所得V3~V4区的产物通过高通量测序,共获得43330条有效序列,以97%的一致性(Identity)将这些序列聚类成为平均280个OTUs。
经统计分析获得序列的α-多样性如表4所示。α-多样性是反映样品内微生物群落的丰富度和均匀度的一个综合指标。评估丰富度的指标主要为种类数目(Observed-species)、测序深度指数(Goods-coverage),Chao1指数和ACE指数;而群落中个体分配上的均匀性,即多样性,主要包括Shannon指数和Simpson指数,通常这2个值越大意味着群落多样性越高。Z3期(S1)样品群落丰度指数Shannon、Simpson和Chao1略高于Z1期(S2)样品,而ACE指数略低于Z1期样品。比较2个时期细菌群落的α-多样性指数可知,Z3期的细菌群落多样性和丰度均高于Z1期(表4)。-test检验结果显示,2组样品的细菌群落的Observed-species、Chao1、ACE和Goods-coverage指数均无显著差异(0.05);而Shannon和Simpson指数则差异显著(<0.05)。
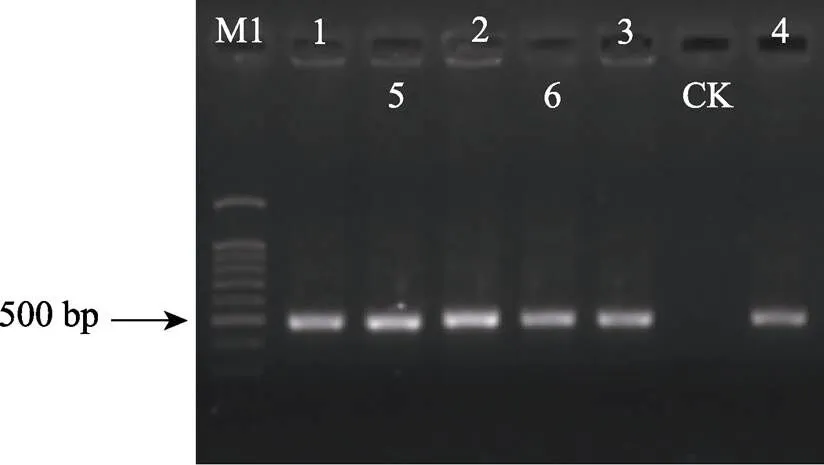
图3 6个样品的PCR扩增产物电泳
M1:100 bp分子量标准; 1~6分别为6份样品的PCR产物;CK:空白对照
M1: 100 bp ladder; 1~6: Six PCR products; CK: Blank control

表4 2组虾苗中总细菌菌群的alpha多样性
注:*表示差异显著(<0.05)
Note: * represent significant difference (<0.05)
2.2.3 样品细菌群落的物种相对丰度分析 根据物种注释结果,选取每个样品和分组在各分类水平上最大丰度排名前10的物种,生成物种相对丰度柱型累加图(图4)显示,2组溞状幼体在菌群门水平上的组成种类差异不大,总菌群的优势门主要包括变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)和厚壁菌门(Firmicutes)。Z3期的平均相对丰度分别为89.5%、8.9%和1.3%,合计占总细菌序列数的99.7%;Z1期的平均相对丰度为72.1%、26.9%和0.7%,合计占总细菌序列数的99.7%。Z3期溞状幼体中变形菌门和厚壁菌门的相对丰度显著高于Z1期,但Z1期中拟杆菌门的相对丰度高于Z3期的样品。
在属水平上的统计分析结果表明(图5),Z3期样品中相对丰度最高的是弧菌属(, 74.3%),相对丰度>1%的包括发光杆菌属(, 2.9%)、欧文维克菌属(, 1.9%)、黏着杆菌属(, 1.4%)、海洋螺菌属(, 1.4%)、深海短杆菌属(, 1.1%)和其他未分类菌(others, 16.1%);Z1期样品中相对丰度高的除了弧菌属(60.5%),还包括黏着杆菌属(21.9%),相对丰度>1%的则是褐指藻杆菌属(, 1.6%)、姜氏菌属(,1.2%)和其他未分类菌(others, 13.1%)。由于此研究仅是对总的16S rDNA序列的V3~V4可变区进行扩增和高通量测序,因此,只能准确到属的水平,而非物种水平。
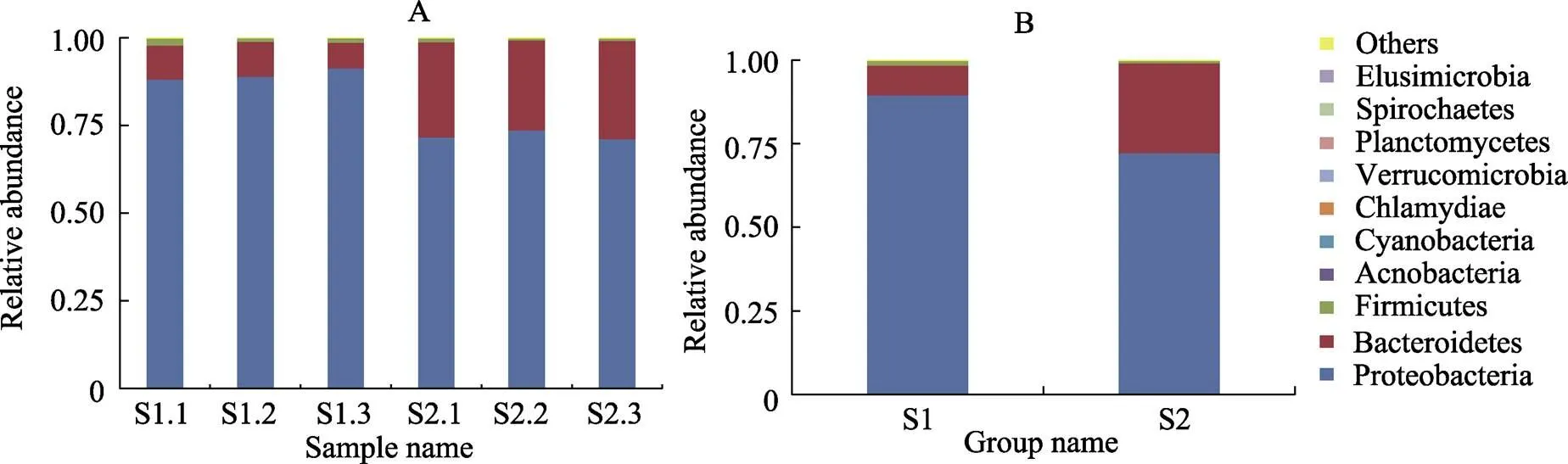
图4 各个样品(A)和分组样品(B)门水平上的物种相对丰度
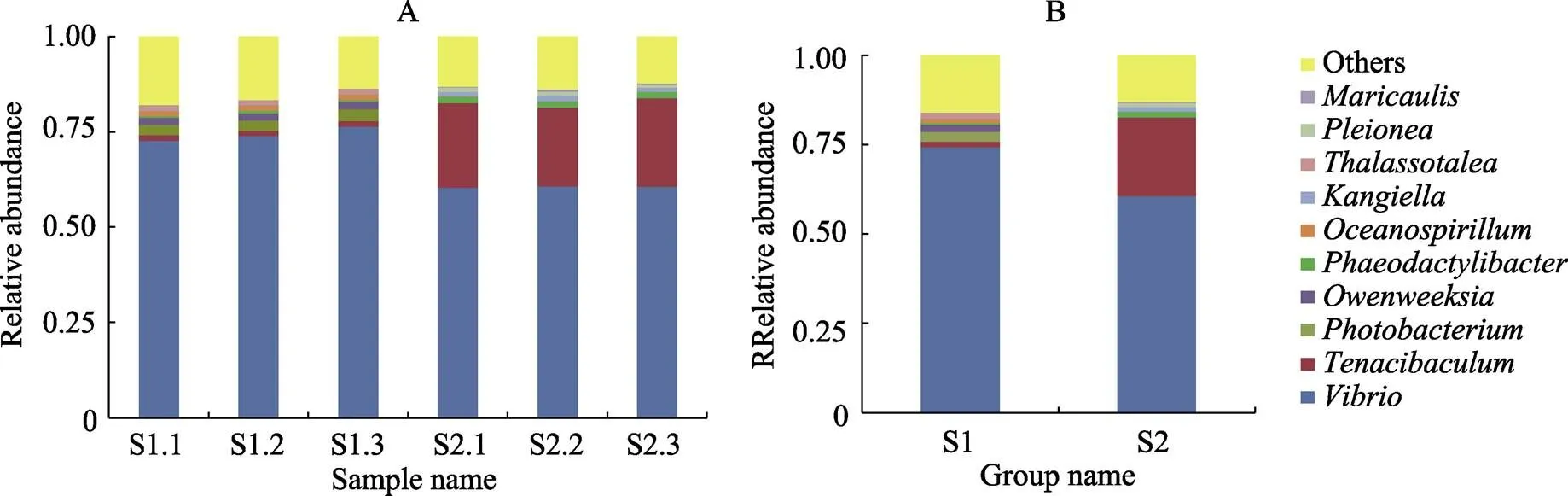
图5 各个样品(A)和分组样品(B)属水平上的物种相对丰度
2.2.4 样品细菌群落结构的差异性分析 从LDA值分布柱状图(图6A)可以看出,在属的水平上,发光杆菌属、弧菌属和黏着杆菌属是2组溞状幼体样品中差异显著的物种。在Z3期样品中,光合杆菌属和弧菌属的LDA值均接近4.8;而对于Z1期样品,在黏着杆菌属的LDA值接近4.8。由进化分支图(图6B)显示,弧菌科(Vibrionaceae)-弧菌目(Vibrionales)、侏囊菌科(Nannocystaceae)-黏球菌目(Myxococcales)和交替单胞菌科(Alteromonadaceae)均属于变形菌门;而黄杆菌科(Flavobacteriaceae)-黄杆菌目(Flavobacteriales)-黄杆菌纲(Flavobacteriia)属于拟杆菌门。
3 讨论
在对虾的育苗期,对虾先由无节幼体变态发育为溞状幼体,溞状幼体分为3期,在此阶段发育至具有完整口器和消化器官,开始摄食。在对虾育苗生产中,溞状期的培育是成败的关键,幼体营养需求的满足能显著影响其变态时间(李绍彬等, 2000),以虾片为饲料主成分时,不同品牌的虾片对育苗效果也可能有显著影响(杨育凯等, 2017)。本研究所针对的育苗场只采用了某品牌的虾片为饲料主成分,且该批虾片在育苗场的4℃~10℃的冷库存放接近半年。采用该虾片投喂的溞状幼体出现了明显的摄食量下降、活力减弱和死亡问题,其中,Z3期的育苗池发病比Z1期的育苗池表现更严重,在采样后Z3期的育苗池因为存活率太低而销毁。组织病理观察发现,Z3的样品在肝胰腺细胞内出现大量不明的棕色团块,疑似饲料沉积物。因此,初步怀疑可能是因为虾片品牌或存储时间过长,发生酸败或其他问题影响了虾片质量,导致其营养未能有效利用,产生细胞内沉积,影响了虾苗的抗病力和存活率(梁萌青等, 1996; Laohabanjong, 2009),而Z1期可能刚开始摄食,受饲料的影响刚开始出现,尚不如Z3严重。
通过分子学手段对对虾已知的多种病原进行检测,均为阴性,说明这次溞状幼体期的病害不是这些感染性病原所致。通过2216E培养基的培养,从Z3和Z1幼体中均分离到溶藻弧菌,2株溶藻弧菌对悉生卤虫幼体表现出致病力,表明该菌存在一定的毒力。研究表明,包括溶藻弧菌在内的多种弧菌容易侵染溞状幼体,且短时间内能导致全部死亡(王景明等, 1992),Hamza等(2018)研究也发现,从发病对虾样品中分离出的溶藻弧菌可使悉生卤虫幼体的死亡率达到97%。然而溶藻弧菌的来源尚不明确,此菌不存在芽孢形式,在干粉饲料制作中如果有污染,其存活的时间也十分有限,因此,饲料中存在溶藻弧菌的可能性很小。
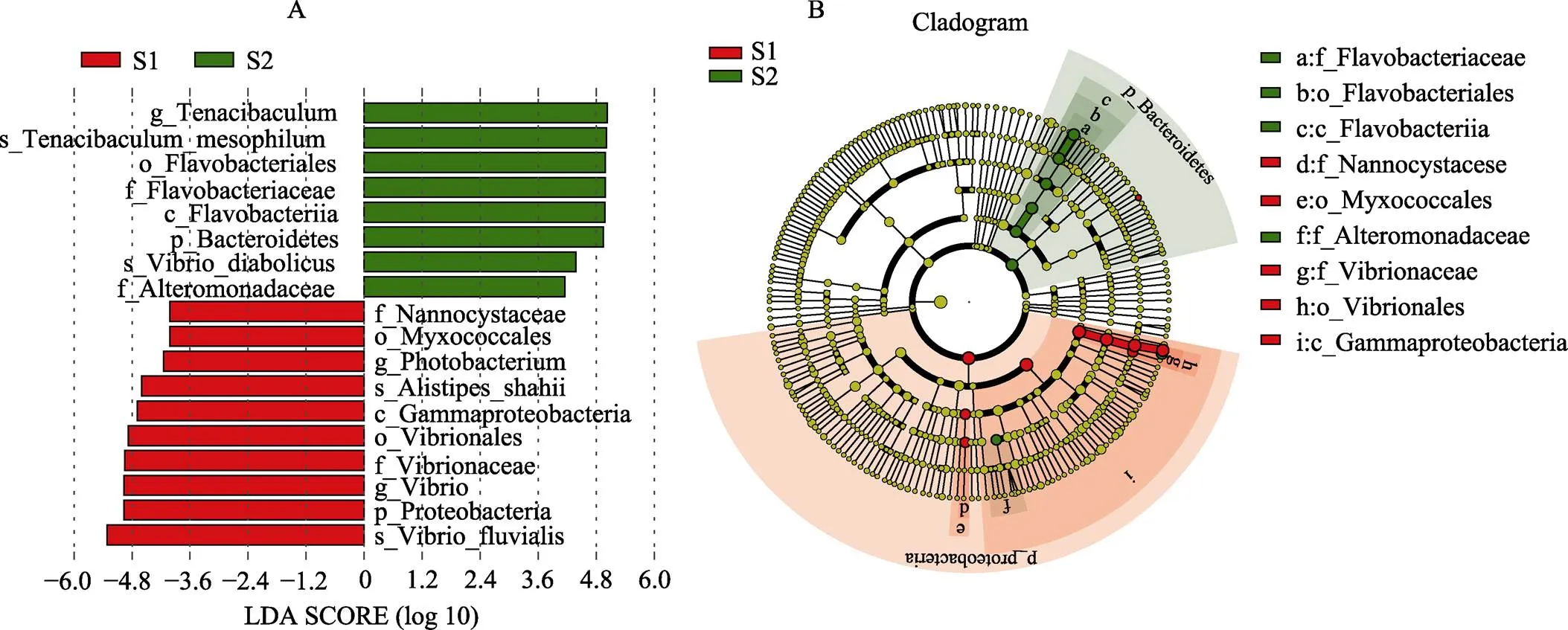
图6 LDA值分布柱状图(A)和进化分支图(B)
LDA值展示2组样品中丰度差异显著的物种(LDA Score>4),柱状图的长度代表差异物种的影响大小。进化分支图由内至外辐射的圆圈代表了由门到种的分类级别。在不同分类级别上的每一个小圆圈代表该水平下的一个分类,小圆圈直径大小与相对丰度大小呈正比。红色圆圈代表Z3期样品(S1)中差异显著的物种,绿色圆圈代表Z1期样品(S2)中差异显著的物种,黄色圆圈代表无显著差异的物种
LDA score (>4) represents the significantly differential abundance in the microbial taxa between samples. The biomarkers from samples were highlighted in the cladogram. From the innermost to outmost, each cycle represents the phylum, class, order, family, genus and species level. Each dot represents a taxon, and its diameter positively correlates with the abundance of the corresponding taxon. The red and green colored dots indicate the biomarkers from Z3 (S1) and Z1(S2) samples, respectively. The yellow dot represents the taxa with nonsignificant changes between groups
宏基因组学方法为未知疾病的病因及其潜在致病微生物分析提供了更全面的微生物组信息。由于大多数微生物在常规的培养基上无法生长,而且采用传统平板培养基分离出的微生物具有营养偏好性,因此,得到的结果无法全面地体现菌群组成的真实情况。高通量测序无需培养,能全面地反映样品的微生物多样性。目前,已有不少对虾体内和养殖水体中菌群组成的研究,但发病虾苗中菌群多样性研究的报道较少。本研究检测到发病对虾溞状幼体主要菌群与报道的健康凡纳滨对虾菌群类似,在门水平均包括变形菌门和拟杆菌门(Rungrassamee, 2013、2014; 吴金凤等, 2016; 唐阳等, 2017),但在属水平上,发病对虾溞状幼体样品中弧菌属和黏着杆菌属相对丰度较高。采用平板培养分离出的优势菌亦为弧菌属的溶藻弧菌,且使用悉生卤虫检验了此菌的致病力,推断弧菌可能是导致这些虾苗发病的原因。有研究显示,加入有益菌(如蜡样芽孢杆菌)可以抑制弧菌等有害菌的生长(唐阳等, 2017),生产中也观察到向溞状幼体期的水体投放益生菌(如交替投放芽孢杆菌和乳酸菌制剂),可提高溞状幼体期的变态率和存活率(林黑着等, 2015)。
黏着杆菌属也是海洋环境中常见的一类致病细菌(Pridgeon, 2012),最早发现可侵染鱼类,比如能导致舌鳎和大菱鲆发生黏着杆菌病(Tenacibaculosis)的海洋黏着杆菌() [曾用名为海洋屈挠杆菌()] (Vilar, 2012; Avendaño-herrera, 2006)。巴西学者发现,海洋黏着杆菌能造成凡纳滨对虾苗期,尤其是仔虾期(Postlarvae)大规模死亡,同时还伴有身体发黑、活力差和吃料少等病症(Mouriño, 2008);但也有研究表明,黏着杆菌在正常的对虾中也存在(张欢欢等, 2016; 唐阳等, 2017),但丰度较低。在本研究的Z1期样品中黏着杆菌的丰度仅次于弧菌,此类细菌也可能直接或者间接导致这批凡纳滨对虾溞状幼体的发病。
虾类病害诊断的目的是确定病害种类、掌握病因和查明病原。但与医学疾病诊断相比,针对病例进行详细剖析的报道少,诊断工作中遇到的多种多样的情况难以找到判断的依据,因此,病例研究的报道是提高对各种病害诊断的准确性的重要支持。本研究首先对对虾育苗期的未知病例进行了详细的背景描述和症状观察,再采用组织病理学、病原检测、分离培养并结合宏基因组测定方法进行分析,报告全面的背景情况和分析数据信息,能为将来该类病害的诊断提供参考。目前,目标基因的宏基因组测序成本降低,此技术能提供微生物组的种属和数量的更全面信息,在疾病诊断方面具有重要意义,因此,对于病因复杂的疾病,应考虑增加宏基因组分析样品的取样,以备为发病宿主的微生物群落分析提供材料。
Alavandi SV, Poornima M. Viral metagnomics: A tool for virus discovery and diversity in aquaculture. Indian Journal of Virology, 2012, 23(2): 88–98
Avendaño-herrera R, Magariños B, Irgang R,. Use of hydrogen peroxide against the fish pathogenand its effect on infected turbot (). Aquaculture, 2006, 257(1–4): 104–110
Bell TA, Lightner DV. A handbook of normal penaeid shrimp histology. Baton Rouge, LA: World Aquaculture Society, 1988
Caporaso JG, Kuczynski J, Stombaugh J,. QIIME allows analysis of high-throughput community sequencing data. Nature Methods, 2010, 7(5): 335–336
Chen DT, Huang J, Wang HL,Selection and optimization of simple and convenient sample solutions for shrimp tissue preservation at normal temperature. Progress in Fishery Sciences, 2015, 36(5): 71–80 [陈大菾, 黄倢, 王海亮, 等. 凡纳滨对虾()组织样品常温保存液的筛选和优化. 渔业科学进展, 2015, 36(5): 71–80]
Edgar RC. UPARSE: Highly accurate OTU sequences from microbial amplicon reads. Nature Methods, 2013, 10(10): 996–998
FAO. Report of the FAO/MARD technical workshop on early mortality syndrome (EMS) or acute hepatopancreatic necrosis syndrome (AHPNS) of cultured shrimp (under TCP/VIE/3304) Hanoi, Vietnam, 25–27 June, 2013, FAO Fisheries and Aquaculture Report No. 1053
Hamza F, Kumar AR, Zinjarde S. Efficacy of cell free supernatant fromin protectingagainstand. Microbial Pathogenesis, 2018, 116: 335–344
Handelsman J, Rondon MR, Brady SF,Molecular biological access to the chemistry of unknown soil microbes: A new frontier for natural products. Chemistry and Biology, 1998, 5(10): 245–249
Jaroenlak P, Sanguanrut P, Williams BA,. A nested PCR assay to avoid false positive detection of the microsporidian(EHP) in environmental samples in shrimp farms. PLoS One, 2016, 11(11): e0166320
Johnston D, Earley B, Cormican P,. Illumina MiSeq 16S amplicon sequence analysis of bovine respiratory disease associated bacteria in lung and mediastinal lymph node tissue. BMC Veterinary Research, 2017, 13(1): 118
Kellenberger E. Exploring the unknown. The silent revolution of microbiology. EMBO Report, 2001, 2(1): 5–7
Laohabanjong R, Tantikitti C, Benjakul S,. Lipid oxidation in fish meal stored under different conditions on growth, feed efficiency and hepatopancreatic cells of black tiger shrimp (). Aquaculture, 2009, 286(3–4): 283–289
Li SB, Zhang X. Effect of nutrition on transformation of zoea stage of white leg shrimp. Mariculture, 2000, 55: 11–13 [李绍彬, 张欣. 南美白对虾蚤状期幼体的营养对其变态的影响. 海水养殖, 2000, 55: 11–13]
Liang MQ, Xu MQ, Yao J,. The effect of acided oil and aflatoxin on the prawns () growth. Journal of Fishery Sciences of China, 1996, 3(4): 48–52 [梁萌青, 徐明起, 姚健, 等. 酸败油脂和黄曲霉毒素对中国对虾()生长的影响. 中国水产科学, 1996, 3(4): 48–52]
Lin HZ, Hong YQ, Yang YK. One kind of white shrimp breeding methods. China patent: CN201510114710.1, 2015 [林黑着, 洪越群,杨育凯,等. 一种南美白对虾的育苗方法. 专利号:CN201510114710.1, 2015]
Liu WL, Mi ZQ, Wang W,. Pathogenic factors research of foot ulcers patients with unknown etiology. Letters in Biotechnology, 2016, 27(3): 318–321, 361 [刘文丽, 米志强, 王伟, 等. 基于16S rDNA宏基因组学技术探究未知病因足部溃烂的致病因素. 生物技术通讯, 2016, 27(3): 318–321, 361]
Mouriño JL, Vinatea L, Buglioneneto C,. Characterization and experimental infection of(Wakabayashi. 1986) in hatcheries of post-larvae ofBoone, 1931. Brazilian Journal of Biology, 2008, 68(1): 173–177
OIE. Manual of diagnostic tests for aquatic animals. World Organisation of Animal Health, 2017, http://www.oie.int/ international-standard-setting/aquatic-manual/access-online/
Pridgeon JW, Klesius PH. Major bacterial diseases in aquaculture and their vaccine development. CAB Reviews Perspectives in Agriculture, Veterinary Science, Nutrition and Natural Resources, 2012, 7(48): doi: 10.1079/ PAVSNNR20127048
Pu SY, Zhang XL, Jiang TJ,. Metagenomics research and its applications in virus detection. Acta Biophysica Sinica, 2014, 30(1): 3–14 [浦绍艳, 张鑫磊, 蒋太交, 等. 宏基因组学研究方法及其在病原体检测中的应用. 生物物理学报, 2014, 30(1): 3–14]
Qiu L, Chen M, Wan X,. Characterization of a new member of, shrimp hemocyte iridescent virus (SHIV), found in white leg shrimp (). Scientific Reports, 2017, 7(1): 11834
Quast C, Pruesse E, Yilmaz P,The SILVA ribosomal RNA gene database project: Improved data processing and web-based tools. Nucleic Acids Research, 2013, 41: D590–D596
Rungrassamee W, Klanchui A, Chaiyapechara S,. Bacterial population in intestines of the black tiger shrimp () under different growth stages. PLoS One, 2013, 8(4): e60802
Rungrassamee W, Klanchui A, Maibunkaew S,. Characterization of intestinal bacteria in wild and domesticated adult black tiger shrimp (). PLoS One, 2014, 9(3): e91853
Schloss PD, Westcott SL, Ryabin T,. Introducing mothur: Open-source, platform-independent, community-supported software for describing and comparing microbial communities. Applied and Environmental Microbiology, 2009, 75(23): 7537–7541
Sun X, Gao Y, Yang YF. Recent advancement in microbial environmental research using metagenomics tools. Biodiversity Science, 2013, 21(4): 393–400 [孙欣, 高莹, 杨云锋. 环境微生物的宏基因组学研究新进展. 生物多样性, 2013, 21(4): 393–400]
Tang Y, Liu WL, Song XL,. Effects of dietary withon the growth rate and intestinal microflora of. Journal of Fisheries of China, 2017, 41(5): 766–774 [唐阳, 刘文亮, 宋晓玲, 等. 饲料中补充蜡样芽孢杆菌对凡纳滨对虾生长及其肠道微生物组成的影响. 水产学报, 2017, 41(5): 766–774]
Vilar P, Failde LD, Bermudez R,. Morphopathological features of a severe ulcerative disease outbreak associated within cultivated sole,(L.). Journal of Fish Diseases, 2012, 35(6): 437–445
Wang JM, Wan SY, Xiao YF,. Studies on the pathogenicity of Vibios to shrimpFishery Science, 1992, 11(12): 10–12 [王景明, 万三元, 肖艳芳, 等. 弧菌对斑节对虾虾苗致病性的研究. 水产科学, 1992, 11(12): 10–12]
Wang Q, Garrity GM, Tiedje JM,. Naive Bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy. Applied and Environmental Microbiology, 2007, 73(16): 5261–5267
Wei S, Zhao H, Xian Y,. Multiplex PCR assays for the detection of,,, andwith an internal amplification control.Diagnostic Microbiology and Infectious Disease, 2014, 79(2): 115–118
Williams ST, Foster PG, Littlewood DT. The complete mitochondrial genome of a turbinid vetigastropod from MiSeq Illumina sequencing of genomic DNA and steps towards a resolved gastropod phylogeny. Gene, 2014, 533(1): 38–47
Wu JF, Xiong JB, Wang X,. Intestinal bacterial community is indicative for the healthy status of. Chinese Journal of Applied Ecology, 2016, 27(2): 611–621 [吴金凤, 熊金波, 王欣, 等. 肠道菌群对凡纳滨对虾健康的指示作用. 应用生态学报, 2016, 27(2): 611–621]
Yan B, Hong K, Xu Y,. Metagenome cloning‒A new approach for novel microbial bioactive compounds discovery. Microbiology, 2004, 32(1): 113–117 [阎冰, 洪葵许云, 等. 宏基因组克隆——微生物活性物质筛选的新途径. 微生物学通报, 2004, 32(1): 113–117]
Yang YK, Lin HZ, Yang QB,. Effect evaluation of three kinds of shrimp flake in the larval rearing of. Journal of Sichuan Agricultural University, 2017, 35(2): 266–272 [杨育凯, 林黑着, 杨其彬, 等. 3种虾片在凡纳滨对虾育苗中的效果评价. 四川农业大学学报, 2017, 35(2): 266–272]
Ye L, Yan YL, Chen QS,. Application of high-throughput sequencing technology in studying matagenomics of intestinal microbiota. Journal of Chinese Institute of Food Science and Technology, 2016, 16(7): 216–223 [叶雷, 闫亚丽, 陈庆森, 等. 高通量测序技术在肠道微生物宏基因组学研究中的应用. 中国食品学报, 2016, 16(7): 216–223]
Zhang BC, Liu F, Bian HH,. Isolation, identification, and pathogenicity analysis of astrain from. Progress in Fishery Sciences, 2012, 33(2) 56–62 [张宝存, 刘飞, 边慧慧, 等. 一株凡纳滨对虾病原菌的分离、鉴定及其致病力分析. 渔业科学进展, 2012, 33(2): 56–62]
Zhang HH, Wang XH, Li C,. Isolation and identification of asp. strain and its role in bioflocs for the shrimp culture system. Progress in Fishery Sciences, 2016, 37(2): 111–118 [张欢欢, 王秀华, 李晨, 等. 一株芽孢杆菌的分离鉴定及在生物絮团对虾养殖中的应用. 渔业科学进展, 2016, 37(2): 111–118]
Zhang Q, Liu Q, Liu S,. A new nodavirus is associated with covert mortality disease of shrimp. Journal of General Virology, 2014, 95(12): 2700–2709
Case Studies: Pathogenic Agent and Microbiome Analysis for Zoea ofSuffering from an Unknown Disease
SUN Jing, WAN Xiaoyuan, YANG Qian, XIE Guosi, DONG Xuan, HUANG Jie①
(Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences; Key Laboratory of Maricultural Organism Disease Control, Ministry of Agriculture and Rural Affairs; Laboratory for Marine Fisheries Science and Food Production Processes, Pilot National Laboratory for Marine Science and Technology (Qingdao); Qingdao Key Laboratory of Mariculture Epidemiology and Biosecurity, Qingdao 266071)
A shrimp hatchery ofwas infected by an unknown disease during the zoea stage with declined feeding, reduced activity, and increased mortality. In order to diagnose and explore the pathogenic factors causing the zoea disease, we collected samples from two diseased rearing ponds at zoea stage 3 (Z3) and zoea stage 1 (Z1), respectively. PCR detection for nine known pathogens, including WSSV (White spot syndrome virus), IHHNV (Infectious hypodermal and hematopoietic necrosis virus),AHPND(Acute hepatopancreatic necrosis disease caused by), TSV (Taura syndrome virus), IMNV (Infectious myonecrosis virus), YHV (Yellow head virus), EHP (), CMNV (Covert mortality nodavirus), and SHIV (Shrimp hemocyte iridescent virus) showed negative results. Histopathological diagnosis showed unknown brown particles in the epithelial cells of the hepatopancreatic tubules. The results of bacterial isolation and identification for potential pathogens resulted in only two isolates ofon 2216E media. The average survival rate of gnotobiotic brine shrimp () nauplii immersion- challenged with these isolates were 58% and 83% respectively. The microbial communities were profiled by high-throughput sequencing of the V3~V4 hypervariable region of 16S rRNA gene using the Illumina HiSeq sequencing platform. Meta-16S-sequencing revealed the microbial diversities and their relative abundance in the zoea stage of. The results showed that Proteobacteria, Bacteroidetes, and Firmicutes were the most dominant phyla in the entire bacterial community from both samples, but their relative abundance was obviously different. At the genus level,was the most abundant genus in both samples, at 74.3% and 60.5%, respectively, and the relative abundance of(21.9%) was the second highest genus in the Z1 stage sample. Collectively, the information regarding the management of larval rearing and the above results of the case study imply that the disease may be caused by an opportunistic infection with some strains ofhaving moderate pathogenicity under the dystrophia of larva caused by quality degradation of larval feed due to the expiration of storage.
Case study;; Zoea; Unknown disease; Microbial diversity
HUANG Jie, E-mail: huangjie@ysfri.ac.cn
* 青岛海洋科学与技术试点国家实验室主任基金(QNLM201706)、中国-东盟海上合作基金项目(2016-2018)、中国水产科学研究院基本科研业务费专项(2017HY-ZD10)和现代农业产业技术体系(CARS-48)共同资助[This work was supported by Pilot National Laboratory for Marine Science and Technology (Qingdao) (QNLM201706), China ASEAN Maritime Cooperation Fund Project (2016-2018), the Fundamental Research Funds for Chinese Academy of Fishery Sciences (2017HY-ZD10), and China Agriculture Research System (CARS-48)]. 孙 静,E-mail: sunjing@ysfri.ac.cn
黄 倢,研究员,E-mail: huangjie@ysfri.ac.cn
2018-06-29,
2018-08-22
S945
A
2095-9869(2019)05-0134-11
10.19663/j.issn2095-9869.20180629001
http://www.yykxjz.cn/
孙静, 万晓媛, 杨倩, 谢国驷, 董宣, 黄倢. 病例研究:未知病因的凡纳滨对虾溞状幼体的病原和微生物组分析. 渔业科学进展, 2019, 40(5): 134–144
Sun J, Wan XY, Yang Q, Xie GS, Dong X, Huang J. Case studies: Pathogenic agent and microbiome analysis for zoea ofsuffering from an unknown disease. Progress in Fishery Sciences, 2019, 40(5): 134–144
(编辑 马璀艳)
