Oncomine数据库的申请及基因信息挖掘
2019-09-27李瑞华田国祥郭晓娟李豹张军吕军
李瑞华,田国祥,郭晓娟,李豹,张军,吕军,5
癌症是一种世界范围内的疾病,严重威胁人类的健康和生命。《世界癌症报告》预测,全球癌症将由2012年的1400万人递增至2025年的1900万人[1]。如何科学地对癌症进行预防控制已成为目前全球性的重大公共卫生问题[2]。根据传统指标如肿瘤大小、临床分期、病理分级、淋巴结转移数目等对癌症患者进行诊疗的预后差异较大。因此,从基因水平出发, 寻找与癌症密切相关的差异表达基因或基因突变对于癌症的精准化治疗显得尤为重要[3]。基因芯片技术是基因水平研究的主要手段之一,将一定数量的DNA片段作为探针按照一定规则有序排列, 固化于固相介质表面, 并生成二维DNA探针阵列[4]。随着精准医学的不断发展,目前基因芯片技术已广泛应用于肿瘤临床治疗和预测[3],随之产生了海量癌基因芯片数据,Oncomine数据库就是一个经典的癌基因芯片数据库,它整合了TCGA、GEO的部分数据,能够帮助研究者筛选一些有价值的靶分子或预测表型,并提供了多种分析工具,可视化地展示癌症和正常组织差异表达分析、共表达分析、元分析等,同时还可以进行药物敏感性、突变或甲基化引起的表达变化等方面的分析。Oncomine数据库的样本量大,质量高,可视化分析效能强,为广大的肿瘤学及生信研究者提供了宝贵的肿瘤基因的相关资料。例如Rugge等[5]对Oncomine中食道癌芯片数据进行差异分析和元分析后,将PDCD4作为目的基因开展后续的研究;Varisli[6]利用Oncomine共表达分析方法和差异分析方法预测Fam83D的功能。
1 Oncomine数据库的申请
研究者可通过Oncomine官方网站申请账号,获得部分数据。首先进入Oncomine数据库官网:https://www.oncomine.org/,首页上方界面展示了数据库数据收录情况:目前Oncomine已收录了715个数据集,包含86 733个样本的芯片数据,界面左侧为登录界面(图1)。新用户点击“Not a user?Register now!”即进入账号注册界面(图2)。依次填写信息进行注册,注意注册邮箱必须是非营利机构(院校或研究机构)邮箱。信息填写完成后,点击“SUBMIT”确认注册。

图1 Oncomine数据库首页
一般5 min内申请者注册邮箱将收到Oncomine数据库的回复邮件,邮件内含有登录账号和密码(图3)。首次登录时会提示修改密码,按提示修改密码后,注册邮箱会收到附有链接的验证邮件,点击链接完成密码修改,即注册完成。

图2 Oncomine数据库账号注册

图3 Oncomine数据库回复邮件
2 Oncomine数据库的基因信息挖掘
2.1 基因表达差异分析 以研究结直肠癌与正常组织的基因表达差异为例,登陆Oncomine数据库后,在界面左侧“Primary Fliter”下依次选择“Analysis Type”、“Differential Analysis”、“Cancervs. Normal Analysis”、“Colorectal Cancer vs.Normal Analysis”,即得到结直肠癌与正常组织的基因表达差异(图4)。通过在界面中间列选择不同的数据子集可分别查看相应的数据,默认显示第一个数据子集的结果,本例中为“Alon Colon”。界面右侧即为比较结果,用渐变色块展示了各个样本中的基因表达情况,其中蓝色表示低表达,红色表示高表达。每一个色块代表一个样本的表达数据,将鼠标移动到色块上时会出现对应数据信息的浮动窗口。还可以通过右上角的下拉菜单选择显示结直肠癌中Overexpression基因以及under-expression基因。

图4 结直肠癌与正常组织的差异表达
2.2 多数据集整合检索基因表达差异分析(元分析) 根据研究目的,在界面中间列勾选所需要的数据集,点击上方的Compare按钮。比如要研究结肠粘液腺癌相关的差异表达基因,可勾选各个结直肠癌研究下“Colon Mucinous Adenocarcinomavs. Normal”相关的样品分析,点击上方的“Compare”,得到元分析结果,即在多个研究结果中,结肠粘液腺癌中高表达的分子(图5)。结果中还展示了中位秩、P值、基因名称,结合元分析结果,选择有研究价值的靶分子进行深入研究。

图5 结肠粘液腺癌基因表达差异元分析
2.3 特定靶分子的检索 以NEBL为例进行检索,在Search栏中输入“NEBL”进行搜索,页面右上角“OTHER VIEWS” 下拉菜单选择“Gene Summary View”,可得到NEBL在膀胱癌、脑癌、乳腺癌、宫颈癌等多种肿瘤中的表达情况(图6)。表格中不同颜色的色块代表表达程度,蓝色表示NEBL在对应的肿瘤中是低表达,红色表示高表达,灰色表示没有数据。色块中的数字代表符合筛选条件的研究数量。点击色块及表格中的浅蓝色字体可直接进入相应研究的数据界面,例如点击图6中表格里浅蓝色字“Bladder Cancer”,可得到NEBL在膀胱癌中的表达数据页面(图7)。
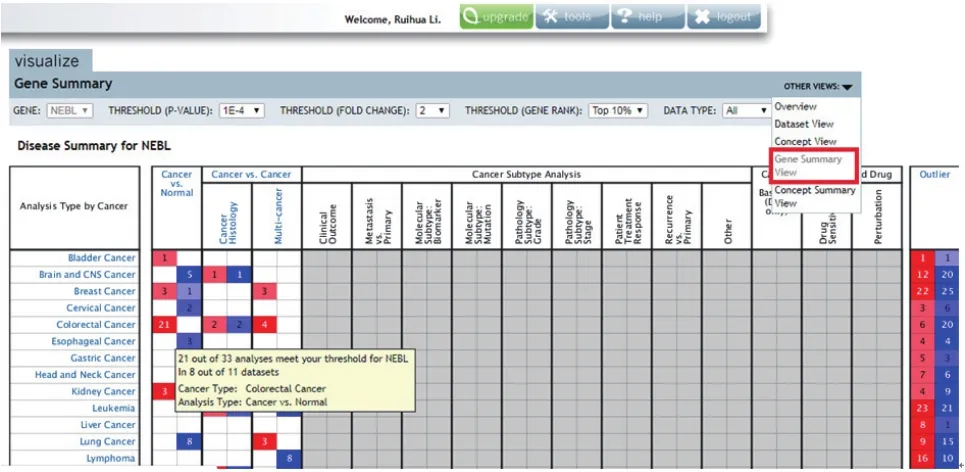
图6 NEBL在多种肿瘤中的表达情况
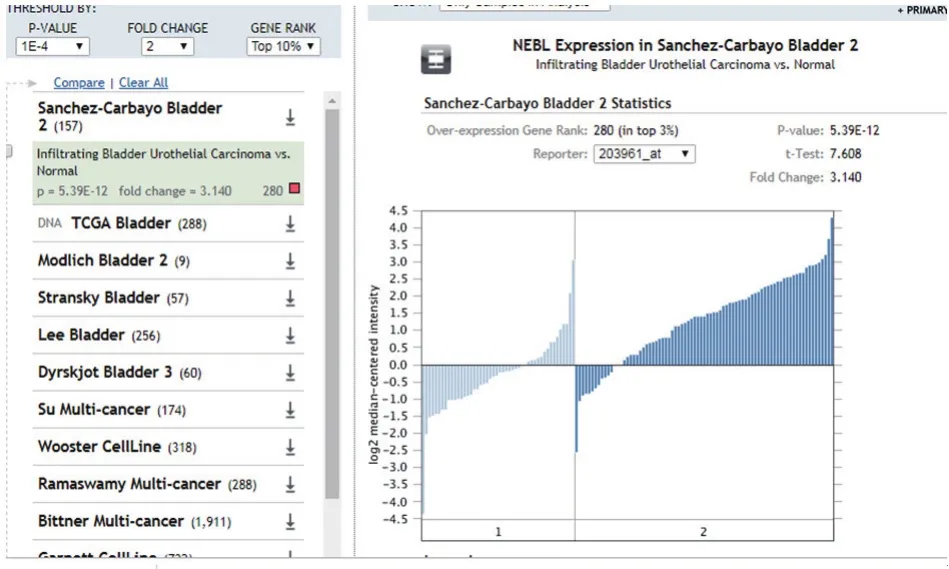
图7 NEBL在膀胱癌中的表达情况
除这种方法外,还可直接在检索特定靶分子在特定肿瘤中的表达,如研究NEBL在结直肠癌中的表达,可在Search栏里输入“NEBL”,在“Primary Filters”里依次选择“Analysis Type”、“Differential Analysis”“Cancervs.Normal Analysis”、“Colorectal Cancervs. Normal Analysis”,然后在中间界面选择要研究的数据集分析,本例选择TCGA下的“Colon Mucinous Adenocarcinomavs. Normal”进行研究,界面上数据集下方还显示该分析的简要结果,包括P值、变化倍数、数值秩,比如NEBL在Colon Mucinous Adenocarcinoma中的表达是正常组织的4.511倍,P值是1.26E-15,秩是66(图8)。通过图形上方的Reporter下拉菜单还可以选择不同的探针。

图8 NEBL在结直肠癌中的表达情况条形图(基于TCGA数据集)
点击图8中椭圆框内示意的按钮,可将单个样品的条形图切换为箱线统计图(图9)。
2.4 多基因共表达分析 仍以NEBL为例,检索结直肠癌中与NEBL共表达的基因,在Search栏输入“NEBL”进行搜索,“Primary Fliters”里依次选择“Analysis Type”、“Coexpression Analysis”,在“Cancer Type”里选择“Colorectal Cancer”,然后在中间列数据集列表里选择“Coexpression”(图10),可得到与目标基因NEBL具有相同表达相关性的分子,按照正相关系数从高到低的顺序排列(图11)。

图9 NEBL在结直肠癌中的表达情况箱线图(基于TCGA数据集)

图10 结直肠癌中与NEBL共表达的基因检索条件

图11 与NEBL具有表达相关性的分子
3 讨论
Oncomine数据库集合了世界上最全的基因组DNA拷贝数、突变、融合基因和mRNA表达的芯片和深度测序结果[7],部分数据免费向公众开放,为相关科研人员提供了极高质量的肿瘤基因芯片数据。本文旨在介绍Oncomine数据库的账号申请及数据挖掘方法,以帮助广大研究者提供高效精准的肿瘤基因分析途径,减少初学者在探索软件过程中浪费时间和精力,提高工作效率。
