青藏高原地区山生柳遗传多样性研究
2019-09-10吴锦杨茜王久利高庆波张发起陈世龙
吴锦 杨茜 王久利 高庆波 张发起 陈世龙


摘要: 利用叶绿体非编码区片段研究分布于青藏高原地区的山生柳居群遗传多样性,对未来山生柳生态环境和青藏高原地区物种丰富度的保护具有指导意义。该研究设计并筛选出cpDNA引物5′trnG2G-3′trnG(UUC)和5′rpS12-rpL20,用扩增出的片段和对应的联合片段进行后续的遗传多样性分析。结果表明:通过山生柳的联合片段检测到35种单倍型,单倍型多态性0.626,核苷酸多态性0.000 85。中性检验Tajima’s D(-2.286 70, P<0.01)和Fu’s Fs(-5.298 05, P<0.02)都是顯著负值,推测山生柳个体数近期经历过扩张。AMOVA分析显示,居群内和居群间遗传变异分别为93.70%和6.30%,表明居群内的变异是山生柳遗传变异的主要来源。居群间遗传分化程度中等偏低(FST=0.063),基因流(Nm)为7.439,说明山生柳各居群的基因交流非常频繁,不同地理居群间存在一定的基因流动。遗传分化系数NST(0.075)大于GST(0.068)和基于遗传距离和单倍型的UPGMA聚类分析,表明山生柳12个居群分为4组且与居群的地理分布没有明显相关性。山生柳是进行有性繁殖还是无性繁殖主要受环境因素的影响,居群内变异是山生柳遗传变异性的主要来源,居群间基因交流频繁。
关键词: 山生柳, 基因流, 青藏高原, 叶绿体DNA, 遗传多样性
中图分类号: ; Q943文献标识码: A文章编号: 1000-3142(2019)09-1180-11
Abstract: We studied the genetic diversity of Salix oritrepha in the Qinghai-Tibetan Plateau using the non-coding region of chloroplast DNA fragments. The study will have guiding significance for the protection of ecological environment and animal husbandry of S. oritrepha and species richness in Qinghai-Tibet Plateau in the future. We designed two useful primers 5′trnG2G-3′trnG (UUC) and 5′rpS12-rpL20. The amplified fragments and their combined fragments were used for subsequent genetic diversity analysis of S. oritrepha. The results showed that we detected 35 haplotypes by the combined sequences, haplotype diversity (Hd) was 0.626, nucleotide diversity (Pi) was 0.000 85. Neutrality tests of Tajima’s D (-2.286 70, P<0.01) and Fus Fs (-5.298 05, P<0.02) showed significantly negative values, which meant that S. oritrepha had expansion recently of its individual numbers. Analysis of molecular variance showed that within-population variance was described as 93.70% of the total variance, while among-population variance was 6.30%, which meant that within-population variance was the main resource of genetic variation of S. oritrepha. The fi-xation index (FST) was 0.063 and number of migrants (Nm) was 7.439, which meant that gene flow was very frequent among different geographical populations. An estimation of non-signification NST (0.075) was bigger than gene differentiation coefficient GST (0.068). UPGMA tree based on genetic distance and haplotypes suggested that all populations were divided into four groups and had no significant correlation with the geographical distribution of the population. The mode of reproduction (sexual or asexual) of S. oritrepha mainly depended on environmental factors. The genetic variability mainly came from within-population and it has a frequently gene flow among populations.
Key words: Salix oritrepha, gene flow, Qinghai-Tibetan Plateau, chloroplast DNA, genetic diversity
山生柳(Salix oritrepha)是中国特有物种,落叶矮小灌木,高10~120 cm,藏药中称朗玛,生于海拔3 000~4 700 m的高山地带,繁衍方式以有性生殖为主,无性繁殖为辅,是青藏高原地区高寒柳灌丛的建群种和优势种,具有极高的药用价值,对生态环境的保护也尤为主要。一方面对山生柳化学成分的研究,证明其可解热、消炎止痛、治疗妇科疾病和提高机体免疫力(封士兰等,2001);另一方面山生柳是深根性灌木,根深长度达到植株纵向高度的4 ~ 5倍,故可以抵抗干旱和低温。由于耐高盐高碱且生长繁殖快速,故能涵养水源、治沙和防沙,使其起到河岸防护、水土保持的作用(陈文业等,2008)。常与高山绣线菊(Spiraea alpina)、金露梅(Potentilla fruticosa)、沙棘(Hippophae rhamnoides)、窄叶鲜卑花(Sibiraea angustata)、大苞柳(Salix pseudospissa)等组成高寒灌丛(王芳等,2010),是藏民牲畜的夏季放牧地。
遗传多样性分析是评价和保护濒危物种的主要指标(Cires et al., 2011),种内遗传多样性越丰富,物种对环境的适应能力就越强,保护了物种和生态系统的多样化,也能减慢因为适应、进化所致的灭亡进程。近几十年来,对青藏高原地区植被的遗传多样性的研究越来越深入(Duan et al., 2011; Khan et al., 2014),这些都对保护青藏高原地区植被丰富度具有重要的指导意义。研究植物遗传多样性的方法中,叶绿体基因组DNA被广泛使用(Demesure et al., 1996;Rungis et al., 2017;Wu, 2016)。叶绿体DNA的结构紧凑简单、分子量小,可從分子角度作比较精准的分析,其比核DNA存在更加稳定、进化更加缓慢且编码序列也保守,故广泛应用于植物遗传多样性的剖析研究(Shaw et al., 2014)。叶绿体DNA在大多数被子植物中为母系遗传,只通过种子进行遗传,不受花粉遗传的影响(Mogensen, 1996)。
目前,对高寒山生柳灌丛的群落特征、在畜牧业中的作用以及在防止沙漠化中起到的作用都有研究(Kang et al., 2015;王纬和王盛,2015;杨路存,2013),但对山生柳遗传多样性的研究较少,郭敏等(2012)的研究表明山生柳种群具有较高的遗传多样性且随着海拔梯度的升高而呈现升高趋势,且遗传变异主要存在于居群内,因其所研究的地理分布范围仅限祁连山,不能准确分析出山生柳遗传结构及种群动态。本研究采集了青藏高原大部分地区的山生柳,设计并筛选出两对特异性针对山生柳的cpDNA引物,用叶绿体非编码区的联合片段对山生柳进行遗传多样性研究,将对山生柳生殖系统的研究、种质资源的保护和生态环境的保护都有重要意义。
1材料与方法
1.1 材料
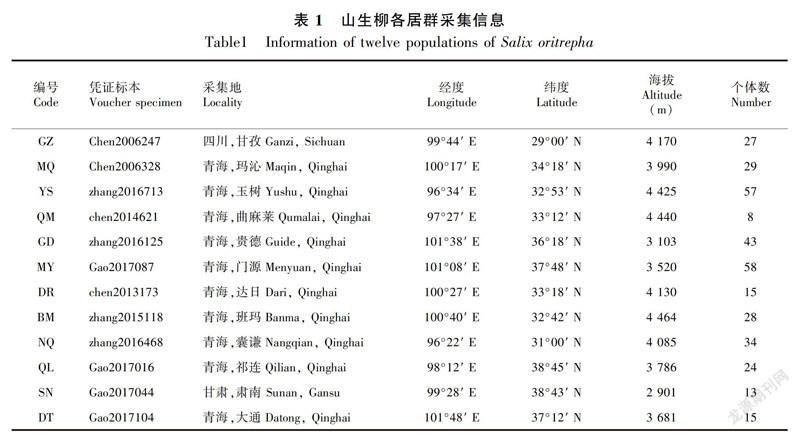
2006—2017年对分布于青藏高原地区的山生柳进行广泛的野外采集。对每个野外自然居群选取8~58个个体,个体间距超过100 m,共收集山生柳12个居群351个个体(表1)。采集山生柳生长良好的新鲜幼嫩叶片,迅速用硅胶进行干燥,之后放于-20 ℃ 长期保存。所采集的凭证标本存放于中国科学院西北高原生物研究所青藏高原生物标本馆(HNWP)中。
1.2 DNA提取与PCR扩增
选取改良的CTAB法从干燥处理的山生柳嫩叶中提取基因组DNA(Porebski et al., 1997)。参照序列选取已上传到NCBI上的Salix arbutifolia、S. babylonica、S. interior、S. oreinoma、S. purpurea、S. suchowensis和S. tetrasperma等柳属植物叶绿体基因组序列,引物片段的选取参考之前的研究(Hamilton, 1999; Scarcelli et al., 2011; Shaw et al., 2005),用软件Primer Premier 5.0(Singh et al., 1998)、BioXM 2.6(黄骥和张红生,2004)和SnapGene 3.2.1(McKeone et al., 2014)进行引物设计,共设计了21对引物用于PCR扩增。扩增反应体系为25 μL:2.5 μL的10×PCR Buffer(含1.5 mmol·L-1 MgCl2)、0.5 μL的10 mmol·L-1 dNTP、正反引物各0.5 μL(10 pmol·L-1)、1个单位的Taq DNA酶(TaKaRa,大连)、15~50 ng的模板DNA,去离子水补齐到25 μL。PCR反应扩增程序:94 ℃预变性3 min;94 ℃变性45 s;60 ℃退火45 s;72 ℃延伸30 s;35个循环;72 ℃延伸5 min;4 ℃保存。PCR反应产物送至生物工程(上海)股份有限公司测序。将获得21对引物在12个居群24个个体中进行PCR扩增预试验,筛选出获得两对遗传多态性较高的引物 [5′trnG2G-3′trnG(UUC)和5′rpS12-rpL20]用于所有个体的扩增测序(表2)。
1.3 数据分析
运用Chromas软件(http://technelysium.com.au/wp/chromas/)和MEGA 7.0.26(Kumar et al., 2018)对所有测序序列进行人工校对,并用MEGA软件对校对好的所有个体的扩增片段进行对位排列。利用DnaSP 6.10.04(Rozas et al., 2017)软件确定山生柳单倍型,并统计中性检验中的Tajima’s D和Fu’s Fs。运用SPSS(George & Mallery, 2003)软件获得基于遗传距离和单倍型分布的UPGMA聚类分析图。利用Arlequin软件包(Excoffier & Lischer, 2010)中的分子变异分析方法(analysis of molecular variance, AMOVA)检测居群内和居群间的遗传变异水平,并对单倍型分布的FST进行评价(1 000次重复置换检测)。根据基因流(Nm)与遗传分化系数(FST)之间的关系计算Nm值和居群平均等位基因数。失配分布(mismatch distribution)分析也在Arlequin中完成。通过PERMUT软件计算居群内平均遗传多样性(average gene diversity within populations,hS)、总遗传多样性(total gene diversity,hT)、居群间遗传分化系数(GST)和(NST),并用U-统计方法对GST和NST进行比较(1 000次重复置换检测)以检测山生柳单倍型的分布特点(Caraux & Pinloche, 2005)。利用TCS软件(Clement et al., 2000)来建立山生柳单倍型之间的TCS简约网络关系图。
2结果与分析
2.1 山生柳遗传多样性分析
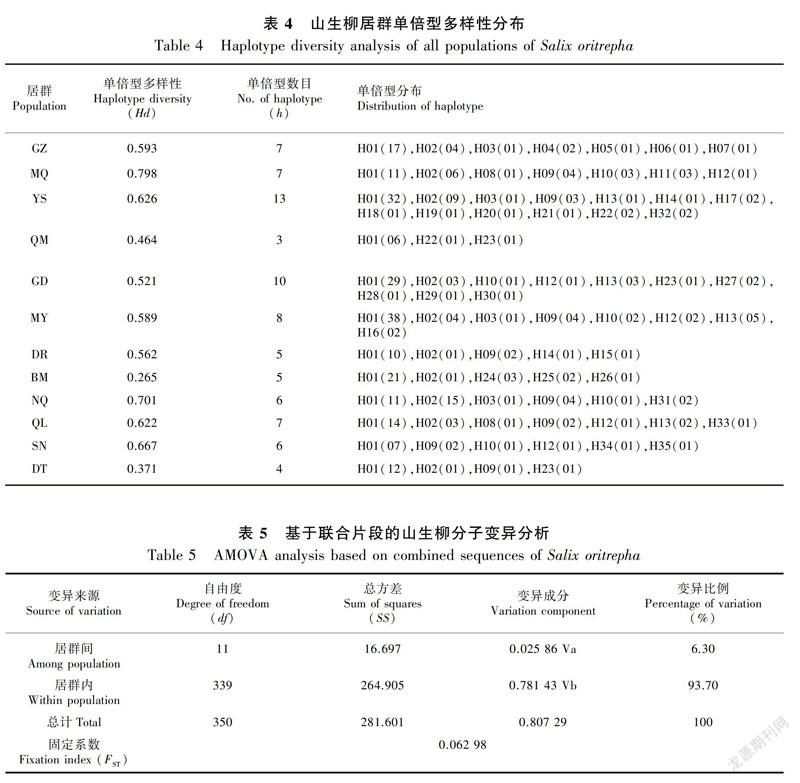
对351个个体的扩增产物进行单向测序,利用MEGA软件对位排列且删除poly结构后,5′trnG2G-3′trnG(UUC)和5′rpS12-rpL20片段矩阵长度分别为481 bp、730 bp,共检测到36个变异位点,其中17个为简约性信息位点,变异率为2.97%。从山生柳的联合片段中检测出35个单倍型(Haplotype, H01~H35),将5′trnG2G-3′trnG(UUC)片段的单倍型序列和5′rpS12-rpL20片段的单倍型序列提交至GeneBank(MK602662~MK602696和MK606373~MK606407)。联合片段的单倍型多样性为0.626 1,单倍型多样性标准差为0.029(表3)。各居群联合片段之间的多态性表现不同,居群的平均等位基因數(Na)依次为1.067、1.078、1.278、1.022、1.656、1.089、1.067、1.100、1.056、1.078、1.089、1.033、1.134。遗传多样性最丰富的是居群YS,其具有最高的核苷酸多样性(0.001 45)、平均核苷酸差异(1.722)和最多的单倍型数目(13)(表4)。
由各居群的单倍型分布可知,仅H01被全部12个居群所共享;H04、H05、H06和H07是GZ所拥有的特有单倍型;H11是MQ所拥有的特有单倍型;H17、H18、H19、H20、H21和H32是YS所拥有的特有单倍型;H27、H28、H29和H30是GD所拥有的特有单倍型;H16是MY所拥有的特有单倍型;H15是DR所拥有的特有单倍型;H24、H25和H26是BM所拥有的特有单倍型;H31是NQ所拥有的特有单倍型;H33是QL所拥有的特有单倍型;H34和H35是SN所拥有的特有单倍型;H02,H03,H08,H09,H10,H12,H13,H14,H16,H17,H22,H23是2~10个居群所共有的单倍型(图1,表4)。
2.2 山生柳遗传结构分析
分子变异分析(AMOVA)结果表明,居群间遗传变异为6.30%,居群内遗传变异为93.70%(P<0.01),说明居群内遗传变异为山生柳遗传变异的主要来源(表5)。遗传分化系数FST=0.062 98,居群间遗传分化很小。根据基因流Nm与FST之间的关系(Kirk & Freeland, 2011),Nm =(1-FST)/2FST,得到Nm为7.439, 基因流很强。用PERMUT和DnaSP软件计算得出山生柳居群内平均遗传多样山生柳各居群间的遗传距离大部分都较小,其中QM和NQ之间出现了最大遗传距离0.977,YS和SN出现了最小遗传距离0.000,大多数居群之间的遗传距离差异性不大,总的平均遗传距离为0.424;居群间的遗传分化指数(FST)显示有18个负值,占27.28%。其中QM和NQ之间出现了最大的FST=0.255 0(表6)。
山生柳12个居群的遗传距离的范围为0.000~1.234,表明山生柳居群内遗传多样性丰富。基于遗传距离的UPGMA聚类分析,得到山生柳的12个居群分为I(YS, SN, MY, DT, QL, BM)、Ⅱ(GZ, MQ, DR)、Ⅲ(GD, NQ)和IV(QM)共四组(图2)。基于单倍型的UPGMA聚类分析,得到12个居群分为I(YS, GD, MY)、II(NQ)、III(BM, GZ)和IV(QM, SN, QM, QL, DT, DR)共四组(图3)。两者均没有表现出与12个居群的地理分布相对应的聚类关系。
2.3 山生柳种群动态
中性检验结果表明,山生柳各个引物片段和联合片段的Tajima’s D(-2.286 70, P<0.01)和Fu’s Fs(-5.298 05, P<0.02)均为显著的负值(表3)。失配分析结果显示,在山生柳分布范围内其分布曲线整体呈现单峰分布, 其SSD(sum of square devia-tions)值 (0.003 8, P=0.68)和HRag (Harpending’s raggedness index)值(0.047 9, P=0.72)均不显著(P>0.05),这与中性检验的结果相一致(图4)。
用山生柳叶绿体基因所检测出的35个单倍型,建立单倍型多样性的简约网络图(图5)。回路结构是趋同演化的结果,矩形框代表祖先(或根部)单倍型即H01(Saltonstall, 2002)。图5中略去了单倍型的频率数值。图5中“o”表示理论分析出的,但没有实际观测到的单倍型。除YS(H32)和DG(H27,H29)的单倍型同其它单倍型关系较远外,大部分单倍型之间仅有一到两步突变,演化关系呈现星状辐射结构(杨玉慧等,2004)。
3讨论
3.1 山生柳的居群遗传多样性
筛选出的山生柳特异性条带为山生柳无性系鉴定提供可操作性。山生柳的联合片段共检测到36个变异位点和17个简约性的信息位点,插入缺失变异位点19个,占总变异的52.78%。插入缺失是山生柳变异位点出现的主要来源,与DNA在进行复制时存在高频率的点突变有关。在残酷的高山图 4基于山生柳单倍型的失配分析山地冰雪寒温带生存环境中,山生柳的DNA复制发生碱基错配的可能性更大,机体的自我修复功能会受到一定阻碍,说明恶劣的生存环境可能在一定程度上影响DNA复制过程(Baccarelli & Bollati, 2009; Chinnusamy & Zhu, 2009; Dubrova, 2003)。
对山生柳居群的联合片段进行数据分析,检测到35个单倍型。数据分析的结果总体呈现出较低的遗传多样性(Hd=0.626 1, Pi=0.000 85)。山生柳每个居群间的单倍型多样性(Hd)表现不同,推测可能是由于在青藏高原地区的山生柳分布范围广,各居群所在的自然生存环境条件具有差异,经过长期不同的生态环境和自然选择,会导致具有一定地理隔离的山生柳居群存在差异的遗传多样性。根据之前对山生柳在祁连山上不同海拔种群的遗传多样性水平的研究结论(郭敏等,2012),推测不同的地理位置、生存环境和海拔高度都体现山生柳不显著的分子水平的差异性,但是差异性不会太大。
3.2 山生柳的居群遗传结构
AMOVA分子变异结果表明,山生柳居群内的遗传变异占总变异的93.70%(P<0.01),居群间的遗传变异仅为6.30%,揭示了山生柳居群内的遗传变异比居群间的遗传变异要大,居群内的遗传变异是山生柳遗传变异的主要来源。推测的原因:(1)35个单倍型中仅有H01为12个居群都拥有的共享单倍型,共有208个个体拥有,所占比例为59.26%,由于H01在居群中是无规律分布,因此山生柳居群间的遗传差异并不明显;(2)35个单倍型中有22个单倍型是各个居群所独有,甚至某一居群会同时固定多个特有单倍型,这就说明山生柳的某些居群内部会有可辨别的遗传变异。特有单倍型比例较高的物种往往不能揭示出明显的谱系地理格局(Geng et al., 2018)。繁育系统和遗传变异分布之间存在着密切的联系(Hogbin & Peakall, 1999),即异交物种的遗传变异大多分布在群体之内,群体间的遗传变异通常占27%以下(赵景奎等,2008),与本研究结果相符。
植物居群的遗传结构受群体进化历史、基因流等影响,群体进化历史一般通过遗传分化指数(FST)来反映,其大小可在一定程度上揭示种群间基因流和遗传漂变的程度(杨艳婷等,2018)。一般来讲,当FST∈[0,0.05),群体间没有遗传分化;当FST∈[0.05,0.15),群体分化程度中等;当FST∈[0.15,0.25),群体间高度分化;FST∈[0.25,1],群体间分化程度非常高(Wright,1950,1970)。山生柳自然居群的遗传分化指数(FST)为0.062 98,表明居群分化程度为中等。山生柳各居群间的遗传分化指数(FST)有19个负值,占28.79%(正常情况下FST∈[0,1]),表明少部分居群间没有分化,但总体上反映出的是大部分居群间出现非常普遍的基因交流(Wright, 1950, 1970, 1982),其中QM和NQ之间的FST值最大为0.255 0。山生柳一般为有性生殖,花粉量大随风飘散,但在严酷的生存环境下会进行无性繁殖,本研究所选的山生柳材料地理分布较广、地理环境高度复杂,导致居群间遗传变异的差异性较大,有的居群几乎无遗传分化(QM, DT),有的居群分化程度非常高(QL),但总体还是表现为分化程度中等偏低。
根据基因流公式,算得Nm=7.438,当Nm=7.438>1,属于较高水平(Wright,1982),表明不同的居群之间存在频繁的基因流动,增大群体间遗传差异的表现一般为自然选择以及遗传漂变,而基因流可以“弱化”群体之间不易察觉的遗传差异使居群间趋向统一。
对山生柳单倍型变异的地理结构检验结果为GST值(0.068)和NST值(0.075)(NST>GST,P>0.05),表明山生柳不同居群间出现了大量的亲缘关系靠近的单倍型,可以证明山生柳不存在明显的谱系地理学的关系。基于遗传距离和单倍型的UPGMA聚类分析,得到山生柳的12个居群各分为四组且与12个居群地理分布没有明显相关性,进一步验证了山生柳居群不具有谱系地理结构。
3.3 山生柳的种群扩张
基因流分析结果证实山生柳的12个居群存在频繁的基因交流,山生柳居群间不存在明显的基因流限制。通过对所有地理居群单倍型进行失配分析,所有的山生柳单倍型呈现只有一个明显顶峰的单峰型曲线,表示不能拒绝居群扩张的假说。中性检验的结果表明,Tajima’s D (-2.286 70, P<0.01)和Fu’s Fs(-5.298 05, P<0.02)均为显著的负值,这与失配分析的单峰型曲线结果相一致。TCS网络图显示H01单倍型为祖先单倍型,由于H01为种群的共享单倍型,故不能确定山生柳种群的扩散中心。TCS网络图呈星状辐射分布,且新衍生出的单倍型与原始单倍型组成一个“星状”辐射结构,说明该群体曾经在遇到瓶颈后迅速扩增(Arbogast, 2000)。通过单倍型多样性与核苷酸多样性分析得到单倍型多样性高(Hd>0.5)而核苷酸多样性低(Pi<0.005),預示着群体曾经历过瓶颈效应后,伴随了迅速的种群扩张与变异的积累。综上所述,推测山生柳在其分布范围或者其个体数近期经历过扩张。
对青藏高原地区相同生境的高山绣线菊(Khan et al., 2014)、金露梅(Ma et al., 2014)、沙棘(Bartish et al., 2006)、窄叶鲜卑花(Duan et al., 2011)等灌木的种群动态研究表明其遗传分组曾经历过快速的种群扩张事件。这与山生柳的种群扩张结果一致,推测物种生境和繁育方式的相似性会出现物种为了营养、繁殖、保护、扩散以及对环境的适应出现协同进化的现象(Morgan et al., 2005; Thompson & Burdon, 1992)。有研究表明山生柳种群可能以有性和无性两种繁殖方式进行更新以巩固其优势地位(李毅等,2002),年龄结构分布图呈正三角形,种群动态表现为增长型(李毅等,2002),这与本研究中居群近期出现过扩张相吻合。花粉和种子的扩散是自然植物种群最主要的基因流方式(李海生和陈桂珠,2004),故山生柳生殖方式的多样化会导致其基因交流频繁,预期未来山生柳居群会处于进一步扩张状态。
山生柳频繁的基因交流和经历过近期扩张,猜测此结果可能的原因如下:(1)cpDNA是母系遗传,估算的基因流大小与花粉传播无关,与其多样化的繁育方式有关;(2)山生柳一般以有性生殖为主,其生存环境是长期积雪(低温)、干燥风大等气象特点,使其种子可以随处传播;(3)由于种子的传播距离远,故青藏高原地区和横断山脉错综复杂地形也只能在很小的程度上阻隔山生柳居群间的基因交流;(4)在严酷环境压力下,山生柳种子活力极低,幼苗补充量受限,从而有性生殖受限,此时主要依托无性繁殖来巩固其物种数量的稳定(陈文业等,2008)。
参考文献:
ARBOGAST BS, 2000. Phylogeography: The history and formation of species [J]. Integr Comp Biol, 41(1): 134-135.
BACCARELLI A, BOLLATI V, 2009. Epigenetics and environmental chemicals [J]. Curr Opin Pediatr, 21(2): 243-251.
BARTISH IV, KADEREIT JW, COMES HP, 2006. Late Quaternary history of Hippophae rhamnoides L. (Elaeagnaceae) inferred from chalcone synthase intron (Chsi) sequences and chloroplast DNA variation [J]. Mol Ecol, 15(13): 4065-4083.
CARAUX G, PINLOCHE S, 2005. PermutMatrix: A graphical environment to arrange gene expression profiles in optimal linear order [J]. Bioinformatics, 21(1): 1280-1281.
CHEN WY, CHEN YX, ZHENG HP, et al., 2008. Ecological habits and seedling raising techniques of Salix oritrepha [J]. Prot For Sci Technol, (3): 135-137. [陈文业, 陈永霞, 郑华平, 等, 2008. 山生柳的生态习性与育苗技术 [J]. 防护林科技, (3):135-137.]
CHINNUSAMY V, ZHU JK, 2009. Epigenetic regulation of stress responses in plants [J]. Curr Opin Plant Biol, 12(2): 133-139.
CIRES E, SAMAIN MS, GOETGHEBEUR P, et al., 2011. Genetic structure in peripheral Western European populations of the endangered species Cochlearia pyrenaica (Brassicaceae) [J]. Plant Syst Evol, 297(1-2): 75-85.
CLEMENT M, POSADA D, CRANDALL KA, 2000. TCS: A computer program to estimate gene genealogies [J]. Mol Ecol, 9(10): 1657-1659.
DEMESURE B, COMPS B, PETIT RJ, 1996. Chloroplast DNA phylogeography of the common beech (Fagus sylvatica L.) in Europe [J]. Evolution, 50(6): 2515.
DUAN YZ, GAO QB, ZHANG FQ, et al., 2011. Phylogeographic analysis of the endemic species Sibiraea angustata reveals a marginal refugium in the Qinghai-Tibet Plateau [J]. Nord J Bot, 29(5): 615-624.
DUBROVA YE, 2003. Radiation-induced transgenerational instability [J]. Oncogene, 22(45): 7087-7093.
EXCOFFIER L, LISCHER H, 2010. Arlequin suite ver 3.5: A new series of programs to perform population genetics analyses under Linux and Windows [J]. Mol Ecol Resour, 10(3): 564-567.
FENG SL, SU C, DUAN SY, et al., 2001. Chemical constituents from Salix oritrepha [J]. Chin J Chin Mat Med, (9): 31-32. [封士蘭, 苏策, 段生玉, 等, 2001. 藏药山生柳的化学成分研究 [J]. 中国中药杂志, (9):31-32.]
GENG JZM, LI Y, JIA LK, et al., 2018. Phylogeography of Saxifraga tangutica Engl. (Saxifragaceae) [J]. Acta Bot Boreal-Occident Sin, 38(2): 370-380. [更吉卓玛, 李彦, 贾留坤, 等, 2018. 唐古特虎耳草谱系地理学研究 [J]. 西北植物学报, 38(2):370-380.]
GEORGE D, MALLERY P, 2003. SPSS for Windows step by step: A simple guide and reference, 16.0 update [J]. Comp Softw, (100): 357.
GUO M, MA YJ, LI Y, 2012. Analysis of genetic diversity in Salix oritrepha from different altitudes in Gansu Qilian mountains using SSR markers [J]. Acta Pratac Sin, 21(5): 114-121. [郭敏, 马彦军, 李毅, 2012. 祁连山不同海拔梯度山生柳遗传多样性的SSR分析 [J]. 草业学报, 21(5):114-121.]
HAMILTON MB, 1999. Four primer pairs for the amplification of chloroplast intergenic regions with intraspecific variation [J]. Mol Ecol, 8(3): 521-523.
HOGBIN PM, PEAKALL R, 1999. Evaluation of the contribution of genetic research to the management of the endangered plant Zieria prostrata [J]. Conserv Biol, 13(3): 514-522.
HUANG Y, ZHANG HS, 2004. Development of nucleotide sequence analysis software based on Windows [J]. Chin J Bioinform, 2(1): 13-17. [黄骥, 张红生, 2004. 基于Windows的核酸序列分析软件的开发 [J]. 生物信息学, 2(1):13-17.]
KANG JJ, ZHAO WZ, ZHAO M, et al., 2015. The cutting reproduction technique of Salix oritrepha and its application on degraded grasslands restoration [J]. J Soil Water Conserv, 70(1): 45-53.
KHAN G, ZHANG FQ, GAO QB, et al., 2014. Molecular phylogeography and intraspecific divergence of Spiraea alpina (Rosaceae) distributed in the Qinghai-Tibetan Plateau and adjacent regions inferred from nrDNA [J]. Biochem Syst Ecol, 57: 278-286.
KIRK H, FREELAND J R, 2011. Applications and implications of neutral versus non-neutral markers in molecular ecology [J]. Int J Mol Sci, 12(6): 3966-3988.
KUMAR S, STECHER G, LI M, et al., 2018. MEGA X: Molecular evolutionary genetics analysis across computing platforms [J]. Mol Biol Evol, 35(6): 1547-1549.
LI HS, CHEN GZ, 2004. Genetic diversity of mangrove plant Sonneratia caseolaris in Hainan Island based on ISSR analysis [J]. Acta Ecol Sin, 24(8): 1657-1663. [李海生, 陳桂珠, 2004. 海南岛红树植物海桑遗传多样性的ISSR分析 [J]. 生态学报, 24(8):1657-1663.]
LI Y, HU ZZ, WANG ZT, 2002. Populations distribution patterns of alpine Salix Oritrepha in East Qilian Mountains [J]. Acta Pratac Sin, (3): 48-54. [李毅, 胡自治, 王志泰, 2002. 东祁连山高寒地区山生柳种群分布格局研究 [J]. 草业学报, (3):48-54.]
MA YZ, LI ZH, WANG X, et al., 2014. Phylogeography of the genus Dasiphora (Rosaceae) in the Qinghai-Tibetan Plateau: Divergence blurred by expansion [J]. Biol J Linn Soc, 111(4): 777-788.
MCKEONE R, WIKSTROM M, KIEL C, et al., 2014. Assessing the correlation between mutant rhodopsin stability and the severity of retinitis pigmentosa [J]. Mol Vis, 20: 183-199.
MOGENSEN HL, 1996. Invited special paper: The hows and whys of cytoplasmic inheritance in seed plants [J]. Am J Bot, 83(3): 383-404.
MORGAN MT, WILSON WG, KNIGHT TM, 2005. Plant population dynamics, pollinator foraging, and the selection of self-fertilization [J]. Am Nat, 166(2): 169-183.
POREBSKI S, BAILEY LG, BAUM BR, 1997. Modification of a CTAB DNA extraction protocol for plants containing high polysaccharide and polyphenol components [J]. Plant Mol Biol Rep, 15(1): 8-15.
ROZAS J, FERRER-MATA A, SANCHEZ-DELBARRIO JC, et al., 2017. DnaSP 6: DNA sequence polymorphism analysis of large data sets [J]. Mol Biol Evol, 34(12): 3299-3302.
RUNGIS D, LAIVINS M, GAILITE A, et al., 2017. Genetic analysis of Latvian Salix alba L. and hybrid populations using nuclear and chloroplast DNA markers [J]. iForest, 10(2): 422-429.
SALTONSTALL K, 2002. Cryptic invasion by a non-native genotype of the common reed, Phragmites australis, into North America [J]. P Natl Acad Sci USA, 99(4): 2445-2449.
SCARCELLI N, BARNAUD A, EISERHARDT W, et al., 2011. A set of 100 chloroplast DNA primer pairs to study population genetics and phylogeny in monocotyledons [J]. PLoS ONE, 6(5): e19954.
SHAW J, LICKEY EB, BECK JT, et al., 2005. The tortoise and the hare II: Relative utility of 21 noncoding chloroplast DNA sequences for phylogenetic analysis [J]. Am J Bot, 92(1): 142.
SHAW J, SHAFER HL, LEONARD OR, et al., 2014. Chloroplast DNA sequence utility for the lowest phylogenetic and phylogeographic inferences in angiosperms: The tortoise and the hare IV [J]. Am J Bot, 101(11): 1987-2004.
SINGH VK, MANGALAM AK, DWIVEDI S, et al., 1998. Primer premier: Program for design of degenerate primers from a protein sequence [J]. Biotechniques, 24(2): 318-319.
THOMPSON JN, BURDON JJ, 1992. Gene-for-gene coevolution between plants and parasites [J]. Nature, 360(6400): 121-125.
WANG F, CHEN WY, CAI RZM, et al., 2010. Gray system theory based analysis of hard branch cuttage of Salix oritrepha in alpine area [J]. Pratac Sci, (9): 86-90. [王芳, 陈文业, 才让卓玛, 等, 2010. 高寒区山生柳硬枝扦插灰色系统理论分析研究 [J]. 草业科学, (9):86-90.]
WANG W, WANG S, 2015. Analysis of plant community diversity of Salix oritrepha shrub meadow in Maqu County [J]. For Gansu, (6): 28-30. [王玮, 王盛, 2015. 玛曲县山生柳灌丛草甸植物群落多样性分析 [J]. 甘肅林业, (6):28-30.]
WRIGHT S, 1950. Genetic structure of populations [J]. Brit Med J, 2(4669): 36.
WRIGHT S, 1970. A Second Installment. (Book Reviews: Evolution and the genetics of populations. The theory of gene frequencies) [J]. J Biosoc Sci, 168(2): 722-723.
WRIGHT S, 1982. The shifting balance theory and macroevolution [J]. Ann Rev Genet, 16(1): 1-20.
WU ZQ, 2016. The whole chloroplast genome of shrub willows (Salix suchowensis) [J]. Mitochondrial DNA A, 27(3): 1-2.
YANG LC, ZHONG ZB, LIU HC, et al., 2013. Community characteristics of alpine shrub Salix oritrepha [J]. Bull Bot Res, 33(6): 653-658. [杨路存, 钟泽兵, 刘何春, 等, 2013. 高寒山生柳灌丛的群落特征研究 [J]. 植物研究, 33(6):653-658.]
YANG YH, ZHANG DX, LI YM, et al., 2004. Mitochondrial DNA diversity and preliminary biogeographic inference of the evolutionary history of the black-spotted pond frog Rana nigromaculata populations in China [J]. Acta Zool Sin, 50(2): 193-201. [杨玉慧, 张德兴, 李义明, 等, 2004. 中国黑斑蛙种群的线粒体DNA多样性和生物地理演化过程的初探 [J]. 动物学报, 50(2):193-201.]
YANG YT, HOU XY, WEI ZW, et al., 2018. Screening and genetic diversity analysis of chloroplast non-coding region in Leymus chinensis [J]. Acta Pratac Sin, 27(10): 147-157. [楊艳婷, 侯向阳, 魏臻武, 等, 2018. 羊草叶绿体非编码区多态性标记筛选及群体遗传多样性 [J]. 草业学报, 27(10):147-157.]
ZHAO JK, XU LA, JIE HF, et al., 2008. RAPD analysis of population genetic diversity of Tamarix chinensis in Yellow River delta [J]. J Nanjing For Univ (Nat Sci Ed), 32(5): 56-60. [赵景奎, 徐立安, 解荷峰, 等, 2008. 黄河三角洲柽柳群体遗传多样性RAPD分析 [J]. 南京林业大学学报(自然科学版), 32(5):56-60.]
