基于卷积神经网络的超声图像左心室分割方法
2019-09-04朱锴付忠良陈晓清
朱锴 付忠良 陈晓清


摘 要:超声图像左心室的分割在临床上对医生的作用巨大。由于超声图像含有大量噪声,轮廓特征不明显,目前的卷积神经网络(CNN)方法对左心室分割容易得到不必要的区域,并且分割目标不完整。为了解决上述问题,在全卷积神经网络(FCNFCN的缩写对吗?不是FCNN吗?请明确。)基础上加入了关键点定位和求取图像凸包方法对分割结果进行优化。首先采用FCN获取初步的分割结果;然后为了去除分割结果中的错误区域,提出一种CNN定位左心室三个关键点的位置,通过关键点筛选掉分割结果中不必要的区域;最后为保证剩余区域能够组合成一个完整的心室,利用求取图像凸包算法将所有有效区域进行合并。实验结果表明,在超声图像左心室分割效果上,所提方法能够在普通FCN的基础上获得很大的提升,在交并比评价标准下,该方法获取的左心室结果能够比传统CNN方法提升近15%。
Abstract:Ultrasound image segmentation of left ventricle is very important for doctors in clinical practice. As the ultrasound images contain a lot of noise and the contour features are not obvious, current Convolutional Neural Network (CNN) method is easy to obtain unnecessary regions in left ventricular segmentation, and the segmentation regions are incomplete. In order to solve these problems, keypoint location and image convex hull method were used to optimize segmentation results based on Fully Convolutional neural Network (FCN). Firstly, FCN was used to obtain preliminary segmentation results. Then, in order to remove erroneous regions in segmentation results, a CNN was proposed to locate three keypoints of left ventricle, by which erroneous regions were filtered out. Finally, in order to ensure that the remained area were able to be a complete ventricle, image convex hull algorithm was used to merge all the effective areas together. The experimental results show that the proposed method can greatly improve left ventricular segmentation results of ultrasound images based on FCN. Under the evaluation standard, the accuracy of results obtained by this method can be increased by nearly 15% compared with traditional CNN method.
Key words: ultrasound image; segmentation; keypoint location; Convolutional Neural Network (CNN); convex hull
0 引言
心臟超声图像是临床医学上评估患者心脏状况的主要手段之一,其中左心室的大小是判断心脏是否正常的重要生理参数。一般来说医生通过在超声图像上手动描绘左心室轮廓来计算左心室容积大小,由于手动描绘轮廓很繁琐,在术中十分不便,因此设计一种全自动的分割算法能给医生带来极大的便利,但目前由于超声图像存在如下几个分割难点:一是相对于其他医学图像,超声图像中存在巨大的斑点噪声,为图像分割带来了很大的干扰;二是每位患者的生理结构虽然大体类似,但是个体差异带来的生理结构不规则变化为分割增加了挑战。
图像分割技术在医学图像中应用很广泛,通过对关键区域的分割提取,能够获取病人重要生理结构的形状、大小等信息。传统医学图像分割方案通常先对图像进行预处理,后采用算法对目标区域分割。传统医学图像分割方法通常分为利用单幅图像本身特征进行的分割方法和采用带标注数据集训练的方法。通常两种方法均需要手工提取图像局部特征来对像素进行聚类或分类。在前一类方法中,有学者采用基于更为快速的基于区域的卷积神经网络(Faster Region-based Convolutional Neural Network, Faster RCNN)结合均值漂移算法的方法对超声图像左心室进行提取[1],也有学者采用规范化切割结合K均值算法对乳房病变区域进行定位[2],还有学者采用局部高斯分布拟合能量模型对超声图像进行定位[3]。此类算法虽然简单,不需要依赖其他图像便可完成,但由于算法仅仅从单一图像提取特征进行分割,图像鲁棒性较差,对图像质量要求较高。后一类方法中,Jiang等[4]采用Haar特征结合AdaBoost方法对乳房超声图像进行分割;Ngo等[5]采用水平集方法对心脏核磁共振图像左心室进行分割;也有学者利用关键点采用基于活动轮廓模型的方法对目标区域进行定位[6-7]。此类方法较为依赖手工特征的选取,并且基于轮廓的方法需要有较好的初始位置选取。
近年来,由于深度学习具有不需要手动设计特征等优势,该方法在图像分类上的成功也带动了将其在图像分割的运用。2014年Long等[8]首次将深度卷积神经网络(Convolutional Neural Network, CNN)运用于图像分割上,提出了一种全卷积的神经网络(Fully Convolutional neural Network, FCN),该方法对图像中每个像素进行类别预测实现像素级别的语义分割,并取得了很好的效果,后有大量学者对FCN进行改进,并在自然图像上得到了有效的验证[9-10]。随着深度学习在自然图像上的成功,有研究者改进FCN提出了U-Net补充U-Net全称。回复无全称对生物医学图像进行分割,尽可能地保留了图像的细节[11],也有学者将FCN结合条件随机场对大脑电子计算机断层扫描图像进行分割,能够有效地对FCN粗糙的分割进行补充[12]:Li等[13]提出了一种基于动态CNN的方案对左心室轮廓进行分割;詹曙等[14]采用反卷积神经网络分割核磁共振图像,大幅减小了分割时间;Jafari等[15]使用U-Net结合长短期记忆网络对超声心动图左心室进行分割。但上述深度学习算法在运用于心室分割中有如下问题:不同于其他医学图像,超声图像很多左心室边界模糊,单纯采用卷积神经网络获取的结果很可能包含很多部分,而不是单一的左心室腔体,目前的区域筛选办法主要是采用普通的阈值将较小的区域筛选掉,并没有有效的筛选方法能选取想要进行合并的区域。
为解决上述问题,本文提出了一种基于FCN的超声图像左心室分割方案,算法结合关键点定位对多余的区域进行筛选,并对余下区域采用图像求凸包算法进行合并,最终得到左心室腔体。实验表明,本文算法能够有效地对超声图像左心室进行提取,与传统的FCN算法相比,在评价标准上获得更高的精度。
1 基于FCN的图像分割算法
基于FCN的图像分割算法是Long等[8]提出的,算法去除了普通CNN的全连接层,改用全卷积的网络结构对图像每个像素进行分类,达到了像素级别的分割效果。算法大体框架如图1,网络结构将是根据VGG16[16]改进的,首先将VGG(Visual Geometry Group)请补充VGG的英文全称的全连接层改为卷积核大小为1×1的卷积层,将全连接层改为了图中conv6和conv7。图像首先经过若干卷积层和池化层进行降采样,其中卷积层后采用线性整流函数(Rectified Linear Unit, ReLU)为激活函数,池化层采用最大池化,后采用反卷积操作将图像特征图映射回原图大小,并对每个像素类别进行预测,算法采用softmax损失函数。
本文采用全卷积网络模型实现经食道超声四腔心切面左心室的初步分割,本文选取上采样8倍的FCN对左心室分割,将图像大小缩放为3×256×256作为输入。
2 利用关键点辅助FCN的左心室提取
2.1 左心室关键点定位
由于采用FCN网络得到的分割结果对于某些心室超声图像会包含多个区域结果,为对这些区域是否为心室区域进行筛选,本文提出了利用关键点定位对区域筛选的方法。在左心室结构中,包含心尖、二尖瓣左右根部三个重要关键点的位置,为对这三个关键点的位置作粗略估计,本文提出利用卷积神经网络回归的方法对关键点的位置进行定位。
如图2所示,本文采用包含8个卷积层和2个全连接层的网络进行关键点定位,输入图像大小为256像素×256像素,经过一系列卷积层和池化层的采样之后,利用2个全连接层输出3个关键点坐标。由于本文定位关键点的位置是辅助图像分割的,并不需要十分精确的关键位置也能够达到辅助分割的效果。采用的损失函数为欧氏损失函数,如式(1):
其中:E代表损失,N为样本数目,为预测值,y为实际值。
2.2 分割区域筛选及合并
由于超声图像轮廓不清晰,实验数据量有限,在实验过程中利用FCN算法得到的结果可能包含的不是同一个区域,而是图像中很多区域。为对这些区域进行筛选和合并,本文采用关键点定位对区域进行筛选,后采用图像求凸算法对区域进行合并。图3展示了区域筛选的实例,假设图3(a)为FCN分割得到的结果,图3(b)为左心室标签和3个关键的位置,希望保留图(a)的区域2和区域3,去除掉区域1,传统的区域筛选算法仅仅通过区域大小设置阈值而删除;但若FCN选取结果某些区域较大而且是错误的,阈值去除区域办法就将得到一些错误的区域,并且在合并这些非连通区域时将得到更为糟糕的结果,为此本文提出的关键点定位算法能够有效解决这种问题,如图(b)所示,本文将定位的三个关键点所构成的三角区域视为筛选区域,筛选时将和三角区域无交集的区域去除,这样一来既能筛选掉不需要的大部分区域,还能保留离分割区域更为接近的区域。即使关键点定位无法达到精准的情况下,大部分无关区域也能够被剔除。
对于筛选过的区域,本文根据求凸算法[17]将剩下相关区域进行合并,得到最终分割结果。如图4所示2个区域合并的流程如下:
1)寻找待合并区域所有边界点,并确定所有关键点纵坐标最小的P。
2)计算其余各点和P构成直线与x轴正向的幅角,并将所有点按幅角大小α排序,α相同的离P近的排在前,即各点相对P按逆时针方向排序,则P后第一个点Ri+1位于凸包上,并将P、Ri+1入栈,按排序将Ri+1后Ri+2入栈,Ri+2为栈顶元素。
3)计算当前栈顶元素Ri+2和排序在栈顶元素的后一元素Ri+3构成射线与Ri+2前的栈内元素构成射线位置关系,若为顺时针旋转,则Ri+2出栈。
4)将Ri+3压栈并视为栈顶元素,重复3)的操作到所有边界点完成,栈内元素即为最后图像边界点。
3 实验及分析
3.1 实验数据
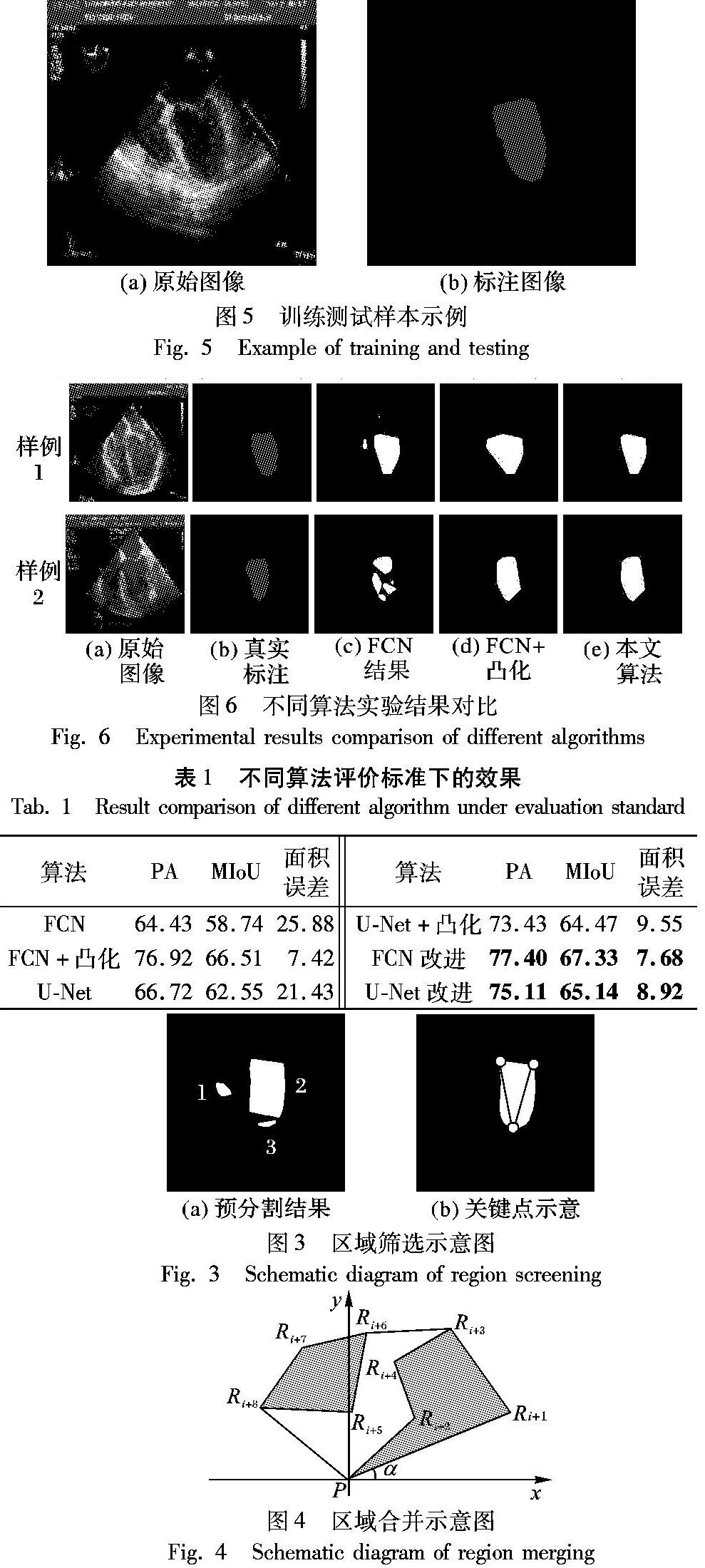
实验数据采集自四川省某医院麻醉科,共获取四腔心切面图像533张,其中248张图像用于训练集制作,285张图像用于測试集制作。所有图像处理为256像素×256像素此句不通顺,请作相应调整。。训练集制作过程中除了将左心室轮廓制作标签,还将左心室心尖及两个二尖瓣跟三个关键点位置标注出。如图5展示了训练集及标签样例。
3.2 评价标准
本文主要采用了像素精度(Pixel Accuracy, PA)和像素交并比(Mean Intersection over Union, MIoU)对分割准确度进行度量,像素精度表达如式(2)所示:
其中:PT代表预测正确的像素数目,PS代表总像素数目。
像素交并比精度如式(3)所示:
其中:PT代表预测正确的像素数目,PTF将正确标记预测为背景的像素数目,PFT代表将背景预测为心室的像素数目。
由于预测类别只有一类,本文在评价标准中并未将背景加入计算,因为背景面积较大,评价效果若将背景加入不能体现分割效果。
此外,由于左心室计算规则和预测区域面积有关,本文还加入了预测区域和真实区域像素数目误差对比,如式(4)所示:
其中PP代表预测左心室像素数目。
3.3 实验及结果
实验于Ubantu系统上进行,代码采用基于Caffe框架实现。在第一步训练FCN网络过程中,本文选用VGG16在自然图像分类结果的模型初始化网络参数,然后对网络参数进行训练微调,学习率固定为10-14,训练轮次105,网络采用带动量的优化方法。在训练关键点网络中,由于网络参数数目较小,本文直接对网络进行训练,学习率为10-3,迭代轮次104。
本文分别对比了仅仅采用FCN的分割结果,利用关键点辅助去除无关区域的结果,以及最后合并区域后的结果。图6展示了基于FCN的几种不同算法带来的实验结果的样例。图中分别展示了原始图像、标注图像、仅仅采用FCN的分割结果、利用FCN和凸化的结果和本文采用FCN结合关键点筛选区域最后合并的结果。从图6中可以看出,仅仅采用FCN对目标区域分割容易造成区域分散,不是同一个区域,并且有的结果容易包含无关区域,而从样例1采用阈值筛选区域合并的结果和本文用关键点筛选区域带来的结果看,仅仅用阈值篩选掉小的无关区域不能保证筛选掉所有无关区域。本文算法带来的结果从视觉上更为接近真实标注的结果。
表1展示了不同算法在评价标准下的结果。从表中看出,只采用FCN得到的结果远低于本文采用关键点筛选区域和凸化带来的效果,而采用关键点筛选区域也比不采用关键点筛选区域带来的效果要好。在只采用FCN时计算区域面积带来的误差也十分巨大,而采用了凸化及筛选之后大幅度减小了区域面积误差。本文还对采用U-Net算法得到的结果进行对比,在只采用U-Net的情况下,检测精度比仅仅采用FCN网络精度高,但由于U-Net获取图像较为完整,利用关键点检测和凸化算法筛选合并区域后效果并不如改进FCN精度高。
表2展示了基于FCN网络的前提下采用本文关键点算法筛选区域和直接采用真实关键点标签来筛选区域带来的结果。从表中可以看出预测关键点位置和真实关键点位置筛选区域效果相差不大。
此外,还采用了对数据进行旋转、对比度变换的数据增广方式进行训练,测试集图像也采用扩充后的图像进行实验。由于FCN在分割过程中得到的结果仅仅是初步结果,因此,如表3所示,可以看到采用扩充后数据训练结果略微好于未采用数据扩充带来的结果。
表4展示了基于FCN网络下程序的运行时间,实验采用机器内存大小为16GB,CPU为Intel Core i5-6600,显卡为GTX Titan X。表中显示了处理每张图像所需平均耗时。从表4中看出,本文算法几乎不需要额外花费过多时间就能大幅度提高图像分割精度。
4 结语
本文提出一种超声图像左心室轮廓提取方案,该方法通过心室关键点定位去除掉FCN网络分割结果带来的冗余区域,后再通过图像凸化合并剩余区域的方法获取左心室。实验中证明,结合图像凸化算法能够将分割结果大幅度优化到更为接近真实结果;此外,关键点的定位筛选区域也能够将部分FCN错分的区域去除,免去了手动设置阈值无法保证每幅图像都能正确筛选的问题。本文提出的方案能够有效克服全卷积神经网络在超声图像上分割单一目标时的不足。
参考文献 (References)
[1] 朱锴,付忠良,陶攀,等.基于自适应均值漂移的超声心动图左心室分割方法[J].生物医学工程学杂志,2018,35(2):273-279.(ZHU K, FU Z, TAO P, et al. Left ventricle segmentation in echocardiography based on adaptive mean shift[J]. Journal of Biomedical Engineering, 2018, 35(2): 273-279.)
[2] SADEK I, ELAWADY M, STEFANOVSKI V. Automated breast lesion segmentation in ultrasound images[J]. ArXiv Preprint, 2016, 2016: 1609.08364.
[3] 朱永杰,邱天爽.基于改进LGDF模型的超声图像自动分割方法[J].大连理工大学学报,2016,56(1):28-34.(ZHU Y J, QIU T S. Automated segmentation method for ultrasound image based on improved LGDF model[J]. Journal of Dalian University of Technology, 2016, 56(1):28-34.)
[4] JIANG P, PENG J, ZHANG G, et al. Learning-based automatic breast tumor detection and segmentation in ultrasound images[C]// Proceedings of the 2012 IEEE International Symposium on Biomedical Imaging. Piscataway, NJ: IEEE, 2012:1587-1590.
[5] NGO T A, LU Z, CARNEIRO G. Combining deep learning and level set for the automated segmentation of the left ventricle of the heart from cardiac cine magnetic resonance[J]. Medical Image Analysis, 2017, 35: 159-171.
[6] 纪祥虎,高思聪,黄志标,等. 基于Centripetal Catmull-Rom曲线的经食道超声心动图左心室分割方法[J]. 四川大学学报(工程科学版),2016,48(5):84-90.(JI X H, GAO S C, HUANG Z B, et al. Left ventricle segmentation in transesophageal echocardiography based on Centripetal Catmull-Rom curve[J]. Journal of Sichuan University (Engineering Science Edition), 2016, 48(5): 84-90.)
[7] VARGAS-QUINTERO L, ESCALANTE-RAMREZ B, CAMARGO M L, et al. Left ventricle segmentation in fetal echocardiography using a multi-texture active appearance model based on the steered Hermite transform[J]. Computer Methods and Programs in Biomedicine, 2016, 137: 231-245.
[8] LONG J, SHELHAMER E, DARRELL T. Fully convolutional networks for semantic segmentation[J]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2017, 39(4): 640-651.
[9] ZHENG S, JAYASUMANA S, ROMERA-PAREDES B, et al. Conditional random fields as recurrent neural networks[C]// ICCV15: Proceedings of the 2015 IEEE International Conference on Computer Vision. Washington, DC: IEEE Computer Society, 2015:1529-1537.
[10] HE K, GKIOXARI G, DOLLAR P, et al. Mask R-CNN[C]// ICCV 2017: Proceedings of the 2017 IEEE International Conference on Computer Vision. Washington, DC: IEEE Computer Society, 2017: 2961-2969.
[11] RONNEBERGER O, FISCHER P, BROX T. U-Net: convolutional networks for biomedical image segmentation[C]// MICCAI 2015: Proceedings of the 2015 International Conference on Medical Image Computing and Computer-Assisted Intervention, LNCS 9351. Berlin: Springer, 2015: 234-241.
[12] GAO M, XU Z, LU L, et al. Segmentation label propagation using deep convolutional neural networks and dense conditional random field[C]// Proceedings of the 2016 IEEE International Symposium on Biomedical Imaging. Piscataway, NJ: IEEE, 2016: 1265-1268.
[13] LI Y, YI G, WANG Y, et al. Segmentation of fetal left ventricle in echocardiographic sequences based on dynamic convolutional neural networks[J]. IEEE Transactions on Biomedical Engineering, 2017, 64(8): 1886-1895.
[14] 詹曙,梁植程,謝栋栋.前列腺磁共振图像分割的反卷积神经网络方法[J].中国图象图形学报,2017,22(4):516-522.(ZHAN S, LIANG Z C, XIE D D. Deconvolutional neural network for prostate MRI segmentation[J]. Journal of Image and Graphics, 2017, 22(4): 516-522.)
[15] JAFARI M H, GIRGIS H, LIAO Z, et al. A unified framework integrating recurrent fully-convolutional networks and optical flow for segmentation of the left ventricle in echocardiography data[C]// Proceedings of the 2018 International Workshop on Deep Learning in Medical Image Analysis and Multimodal Learning for Clinical Decision Support. Berlin: Springer, 2018: 29-37.
[16] SIMONYAN K, ZISSERMAN A. Very deep convolutional networks for large-scale image recognition[J]. ArXiv Preprint, 2014, 2014: 1409.1556.
[17] GRAHAM R L. An efficient algorithm for determining the convex hull of a finite planar set[J]. Information Processing Letters, 1978, 7(1): 53-55.
