大肠杆菌持留菌株与抗药菌株生长优势的比较
2019-09-03胡明付晓杰张庆齐静骆延波李璐璐刘玉庆
胡明 付晓杰 张庆 齐静 骆延波 李璐璐 刘玉庆

摘要:以64倍最小抑菌浓度(minimum inhibitory concentration,MIC)的氨苄西林、环丙沙星、多粘菌素三种抗菌药物分别处理大肠杆菌ATCC 25922,去除药物后,保持原始菌株药物敏感性的存活菌株为持留菌株;另用浓度递增的亚致死量的三种抗菌药物培养基连续对大肠杆菌ATCC 25922进行分批培养,获得能够耐受一定药物浓度的抗药性增强的菌株,去除药物后,菌株稳定的MIC值仍显著高于原始菌株,为抗药菌株。以稳定的MIC范围为检测指标,比较原始菌株的持留菌株和抗药菌株后续的生长优势。结果表明:经过一段时间的混合培养,持留菌株在95%以上,数量上占绝对优势,说明持留菌株比抗药菌株具有更强的生长优势,由此引发对抗药性回复的重新思考,自然界中持留菌株的特性、形成及存在可能为抗药性的逆转预留了一条途径。
关键词:抗药性;持留性;抗药菌株;持留菌株;最小抑菌浓度(MIC);生长优势
中图分类号:S852.61文献标识号:A文章编号:1001-4942(2019)07-0108-05
抗菌药物的发现和使用大大降低了细菌感染性疾病导致的死亡率,但随着抗菌药物的广泛应用,人类和动物病原菌的抗药性问题凸显并引起全球高度关注[1]。抗菌药物使用之初就有相关抗药性的报道,至今有关细菌抗药性的起源、发展和传播已有了相当的积累。研究表明细菌抗药性可以通过多种机制产生,如编码靶蛋白的基因突变、药物渗透降低、细菌外排泵的外排功能激活、靶蛋白旁路、非酶的靶位保护、酶靶位修饰等,细菌通过整合各种可能的机制形成抗药性表型,并且可遗传的抗药性能够通过可移动遗传因子进行水平转移[2],尤其在抗菌药物选择压力下,越来越多的“超级细菌”逐渐产生,对人类健康造成巨大威胁[3]。
细菌的持留性(persistance)不同于抗药性,表型上,持留菌虽然也能够耐过远高于MIC的药物浓度得以存活,却保留了原始菌株的药物敏感特性。早在1944年,Bigger发现青霉素并不能完全杀死金黄色葡萄球菌,其中极小一部分可以存活,认为其表现为休眠状态、非可遗传表型[4]。抗菌药物可以杀死大部分细菌,但总有一小部分不能被抗菌药物杀灭,当这些存活的亚群在同样的抗菌药物条件下生长,会重复这种异质特性,这种现象叫做细菌持留性,存活的细菌叫做持留菌[5]。持留菌的形成机制尚不明确,目前已有的研究包括持留性相关基因如hipA[6]、毒素/抗毒素(TA)系统[7]和外排泵系统[8]等,与细菌抗药性一样,持留性也是多因素共同参与形成的。
持留菌和抗药菌的本质和形成机制不同,但共存于抗菌药物压力下的细菌群体中。在对持留菌的研究中,普遍认为持留菌在抗药性中起到了至关重要的“帮凶”作用,研究思路多为阻断持留菌的形成和阻止进入休眠状态[6-8]。而本文思路是从生态角度审视抗菌药物使用过程中,休眠的持留菌在抗药性回复中可能的作用及当去除药物后二者在菌群后续繁殖中各自发挥怎样的作用。在体外条件下采用氨苄西林、环丙沙星和多粘菌素三种不同类型的常用抗菌药物,以不同方式对原始菌株进行处理,获得原始菌株的持留菌株和抗药菌株,比较其药敏特性和相同生态环境中的生长优势,并探讨持留菌存在的意义及其在抗药性回复中潜在的作用。
1 材料与方法
1.1 试验材料
菌株:药物敏感性试验质控菌株大肠杆菌ATCC 25922,由山东省畜禽疫病防治与繁育重点实验室保存。
試剂与药品:氨苄西林、环丙沙星和多粘菌素,购自中国药品生物制品检定所,分析纯;MH液体培养基(MHB)、MH固体培养基(MHA),购自北京陆桥技术有限责任公司。
1.2 试验方法
1.2.1 MIC测定 根据美国临床和实验室标准协会(Clinical and Laboratory Standards Institute,CLSI)建立的标准药敏方法,采用微量肉汤稀释法[9],测定原始菌株、持留菌及抗药菌株对氨苄西林、环丙沙星、多粘菌素三种抗菌药物的最小抑菌浓度(minimum inhibitory concentration,MIC)。
1.2.2 高药物浓度下获得持留菌株 用微量肉汤稀释法测定大肠杆菌ATCC 25922 MIC后,在96孔板上,将氨苄西林、环丙沙星和多粘菌素的药物浓度分别设置为各种药物MIC的64倍(氨苄西林256μg/mL,环丙沙星1μg/mL,多粘菌素32μg/mL),按照药敏试验方法的接种量接入稳定期大肠杆菌ATCC 25922,每种药做18个重复孔,37℃静置培养。分别于6 d内每天取3个重复样,将每孔液体共200μL全部取出,10 000 r/min 离心5 min,用200μL生理盐水洗涤2次,加50μL生理盐水重悬后涂布MHA平板,培养24 ~ 48 h,计算菌落数,测定MIC。
1.2.3 在递增的亚致死量药物浓度下分批传代获得抗药菌株 取大肠杆菌ATCC 25922接种于装有50 mL MHB的三角瓶中(接种量为1%,V/V),氨苄西林、环丙沙星和多粘菌素的终浓度分别为各自的1/2 MIC,12 h后转接到下一个三角瓶中(药物浓度不变),培养12 h后再转接到药物浓度倍增的三角瓶中;药物浓度由1/2 MIC连续倍比递增至64倍MIC,每一浓度传代2次,第二次培养细菌生长后划线于含相同浓度药物的MHA平板,过夜培养,挑取生长良好的单菌落并测定MIC。然后将其在无药MHB中连续传代直至MIC稳定,获得一系列MIC稳定的抗药性增强的菌株。
1.2.4 持留菌株与抗药菌株的生长曲线测定 将获得的原始菌的持留菌株和抗药菌株在岛津2500 UV-VIS光谱仪中37℃培养900 min,每15 min在线连续测量OD600,比较不同菌株的生长曲线是否存在差异。
1.2.5 持留菌株和抗药菌株的MIC范围 分别选取氨苄西林、环丙沙星和多粘菌素三种药物原始菌的持留菌株和抗药菌株,挑取单菌落分别接种于等体积的MHB中,以同样的条件振荡培养16 ~ 18 h,各取菌液1 mL稀释至适当浓度,涂布于MHA平板获得单菌落,各取100个单菌落进行MIC测定[9],统计不同菌株各自的MIC分布范围。
1.2.6 持留菌株和抗药菌株生长优势的比较 根据1.2.5中测定的MIC频率分布结果,选取各自MIC范围没有交叉的持留菌株和抗药菌株,等比例接种培养基混合培养,以各自MIC为检测指标统计二者组成比例的变化。具体方法:挑取氨苄西林、环丙沙星和多粘菌素的持留菌株和抗药菌株的单菌落,分别接种于MHB中,37℃、200 r/min振荡培养12 h,用MHB稀释,于紫外-可见光分析仪(GE Ultrospec 3100 pro)检测OD600,调节至0.08 ~ 0.10范围内,尽量调整稀释倍数使其最终达到相同的OD600值,确保小数点后两位相同。以此浓度各取500μL,同时接种于含有100 mL MHB的三角瓶中,等比例接种后混合振荡培养,每天取1 mL转接入新的100 mL MHB中,连续转移传代7 d甚至更长时间。每次培养后取混合菌液稀释至合适浓度,涂布获得单菌落100株,测定其MIC。以1.2.5中测定的MIC频率分布为指标,统计菌液中持留菌株和抗药菌株来源的大肠杆菌的比例。
2 结果与分析
2.1 高药物浓度下获得持留菌株
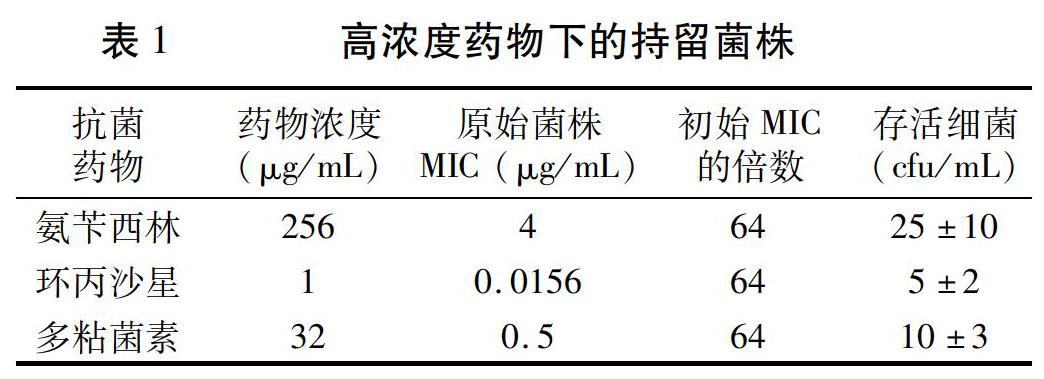
在高药物浓度的培养条件(氨苄西林256μg/mL,环丙沙星1μg/mL,多粘菌素32μg/mL)下,持续作用6 d,依然能够检测到存活的细菌。按照接种量104 cfu/mL数量级计算,存活率在0.1%左右(表1)。此试验结果表明,即使在有限的空间内,只要初始接种量达到一定数量,高于致死浓度的抗菌药物并不能完全杀死细菌,总会有一小部分细菌存活下来。
在药物氨苄西林浓度为256μg/mL条件下,先后共收集存活的细菌134株,测定其MIC,90%以上的菌株与原始菌株持平,少数略高于原始菌株一个或两个梯度,均远远低于其生存环境的药物浓度。环丙沙星和多粘菌素作用下的群体也存在同样的现象(数据略)。这些在高浓度药物作用下存活的细菌,其MIC却保持不变,为持留菌。致死量的抗菌药物不能将其杀灭,说明持留菌通过某种机制耐过了高浓度的药物,并且保持了原有的药物敏感特性。随机选取其中一株氨苄西林的持留菌,在氨苄西林浓度为256μg/mL的环境中得以存活,测定其分裂繁殖的后代实际MIC为4μg/mL,经传代后MIC保持不变,命名为菌株P-A,是原始菌株持留菌株繁殖后代的纯培养。
用同样的方法获得环丙沙星和多粘菌素作用下的持留菌株,分别耐过的药物浓度为1μg/mL和32μg/mL,保持原始菌株的MIC分别为0.0156μg/mL和0.5μg/mL不变。
2.2 在递增的亚致死量药物浓度下传代获得抗药菌株
在药物选择压力不断提高的试验条件下获得一系列抗药性增强的菌株,能够在相应药物浓度下继续生长繁殖。这些菌株的MIC值远远高于原始菌株MIC。由于存在多种形成机制,在药物去除后有些抗药机制关闭,而有些抗药机制是稳定的,因此,在无药培养基中连续传代后,抗药菌株的MIC值有不同程度的回落,但都难以回复到原始菌株的敏感性。
经递增的亚致死量氨苄西林连续培养后,选取一株MIC稳定的耐过氨苄西林256μg/mL的抗药菌株,传代后MIC稳定在128μg/mL,是原始菌株MIC的32倍,命名为R-A。
用同样的方法获得环丙沙星和多粘菌素的抗药菌株,选取耐过1μg/mL环丙沙星和32μg/mL多粘菌素的抗药菌株,稳定的MIC分别为0.5μg/mL和8μg/mL。
2.3 持留菌株和抗药菌株的MIC范围
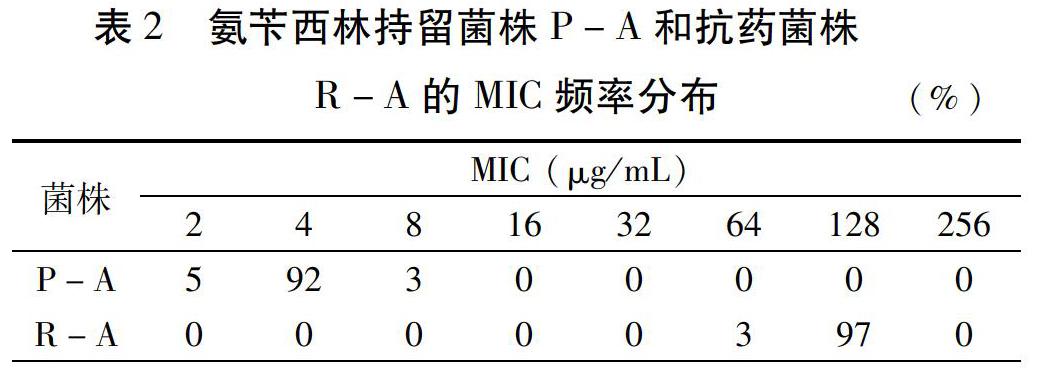
MIC值表示菌株对药物的敏感程度,是一个群体概念。由于细菌群体具有异质性,纯培养的细菌群体中小亚群的MIC也可能有所差异。因此,MIC并非一个准确的数值,而是一个范围。为了后续比较生长优势,先检测各类菌株的MIC范围,分别取100个单菌落测定MIC分布情况。氨苄西林药物处理的菌株MIC统计结果如表2,P-A的MIC分布峰值为4μg/mL,MIC与原始菌株持平;R-A的MIC分布峰值为128μg/mL,是原始菌株MIC的32倍。经过传代培养证明,两个菌株的MIC范围是稳定的表型指标,可以作为判定菌株性质的指标。对环丙沙星和多粘菌素处理菌株的稳定MIC范围也按同样的方法进行统计(数据略)。
2.4 持留菌株和抗药菌株生长优势的比较
经检测,不同药物作用下筛选的两类菌株的生长曲线并无明显差异(生长曲线图略),因此可采取混合培养的方式,以各自MIC为衡量指标比较其生长优势。
氨苄西林处理的P-A和R-A菌株MIC范围无相互交叉,将二者等比例混合接种培养,根据稳定的MIC范围统计其比例变化。如图1所示,混合培养后,持留菌株P-A所占比例逐渐升高,到第4 d增至70%以上,至第7 d抗药菌株R-A比例低于5%,菌株P-A数量占绝对优势。此结果表明,在没有药物的培养条件下,P-A具有更强的生长优势。随机挑选氨苄西林作用下存活的其它持留菌株和抗药菌株,用同样的方法对比其生长优势,也有类似的结果(数据略)。另外两种药物环丙沙星和多粘菌素的持留菌株和抗药菌株,生长优势的比较也存在类似结果(数据略),最终都能体现出持留菌株具有更强的生长优势。本研究中共检测8组菌株的对比,未检测到例外情况。
3 讨论与结论
3.1 细菌抗药性的意义
抗菌药物的广泛应用是筛选与富集抗药菌株的主要推动力,却并非抗药性产生的根源[10]。抗药基因广泛而普遍地存在于环境中—病原菌、共生菌及多种环境微生物[11]。越来越多的研究表明,抗药性是细菌的一个自然属性[12],彻底消除抗药性是不可能的。由于细菌遗传物质的自发突变、药物压力的筛选和诱导、复杂多样的抗药性机制、不可避免地与周围环境微生物接触等众多因素,即使在有限的空间内和特定條件下也难以完全消除细菌抗药性。从另一个方面看,抗药性对于细菌本身而言则意味着物种能够克服环境抗菌药物压力得以延续和存活,对环境微生物群落结构的多样性和稳定性起到了重要作用,对动物(包括人)免疫系统的形成、健康乃至整个生态环境都具有重大影响[13,14]。
細菌的抗药性为菌株提供了额外的生物学特性,帮助提高微生物对特定环境的适应性,同时多数情况下会付出一定的抗性代偿,包括生长繁殖能力降低。因此,理论上原始菌株比抗药菌株具有更强的生长优势,但在抗菌药物压力下具有更强生长优势的原始敏感菌被杀死,而抗药菌株的敏感性回复则是复杂漫长的过程[15]。
3.2 持留菌的存在及在抗药性回复中的潜力
在林林总总的抗药表型和抗药机制中,持留菌以其独特的机制引起人们的注意。持留菌通过“休眠-恢复生长-增殖”的方式维持自身的生存和菌体结构稳定,巧妙躲过外界恶劣环境的影响而存留下来,但依然保持着原始菌株的特性。比较持留菌株与抗药菌株的生长优势,可引发对抗药性回复问题的重新思考。本文研究结果表明当去除抗菌药物压力后,抗菌药物压力下存活的持留菌株比抗药菌株具有更强的生长优势,这一结论尚未见其它类似文献。虽然持留菌的作用尚未经过全面验证,但面对生态中严峻的抗药性问题,持留菌的存在和特性还是提供了一线希望。在环境中抗菌药物压力不断增大的过程中,存留了一批批“沉睡”的“种子”——持留菌。当药物压力逐渐降低,人们对于抗药性水平降低的期待不仅停留在抗药菌株的敏感性恢复[10],也在于预留的持留菌“苏醒”并恢复活力,以更为原始的菌株特性与现存的抗药性细菌共同竞争,而多数抗药性细菌可能由于其抗性代偿而处于劣势。只有环境中菌群的抗药性水平整体回落才能根本解决抗药性问题,并非对抗药菌斩尽杀绝,也非阻断所有抗药性机制。本研究结论表明,持留菌的存在和形成机制保留了原始菌株的生长优势,随着抗菌药物使用水平的降低,可能在抗药性的逆转中起到一定作用。
参 考 文 献:
[1]Laxminarayan R, Duse A, Wattal C, et al. Antibiotic resistance - the need for global solutions[J]. The Lancet Infectious Diseases, 2013, 13(12): 1057-1098.
[2]Wax R G, Lewis K, Salyers A A, et al. Bacterial resistance to antimicrobials[M]. Second Edition. New York: CRC Press, 2007.
[3]Kuehn B M. Antibiotic-resistant “superbugs” may be transmitted from animals to humans[J]. The Journal of American Medical Association, 2007, 298(18): 2125-2126.
[4]Bigger J W. Treatment of staphylococcal infections with penicillin by intermittent sterilization[J]. The Lancet, 1944, 244(6320): 497-500.
[5]Balaban N Q, Merrin J, Chait R, et al. Bacterial persistence as a phenotypic switch[J]. Science, 2004, 305(5690):1622-1625.
[6]Moyed H S, Bertrand K P. hipA, a newly recognized gene of Escherichia coli K-12 that affects frequency of persistence after inhibition of murein synthesis[J]. Jorunal of Bacteriology, 1983, 155(2): 768-775.
[7]Gerdes K,Maisonneuve E. Bacterial persistence and toxin-antitoxin loci[J]. The Annual Review of Microbiology,2012,66:103-123.
[8]Pu Y, Zhao Z, Li Y, et al. Enhanced efflux activity facilitates drug tolerance in dormant bacterial cells[J]. Molecular Cell, 2016, 62(2): 284-294.
[9]CLSI. Performance standards for antimicrobial disk susceptibility tests. 27th edition. CLSI document M100[S]. Wayne: Clinical and Laboratory Standards Institute, 2017.
[10]Hughes D. Selection and evolution of resistance to antimicrobial drugs[J]. IUBMB Life, 2014, 66(8): 521-529.
[11]Allen H K, Donato J, Wang H H, et al. Call of the wild: antibiotic resistance genes in natural environments[J]. Nature Reviews Microbiology, 2010, 8(4): 251-259.
[12]Martinez J L. The role of natural environments in the evolution of resistance traits in pathogenic bacteria[J]. Proceedings of the Royal Society B: Biological Sciences, 2009, 276(1667): 2521-2530.
[13]Sommer M O A, Dantas G, Church G M. Functional characterization of the antibiotic resistance reservoir in the human microfiora[J]. Science, 2009, 325(5944): 1128-1131.
[14]Whitman W B, Coleman D C, Wiebe W J. Prokaryotes: the unseen majority[J]. Proceedings of the National Academy of Sciences of the United States of America, 1998, 95(12): 6578-6583.
[15]Andersson D I, Hughes D. Antibiotic resistance and its cost: is it possible to reverse resistance?[J]. Nature Reviews Microbiology, 2010, 8(4): 260-271.
