PLUG引物和IT引物在顶芒山羊草分子标记建立中的通用性分析
2019-09-03宫文萍李凯程敦公武智民李洪振韩冉李豪圣刘成刘建军
宫文萍 李凯 程敦公 武智民 李洪振 韩冉 李豪圣 刘成 刘建军

摘要:分子标记是检测和追踪小麦背景中近缘物种染色体的重要手段。依据禾本科物种基因共线性原则,基于基因序列设计的引物在不同物种中可能具有通用性。本研究对水稻PLUG引物和簇毛麦IT引物在顶芒山羊草中的通用性进行了分析,并建立了顶芒山羊草染色体特异IT标记。供试的397对PLUG引物和841对IT引物对小麦-顶芒山羊草双二倍体与中国春的扩增结果显示,377对(95.0%)PLUG引物和593对(70.5%)IT引物可以在供试材料中扩增出清晰条带。因此,PLUG引物在顶芒山羊草中的通用性优于IT引物。相比小麦对照,PLUG引物和IT引物能在小麦-顶芒山羊草双二倍体中扩增出多态性条带的引物数分别有17对和5对,分别占供试PLUG引物和IT引物的4.3%和0.6%。利用获得的IT多态性引物对一套小麦-顶芒山羊草附加系和一套小麦-卵穗山羊草附加系进行扩增,结果发现仅引物CINAU1060、CINAU1465和CINAU1517能在相应附加系中扩增出大小分别约为300、480 bp和320 bp的多态性标记,为小麦背景中顶芒山羊草染色质的鉴定提供了新的检测手段。然而,本研究中IT引物建立顶芒山羊草分子标记的比率仅为0.4%,低于我们之前用PLUG引物建立顶芒山羊草分子标记的比率(8.9%)。
关键词:顶芒山羊草;分子标记;PLUG引物;IT引物;通用性
中图分类号:S512.103.53文献标识号:A文章编号:1001-4942(2019)07-0010-07
Abstract Molecular marker plays an important role in detecting and tracking chromosomes of related species in wheat background. According to the principle of gene collinearity in Gramineae species, primers designed based on gene sequences might be universal in different species. In this research, the universality of PLUG primers for Oryza sativa and IT primers for Dasypyrum villosum were analyzed in Aegilops comosa, and chromosome specific IT markers of Ae.comosa were established. The amplification results of 397 pairs of PLUG primers and 841 pairs of IT primers in wheat-Ae.comosa amphiploid and Chinese Spring showed that 377 pairs (95.0%) of PLUG primers and 593 pairs (70.5%) of IT primers could amplify clear bands in the tested materials. Therefore, the universality of PLUG primers in Ae.comosa was better than that of IT primers. Compared with Chinese Spring, the number of PLUG and IT primers that could amplify polymorphic bands in wheat-Ae.comosa amphiploid was 17 pairs and 5 pairs, which accounted for 4.3% and 0.6% of the PLUG and IT tested primers, respectively. In this research, a set of wheat-Ae.comosa addition and a set of wheat-Ae.geniculata addition were amplified with the obtained IT polymorphic primers. The results showed that 300, 480 bp and 320 bp in length polymorphism bands could be amplified by primers CINAU1060, CINAU1465 and CINAU1517 in relative additions, respectively, which provided new method for the identification of Ae.comosa chromatin in wheat background. However, the ratio for molecular marker development of Ae.comosa using IT primers was 0.4%, which was much lower than that of PLUG primers (8.9%).
Keywords Aegilops comosa; Molecular marker; PLUG primer; IT primer; Universality
小麥(Triticum aestivum L.)是世界上播种面积最大、总产量最多和分布范围最广的重要粮食作物,但其病害如小麦条锈病、叶锈病和白粉病等严重阻碍了小麦生产[1]。小麦近缘物种抗病性优异,从小麦近缘物种中将抗病基因导入小麦是改良小麦抗病性的重要方法之一。小麦近缘物种如山羊草属(Aegilops)中的顶芒山羊草(Ae. comosa,也作Triticm comosum,2n=2x=14,基因组MM),起源于欧洲南部的希腊和土耳其等国,一年生草本植物,高抗小麦条锈病[2]、秆锈病[2]、叶锈病[3]和白粉病[4],是小麦抗病育种的优异基因源。小麦-近缘物种双二倍体是向普通小麦转育顶芒山羊草抗病性的重要桥梁[5,6],目前,本试验室已鉴定获得小麦-顶芒山羊草双二倍体[7]以及该双二倍体与普通小麦的杂交后代材料,然而,分子标记的缺乏极大地限制了这批杂交后代材料的精确鉴定。
分子标记对小麦背景中近缘物种染色体的鉴定和追踪起到重要作用。研究证明,可依据禾本科物种基因共线性原则,根据已测序物种的基因序列开发引物用于建立另一未测序物种的分子标记[3,6]。基于水稻和簇毛麦基因序列的PLUG引物[8]和IT引物[9]已经被分别开发出来。目前,PLUG标记不仅已经被用于识别小麦与外源染色体的同源群[10-12],还被用于标记辅助选择(MAS)、比较基因组学、异源染色体的追踪和基因分型[10-13]。近年来,IT引物也已被用于鉴定小麦背景中的簇毛麦染色质[9,14]。然而,这两类引物在顶芒山羊草中的通用性还不清楚。本研究利用PLUG引物和IT引物对小麦-顶芒山羊草双二倍体及对照小麦进行PCR扩增,比较了两类引物在顶芒山羊草中的通用性,进而利用一套小麦-顶芒山羊草附加系为材料,对获得的多态性片段进行染色体定位,建立了顶芒山羊草IT分子标记,为小麦背景中顶芒山羊草染色质的检测提供了新的手段。
1 材料与方法
1.1 试验材料
24份供试材料的相关信息列于表1,其中,小麦中国春由电子科技大学(University of Electronic Science and Technology of China, UESTC)杨足君教授提供。序号2—13的材料由英国John Innes Centre(JIC)的Reader S.M.教授提供。序号14—20的材料由美国堪萨斯州立大学小麦遗传与基因资源中心(Wheat Genetics Resource Center, WGRC)的Friebe B.教授提供。序号21—24的材料由本实验室(Current Lab, CL)育成。
1.2 试验方法
1.2.1 DNA提取
供试材料基因组总DNA用快捷型植物基因组DNA提取试剂盒(天根生化科技(北京)有限公司)提取。取约100 mg的叶片放入2 mL离心管(提前加入直径2 mm的钢珠)中,液氮中速冻,然后用SCIENTZ-192组织研磨机在30 Hz频率下将叶片打碎至粉末,加入400 μL缓冲液FP1和6 μL的TNase,漩涡振荡1 min,室温放置10 min。然后加入130 μL缓冲液FP2,轻轻混匀,涡旋振荡1 min。12 000 r·min-1离心5 min,将上清转移至新的离心管中。向上清液中加入70%体积的异丙醇,充分混匀,12 000 r·min-1离心2 min,弃上清,保留沉淀。加入500 μL 70%乙醇洗涤DNA,12 000 r·min-1离心2 min,弃上清,再次用500 μL 70%乙醇洗涤一次,步骤与第一次相同。晾干后加入TE溶解DNA备用。
1.2.2 PLUG引物与IT引物的PCR扩增及电泳分析
397对PLUG引物根据水稻基因序列设计,染色体第一到第七同源群引物分别为51、55、63、54、61、59、54对,引物序列及PCR扩增程序参照文献[11],PLUG引物由成都瑞信生物公司合成。841对IT引物根据簇毛麦基因内含子区多态性设计[9,14],染色体第一到第七同源群引物分别为135、175、120、89、140、71、111对,扩增程序参照文献[9]。IT引物由青岛擎科天成生物技术有限公司合成。
PLUG-PCR和IT-PCR产物在2%的琼脂糖凝胶中电泳,电泳后凝胶在紫外凝胶成像仪GDS-Gel Dol 2000下扫描照相。用凝胶成像系统的Quality One软件进行条带统计,同时人工统计条带与软件统计结果相互核实印证。
2 结果与分析
2.1 PLUG引物和IT引物的扩增结果
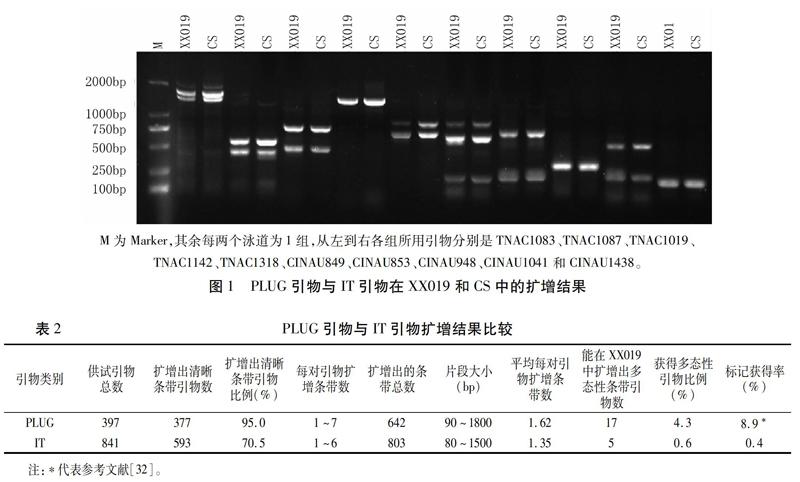
用397对PLUG引物对小麦-顶芒山羊草双二倍体和中国春基因组DNA进行PCR扩增,结果显示,377对(95.0%)引物可以在供试材料中扩增出清晰条带,其中,引物TNAC1083、TNAC1087、TNAC1019、TNAC1142和TNAC1318的扩增效果如图1所示。每对PLUG引物的扩增条带数1~7条不等,片段大小90~1 800 bp,共扩增出642条清晰条带,平均扩增条数为1.62条(表2)。
用841对IT引物对小麦-顶芒山羊草双二倍体和中国春基因组DNA进行PCR扩增,结果显示, 593对(70.5%)引物能在供试物种中扩增出清晰条带,其中,引物CINAU849、CINAU853、CINAU948、CINAU1041和CINAU1438的扩增效果如图1所示。每对IT引物的扩增条带数1~6条不等,片段大小80~1 500 bp,共扩增出803条清晰条带,平均扩增条数为1.35条(表2)。
2.2 顶芒山羊草特异PLUG和IT片段
能在供试材料中扩增出清晰条带的377对PLUG引物中,相比对照小麦中国春、济麦23、济麦44、济麦229和济麦262,TNAC1017、TNAC1204和TNAC1364等17对引物能在小麦-顶芒山羊草双二倍体中扩增出多态性条带,其中,引物TNAC1296和TNAC1920各扩增出2个多态性条带,其余引物均扩增出1个多态性条带,共计获得19个顶芒山羊草的特异性片段,获得多态性引物比例为4.3%(17/397)。所获19个多态性片段大小220~1 400 bp不等,目前这些多态性标记已经被应用于小麦-顶芒山羊草杂交种质的鉴定中[15]。
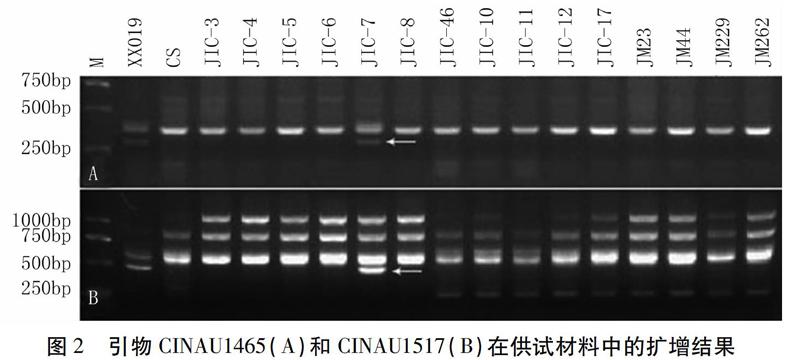
能在供试材料中扩增出清晰条带的593对IT引物中,相比中国春、济麦23、济麦44、济麦229和济麦262,CINAU883、CINAU1060、CINAU1067、CINAU1465和CINAU1517共5对引物能在小麦-顶芒山羊草双二倍体中扩增出多态性条带,5对引物分别扩增出1个多态性条带,共获得5个顶芒山羊草的特異性片段,获得多态性引物比例为0.6%(5/841)。其中,CINAU1465和CINAU1517的扩增结果如图2所示。
2.3 顶芒山羊草染色体特异IT分子标记的建立
因为顶芒山羊草的M染色体组是卵穗山羊草Mg染色体组的供体[16],所以本研究同时利用小麦-顶芒山羊草2M-7M附加系和小麦-卵穗山羊草1Mg-7Mg附加系对上述多态性片段外进行染色体定位。利用引物CINAU883、CINAU1060、CINAU1067、CINAU1465和CINAU1517对上述两套附加系进行扩增,结果发现,根据簇毛麦2VL上基因序列设计的引物CINAU1060仅能在顶芒山羊草2M和卵穗山羊草2Mg染色体扩增出片段长度约为300 bp的目标片段(表3);根据簇毛麦5VS上基因序列设计的引物CINAU1465仅能在顶芒山羊草6M(表3,图2A)和卵穗山羊草5Mg染色体扩增出片段长度约为480 bp的目标片段(表3);根据簇毛麦6VL上基因序列设计的引物CINAU1517仅能在顶芒山羊草6M(表3,图2B)和卵穗山羊草5Mg染色体扩增出片段长度约为320 bp的目标片段(表3)。
本研究筛选了841对IT引物,获得顶芒山羊草2M染色体特异标记1个和6M染色体标记2个(表3),获得标记比例约为0.4%(3/841),远低于我们之前利用PLUG引物建立顶芒山羊草染色体特异标记8.9%的比例(表2)。本研究中,CINAU1465和CINAU1517均分别能在顶芒山羊草6M和卵穗山羊草5Mg染色体上扩增出特异目标多态性片段,这表明顶芒山羊草和卵穗山羊草染色体可能存在5/6重组。
3 讨论
3.1 水稻PLUG引物在小麦族不同物种中的通用性及分子标记建立中的应用
Ishikawa等[8,16]最早利用水稻单拷贝基因序列开发设计获得PLUG引物,用于有效分选基因产物,是大规模标记开发的有效工具。其后,Li等[17]建立了长穗偃麦草染色体第七同源群的PLUG标记,用于鉴定小麦-长穗偃麦草附加系材料1—27,分子标记获得比例为5.4%。Li等[18]建立了帝国黑麦染色体特异PLUG标记79个, 分子标记获得比例为54.9%。Lang[10]和Hu[19]等分別筛选PLUG引物建立了中间偃麦草特异PLUG标记,用于鉴定小麦-中间偃麦草杂交种质,分子标记获得比例分别为82.0%和84.9%。Li[20]和Liu[21]等分别建立了多年生簇毛麦染色体不同同源群PLUG标记用于鉴定小麦-多年生簇毛麦染色体系,分子标记获得比例41.7%和10.7%。Liu等[22]建立了无芒山羊草染色体特异PLUG标记,分子标记获得比例为6.9%。上述实例表明,PLUG引物在小麦族不同物种中的通用性及分子标记建立获得率相差较大,上述物种中,PLUG引物在黑麦和偃麦草标记建立方面优于其他物种,且对同一物种而言随着PLUG引物对用量的多少其建立物种分子标记的获得比例也可能发生变化。因为本研究发现,供试的397对PLUG引物中,有17对可以用于建立顶芒山羊草染色体特异分子标记,获得多态性比例仅为4.3%,而筛选更多量的PLUG引物(526对)建立顶芒山羊草分子标记的比例反而增加到了8.9%,这可能是因为水稻PLUG与小麦族物种不同染色体同源群EST同源性有所差异造成的。
3.2 簇毛麦IT引物在顶芒山羊草中的通用性及分子标记建立中的应用
基于基因内含子序列开发IT引物并建立物种分子标记的工作已经在马铃薯和龙葵[23]、豌豆[24]、高粱[25]和燕麦[26]等物种中报道。然而,在小麦野生近缘种中却鲜有报道。近年来,小麦野生近缘种山羊草IT引物[27] 、簇毛麦IT引物[9,14]已逐渐被开发出来。Sheikh等[27]开发了135对IT引物,建立了粘果山羊草和易变山羊草染色体特异标记29个,标记获得率为21.5%。Zhang[9]等利用开发的1 624对IT引物建立了簇毛麦染色体特异标记841个,标记获得率为51.8%。Wang[14]等利用流式细胞仪对簇毛麦4VS染色体臂进行分拣并测序,根据测序结果开发了IT引物359对,建立了簇毛麦4VS特异性标记232个,标记获得率为64.62%。本研究利用上述簇毛麦的841对IT引物对小麦对照和小麦-顶芒山羊草双二倍体等物种进行扩增,结果发现,这批IT引物能在供试物种中扩增出清晰条带的占70.5%,然而,仅3对可以用于建立顶芒山羊草染色体特异IT标记,标记获得率仅为0.4%,这可能是因为本研究所用琼脂糖凝胶的分辨率远低于Zhang[9]和Wang[14]等所用聚丙烯酰胺凝胶所致。目前,我们正在开展利用聚丙烯酰胺凝胶建立顶芒山羊草染色体特异IT标记工作。
3.3 顶芒山羊草特异标记的建立
对不同引物进行物种间通用性分析,不仅可以用于评价物种间亲缘关系[27],还可用于物种分子标记的建立[15-21]。在顶芒山羊草特异标记建立方面,Teoh等[28]通过C带技术获得不同来源顶芒山羊草的标准核型图,即建立了顶芒山羊草细胞遗传标记。Wang等[29]从顶芒山羊草中发现一类新的对小麦面团品质具有正面效应的低分子麦谷蛋白基因并开发了分子标记。翁跃进等[30-31]建立了顶芒山羊草M染色体组RFLP标记40个,并将其应用于小麦-顶芒山羊草4M(4D)异代换系的鉴定。Liu等利用水稻PLUG引物对顶芒山羊草染色体组分子标记进行开发,获得顶芒山羊草染色体特异标记44个[32]。本研究首次利用IT引物对顶芒山羊草进行扩增,获得顶芒山羊草新型IT分子标记3个,为检测小麦背景中顶芒山羊草染色质提供了新的检测方法。
4 结论
PLUG引物在顶芒山羊草中的通用性优于IT引物;顶芒山羊草染色体特异IT标记CINAU1060、CINAU1465和CINAU1517的建立为小麦背景中顶芒山羊草染色质的鉴定提供了新的检测手段。
参 考 文 献:
[1] Mcintosh R A, Miller T E, Chapman V.Cytogenetical studies in wheat Ⅻ. Lr28 for resistance to Puccinia recondita and Sr34 for resistance to Puccinia graminis tritici[J].Zeitschrift Pflanzenzuhtung, 1982, 89(4):295-306.
[2] Riley R, Chapman V, Johnson R. Introduction of yellow rust resistance of Aegilops comosa in wheat by genetically induced homoeologous recombination[J]. Nature, 1968, 217(5126):383-384.
[3] 刘成, 宫文萍, 李光蓉, 等.小麦-无芒山羊草创新种质资源的鉴定[C]∥中国作物学会.第六届全国小麦基因组学及分子育种大会论文集.2015:164.
[4] Eser V. Characterisation of powdery mildew resistant lines derived from crosses between Triticum aestivum and Aegilops speltoides and Ae.mutica[J]. Euphytica, 1998, 100:269-272.
[5] 蔣华仁, 戴大庆, 孙东发. 小麦特异种质资源的创新研究[J]. 四川农业大学学报, 1992, 10(2):255-259.
[6] Gong W P, Li G R, Zhou J P, et al. Cytogenetic and molecular markers for detecting Aegilops uniaristata chromosomes in a wheat background[J]. Genome, 2014, 57(9):489-497.
[7] 宫文萍, 韩冉, 宋健民, 等. 顶芒和无芒山羊草育种价值及细胞学标记[J]. 核农学报, 2017, 31(10):1889-1895.
[8] Ishikawa G, Nakamura T, Ashida T, et al. Localization of anchor loci representing five hundred annotated rice genes to wheat chromosomes using PLUG markers[J]. Theoretical and Applied Genetics, 2009, 118(3):499-514.
[9] Zhang X D, Wei X, Xiao J, et al. Whole genome development of intron targeting (IT) markers specific for Dasypyrum villosum chromosomes based on next-generation sequencing technology[J]. Molecular Breeding, 2017, 37:115.
[10]Lang T, La S X, Li B, et al. Precise identification of wheat-Thinopyrum intermedium translocation chromosomes carrying resistance to wheat stripe rust in line Z4 and its derived progenies[J]. Genome, 2018, 61(3):177-185.
[11]Lei M P,Li G R,Zhang S F,et al.Molecular cytogenetic characterization of a new wheat Secale africanum 2Ra(2D) substitution line for resistance to stripe rust[J].Journal of Genetics,2011,90(2):283-287.
[12]Zhan H X, Li G R, Chang Z J, et al. Molecular identification of a new wheat-Thinopyrum intermedium cryptic translocation line for resistance to powdery mildew[J]. IJBBB,2013, 4(3):376-378.
[13]Hu L J, Li G R, Zeng Z X, et al. Molecular cytogenetic identification of a new wheat-Thinopyrum substitution line with stripe rust resistance[J]. Euphytica, 2011, 177(2):169-177.
[14]Wang H Y, Dai K L, Xiao J, et al. Development of intron targeting (IT) markers specific for chromosome arm 4VS of Haynaldia villosa by chromosome sorting and next-generation sequencing[J]. BMC Genomics, 2017, 18:167-175.
[15]Schneider A, Molnar I, Molnar L M. Utilisation of Aegilops (goatgrass) species to widen the genetic diversity of cultivated wheat[J]. Euphytica, 2008, 163(1):1-19.
[16]Ishikawa G, Yonemaru J, Saito M, et al. PCR-based landmark unique gene (PLUG) markers effectively assign homoeologous wheat genes to A, B and D genomes[J]. BMC Genomics, 2007, 8(1):135.
[17]Li X J,Hu X G,Hu T Z,et al.Identification of a novel wheat-Thinopyrum ponticum addition line revealed with cytology, SSR, EST-SSR, EST-STS and PLUG markers[J]. Cereal Research Communications, 2015, 43(4):1-10.
[18]Li J J, Endo T R, Saito M, et al. Homoeologous relationship of rye chromosome arms as detected with wheat PLUG markers[J]. Chromosoma, 2013, 122(6):555-564.
[19]Hu L J, Li G R, Zhan H X, et al. New St-chromosome specific molecular markers for identifying wheat-Thinopyrum intermedium derivative lines[J]. J.Genet., 2012, 91:e69-e74.
[20]Li G R, Zhao J M, Li D H, et al. A novel wheat-Dasypyrum breviaristatum substitution line with stripe rust resistance[J]. Cytogenetic and Genome Research, 2014, 143(4):280-287.
[21]Liu C, Li G R, Yan H F, et al. Molecular and cytogenetic identification of new wheat-Dasypyrum breviaristatum additions conferring resistance to stem rust and powdery mildew[J]. Breeding Science, 2011, 61(4):366-372.
[22]Liu C, Li G R, Gong W P, et al. Molecular and cytogenetic characterization of a powdery mildew-resistant wheat-Aegilops mutica partial amphiploid and addition line[J]. Cytogenetic and Genome Research, 2016, 147(2/3):186-194.
[23]Poczai P, Cernak I, Gorji A M, et al. Development of intron targeting (IT) markers for potato and cross-species amplification in Solanum nigrum (Solanaceae) [J]. American Journal of Botany, 2010, 97(12):e142-e145.
[24]Jain S L, Weeden N F, Portwe L D, et al. Finding linked markers to En for efficient selection of pea enation mosaic virus resistance in pea[J]. Crop Science, 2013, 53(6):2392-2399.
[25]Jaikishan I, Rajendrakumar P, Madhusudhana R,et al. Development and utility of PCR-based intron polymorphism markers in sorghum [Sorghum bicolor (L.) Moench][J]. Journal of Crop Science and Biotechnology, 2015, 18(5):309-318.
[26]Holland J B, Hekkand S J, Sharopova N, et al. Polymorphism of PCR-based markers targeting exons, introns, promoter regions, and SSRs in maize and introns and repeat sequences in oat[J]. Genome, 2001, 44(6):1065-1076.
[27]Sheikh I, Sharma P, Verma S K, et al. Development of intron targeted amplified polymorphic markers of metal homeostasis genes for monitoring their introgression from Aegilops species to wheat[J]. Molecular Breeding, 2018, 38(4):47.
[28]Teoh S B,Miller T E,Readet S M.Intraspecific variation in C-banded chromosomes of Aegilops comosa and Aegilops speltoides[J]. Theoretical and Applied Genetics, 1983, 65(4):343-348.
[29]Wang K, Gao L Y, Wang S L, et al. Phylogenetic relationship of a new class of LMW-GS genes in the M genome of Aegilops comosa[J]. Theoretical and Applied Genetics, 2011, 122(7):1411-1425.
[30]翁躍进, 贾继増, 董玉琛. 小麦M染色体组的RFLP标记[J]. 农业生物技术学报,1997(3):211-215.
[31]翁跃进, 贾继增, 董玉琛. 利用RFLP分子标记鉴定小麦-顶芒山羊草异代换系[J]. 遗传学报, 1997, 24(3):248-254.
[32]Liu C, Gong W P, Han R, et al. Characterization, identification and evaluation of a set of wheat-Aegilops comosa chromosome lines[J]. Scientific Reports, 2019, 9(1):4773.
