自然发酵腐乳中细菌多样性评价
2019-08-27李娜崔梦君马佳佳余海忠张振东郭壮赵慧君
李娜,崔梦君,马佳佳,余海忠,张振东,郭壮,2,赵慧君,*
(1.湖北文理学院食品科学技术学院鄂西北传统发酵食品研究所,湖北 襄阳 441053;2.恩施市公共检验检测中心,湖北 恩施 445000)
腐乳,又称豆腐乳,是一种中国传统的大豆发酵食品,也是由微生物作用的代表性豆制品[1]。根据加工方式不同,腐乳可分为细菌型腐乳、霉菌型腐乳、酶法发酵以及自然发酵腐乳[2]。在腐乳自然发酵过程中,由于制作环境和人工因素等的影响,导致其蕴含的微生物的种类丰富多样,从而使腐乳具有别样风味。众多学者对腐乳中微生物群落构成进行了研究,其中程永强等[3]在低温发酵腐乳中发现1 株嗜低温的毛霉——黄色毛霉(Mucor flavus),同时姚翔等[4]在益阳自然发酵腐乳中分离出总状毛霉(Mucor racemosus)和鲁氏毛霉(Mucor roxianus),而鲁菲等[5]在青方腐乳中分离出植物乳杆菌(Lactobacillus plantarum)和短小奇异菌,然而关于湖北地区腐乳微生物多样性的研究较少。
变性梯度凝胶电泳(polymerase chain reaction-denaturing gradient gel electrophoresis,PCR-DGGE)技术是一种可以对微生物的群落结构及遗传多样性进行连续分析的技术[6],同时具有重复性好、检测结果可靠以及应用范围广等优点[7],目前应用于葡萄酒[8]、香肠[9]和奶酪[10]等领域。Illumina Miseq 高通量测序技术可以从宏基因组层面对样品中的微生物多样性进行全方位且客观的分析评价,同时克服了传统微生物学手段耗时长、工作量大等缺点[11],广泛应用于研究肠道菌群[12]、发酵食品[13]以及环境检测[14]等方面。
本试验以恩施地区自然发酵腐乳为研究对象,利用PCR-DGGE 结合Illumina Miseq 高通量测序技术相结合的手段对腐乳中的微生物群落组成及多样性进行解析,同时利用传统微生物学方法分离鉴定腐乳中的乳酸菌。通过本研究的开展,可促进腐乳产业化生产,为大豆发酵食品领域提供一个重要的理论支持。
1 材料与方法
1.1 材料与试剂
样品:采购于恩施市菜市场。
三羟甲基氨基甲烷(分析纯)、乙酸(分析纯),乙二胺四乙酸(分析纯)、丙烯酰胺(分析纯)、甲叉双丙烯酰胺(分析纯)、去离子甲酰胺(分析纯)、(分析纯)、过硫酸铵(分析纯)、四甲基乙二(分析纯)、乙醇(分析纯)、冰醋酸(分析纯)、甲醛(分析纯)、硝酸银(分析纯)、氢氧化钠(分析纯)、MRS 合成培养基:国药集团化学试剂有限公司;D5625-01 DNA 提取试剂盒、DNA marker、PCR 清洁试剂盒:北京科博汇智生物科技发展有限公司;2xPCR mix:南京诺唯赞生物科技有限公司;rTaq、dNTP mix、pMD18-T vector:大连宝生物技术有限公司;正向引物338F(加入7 个核苷酸标签barcodes)和反向引物806R、PCR 引物合成和测序:武汉天一辉远生物科技有限公司。
1.2 仪器与设备
VeritiTM 96-well thermal cycler PCR 仪、NanoDrop 2000/2000c:美国 Thermo Fisher 公司;DCodeTM System:美国Bio Red 公司;DYY-12 电泳仪:北京六一仪器厂;Miseq PE300 高通量测序平台:美国Illumina 公司;R920 机架式服务器:美国 DELL 公司;CT15RE 冷冻离心机:日本HITACHI 公司;Bio-5000 plus 扫描仪:上海中晶科技有限公司;Whitley DG250 厌氧工作站:英国DWS 公司。
1.3 试验方法
1.3.1 样品宏基因组提取与检测
采用试剂盒方法提取腐乳样品中的宏基因组,用0.8%琼脂糖凝胶进行电泳检测,测定各样品宏基因组DNA 浓度。
1.3.2 PCR-DGGE
将宏基因组DNA 浓度调整为一致后作为模板细菌16S rRNA V3 区域基因片段PCR 扩增。采用25 μL体系进行 PCR 扩增:10×PCR Buffer(含 Mg2+)2.5 μL,dNTP 2 μL,上下游引物各 0.5 μL,rTaq 0.5 μL,模板1 μL,灭菌超纯水补充至25 μL。其中上游引物为ALL-GC-V3F(5’-CGCCCGGGGCGCGCCCCGGGCG GCCCGGGGGCACCGGGGGCCTACGGGAGGCAGCAG-3’),下游引物为 ALL-V3R(5’-ATTACCGCGGCTGCTGG-3’)。扩增程序:95 ℃ 4 min,95 ℃ 30 s,55 ℃30 s,72 ℃ 30 s,30 个循环,72 ℃ 10 min。扩增结束后,PCR 扩增产物用2%的琼脂糖凝胶电泳检测。
采用8%的聚丙烯酰胺(丙烯酰胺∶甲叉双丙烯酰胺=38.93 ∶1.07,质量比)、变性范围为35%~52%(100%变性剂:尿素42 g,去离子甲酰胺40 mL)的凝胶进行分析。将凝胶置于温度为60 ℃的0.5 x TAE 电泳缓冲液中,于每个胶孔点样10 μL,先电压120 V,持续80 min 后,电压80 V,持续13 h。电泳结束后,采用硝酸银法染色,使用扫描仪对电泳图进行观察拍照,找出各泳道优势条带并切胶,将胶块捣碎于50 μL 无菌超纯水中,4 ℃静置过夜。用不含GC 夹的引物(ALL-V3F 和ALL-V3R)将回收胶块进行PCR 扩增,扩增体系及条件同DGGE 扩增。用清洁试剂盒纯化PCR 产物,并与载体(PMD18-T)连接后转化到感受态细胞中进行克隆培养,筛选阳性克隆进行测序。使用BioEdit 软件将去除载体序列后在NCBI 中进行同源性比对。
1.3.4 样品细菌16S rRNA PCR 扩增及Miseq 高通量测序
参考王玉荣等[15]方法进行样品细菌16S rRNA PCR扩增及Miseq 高通量测序。采用20 μL 扩增体系:5×PCR 缓冲液 4 μL,dNTP mix 2 μL,上游引物 338F(5’-ACTCCTACGGGAGGCAGCA-3’)和下游引物 806R(5’-GGACTACHVGGGTWTCTAAT-3’)各 0.8 μL,rTaq 酶0.4 μL,模板 10 ng,灭菌超纯水补充至 20 μL。扩增条件为:95 ℃预变性 3 min,95 ℃变性 30 s,55 ℃退火 30 s,72 ℃延伸 45 s,共运行 30 个循环,72 ℃延伸 10 min。扩增结束后用1.0%琼脂糖凝胶电泳检测PCR 扩增产物,合格后进行高通量测序。
1.3.5 序列拼接及质量控制
参照于丹等[16]和陈泽斌等[17]的方法,在除去不合格序列、barcode 序列和引物序列的基础上,将数据序列进行拼接。同时利用PyNAST 软件将所有的序列对齐,采用UCLUST 算法将相似度>97%序列划分为一个操作单元(operational taxonomic units,OTU),从而进行物种鉴定和相对含量分析,对腐乳中的微生物多样性进行解析。
1.3.6 腐乳中乳酸菌的分离与鉴定
腐乳样品中乳酸菌的分离采用倍比稀释涂布法。将各样品稀释液涂布于改良MRS 固体培养基(含1.5%CaCO3)上,置于37 ℃厌氧培养48 h,挑选具有不同特征且透明圈现象明显的菌落划线纯化。纯化后的菌株进行革兰氏染色镜检、过氧化氢试验及冻存。参考文献[18]提取各菌株DNA,并参照张晓辉等[19]方法使用通用正向引物 27F(5’-AGAGTTTGATCCTGGCTCAG-3’)和反向引物 1495R(5’-CTACGGCTACCTTGTTACGA-3’)进行PCR 扩增和测序。测序结果分析同1.3.3。
1.4 数据处理
通过Origin 8.5 软件对DGGE 图谱特征条带序列进行统计,同时对样品的稀释曲线及香农指数曲线(shannon diversity index curve)的作图。系统发育树由BioEdit 软件和MEGA 5.0 软件共同绘制。使用Office 2016 绘制平均相对含量>5%的属水平饼图。Venn 图由在线绘图网页(http://bioinfogp.cnb.csic.es/tools/venny/index.html)进行绘制。相对含量>1.5%的核心OTU热图由Matlab 2010b 绘制。
2 结果与分析
2.1 腐乳中细菌DGGE图谱及分析
本研究首先使用变性梯度凝胶电泳技术对样品16SrRNAV3 区域细菌群落组成进行研究,如图1所示。
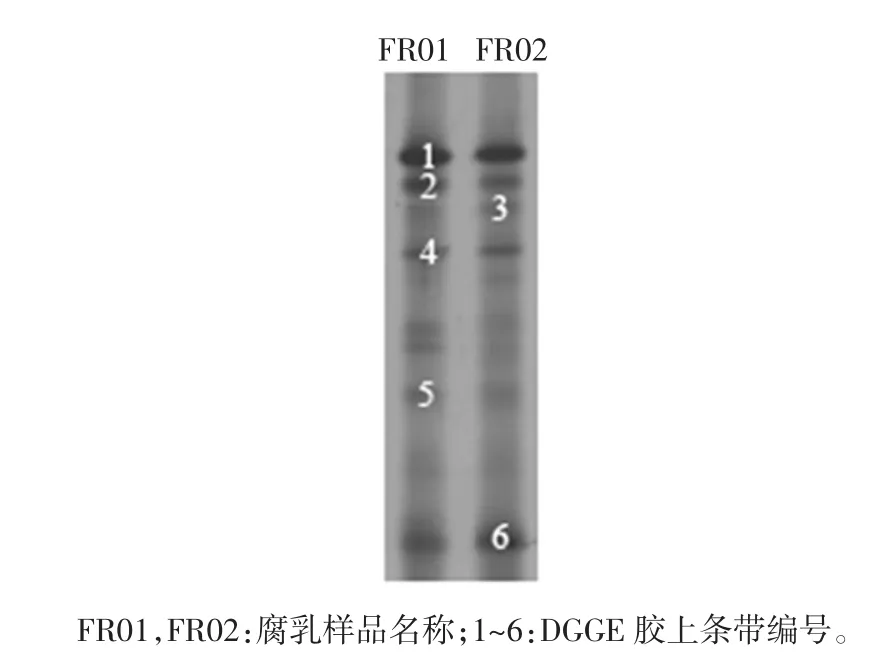
图1 腐乳细菌PCR-DGGE 图谱Fig.1 PCR-DGGE analysis of bacteria in sufu
由图1可知,图谱中共出现6 条明亮的条带,同时条带 1、2、4、5 和 6 是两个样品的共有条带,说明不同腐乳样品中存在一些共有的细菌种群,其中条带1 最亮且存在于两个样品中,说明此条带所对应的细菌在腐乳中发挥着重要的作用。值得一提的是各条带亮度不同,条带2 和条带5 在FR01 中亮度较高,条带6 在FR02 中亮度较高,表明在细菌种群丰富度存在差异。而条带3 仅存在FR02 样品中,说明不同腐乳样品中存在着不同的细菌菌群。进一步将各条带进行序列分析,结果如表1所示。

表1 腐乳细菌DGGE 比对结果Table 1 Blast results of bacteria DGGE in sufu
由表1可知,各条带序列与数据库中16S rRNA序列均具有较高的相似度。其中条带1 和2 为植物乳杆菌(Lactobacillus plantarum),条带3 为嗜盐芽孢杆菌属细菌(Halobacillus karajiensis),条带 4 为不动杆菌属细菌(Acinetobacter oryzae),条带5 为荧光假单胞杆菌(Pseudomonas fluorescens),条带6 为嗜盐四联球菌(Tetragenococcus halophilus)。由此可知,腐乳样品中微生物构成具有多样性,而且隶属于乳杆菌属(Lactobacillus)的植物乳杆菌(Lactobacillus plantarum)为优势细菌。陈颖慧[20]利用PCR-DGGE 技术对4 种不同品牌腐乳中的细菌多样性进行了研究,结果发现乳酸菌属、藤黄微球菌(Micrococcus luteus)和屎肠球菌(Enterococcus Faecium)在各腐乳样品中均存在,进一步发现乳酸杆菌属(Lactobacillus)为优势细菌。陈浩等[21]利用构建16S rRNA 基因文库的方法对豆酱样品进行研究, 结果发现嗜盐四联球菌(Tetragenococcus halophilus)为优势细菌,同时不动杆菌(Acinetobacter baylyi)也被检测到存在于样品中。王夫杰等[22]在青方腐乳中分离出植物乳杆菌(Lactobacillus plantarum)和干酪乳杆菌(Lactobacillus casei)等乳酸菌。这与上述结论相一致。
本研究进一步将各条带序列与模式菌株序列进行系统发育树的构建,结果图2所示。

图2 腐乳中细菌系统发育树Fig.2 Phylogenetic tree of bacteria in sufu
由图2可知,系统发育树被分为2 大分支,其中条带1 和2 与Lactobacillus plantarum 聚为一类,条带6与Tetragenococcus halophilus 聚为一类,条带 3 与Halobacillus karajiensis 聚为一类。而条带4 和条带5聚集在另外一支上,其中条带4 和Acinetobacter oryzae聚为一类,条带5 和Pseudomonas fluorescens 聚为一类。由此可知。不同腐乳样品中细菌群落构成存在一定的差异性。
2.2 序列丰富度及多样性分析
通过Miseq 高通量测序发现,两个样品共产生78 412 条高质量16S rRNA 序列。本研究采用两步UCLUST 算法分别以100%和97%的相似度进行序列划分并建立OTU,首先根据100%相似度进行序列划分得到33 254 条序列,根据97%相似度进行OTU 划分后得到2 317 个OTU,平均每个样品1 158 个OTU。当样品测序量为36 219 条序列时,FR02 样品具有最大的细菌物种丰富度同时细菌多样性最高,其Chao 1指数为611,Shannon 指数为5.64。进一步通过稀疏曲线和香农指数曲线图对各样品产生的数据量来判定是否满足后续生物信息学分析,其结果如图3所示。
由图3A 可知,随着测序量不断的增加,各样品被发现OTU 的数量也随之增加,而由图3B 可知,当序列数达到10 000 条时,各样品的香农多样性曲线已处于饱和状态,由此可知随着测序序列数的增加,尽管会有新的细菌种系型出现,但其多样性不再发生变化,可以反映样品中绝大多数微生物物种信息。因而本研究中每个样品产生的序列数是可以将样品中细菌微生物多样性表现出来,同时可以满足后续生物信息学分析需求。
2.3 基于不同分类地位腐乳样品核心细菌菌群相对含量分析
纳入本研究的序列被鉴定为14 个门,24 个纲,53 个目,84 个科,145 个属,其中只有5.3%的序列不能鉴定到属水平。研究发现腐乳样品中平均相对含量>1%的细菌门为变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)和硬壁菌门(Firmicutes),其含量分别为80.45%、10.7%和6.81%。同时在2 个样品中隶属于变形菌门(Proteobacteria)的细菌相对含量分别为77.29%和83.61%,隶属于拟杆菌门(Bacteroidetes)的细菌相对含量为17.19%和4.22%,而隶属于硬壁菌门(Firmicutes)的细菌相对平均含量为3.76%和9.87%。由此可知,在门水平上各样品中的微生物存在差异,本试验进一步对各样品在属水平上进行分析,如图4所示。

图3 稀疏曲线图和香农指数曲线图Fig.3 Rarefaction curve and Shannon diversity index curve

图4 腐乳中优势细菌属平均相对含量比较分析Fig.4 Comparative analysis the dominant bacterial genera with average relative abundance in sufu
由图4可知,腐乳样品中平均相对含量>5%的细菌属包括绿脓杆菌属(Pseudomonas)、不动杆菌属(Acinetobacter)、鞘氨醇杆菌属(Sphingobacterium)、布丘氏菌属(Buttiauxella)和草螺菌属(Herbaspirillum),其平均相对含量分别为33%、10.56%、8.82%、6.57%和6.32%。然而各细菌属在2 个样品中的相对含量均存在很大差异,其中绿脓杆菌属(Pseudomonas)的相对含量分别为22.56 %和43.46 %,同时不动杆菌属(Acinetobacter)的相对含量分别为18.68%和2.43%,鞘氨醇杆菌属(Sphingobacterium)的相对含量分别为16.77%和0.88%,这与PCR-DGGE 结果相一致。刘亚栋[23]利用16S rDNA 测序的方法对腐乳中的微生物多样性进行鉴定分析,结果发现变形菌门(Proteobacteria)、硬壁菌门(Firmicutes)、放线菌门(Actinobacteria)和拟杆菌门(Bacteroidetes)为腐乳样品中的优势细菌门,进一步分析发现四联球菌属(Tetragenococcus)、盐单胞菌属(Halanaerobium)、Rummeliibacillus 属和乳酸杆菌属(Lactobacillus)、不动杆菌属(Acinetobacter)和假单胞菌属(Pseudomonas)均为腐乳汇样品中的优势属,这与本文结论一致。
本试验进一步统计了OTU 在两个样品中出现次数,如图5所示。

图5 基于OTU 水平的Venn 图Fig.5 Venn diagram based on OTU level
由图5可知,在两个腐乳样品中出现1 次的OTU数量分别为1 144 个和1 432 个,分别占OTU 总数的43.8%和54.9%,序列数分别为2 183 条和5 426 条。同时核心OTU 共有34 个,占OTU 总数的1.3%,包含70 802 条序列。进一步分析发现有9 个核心OTU 的相对含量>1.5%,结果如图6所示。

图6 相对含量>1.5%的核心OTU 热图Fig.6 Heat map of the relative abundance more than 1.5%of cores OTUs
由图6可知,OTU874 和 OTU1711 隶属于绿脓杆菌属(Pseudomonas),OTU1671 和 OTU894 隶属于不动杆菌属(Acinetobacter),OTU577 隶属于沙雷菌属(Serratia),OTU1973隶属于短波单胞菌属(Brevundimonas),OTU1907 隶属于草螺菌属(Herbaspirillum),OTU635 隶属于鞘氨醇杆菌属(Sphingobacterium),而OTU346 未鉴定在属水平,只鉴定在Enterobacteriaceae。同时由图6可以发现,OTU874、OTU635 和 OTU1671 在FR01 中含量较高,其相对含量分别为16.89 %、16.73 %和 11.32 %,同时 OTU874 和 OTU1907 在FR02 中含量较高,其相对含量分别为28.56%和11.85%。
2.4 腐乳中乳酸菌分离鉴定结果及系统发育分析
通过传统微生物培养手段结合16S rDNA 测序方法对腐乳样品中的乳酸菌进行分离与鉴定,其鉴定结果和数据库中模式菌的系统发育树如图7所示。
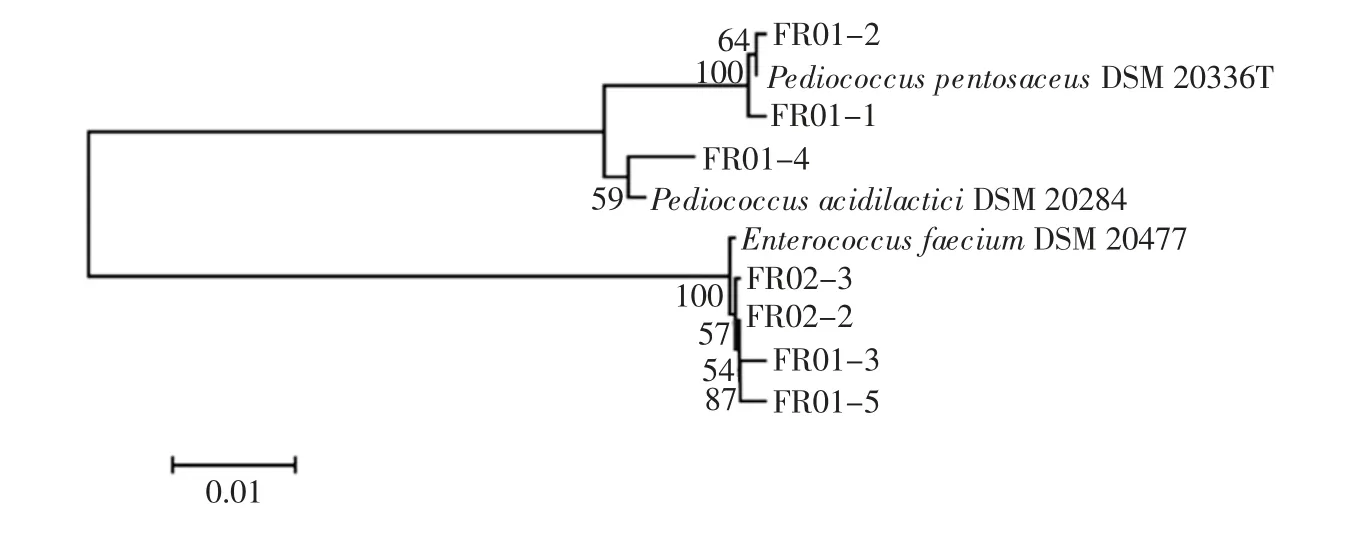
图7 腐乳中乳酸菌系统发育树Fig.7 Phylogenetic tree of Lactobacillus in sufu
由图7可知,在腐乳样品中共分离出7 株乳酸菌,其中2 株为戊糖片球菌(Pediococcus pentosaceus),1株为乳酸片球菌(Pediococcus acidilactici),4 株为屎肠球菌(Enterococcus faecium)。由此可知,腐乳中乳酸菌种类具有多样性。
3 结论
本文使用PCR-DGGE 技术和Illumina Miseq 第二代高通量测序技术相结合的手段对恩施地区腐乳中的微生物群落组成及多样性进行解析,同时利用传统纯培养的方法对其乳酸菌资源进行发掘。结果表明:隶属于变形菌门的绿脓杆菌属、不动杆菌属、鞘氨醇杆菌属、布丘氏菌属和草螺菌属为腐乳样品中的优势细菌属,同时PCR-DGGE 技术与传统微生物培养方法显示腐乳中的微生物资源较为丰富且具有多样性。通过本研究的开展,可为传统大豆发酵食品提供优秀的菌种资源,同时更好地促进其产业化生产。
