葡萄miR159s靶基因的鉴定及其应答GA在果实不同组织的调控作用
2019-08-27张文颖韩旭朱旭东解振强纠松涛黄雨晴贾海锋房经贵王晨
张文颖,韩旭,朱旭东,解振强,纠松涛,黄雨晴,贾海锋,房经贵,王晨
葡萄miR159s靶基因的鉴定及其应答GA在果实不同组织的调控作用
张文颖1,韩旭1,朱旭东1,解振强2,纠松涛3,黄雨晴1,贾海锋1,房经贵1,王晨1
(1南京农业大学园艺学院,南京 210095;2江苏农林职业技术学院,江苏句容 212400;3上海交通大学农业与生物学院,上海 200240)
【】鉴定葡萄miR159家族成员及其靶基因,明确葡萄miR159家族成员及其靶基因应答外源赤霉素(GA)在无核葡萄果实不同组织发育过程中的作用。【】以‘白罗莎里奥’葡萄(Rosario Bianco)为试材,利用miR-RACE和PCR技术克隆鉴定VvmiR159a/b/c的成熟体及前体序列;通过PsRNATarget软件预测VvmiR159s的靶基因,利用生物信息学软件对其进行系统进化及保守结构域分析;通过启动子作用元件分析预测VvmiR159s及其靶基因的潜在功能;采用RLM-RACE和PPM-RACE验证VvmiR159s对靶基因的裂解作用;利用qRT-PCR鉴定其应答外源GA在葡萄果实不同组织发育过程中的时空表达模式。【】从‘白罗莎里奥’葡萄果实中克隆获得VvmiR159a/b/c的成熟体序列、前体序列并折叠发夹结构。鉴定到VvmiR159s的四条靶基因(、、、),其中VvmiR159s与匹配程度最高,与匹配程度最低。靶基因系统进化及保守结构域分析显示,4条靶基因与其他物种同源基因具有较高同源性。VvmiR159s前体基因及其靶基因启动子的激素响应元件均以赤霉素和水杨酸响应元件为主,表明其可能主要通过应答这两种激素参与调控葡萄的生长发育过程。VvmiR159s在不同组织中的表达具有时空特异性,其中VvmiR159c在果皮和果肉中的表达趋势不同,在果皮中VvmiR159a/b/c与的表达水平呈负相关;而在果肉中,VvmiR159a/b与和的表达模式相反。此外,在葡萄果皮、果肉中GA处理可显著下调VvmiR159a/c的表达,但却在幼果果肉中上调VvmiR159b的表达。表明葡萄miR159家族成员在果实不同组织中通过不同模式应答GA信号参与果实发育的调控。【】葡萄miR159家族含有VvmiR159a/b/c 3个成员;3个成员均可切割、、及4个靶基因;VvmiR159a/b/c及其4个靶基因可能以不同的GA应答模式参与调控葡萄果皮与果肉的发育。
葡萄;miR159;靶基因;GA;进化特性;基因表达
0 引言
【研究意义】MiR159是一类非常保守的miRNA,近年的研究揭示其在植物的生长发育、激素信号的应答及逆境响应等方面具备重要的调节作用[1-3]。葡萄(L.)是世界性的重要果树作物,无核是鲜食和制干葡萄的优良性状之一[4-5],而赤霉素(gibberellic acid,GA)是诱导葡萄无核的关键激素[6],但miR159在GA诱导葡萄无核果实发育过程中的作用尚未明确。鉴定葡萄中miR159家族成员及其靶基因,明确其在葡萄果实不同组织中应答GA的表达模式,可从非编码基因角度为今后葡萄的遗传改良育种提供新的理论依据。【前人研究进展】MiR159家族是一个在植物中分布广泛,相对较为古老的miRNA家族[7]。研究表明,miR159家族成员在不同物种中的数量差异很大,其在不同组织的表达水平也存在显著差异[3,8]。Achard等[9]在miR159调控拟南芥花器官发育的研究中发现miR159能够响应GA信号,调节开花时间并干扰花药发育,在大麦中发现其与花药长度和雄性不育有关[10],在水稻中也发现相同作用[11]。葡萄方面,笔者课题组前期研究鉴定了大量的葡萄miRNAs[12-16],其中葡萄miR159的研究主要集中在花发育上,如王梦琦等[16]发现赤霉素能够抑制miR159对靶基因的裂解作用,影响下游基因的表达,从而调控葡萄的开花过程。同时,笔者也发现葡萄miR159家族在果实中具有一定的表达。【本研究切入点】笔者课题组前期的研究表明miR159s能够响应GA信号调控葡萄花的发育,但葡萄miR159家族成员及其靶基因的系统鉴定,以及miR159s介导其靶基因应答GA信号调控葡萄无核果实发育的研究尚无相关报道,miR159s是否参与了葡萄无核果实的发育及其调控方式尚未明确。【拟解决的关键问题】以miRBase数据库为基础,鉴定葡萄miR159家族成员及其靶基因,明确葡萄miR159家族成员及其靶基因在果皮、果肉中的表达特征以及应答外源GA的时空表达模式。
1 材料与方法
1.1 材料与处理
本试验以6年生优质‘白罗莎里奥’葡萄为试验材料,于花前10 d(2017年5月1日)用50 mg·L-1的GA3浸蘸葡萄花序30 s,设置清水处理作为对照。每组处理随机选定12株长势较为一致的植株,每4株为1个重复,共设3次重复。分别于幼果期(花后10 d,5月28日)、硬核期(花后35 d,6月22日)、第2次膨大期(花后60 d,7月17日)、近成熟期(花后85 d,8月11日)采集葡萄果实样品,将果皮、果肉分离,用液氮冻样,储存于-80℃冰箱备用。
M-MLV反转录试剂盒购自Takara公司,荧光定量PCR试剂盒购自TRAN全式金生物公司,引物由上海生工生物工程股份有限公司合成,引物序列见表1。
1.2 葡萄miR159家族成员成熟体序列及前体序列的克隆鉴定
在miRBase数据库(http://www.mirbase.org/)搜索并下载葡萄miR159a/b/c(VvmiR159a/b/c)的前体与成熟体序列。基于miRBase中VvmiR159a/b/c的成熟体序列,参照Wang等[13]的方法设计特异引物,利用miR-RACE技术在‘白罗莎里奥’葡萄果实中克隆VvmiR159a/b/c的成熟体序列,并测序鉴定其精确序列。利用下载的VvmiR159a/b/c前体序列比对葡萄基因组,并分别向上、下游扩展,预测葡萄miR159a/b/c的前体基因序列,设计特异引物进行PCR扩增(表1),反应体系为50 μL:上、下游引物各2 μL,cDNA 2 μL,10×PCR buffer(Mg2+plus)5 μL,dNTPMixture 4 μL,Ex-酶0.50 μL,ddH2O 34.5 μL。反应程序:95℃5 min;95℃30 s,58℃30 s,72℃1 min,35个循环;72℃10 min,4℃下保存。产物用1.5%的琼脂糖凝胶电泳检测后回收目的片段,连接至pMD19T载体进行TA克隆,DNA测序由上海生工生物工程股份有限公司完成。
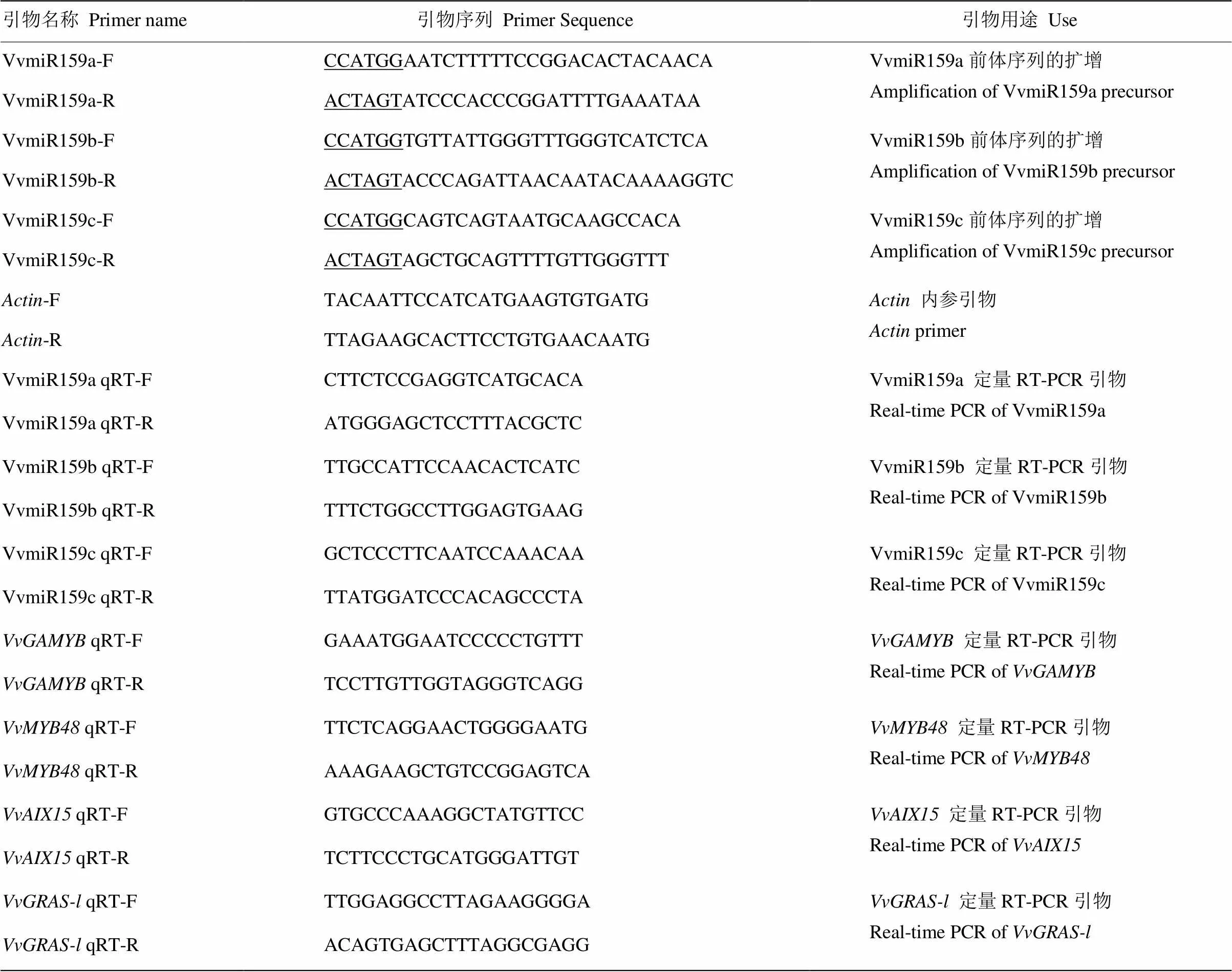
表1 引物序列及用途
下划线部分CCATGG和ACTAGT分别为添加的I、Ⅰ酶切位点
The underlined sequences CCATGG and ACTAGT in the primers represent restriction enzyme sites ofⅠ andⅠ, respectively
1.3 葡萄miR159s靶基因预测及生物信息学分析
基于鉴定的VvmiR159a/b/c成熟体序列,通过psRNATarget在线软件(http://plantgrn.noble.org/ psRNATarget/analysis)预测其靶基因。在葡萄基因组数据库Grape Genome(http://genomes.cribi.unipd.it/grape/)对预测的靶基因进行功能注释。
根据预测的4个靶基因cDNA编码的氨基酸序列,分别与拟南芥(Arabidopsis thaliana)、杨树(Populus trichocarpa)、水稻(Oryza sativa Japonica Group)、胡桃(Juglans regia)、桃(Prunus persica)、大豆(Glycine max)、梅(Prunus mume)、番茄(Solanum lycopersicum)和甜樱桃(Prunus avium)进行同源比对。使用MEGA 7.0.21在线软件构建系统进化树;利用在线工具MEME(http://meme-suite.org/tools/meme)鉴定蛋白保守基序,其参数设置基序数量15个,其余参数为默认条件;采用在线数据库(CDD,https//www.ncbi.nlm.nih.gov/Structure/cdd/cdd.shtml)进行保守结构域分析。
1.4 VvmiR159a/b/c及其靶基因启动子作用元件分析
从葡萄基因组中找出VvmiR159a/b/c前体基因上游1 500 bp及4个靶基因转录起始位置上游1500 bp的序列,参考FAN等[17]的方法,利用PlantCARE在线数据库(http//bioinformatics.psb.ugent.be/webtools/plantcare/html/),进行启动子顺式作用元件分析。
1.5 RLM-RACE和PPM-RACE扩增确定VvmiR159s剪切靶基因位点
用于PPM-RACE反应的模板为加ploy(A)尾巴的mRNA反转录合成的cDNA,通用引物GeneRACE 3′(5′-ATTCTAGAGGCCGAGGCGGCCGACATG-3′)。用于RLM-RACE反应的模板为加接头的mRNA反转录成的cDNA,通用引物GeneRACE 5′(5′-GGAC ACTGACATGGACTGAAGGAGTA-3′)。详细步骤参考Wang等[14]的方法。
1.6 VvmiR159家族成员及其靶基因的表达分析
采用CTAB法提取葡萄不同发育时期不同组织的总RNA,并以提取的总RNA为模板依照M-MLV反转录试剂盒说明书合成cDNA。利用Primer3 Input(http://bioinfo.ut.ee/primer3-0.4.0/primer3/)设计引物(表1),分别以mRNA和小分子RNA反转录的cDNA为模板,以为内参基因,对VvmiR159s前体基因及4个靶基因进行qRT-PCR检测。扩增体系及程序根据试剂盒说明书进行。3次生物学重复,根据公式2-ΔΔCt计算各基因的相对表达量,采用DPS 7.05软件对结果进行统计及差异显著性分析。
2 结果
2.1 GA处理对不同时期葡萄果实发育的影响
GA3处理后,‘白罗莎里奥’葡萄果实纵径变长,各时期果实无明显的果核,纵切后观察到种子区域仅留下一条木质化的细线(图1),果实无核率达到92%。而清水处理的葡萄种子正常发育,种子中胚发育正常,且存在明显的木质化过程(图1)。可见GA3处理可高效诱导‘白罗莎里奥’葡萄果实无核。
2.2 葡萄miR159家族成员成熟体及前体序列的鉴定
为探究miR159家族成员在‘白罗莎里奥’葡萄中的真实序列,对其成熟体和前体序列进行克隆鉴定(图2)。通过miR-RACE克隆获得VvmiR159a/b/c的成熟体序列(图2-A),其中miR159a/b的成熟体序列比miRBase中的序列少一个碱基‘C’,而miR159c的序列与miRBase中的同源序列相同(图2-C)。克隆测序鉴定VvmiR159a/b/c的前体序列分别为495、498和555 bp(图2-B),且3种miRNA均包含完整的茎环结构,且VvmiR159a/b/c成熟体都位于其茎环结构的3′茎臂中(图2-D),表明VvmiR159家族的3个成员在其前体上的位置高度保守。

10DAF:花后10 d;35DAF:花后35 d;60DAF:花后60 d;85DAF:花后85 d

A:VvmiR159a/b/c 3'-miR-RACE和5'-miR-RACE的PCR产物;B:VvmiR159a/b/c前体序列的PCR产物;C:VvmiR159s克隆序列与MiRBase中的同源序列比较;D:VvmiR159s前体的二级茎环结构
2.3 葡萄VvmiR159家族靶基因预测分析
基于克隆鉴定的VvmiR159a/b/c成熟体序列,利用psRNATarget在线软件从葡萄全基因组水平预测到其4条靶基因、、和(表2),分析发现其靶区均在4个靶基因的CDS区,且其作用方式均为裂解作用(表2)。进一步比较VvmiR159s与靶基因的匹配程度,其中VvmiR159a/b与匹配程度最高,VvmiR159a/b与错配最多(图3),表明它们之间的作用强度存在潜在差异。
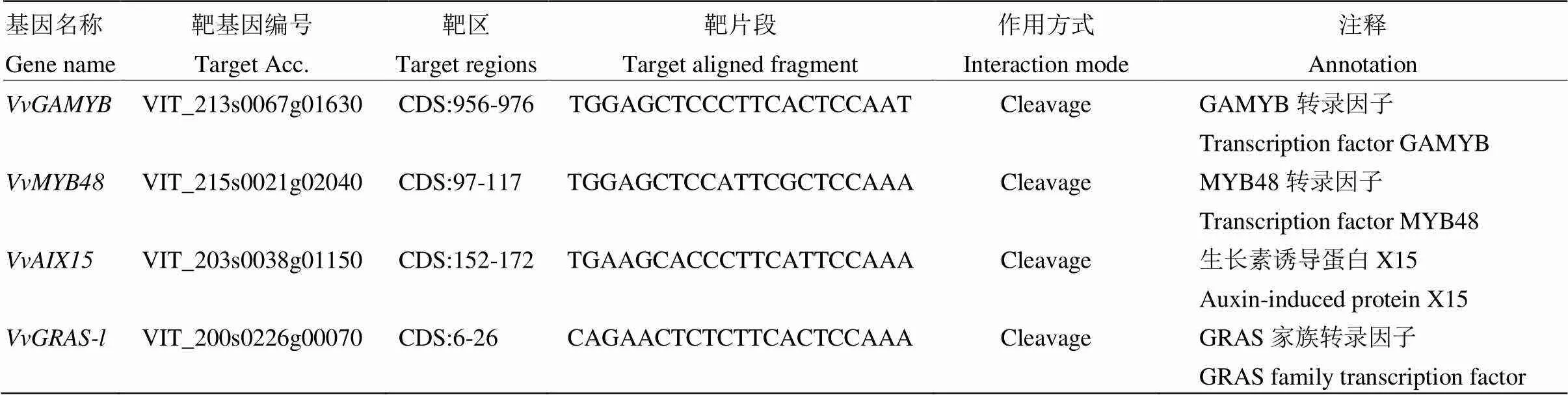
表2 VvmiR159a/b/c的靶基因预测

图3 VvmiR159s与靶基因的匹配程度
2.4 靶基因系统发育进化分析
利用MEGA 7.0.21对同源比对结果进行进化分析,对比发现VvGAMYB与核桃的亲缘关系较近(图4-A),VvMYB48和大豆的同源性较高(图4-B),VvAIX15与杨树的亲缘关系最近(图4-C),而VvGRAS-1与拟南芥和水稻具有较近的亲缘关系(图4-D),且4个靶基因均与桃的亲缘关系较远(图4)。

图4 靶基因的进化树分析
2.5 靶蛋白保守结构域分析
根据系统进化结果,对葡萄和其他物种的同源靶蛋白进行保守结构域分析,结果表明4个靶蛋白在不同物种间的结构域高度保守。其中除番茄SlGAMYB外,其余物种GAMYB蛋白均含有R2R3结构域和Box1、Box2、Box3结构域(图5-A);10个物种的MYB48蛋白均含有R2R3结构域(图5-B);除水稻OsAIX15外,其余物种均含有Auxin_inducible结构域(图5-C);GRAS-1蛋白均含有GRAS保守结构域(图5-D),表明4个靶蛋白在不同物种间可能具有一定的功能保守性。
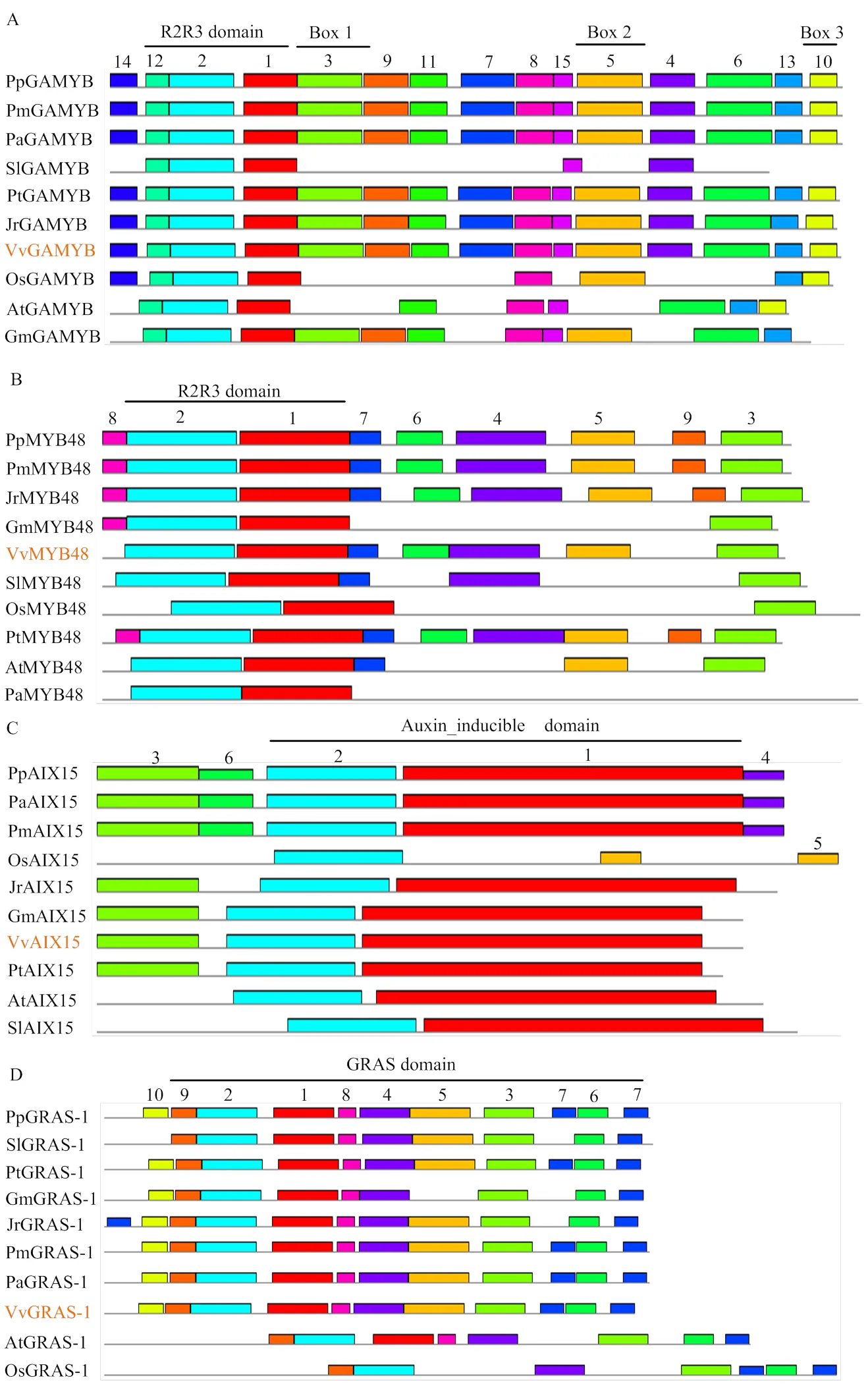
图5 靶蛋白保守结构域分析
2.6 VvmiR159s前体基因及其靶基因启动子作用元件分析
葡萄VvmiR159s前体基因及其靶基因的启动子作用元件除基本作用元件TATA-box和增强子CAAT-box外,主要包括光信号响应、激素响应、组织特异性响应、胁迫响应及周期节律相关元件(图6-B)。对比VvmiR159s前体基因及其靶基因启动子不同作用元件的个数发现,光响应元件数量最多,胁迫响应和组织特异性响应元件次之,而周期节律相关元件数量最少,甚至在部分启动子中缺少周期节律相关元件,激素响应元件数量较少但广泛分布在不同启动子中(图6-B)。
为认识VvmiR159家族成员及其靶基因对激素作用的响应情况,进一步对比了它们启动子的激素响应元件(图6-A)。其中GA响应元件广泛存在于之外的启动子中,水杨酸(salicylicacid,SA)应答元件分布在以外的启动子序列之中,乙烯(ethylene,ET)作用元件只分布在VvmiR159a和VvmiR159b中,脱落酸(abscisic acid,ABA)应答元件仅存在于中,生长素响应元件仅存在于中,表明VvmiR159s及其靶基因可能分别通过应答不同激素参与调控葡萄果实的发育。

A:VvmiR159s及其4个靶基因启动子不同类型顺式作用元件汇总;B:VvmiR159s及其4个靶基因启动子中激素相关的顺式作用元件
2.7 VvmiR159s裂解4个靶基因mRNA的剪切位点验证
利用RLM-RACE和PPM-RACE技术对VvmiR159s剪切4个靶基因的作用位点进行了鉴定。检测到VvmiR159s在上只有1个剪切位点,在和上有2个剪切位点,在上有3个剪切位点,但4个靶基因的裂解位点均位于其miRNA 5′端第9—12位碱基之间所匹配的靶点上(图7),证明4个靶基因是VvmiR159s的真实靶基因。
2.8 果实发育过程中VvmiR159s及其靶基因的时空表达特征
VvmiR159s及其4个靶基因在不同发育时期果皮及果肉中的表达水平存在时空特异性(图8)。VvmiR159a/b/c在葡萄果皮中的表达趋势基本一致,均为先升后降,在硬核期(花后35 d)表达量最高。而靶基因在果皮中的表达趋势与VvmiR159s具有明显差异,为先降后升,在花后35、60 d的表达量很低,与VvmiR159a/b/c呈现一定的负相关(图8)。在葡萄果肉中,VvmiR159a/b均呈现先升后降的表达趋势,在花后60 d表达量最高,而VvmiR159c的表达趋势为逐渐降低(图8),说明VvmiR159s的表达具有组织特异性。对比发现,在果皮中,VvmiR159a/b/c与的表达不呈现典型的负相关,而呈现正相关,在果肉中,的表达水平与VvmiR159a/b也呈正相关,表明VvmiR159a/b/c对的负调控作用不明显。而果肉中和的表达水平均与VvmiR159a/b呈现不同程度的负相关(图8),说明VvmiR159a/b可能通过抑制和的表达参与调控葡萄果肉的发育。

箭头表示剪切位点,数字代表克隆PCR产物在不同位置的切割片段的频率
2.9 VvmiR159及其靶基因在葡萄果实不同组织中应答GA的模式鉴定
为认识VvmiR159s及其靶基因在葡萄果实发育过程中应答GA的模式,对其在GA3处理与对照中的表达水平进行了研究(图9)。在果皮中,GA3处理抑制了VvmiR159s及4个靶基因的表达,尤以果实硬核期(花后35 d)和第二次膨大期(花后60 d)的下调水平最为显著;且GA3处理后,VvmiR159a/ b/c和的表达水平呈现一定的负相关(图9)。在果肉中,除VvmiR159b和在特定时期被GA3上调表达外,其他基因均被GA3处理显著下调(图9),表明VvmiR159s及靶基因对赤霉素的应答模式存在差异,同时也说明VvmiR159s及其靶基因可能以不同的GA3应答模式参与调控葡萄果皮与果肉的发育过程。
3 讨论
在miRNA最初被发现的十多年里,植物miR159并没有受到足够的重视,针对miR159的研究主要是通过真菌、水生植物等生物的表达谱进行[18]。自从拟南芥中首个miRNA成功克隆后[19],植物miRNA的研究得到了空前的发展,关于植物miR159的报道也日益增多[20-21]。研究表明,miR159家族在不同植物如拟南芥[22]、小麦[23]、水稻[24]、番茄[25]中具有高度保守性,且在植物的器官发生、花芽分化、激素响应和逆境生理等方面具有重要作用。但相关研究主要集中在miR159调控花器官的发育上,在果实方面的研究较少。
MiR159家族成员在不同物种间的数量分布差异很大,拟南芥miR159家族有4个成员,水稻中有7个成员,而番茄和烟草中均只有1个成员[26-28]。本研究发现miR159在葡萄中含有3个成员(VvmiR159a/b/c),与拟南芥中的成员数量相同。研究表明,植物miR159家族成员的靶基因主要为转录因子、转座因子、假定蛋白、C3HC4锌结合结构域蛋白、表达蛋白及一些其他未知蛋白等[26],但不同物种间的靶基因存在差异,如拟南芥、水稻和玉米含有10—24个靶基因[29],而本研究在葡萄miR159家族中鉴定了4个靶基因(、、和),这说明不同物种miR159的靶基因并不完全相同,其中值得注意的是,转录因子是GA信号转导途径中的一个正向调控因子,在植物开花和种子发育过程中具有重要作用[16]。启动子作用元件分析表明VvmiR159s及其4个靶基因的光响应作用元件数量最多,推测可能与光合作用是植物所必需的有关,进一步分析发现,除外,其他启动子均含有GA响应元件,表明VvmiR159s及其靶基因可能参与GA信号传导调控葡萄无核果实的发育。

*代表差异显著(P<0.05)。下同 * represented significant difference (P<0.05). The same as below
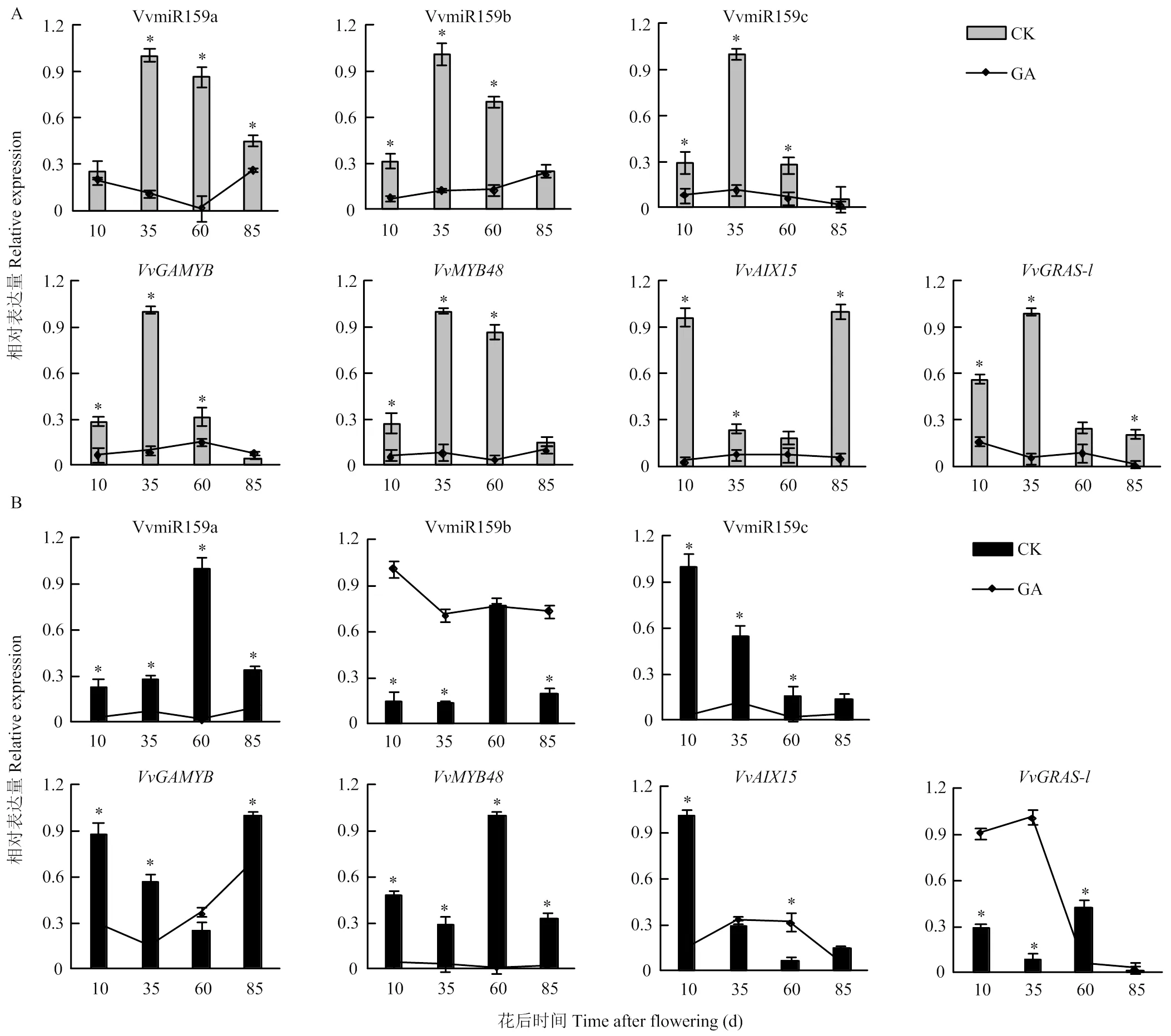
A:果皮Berry pericarp;B:果肉Berry flesh
笔者课题组之前的研究表明,大约85.2%的已知miRNA在其靶基因上具有单个剪切位点,而其余14.8%的已知miRNA具有2—3个剪切位点,并且这些剪切位点主要定位于其miRNA 5′端第9—11个核苷酸位置[15],本研究发现,葡萄miR159家族4个靶基因的裂解位点均位于其miRNA 5′端第9—12位碱基之间所匹配的靶点上,类似于之前的报道,即miRNA在其5'末端第9、10、11、12位特定位点剪切靶标[14-15]。李贺等[30]在草莓中对miR159等6种miRNA在不同组织中的表达差异研究发现,miR159在草莓根系、花托、茎尖和叶片4种器官中表达差异不大。而在本研究中对miR159a/b/c及靶基因的表达分析发现,在不同时期果皮及果肉中,VvmiR159s各成员的表达量具有显著差异,说明miR159在不同物种中的表达可能具有特异性。在对葡萄花器官的研究中发现,GA处理能够抑制miR159的表达[16],在草莓中,GA3处理导致的表达下调[31]。本研究也发现,除VvmiR159b在果肉中被GA3上调表达外,其余成员在果皮、果肉中被GA3处理后均下调其表达,表明miR159参与赤霉素调控过程不仅可能具有时空特异性,还可能具有组织特异性。本研究结果为进一步开展VvmiR159s及其靶基因如何响应GA信号调控葡萄无核果实发育的分子机制研究奠定了重要基础。
4 结论
从‘白罗莎里奥’葡萄果实中克隆获得miR159家族VvmiR159a/b/c 3个成员;3个成员均可剪切、、及4个靶基因,其中VvmiR159a/b/c对和的负调控作用最为明显。此外,VvmiR159a/b/c及其4个靶基因可能以不同的GA应答模式参与调控葡萄无核果实的发育。
[1] GUO C K, XU Y M, SHI M, LAI Y M, WU X, WANG H S, ZHU Z J, POETHIG S, WU G. Repression of miR156 by miR159 regulates the timing of the juvenile-to-adult transition in., 2017, 29: 1293-1304.
[2] ZHENG Z H, REICHEL M, DEVESON I, WONG G G, LI J Y, MILLAR A. Target RNA secondary structure is a major determinant of miR159 efficacy., 2017, 174(3): 1764.
[3] XUE T, LIU Z H, DAI X H, XIANG F N. Primary root growth inis inhibited by the miR159 mediated repression of,and., 2017, 262: 182.
[4] WANG X C, ZHAO M Z, WU W M, KORIR N K, QIAN Y M, WANG Z W. Comparative transcriptome analysis of berry-sizing effects of gibberellin (GA3) on seedlessL., 2017, 39(5): 493-507.
[5] ACHEAMPONG A K, ZHENG C L, HALALY T, GIACOMELLI L, TAKEBAYASHI Y, JIKUMARU Y, KAMIYA Y, LICHTER A, OR E. Abnormal endogenous repression of GA signaling in a seedless table grape cultivar with high berry growth response to GA application., 2017, 8: 850.
[6] WANG M Q, SUN X, WANG C, CUI L W, CHEN L D, ZHNAG C B, SHANGGUAN L F, FANG J G. Characterization of miR061 and its target genes in grapevine responding to exogenous gibberellic acid., 2017, 17(5): 537-549.
[7] LI H, WANG Y, WANG Z, GUO X, WANG F, XIA X J, ZHOU J, SHI K, YU J Q, ZHOU Y H. Microarray and genetic analysis reveals that csa-miR159b plays a critical role in abscisic acid-mediated heat tolerance in grafted cucumber plants., 2016, 39(8): 1790-1804.
[8] ZHAO Y F, WEN H L, TEOTIA S, DU Y X, ZHNAG J, LI J Z, SUN H Z, TANG G L, PENG T, ZHAO Q Z. Suppression of microRNA159 impacts multiple agronomic traits in rice (L.)., 2017, 17(1): 215.
[9] ACHARD P, HERR A, BAULCOMBE D C, HARBERD N P. Modulation of floral development by a gibberellin-regulated microRNA., 2004, 131(14): 3357-3365.
[10] MURRAY F, KALLA R, JACOBSEN J V, GUBLER F. A role forin anther development., 2003, 33(3): 481-491.
[11] MATSUOKA M. GAMYB controls different sets of genes and is differentially regulated by microRNA in aleurone cells and anther., 2006, 47(3): 427-444.
[12] WANG C, SHNAGGUAN L F, NICHOLAS K K, WANG X C, HAN J, SONG C N, FANG J G. Characterization of microRNAs identified in a table grapevine cultivar with validation of computationally predicated grapevine miRNAs by miR-RACE., 2011, 6(7): e21259.
[13] WANG C, WANG X C, NICHOLAS K K, SONG C N, ZHANG C Q, LI X Y, HAN J, FANG J G. Deep sequencing of grapevine flower and berry short RNA library for discovery of novel microRNAs and validation of precise sequences of grapevine microRNAs deposited in miRBase., 2011, 143(1): 64-81.
[14] WANG C, HNA J, NICHOLAS K K, WANG X C, LIU H, LI X Y, LENG X P, FANG J G. Characterization of target mRNAs for grapevine microRNAs with an integrated strategy of modified RLM-RACE, newly developed PPM-RACE and qPCRs., 2013, 170(10): 943-957.
[15] WANG C, LENG X P, ZHANG Y Y, KAYESH E, ZHANG Y P, SUN X, FANG J G. Transcriptome-wide analysis of dynamic variations in regulation modes of grapevine microRNAs on their target genes during grapevine development., 2014, 84: 269-285.
[16] 王梦琦, 解振强, 孙欣, 李晓鹏, 朱旭东, 王晨, 房经贵. 葡萄miR159及其靶基因在花发育过程中的作用分析. 园艺学报, 2017, 44(6): 1061-1072.
WANG M Q, XIE Z Q, SUN X, LI X P, ZHU X D, WANG C, FANG J G. Function analysis of miR159 and its target genein grape flower development., 2017, 44(6): 1061-1072. (in Chinese)
[17] FAN S, ZHANG D, XING L B, QI S Y, DU L S, WU H Q, SHAO H X, LI Y M, MA J J, HAN M Y. Phylogenetic analysis ofgene family and characterization of its expression in response to flower induction in., 2017, 292(4): 755-771.
[18] REINHART B J, WEINSTEIN E G, RHOADES M W, BARTEL B, BARTEL D P. MicroRNAs in plants., 2002, 16(13): 1616-1626.
[19] CARRINGTON J C, LLAVE C, XIE Z, KASSCHAU K D, CARRINGTON JC. Cleavage of scarecrow-like mRNA targets directed by a class ofmiRNA., 2002, 297(5589): 2053-2056.
[20] RHOADES M W, REINHART B J, LIM L P, BURGE C B, BARTEL B, BARTEL D P. Prediction of plant microRNA targets., 2002, 110(4): 513-520.
[21] PATADE V Y, SUPRASANNA P. Short-term salt and PEG stresses regulate expression of microRNA, miR159 in sugarcane leaves., 2010, 13(3): 177-182.
[22] MAHER C, STEIN L, WARE D. Evolution ofmicroRNA families through duplication events., 2006, 16(4): 510-519.
[23] FENG H, ZHANG Q, LI H Y, WANG X J, DUAN X Y, WANG B, KANG Z S. VsiRNAs derived from the miRNA-generating sites of pri-tae-miR159a based on the BSMV system play positive roles in the wheat response tof. sp.through the regulation ofexpression., 2013, 68(7): 90-95.
[24] GUO C, LI L, WANG X F, LIANG C. Alterations in siRNA and miRNA expression profiles detected by deep sequencing of transgenic rice with siRNA-mediated viral resistance., 2015, 10(1): e0116175.
[25] NAQVI A R, HAQ Q M, MUKHERJEE S K. MicroRNA profiling of tomato leaf curl new delhi virus (tolcndv) infected tomato leaves indicates that deregulation of miR159/319 and miR172 might be linked with leaf curl disease., 2010, 7(1): 1-16.
[26] 陈旭, 王嘉毅, 张梓浩, 王天池, 赖钟雄, 林玉玲. 植物miR159家族成员分子特性及其进化规律研究. 西北植物学报, 2017, 37(7): 1286-1295.
CHEN X, WANG J Y, ZHNAG Z H, WANG T C, LAI Z X, LIN Y L. Molecular characteristics and evolution of miR159 family member., 2017, 37(7): 1286-1295. (in Chinese)
[27] BURKLEW C E, XIE F, ASHLOCK J, ZHNAG B H. Expression of microRNAs and their targets regulates floral development in tobacco ()., 2014, 14(2): 299-306.
[28] BUXDORF K, HENDELMAN A, STAV R, LAPIDOT M, ORI N, ARAZI T. Identification and characterization of a novel miR159 target not related toin tomato., 2010, 232(5): 1009-1022.
[29] WANG Y G, AN M, ZHOU S F, SHE Y H, LI W C, FU F L. Expression profile of maize microRNAs corresponding to their target genes under drought stress., 2014, 52(11/12): 474-493.
[30] 李贺, 印东升, 王志刚, 黄飞飞, 常琳琳, 张志宏. 草莓不同器官microRNA表达差异研究. 果树学报, 2009, 26(5): 632-637.
LI H, YIN D S, WANG Z G, HUANG F F, CHANG L L, ZHANG Z H. Study on the difference in expression profiles of microRNA among different organs of strawberry.2009, 26(5): 632-637. (in Chinese)
[31] CSUKASI F, DONAIRE L, CASANAL A, MARTINEZ-PRIEGO L, BOTELLA M A, MEDINA-ESCOBAR N, LLAVE C, VALPUESTA V. Two strawberry miR159 family members display developmental- specific expression patterns in the fruit receptacle and cooperatively regulate., 2012, 195(1): 47-57.
Identification of the Target Genes of VvmiR159s and their Regulation in Response to GA in Different Tissues of Grape Berry
ZHANG WenYing1, HAN Xu1, ZHU XuDong1, XIE ZhenQiang2, JIU SongTao3, HUANG YuQing1, JIA HaiFeng1, FANG JingGui1,WANG Chen1
(1College of Horticulture, Nanjing Agricultural University, Nanjing 210095;2Jiangsu Vocational College of Agriculture and Forestry, Jurong 20140, Jiangsu;3Institute of Agriculture and Biology, Shanghai Jiao Tong University, Shanghai 200240)
【】The main objective of the present research was to identify the miR159 family members and their target genes from grapevine genome, to validate the role of VvmiR159 family members and their target genes in response to exogenous GA in the development of different tissues of seedless grape berries. 【】The mature and precursor sequences of VvmiR159a/b/c were cloned and identified by miR-RACE and PCR techniques from grapevine cv. ‘Rosario Bianco’. The target genes of VvmiR159s were predicted by PsRNATarget software, and the phylogenetic and conserved domain analysis was performed by bioinformatics tools. The potential functions of VvmiR159s and their target genes were predicted by-elements analysis of their promoters. RLM-RACE and PPM-RACE verified the cleavage roles of four target genes by VvmiR159a/b/c, and the qRT-PCR method was used to detect their temporal and spatial expression patterns in different tissues of grape berry induced by exogenous GA application 【】The mature and precursor sequences of VvmiR159a/b/c were cloned from the ‘Rosario Bianco’ and further folded into the typical hairpin structures. Four target genes (,,, and) of VvmiR159s were identified, of which VvmiR159s strongly matched withthe most andwas weak. Phylogenetic evolution and conserved domain analysis of target genes showed that the four target genes had high homology with other species. All VvmiR159s and four target genes had the main hormone related-elements responsive to GA and SA, indicating that they might be involved in the regulation of grape berry growth and development mainly by responding to corresponding hormones. The expression of VvmiR159s in different tissues was spatial-temporal-specific. The expression of VvmiR159c was deviated in berry pericarp and flesh, and the expression level of VvmiR159a/b/c was negatively correlated withandexpression pattern. In addition, GA treatment VvmiR159a/c was significantly down-regulated in grape pericarp and flesh, but VvmiR159b up-regulated in young fruit flesh. These results indicated that different members of the VvmiR159 family participated in the regulation of fruit development through different modes of response to GA signaling in different tissues of the fruit. 【】The grape miR159 family contains three members of VvmiR159a/b/c; all of three members can cleave four target genes:,,and; VvmiR159a/b/c and their four target genes may participate in the regulation of grape pericarp and flesh development in different GA-responsive modes.
grape; miR159; target gene; GA; evolutionary characteristic; gene expression
2019-02-18;
2019-05-16
国家自然科学基金青年基金(31301759)、江苏省自然科学基金(BK20180113)、中央高校基本科研业务费自主创新重大专项项目(自然科学)(KYTZ201602)
张文颖,E-mail:2016104035@njau.edu.cn。
王晨,E-mail:wangchen@njau.edu.cn
(责任编辑 赵伶俐)
