人转运蛋白SR2的原核表达及其与HIV-1整合酶体外相互作用的检测
2019-08-21许晓双张大为
许晓双, 张大为, 孔 韧
(江苏理工学院 生物信息与医药工程研究所, 常州 213001)
HIV-1 整合酶(Integrase,IN)是HIV-1病毒复制过程中的关键酶,能够介导逆转录后形成的病毒cDNA与宿主细胞基因组的整合,也是研究抗艾滋病药物的重要靶标[1]。在已上市的药物中,雷特格韦(Raltegravir,RAL)、埃替格韦(Elvitegravir,EVG)和度鲁特韦(Dolutegravir,DTG)是IN催化位点抑制剂,其分子机制是结合于整合酶的催化活性口袋从而抑制整合酶的链转移反应活性[2]。近年来,由于已上市药物存在越来越严重的耐药性问题,迫切需要进行基于新机制的抗艾滋病药物研发。
HIV-1自身编码的蛋白质数量有限,需要依赖宿主细胞蛋白及其信号通路完成自身复制。人转运蛋白SR2(tansportin-SR2,TRN-SR2)是β-核素蛋白,是近年来发现的一种HIV-1整合酶辅助因子,主要参与HIV-1生命周期中整合前复合体PIC(pre-integration complex)核输入过程,促进HIV感染。通过核孔蛋白复合物将HIV-1 PIC及病毒辅助蛋白运送到细胞核的过程称为核输入过程,是一个多因子参与的复杂过程[3]。HIV-1 PIC包括多种病毒的基质蛋白、IN、病毒蛋白R、cDNA以及多种细胞辅助因子,如晶状体上皮细胞衍生生长因子(LEDGF /p75)、输入蛋白α和转运蛋白SR2[3]。因此,HIV-1核输入过程主要依赖于PIC成分或单独HIV-1蛋白与细胞核输入因子之间的蛋白质-蛋白质相互作用[4]。Demeulemeester等[5]基于TRN-SR2与HIV-1 IN蛋白-蛋白相互作用利用AlphaScreen高通量筛选技术从25 608个小分子中找到2个活性化合物,它们能够阻断HIV-1核输入过程,提示TRN-SR2与HIV-IN的相互作用是抗HIV治疗的有效靶点。因此,寻找和设计开发TRN-SR2与HIV-1 IN相互作用的高效抑制剂具有重要的发展前景和研究意义。
为了开展以TRN-SR2与HIV-1 IN相互作用为靶点的抑制剂筛选研究,本文将人源蛋白TRN-SR2进行原核表达纯化,从而获得可溶表达且具有活性的重组TRN-SR2蛋白,并利用生物膜干涉技术(Bio-Layer Interferometry,BLI)检测TRN-SR2与HIV-1 IN体外相互作用,同时采用均相时间分辨荧光(Homogeneous time resolved fluorescence,HTRF)技术和交叉滴定实验确定TRN-SR2与HIV-1 IN相互作用的最适反应浓度。本文将为后续TRN-SR2与HIV-1 IN相互作用为靶点的抑制剂的开发提供研究基础。
1 材料与方法
1.1 材料
1.1.1菌株及质粒
大肠杆菌(Escherichiacoli)菌株Rosetta(DE3)、DH5α购自北京全式金生物技术有限公司。pGST-TRN-SR2重组质粒由台湾生物医学研究所的Woan-Yuh Tarn博士馈赠[5]。
1.1.2试剂与仪器
GST纯化柱填料购自常州天地人和生物科技有限公司,黑色96孔板、384孔聚丙烯浅孔微孔板购自Perkin Elmer(美国),Anti-6His-XL665受体磁珠、Anti-GST-Cryptate供体磁珠购自Cisbio公司(法国),其他试剂均为国产分析纯。微孔板恒温振荡器BE-9008(其林贝尔仪器制造有限公司),多功能酶标仪Envision 2102 multilabel reader(珀金埃尔默仪器有限公司PerkinElmer Life Sciences),ForteBio Octet Red 96分子相互作用分析仪(美国)。
1.2 方法
1.2.1TRN-SR2的表达纯化
重组质粒pGST-TRN-SR2的载体为pGEX-2T,将重组质粒pGST-TRN-SR2转化到大肠杆菌DH5α感受态细胞,第2天挑取单个克隆进行DNA序列测定,测序工作在苏州金维智公司完成,测序结果正确。然后将重组质粒pGST-TRN-SR2的转化大肠杆菌Rosetta(DE3)菌株,37 ℃培养过夜,将单个克隆37 ℃振荡培养过夜,然后扩大培养至菌液在600 nm下的吸光值达到0.6,添加0.5 mmol/L IPTG 28 ℃诱导表达过夜,8000 r/min离心收集菌体。裂解液(50 mmol/L Tris-HCl,pH 7.5,150 mmol/L NaCl,5 mmol/L DTT)重悬菌体,超声破碎后离心收集上清。利用GST亲和色谱柱纯化TRN-SR2蛋白,用洗脱buffer(50 mmol/L Tris-HCl,pH 7.5,150 mmol/L NaCl,5 mmol/L DTT,20 mmol/L reduced glutathione)洗脱目的蛋白,采用SDS-PAGE检测蛋白表达纯化结果,并使用BCA蛋白浓度测定试剂盒测定蛋白质含量。HIV-1 IN的表达纯化参考文献[6]。
1.2.2 BLI体外检测TRN-SR2与HIV-1 IN的相互作用
在Octet RED96系统中操作BLI实验,反应体积为200 μL,反应温度为30 ℃。实验方法如下:1)基线平衡。首先将Streptavidin (SA)标记的探针放入PBST(0.05% Tween,pH 7.5)中浸湿20 min,在系统中进行第一次基线(Baseline)平衡,时间120 s。2)蛋白固定。将探针转移入含有500 nmol/L biotin标记的终浓度为5 μmol/L HIV-1 IN的PBST中进行HIV-1 IN蛋白固定(Loading),时间600 s。3)结合测定。将固定后含有biotin的HIV-1 IN探针重新转移到PBST中进行第2次基线(Baseline)平衡,时间120 s;然后将探针转移至终浓度5 μmol/L TRN-SR2的PBST中进行结合(Association)测定,时间150 s。4)蛋白解离。将结合后的探针再次转移到PBST中进行解离(Dissociation)实验,时间300 s。
1.2.3基于HTRF确定TRN-SR2与HIV-1 IN相互作用最适浓度
采用交叉滴定实验考察两个蛋白在384孔板中的最适反应浓度,实验中所用蛋白和化合物均用反应Buffer(25 mmol/L Tris-HCl pH 7.5,1 mg/mL BSA,2 mmol/L MgCl2,150 mmol/L NaCl,0.1% NP40)稀释。实验设置HIV-1 IN与TRN-SR2的起始浓度为320 nmol/L,并依次梯度稀释至160、80、40、20和10 nmol/L,以0 nmol/L为对照。将4 μL HIV-1 IN、4 μL TRN-SR2、2 μL Buffer依次加入到384孔板中混匀,25 ℃,300 r/min反应30 min。反应结束后将5 μL 0.8 nmol/L的GST-Cryptate供体磁珠和5 μL 4 nmol/L的Anti-6His-XL665受体磁珠(含100 mmol/L KF)混匀加入到384孔板中,继续反应1 h后使用Envision 2102 multilabel reader酶标仪进行检测[6]。仪器检测条件:以320 nm为激发光,读取665 nm和620 nm处的发射光,分别计算各孔665 nm和620 nm处荧光强度的比值(Ration 665/620)。
1.2.4数据处理
结合-解离曲线、动力学参数使用Data analysis(ForteBio,version 7.1)进行数据分析。KD值是基于结合-解离曲线并通过nonlinear global fitting model模拟计算得出解离常数(Kd)与结合常数(Ka)的比值,该值可反映分子间亲和力的大小。HIV-1 IN与TRN-SR2浓度均为5 μmol/L,重复3次计算平均值。使用软件Graphpad 6.0作柱状图。
2 结果与分析
2.1 TRN-SR2与IN-HIS的表达纯化结果
图1为TRN-SR2在大肠杆菌Rosetta(DE3)中的表达结果,TRN-SR2在经0.5 mmol/L的IPTG诱导后,目的蛋白(约130 ku)蛋白量明显增多,再通过20 mmol/L GSH洗脱,SDS-PAGE电泳检测在130 ku左右有明显的TRN-SR2蛋白条带,使用BCA法测得TRN-SR2蛋白的浓度约为0.8 mg/mL。
2.2 BLI体外检测TRN-SR2与HIV-1 IN的相互作用结果
图2为BLI体外检测5 μmol/L TRN-SR2与HIV-1 IN相互作用的结合-解离曲线,曲线中的横坐标是结合解离持续时间,纵坐标为相位移变化(代表结合信号的强弱)。图2中TRN-SR2与HIV-1 IN在Octet系统Association步骤中曲线明显升高,说明TRN-SR2与HIV-1 IN亲和力较高,结合效果较好。通过结合-解离曲线计算出TRN-SR2与HIV-1 IN的KD值为45.2 nmol/L。
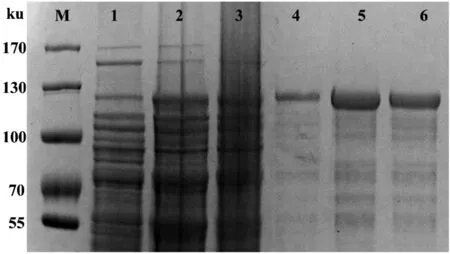
1:诱导前;2:诱导后;3:上清;4-6:20 mmol/L GSH洗脱;M:Marker
图1SDS-PAGE检测TRN-SR2的表达纯化结果
Figure 1 The expression and purification results of TRN-SR2 was detected by SDS-PAGE
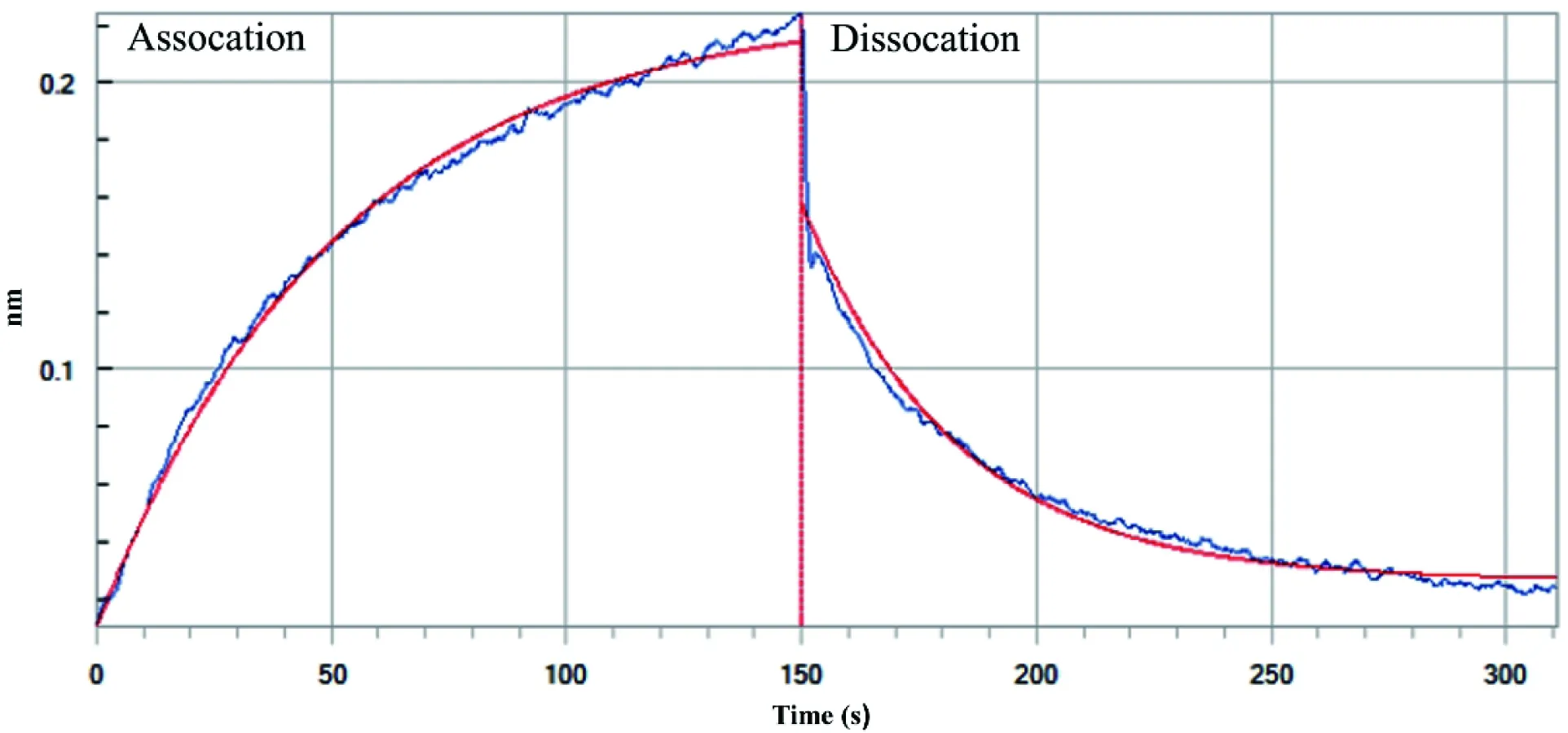
图2 TRN-SR2与HIV-1 IN相互作用的结合-解离曲线(平滑的为拟合曲线)
2.3 TRN-SR2与HIV-1 IN相互作用的最适浓度
图3为不同浓度TRN-SR2与HIV-1 IN相互作用的HTRT交叉滴定实验结果。结果表明:当TRN-SR2蛋白浓度不变时,荧光信号值随着HIV-1 IN蛋白浓度呈现抛物线状变化。同样,当HIV-1 IN蛋白浓度不变时,荧光信号值随着TRN-SR2蛋白浓度也呈现抛物线状变化。TRN-SR2蛋白浓度为20 nmol/L和HIV-1 IN蛋白浓度为40 nmol/L时,信号值达到最大,实验灵敏度最高。
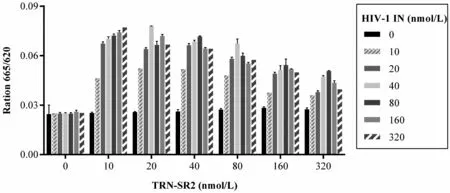
图3 不同浓度TRN-SR2与HIV-1 IN相互作用的HTRT实验结果
3 讨论与结论
目前上市治疗HIV的药物虽然能够有效降低患者体内病毒的数量,但长期服用均有不良反应发生,而耐药毒株的快速出现也使艾滋病的治疗形势更加严峻,因此研究者们迫切希望找到新型抗HIV疗法和以新作用机制为靶点的抗病毒药物[3]。HIV整合前核输入过程中TRN-SR2与HIV-IN的相互作用已成为抗HIV-1 IN药物筛选的新靶点。本文通过原核表达纯化能够简单快速得到大量的重组TRN-SR2蛋白用于后续的抑制剂筛选实验研究,同时也降低了经济成本。
生物膜干涉技术是一种快速检测分子间相互作用的方法,具有准确度高、高通量和速度快等优点[7]。HIV-1 IN晶体结构研究表明[8-10],二聚化的整合酶CCD形成的结合口袋是与其他辅助因子发生相互作用关键位置。因此本文原核表达纯化了HIV-1 IN-CCD蛋白,并通过BLI检测其与TRN-SR2相互作用,计算出TRN-SR2与HIV-1 IN的KD值为45.2 nmol/L。Stephanie等[11]通过AlphaScreen的方法确定His6-IN/GST-TRN-SR2相互作用的KD值为16.8 nmol/L。根据BLI原理,KD值即为亲和力常数,它只与分子间相互作用大小有关,与分子浓度无关,其数值越小代表的分子间亲和力越强[12]。行业内比较认可的亲和力差异的评价标准为:“KD值差异10倍”可认定2种分子与配体间的亲和力存在明显差异,因此本文测得KD值45.2 nmol/L与文献报道的16.8 nmol/L没有明显差异[12-13]。根据计算公式,当KD值为10-6mol/L时,解离百分比约为0.1%,因此TRN-SR2与HIV-1 IN的KD值为45.2 nmol/L同时也说明两者结合后的解离百分比小于0.1%。以上结果表明TRN-SR2与HIV-1 IN在体外相互作用结合效果较好,可用于后续的抑制剂筛选研究。
HTRF是常用于高通量药物筛选的方法,具有稳定、经济、操作简便的特点[14-15],可用于TRN-SR2与HIV-1 IN相互作用抑制剂的活性评价。本文通过交叉滴定实验确定筛选体系中TRN-SR2和HIV-1 IN蛋白最适反应浓度分别为20 nmol/L和40 nmol/L,这将为开展以TRN-SR2与HIV-1 IN相互作用为靶点的抑制剂筛选研究提供良好基础。
