DNA条形码技术鉴定黄颊长臂猿与白颊长臂猿
2019-08-19孙伟东傅兆水
孙伟东 陈 蓉 傅兆水
(南京市红山森林动物园管理处,南京,210028)
长臂猿科(Hylobatidae)分为4个属(Nomascus,Symphalangus,Hylobates,Hoolock)17个种,其中我国分布有3属6种[1]。冠长臂猿属IUCN濒危物种,其家族成员中的北部白颊长臂猿(Nomascusleucogenys)、南部白颊长臂猿(Nomascussiki)、黄颊长臂猿(Nomascusgabriellae)间的形态差异极小,外观不易区分。其中,动物园饲养较多的黄颊长臂猿和白颊长臂猿体毛长而粗糙,雄性均以黑色为主,混有不明显的银色,区别仅为黄颊长臂猿面颊的两旁从嘴角至耳朵的上方各有一块橘红色或黄色的毛,白颊长臂猿为白色或黄色的毛。两个物种的雌性腹部均没有黑色的毛,仅黄颊长臂猿雌性体毛通常为棕黄色,白颊长臂猿雌性体毛为橘黄色至乳白色。通过黄颊长臂猿和白颊长臂猿外形来区分是非常困难的,由于仅有地域隔离而没有生殖隔离,导致迁地保护中出现了黄颊长臂猿和白颊长臂猿的杂交现象。由于上述原因,存在黄颊长臂猿和白颊长臂猿种间杂交等问题,长此以往,对该物种的迁地保护是极其不利的。 因此,需要借助分子遗传学的方法辅助鉴定长臂猿的物种,以避免这一现象的产生。
mtDNA分子标记作为一种母系遗传的生物大分子,是研究物种内遗传多样性及种上系统分类关系的有力工具,特别是线粒体细胞色素 C 氧化酶亚基I(cytochrome C oxidase subunit I,COI),是目前研究种和种下水平分子系统的首选标记。本文利用mtDNA物种鉴定技术,设计合成冠长臂猿属COI序列的引物,对21只长臂猿进行物种鉴定,NJ树显示2只雄性长臂猿为南部白颊长臂猿,其余19只均为黄颊长臂猿。该方法能有效区分冠长臂猿属下的黄颊长臂猿和白颊长臂猿,为这几个物种的迁地保护提供管理依据。
1 材料与方法
1.1 材料
来自南京红山森林动物园圈养条件下的21只长臂猿(基本信息见表1),采集带毛囊的毛发样本放于-20℃保存。

表1 21只长臂猿基本信息Tab.1 Basic information of 21 gibbons
1.2 主要试剂与仪器
微量基因组提取试剂盒、TaqPlus MasterMix、D2000 DNA Marker、GeneGreen核酸染料购自天根生化科技(北京)有限公司。引物均由金斯瑞生物科技有限公司合成。
梯度PCR仪购自杭州龙扬仪器公司。凝胶成像系统购自上海欧翔仪器公司。水平电泳仪购自北京君意东方有限公司。高速冷冻仪离心机购自上海沪粤明科学仪器有限公司。
1.3 基因组DNA提取
按微量基因组提取试剂盒说明书提取长臂猿毛囊中的基因组DNA。提取后的基因组DNA样本于-20℃保存,备用。
1.4 引物设计和筛选
根据NCBI数据库中,相近物种的COI序列进行比对,设计出3对引物(COI F1/R1,COI F2/R2,COI F3/R3),序列见表2,进行PCR筛选。

表2 引物序列Tab.2 Primer sequence
1.5 PCR扩增
利用引物(COI F1/COI R1)进行扩增。反应体系为:Mix 12.5 μL,COI F 0.5 μL,COI R 0.5 μL,双蒸水6.5 μL,DNA模板5 μL。反应条件为:94℃ 5 min;94℃ 1 min,56℃ 30 s,72℃ 1 min,35个循环;72℃ 10 min。
1.6 电泳
取10 μL PCR扩增产物,在2%的琼脂糖凝胶上电泳30 min,通过凝胶成像仪进行拍照记录结果。电泳结果中,目标条带大小约750—500 bp。
1.7 序列测序与分析
将PCR产物送至金斯瑞生物技术有限公司进行克隆测序。测序获得序列利用DNAstar软件包中的SeqMen软件进行拼接,舍弃两端低信号序列,结合测序峰图,对序列内部位点进行人工校对。确定序列后,将校准后的序列输出为FASTA格式的文本文件,利用BLAST比对确认,获得序列为目的序列。将目的序列与黄颊长臂猿N.gab1(HQ622807.1)、黄颊长臂猿N.gab2(HQ622806.1)、北方白颊长臂猿N.leu(KC757404.1)、南方白颊长臂猿 N.siki(HQ622805.1)采用Clustalx软件对基因序列进行比对分析。将其比对结果转换成Mega格式,用Mega 4软件计算序列间碱基对差异、转换、颠换,采用Maximum composite likelihood等模型,构建NJ树。NJ树分支的置信度均采用自展法1 000次重复检验。
2 结果
2.1 RCR检测方法的建立
以长臂猿线粒体DNA为模板,用3对引物进行PCR扩增,结果引物COI F1/R1能扩增与预期一致的特异性DNA 片段(约500 bp)(图1)。进一步切胶回收DNA测序,并进行序列分析,确证2条特异性引物间的序列为COI序列(515 bp)。
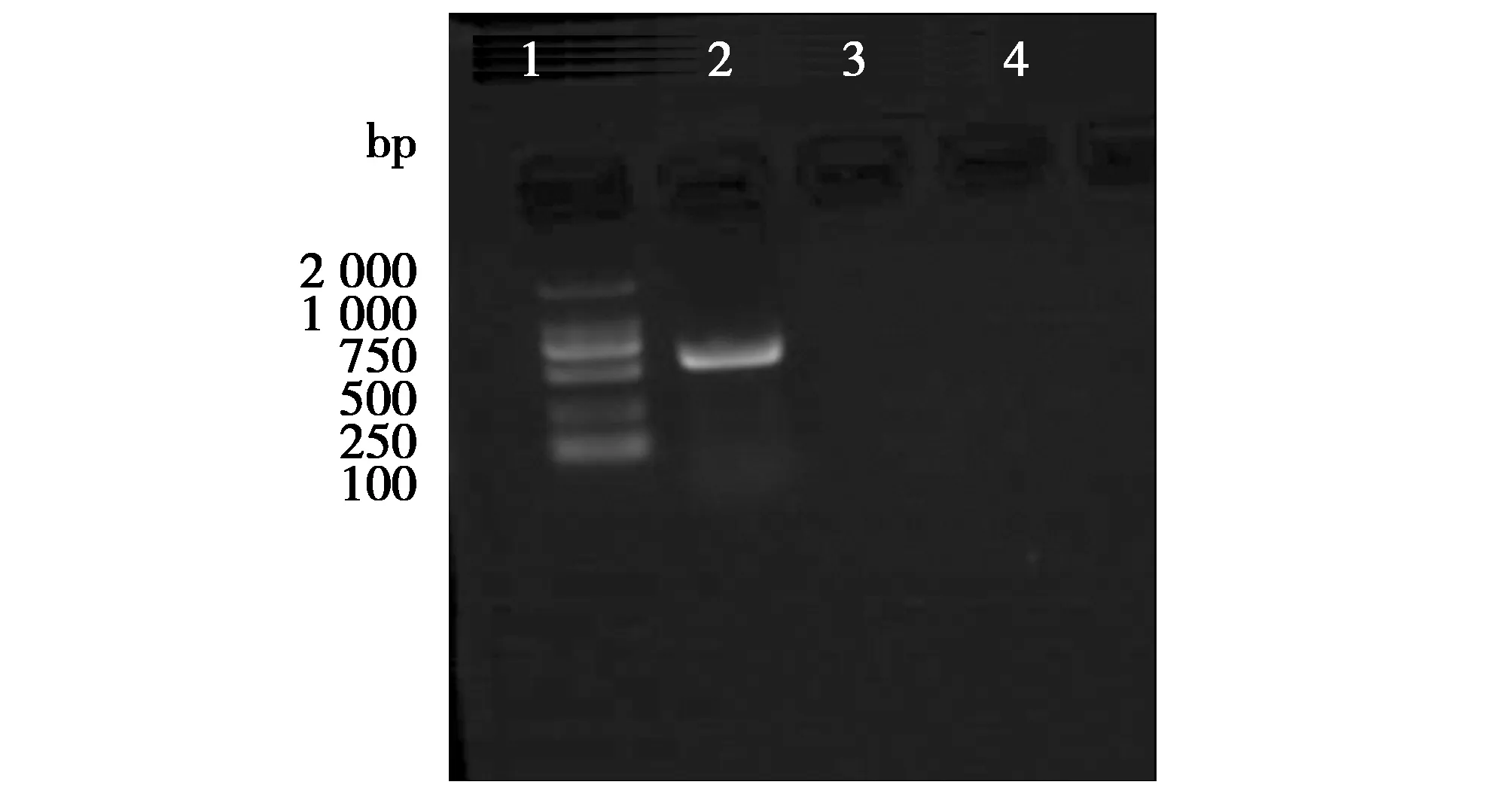
图1 COI引物建立Fig.1 COI primer establishment注:1.D2000Maker;2.引物COI F1/R1;3.引物COI F2/R2;4.引物COI F3/R3Note:1.D2000Maker.2.Primer COI F1/R1.3.Primer COI F2/R2.4.Primer COI F3/R3
2.2 COI PCR扩增结果
利用COI F1/R1对21只冠长臂猿进行PCR扩增,均扩增出515 bp的目的条带(图2,图3)。
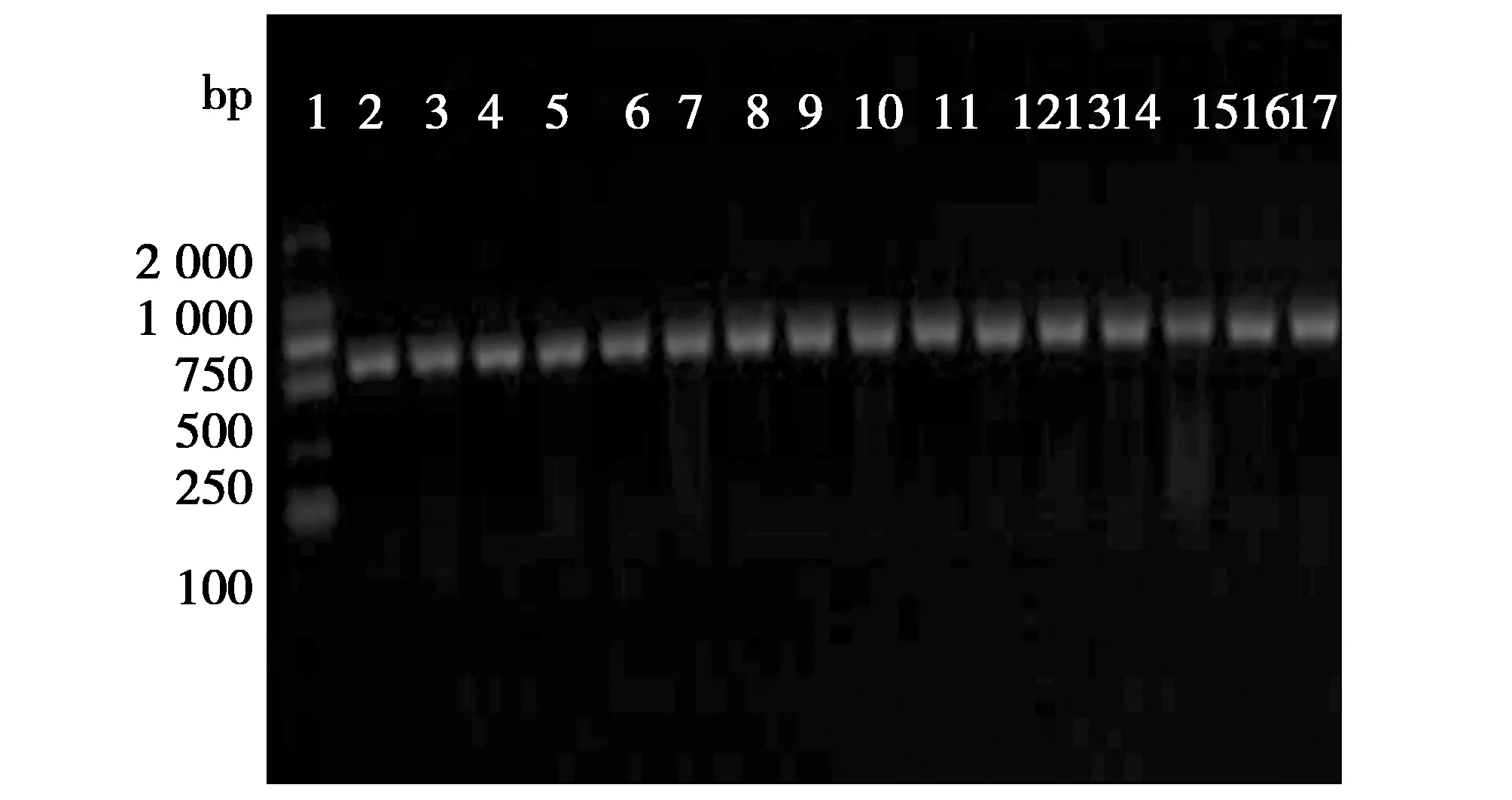
图2 COI PCR扩增结果Fig.2 The amplification results of COI PCR注:1.D2000Maker;2-17.样本1-16号Note:1.D2000Maker.2-17.Sample 1-16
2.3 进化树分析
测序获得序列利用DNAstar软件包中的SeqMen软件进行拼接,校对。采用Clustalx软件对基因序列进行比对分析。用Mega4软件采用Maximum composite likelihood等模型,构建NJ树。系统树分支的置信度均采用自展法1 000次重复检验。

图3 COI PCR扩增结果Fig.3 The amplification results of COI PCR注:1.D2000Maker;2-6.样本17-21号Note:1.D2000Maker.2-6.Sample 17-21
如图4可见,长臂猿Lianlian和Dazai与N.siki在一个分支上,其余19只长臂猿均在N.gab分支上。其中,可以发现种群中的Dahuang家族成员(Yuanyuan,Dongzhi,Lele,Xiatian)和Erhuang家族成员(Fangfang,Yuanbao,Wuming,Yaqing)在分支上比较接近。进化树上,黄颊长臂猿和白颊长臂猿均有明显的分支,能够区分这几个物种。
3 讨论
随着标准数据库的建立,基于COI基因的DNA barcode在动物分类学和生态学中得到了广泛应用[2]。由于常采用基于形态特征的工作方式,传统分类学要将这些物种纳入记录,而获取相关知识有许多困难。在记录新物种的工作中引入COI基因以后可以快速给物种分群,在一定条件下还可以快速判定物种样品是否来源于已知物[3]。这种快速判断在某些情况下显得尤为重要。此外,因为传统分类学大多数情况下有更加关注成年个体的倾向,对幼体经常描述不够详细,导致对许多物种辨别能力不足,DNA barcode方法则没有这种限制。另外,在缺乏形态变化的动物类群中,依赖形态特征的工作方式往往会导致混乱和错误[4]。
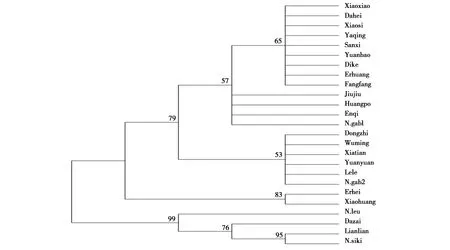
图4 长臂猿NJ进化树Fig.4 The NJ evolution tree of the gibbons
目前,本文通过近物种COI序列进行引物设计和筛选,建立了对黄颊长臂猿和白颊长臂猿的鉴定方法。 运用该方法对21只冠长臂猿进行物种鉴定,NJ树结果显示2只雄性长臂猿在南部白颊长臂猿的分支上,同时,对其外观进行观察,体毛为黑色,唯两颊各具一大块白毛,符合白颊长臂猿的外形特征。其余19只长臂猿与黄颊长臂猿在一个分支上,经COI序列鉴定为黄颊长臂猿,比对其外观,符合黄颊长臂猿特征。NJ树上,冠长臂猿属的各物种均有明显的分支,能够明显区分。因此,将COI序列鉴定与外形特征结合,能准确对黄颊长臂猿和白颊长臂猿进行物种鉴定。
根据物种鉴定结果,建议将这2只白颊长臂猿与黄颊长臂猿分开饲养管理,防止因种间杂交造成的濒危物种基因污染。此外,这2只白颊长臂猿(Dazai和Lianlian)已成年,可以考虑为其引进雌性白颊长臂猿进行配对繁殖。
种群管理对物种保护非常重要,其管理的目标应是维持种群的多样性达到可持续发展。随着技术水平的推进,管理种群不应只停留在宏观的记录和调控上,应逐步运用遗传学、生态学等原理,结合分子技术来了解和运用表观现象后的科学原理。尤其是大种群或群居动物,作为在物种保护中起重要作用的动物园,需要对物种的过去(来源——是否有亲缘关系)、现在(种或亚种类型、种群的遗传多样性)有充分的了解,才能对其未来(是否有足够的遗传多样性来对抗疾病、来繁殖配对等)进行规划。这些都必须而且一定要建立在科学技术的基础上。
