黄野螟蜕皮激素受体基因HvEcR的鉴定及表达分析
2019-07-29吕子豪李治兴
吕子豪,李治兴,林 同
(华南农业大学 林学与风景园林学院,广州 510642)
黄野螟(HeortiavitessoidesMoore)是一种寡食性的食叶害虫[1-2],其寄主为国家二级重点保护野生植物土沉香(AquilariasinensisSprenger)[3]。该虫生长季节虫口爆发迅速,数日便可把叶片吃光,造成枯苗、死苗,严重影响植株生长,制约结香的产量与质量,造成极大的经济损失[1,4]。近年来,国内外关于黄野螟的研究大多集中在生物学特性、行为学、农药防治技术方面,关于昆虫生长调节剂防治及其分子方面的研究鲜见报道。
蜕皮激素核受体EcR是核受体超家族成员[5],最早在果蝇(Drosophilamelanogaster)中得到鉴定[6],具有蜕皮激素(Ecdysteroid hormones, EH) 配体专一性[7], EH通过其进行信号转导。高20E浓度条件下,EcR与另一个蜕皮激素受体——超气门蛋白(Ultraspiracle,USP)会被诱导形成异二聚体,进而激活一系列早期应答基因与晚期基因的表达[8-11]。因此,EcR在昆虫蜕皮和变态过程中具有重要作用。
蜕皮是由数种激素协同调控的昆虫生长发育所必需的复杂生命过程[12],蜕皮激素与保幼激素(Juvenile hormone,JH)的协同作用在此过程中发挥关键作用。蜕皮激素需经过一系列羟基化作用,被激活为具有更高活性的20-羟基蜕皮酮(20E)才能发挥作用[13-16]。当JH滴度较高时,20E引起昆虫脱皮,当JH滴度较低,20E诱导昆虫进入下一龄期或发生变态[17]。EcR接受20E信号后,一系列蜕皮级联反应将被启动。因此,研究昆虫的EcR基因具有相当的分子生物学意义。本研究利用RT-qPCR技术检测HvEcR基因在黄野螟各发育阶段及幼虫组织中的转录水平,同时明确HvEcR的表达量对20E胁迫的响应,丰富了EcR的分子生物学信息。
1 材料与方法
1.1 材料和主要试剂
于广州天鹿湖森林公园采集虫卵及1至5龄试虫,饲喂新鲜土沉香叶片,室内饲养条件为:温度26 ℃,相对湿度75%,光期14 h,暗期10 h。20E试剂购自上海源叶生物科技有限公司。总RNA提取试剂盒(E.Z.N.ATMTotal RNA KitⅡ)购自OMEGA公司;反转录试剂盒(PrimeScript RT reagent Kit With gDNA Eraser)及实时荧光定量试剂盒(TB GreenTMPremix Ex TaqTM)均购自TaKaRa公司。
1.2 试验方法
1.2.1 样品处理 野外采集工作后,立即用液氮处理卵(3片)及各虫态幼虫(各5头),存放于 -80 ℃冰箱中;其余个体饲养至老熟幼虫时,转移到铺有2 cm厚沙土(相对湿度为50%)的塑料养虫盒中,待其化蛹羽化后,用同样方法收集蛹及成虫(各5头)。选取40头健康的4~5龄幼虫,经清洗、消毒后,在腊盘上解剖,获取幼虫体壁、头、脂肪体、中肠、马氏管等组织样品,立即置于无菌去酶的冻存管中,经液氮速冻后,存放于 -80 ℃冰箱。
1.2.2 蜕皮激素胁迫处理 先用DMSO将20E稀释至10 mg/mL,存放于-20℃冰箱,备用。试验时用1×PBS将20E稀释至30 ng/ μL和150 ng/ μL 2个质量浓度。采用注射法对4龄幼虫进行蜕皮激素胁迫,用φ=75%酒精擦拭虫体,虫体置于冰上麻醉后,从腹部侧面注射。每头虫子注射1 μL液体,对照组为等量稀释的DMSO。注射后继续用沉香叶片饲养于人工气候箱中,分别于注射后24、48、72 h取样。
1.2.3 RNA提取与cDNA合成 严格按照E.Z.N.ATMTotal RNA KitⅡ试剂盒说明书提取各样本总RNA,经琼脂糖凝胶电泳以及微量紫外分光光度计(Nanodrop 2000)检测合格后,置于 -80 ℃冰箱保存,备用。严格按照PrimeScript RT reagent Kit With gDNA Eraser试剂盒说明书,将总RNA(0.8 μg)逆转录为第一链cDNA,稀释20倍后,置于-20 ℃冰箱保存,备用。
1.2.4 基因表达 从黄野螟转录组文库筛选出具有完整ORF框的EcR基因序列,用实时荧光定量PCR反应(RT-qPCR)检测其表达量,所用内参基因为β-actin;所用目的基因引物为:Forward:5′-GCCAACCAGCAGTTCCTCATCG-3′和Reverse:5′-CGTCCTCTTCCTCCGTTGATTGC-3′;阴性对照为无菌超纯水处理;反应体系包括cDNA模板 20 μL,TB Green Premix ExTaq 10.0 μL,上游引物0.4 μL,下游引物0.4 μL,ddH2O 7.2 μL。反应程序为:95 ℃预变性5 min;95 ℃解链10 s,60 ℃延伸20 s,共进行40个循环。每个样本设置3个技术重复,根据反应结束后的Ct值,使用2-△△Ct相对定量法计算HvEcR相对表达量。用Excel进行统计分析,采用SPSS 18.0进行单因素方差分析与邓肯分析。
1.3 生物信息学分析、同源比对及系统发育树构建
在multalin网站(http://multalin.toulouse.inra.fr/multalin/cgi-bin/multalin.pl)进行氨基酸序列同源比对,采用ExPASy-ProtPram软件进行蛋白质理化性质预测,采用PSORT在线工具(https://psort.hgc.jp/cgi-bin/runpsort.pl)预测亚细胞定位,采用NPS在线工具(https://npsa-prabi.ibcp.fr/cgi-bin/secpred_gor4. pl)预测蛋白质二级结构,采用SWISS-MODEL在线工具(https://www.swissmodel.expasy.org/interactive/yrSsgx/models/)模拟EcR蛋白高级结构,采用ClustalX软件和MEGA 5.0软件构建系统发育进化树。
2 结果与分析
2.1 HvEcR基因序列及蛋白高级结构分析
从黄野螟转录组文库中筛选出具有完整ORF框的目的片段,将其命名为HvEcR(登录号:MH588318),序列全长1 734 bp,包含1 638 bp长的ORF框,共编码545个氨基酸。使用ProtPram软件对HvEcR的理化性质进行分析,结果表明,该蛋白的分子质量为61.70 ku,理论等电点为5.89,脂肪指数为73.71,不稳定性指数为68.47,为不稳定蛋白,总平均疏水性为 -0.442,为亲水性蛋白。NetPhos 3.1软件的预测结果为:HvEcR含丝氨酸位点16个,苏氨酸和酪氨酸位点各2个(得分大于0.9)。
Signal P 4.1软件预测结果为:HvEcR不存在信号肽,为非分泌蛋白。根据PSORTII软件预测的亚细胞定位结果可知,HvEcR位于线粒体的可能性最大,为60.9%,其次是细胞核和细胞质,分别有26.1%和8.7%的可能性,位于细胞骨架可能性则较低,仅为4.3%。TMpred软件跨膜区分析结果为,无得分大于500的跨膜螺旋。TMHMM 软件基于隐马尔可夫模型(hidden Markov model)的跨膜螺旋预测结果为,HvEcR被标记在外部,不含跨膜螺旋。蛋白的疏水性越强,其疏水区为跨膜区的可能性就越大,HvEcR无跨膜区,符合上文的亲水性预测结果。
Conserved Domains软件分析表明(图1),HvEcR含有EcR家族典型的DNA结合域(DNA binding domain,DBD)和配体结合域(Ligand binding domain,LBD)。SMART软件分析显示,HvEcR由6个功能结构域组成,分别为两段锌指结构域(zinc finger,ZF),3个低密度复杂区(low complexity region,LCR)和1个配体结合域(图2)。这两个软件的分析结果进一步证明筛选出的HvEcR基因与其他昆虫的EcR一样,均为核受体家族成员。经SOPMA和SWISS-MODEL软件进行蛋白质结构预测和建模,结果显示,HvEcR编码的蛋白主要由α-螺旋与无规则螺旋组成,它们分别占36.15%和48.62% (图3)。
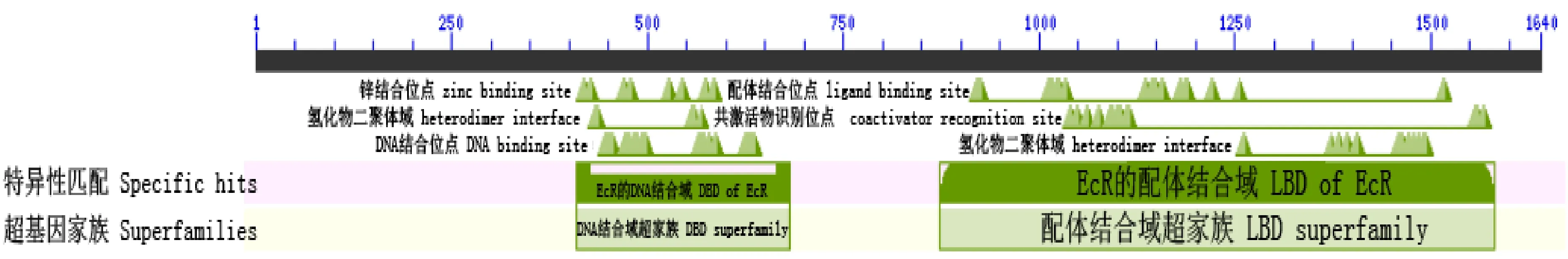
图1HvEcR蛋白序列的保守结构域预测Fig.1 Putative conserved domain of HvEcR

低密度复杂区以大括号标注;锌指结构域以阴影标注;配体结构域以方框标注;终止密码子TAG以星号标注 LCRs are marked in braces; ZFs are marked with shadow; LBD is marked with boxes; the termination codon is marked with asterisk
图2 黄野螟EcR基因核苷酸序列及其编码的氨基酸序列
Fig.2 Nucleotide and deduced amino acid sequences ofEcRgene fromH.vitessoides
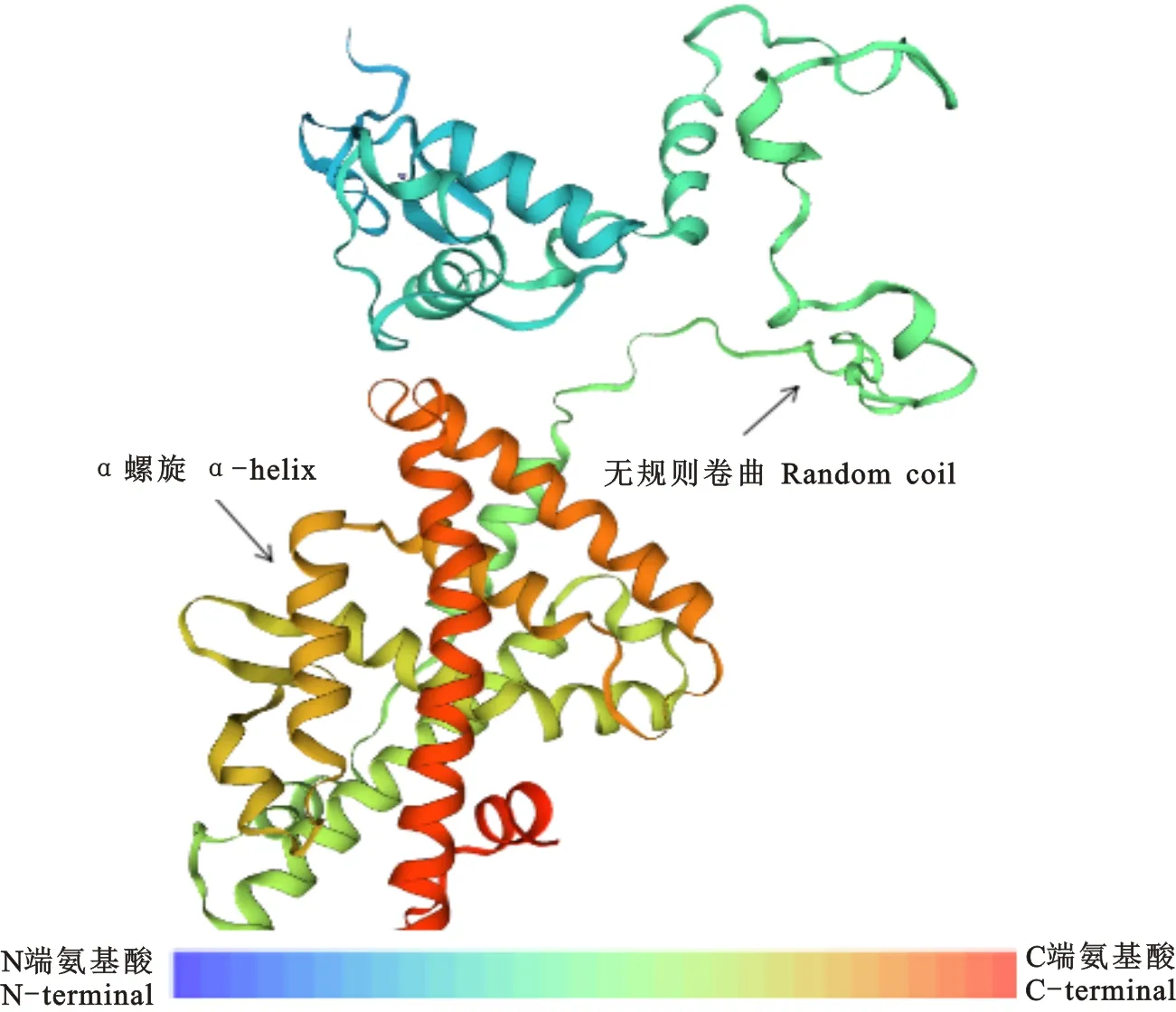
图中彩虹条表示从N端到C端的氨基酸 The rainbow bar represents the amino acid from N-terminal to C-terminal
图3HvEcR的三维结构
Fig.3 Three-dimensional structure ofHvEcR
2.2 同源比对及系统发育树
在NCBI网站上进行同源搜索,用multalin软件进行同源比对,结果表明,HvEcR序列与三化螟和二化螟的同源性最高,分别高达96%和92%(图4)。基于鳞翅目12种昆虫的EcR氨基酸序列,采用NJ法构建系统发育树,结果表明,黄野螟与水稻三化螟和二化螟的亲缘关系最近,结果与同源性比对一致(图5)。
2.3 黄野螟HvEcR基因的时空动态表达
黄野螟发育阶段表达谱显示(图6),以HvEcR在卵期的表达量为对照,HvEcR的转录水平在各发育时期差异显著,在成虫时达到峰值,为对照的12倍;其次是5龄幼虫时,为对照的 5.68倍。在3龄幼虫时表达量最低,仅为卵的 0.22。
由图7可知,HvEcR在黄野螟幼虫各组织中均有表达,在体壁中表达量最低,设为对照;在脂肪体中表达较高,为对照的4.54倍。其次,HvEcR在头与马氏管中也有较高表达,分别为对照的2.85倍和2.49倍。
用质量浓度分别为30 ng/μL、150 ng/μL的蜕皮激素溶液和等量稀释的DMSO溶液处理黄野螟4龄幼虫,于处理后24 h、48 h和72 h检测HvEcR基因的表达情况。RT-qPCR结果显示(图8),在两种质量浓度处理24 h后,均能引起HvEcR基因的表达量上调。在注射质量浓度为150 ng/μL,处理时间为48 h时,HvEcR表达量上调最为明显,为对照的4.10倍。结果表明,HvEcR基因的表达受20E调控。
3 讨 论
本研究鉴定1条黄野螟HvEcR基因,其编码的氨基酸含2个锌指结构域、1个配体结构域和3个低密度复杂区等6个保守结构域,具有与其他昆虫EcR相似的典型的基因家族特征。HvEcR与其他鳞翅目昆虫EcR的亲缘关系较近,在进化过程中十分保守。
作为蜕皮激素的受体,HvEcR的表达水平可能与20E滴度密切相关[18],本研究结果显示,黄野螟HvEcR的表达水平可能受20E的诱导调节(图8)。蜕皮激素与保幼激素共同参与每一龄幼虫的组织重建和表皮形成[19]。RT-qPCR结果显示,HvEcR在卵期及1~4龄幼虫时表达量较低且相对稳定,到5龄时达到小高峰,到蛹期又降低,与昆虫不同发育时期体内蜕皮激素的变化趋势一致[20-21]。因此,推测HvEcR对黄野螟末龄幼虫的发育起重要作用,可能与组织重建和预蛹发育有关[22]。蜕皮激素参与昆虫成虫的卵子发生和生殖过程[17],在黄野螟成虫体内,HvEcR有高表达,推测作为蜕皮激素受体的EcR也与该过程有关[23]。
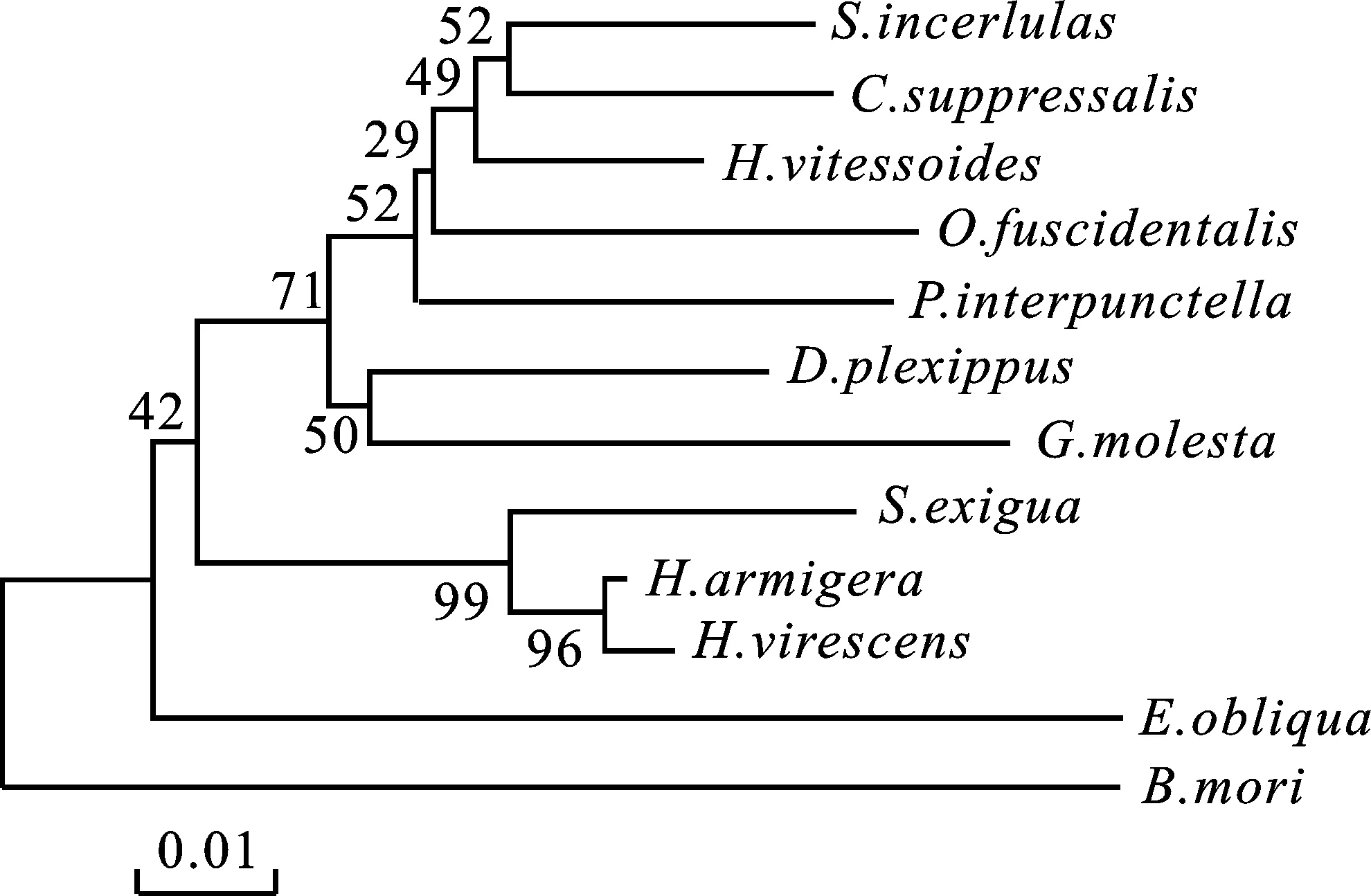
图5 基于昆虫EcR氨基酸序列构建的系统发育树Fig.5 Phylogenetic relationships of insects based on amino acids of EcR
EcR存在昆虫不同组织中,参与20E介导下的组织重建,如EcR在埃及伊蚊 (Aedesaegypti)幼虫中参与组织溶解过程,在家蚕和水稻二化螟幼虫脂肪体中特异性表达,在果绳中参与神经元重建[24-27]。对黄野螟幼虫不同组织的HvEcR转录水平进行检测,发现其主要在脂肪体中表达,为对照的4.54倍。脂肪体是昆虫重要的能量贮备中心,且在物质代谢中扮演关键角色,作为蜕皮激素受体,HvEcR可能与黄野螟营养储存及能量代谢有关。除在脂肪体表达外,HvEcR在马氏管和头部中也有较高表达,但其功能需进一步澄清。
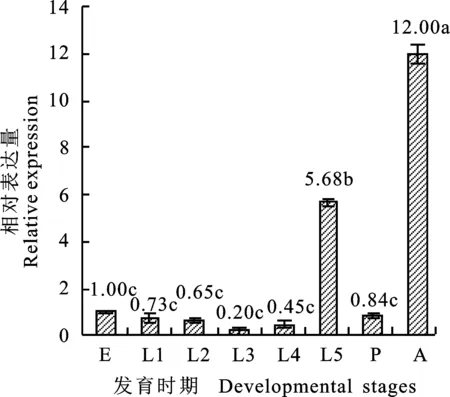
数据为“平均数±标准差”;柱上字母代表差异显著(P< 0.05);图7、图8同 Data in the figure are represented as mean ± SD;different letters mean significant difference on the bar (P<0.05). The same as Fig.7 and 8;E.卵 Egg;L1、L2、L3、L4、L5.1~5龄幼虫 Larve stage 1-5;P.蛹 Pupal;A.成虫 Adult
图6HvEcR基因在黄野螟各发育时期的表达
Fig.6 The expression level ofHvEcRindevelopmental stages ofH.vitessoides
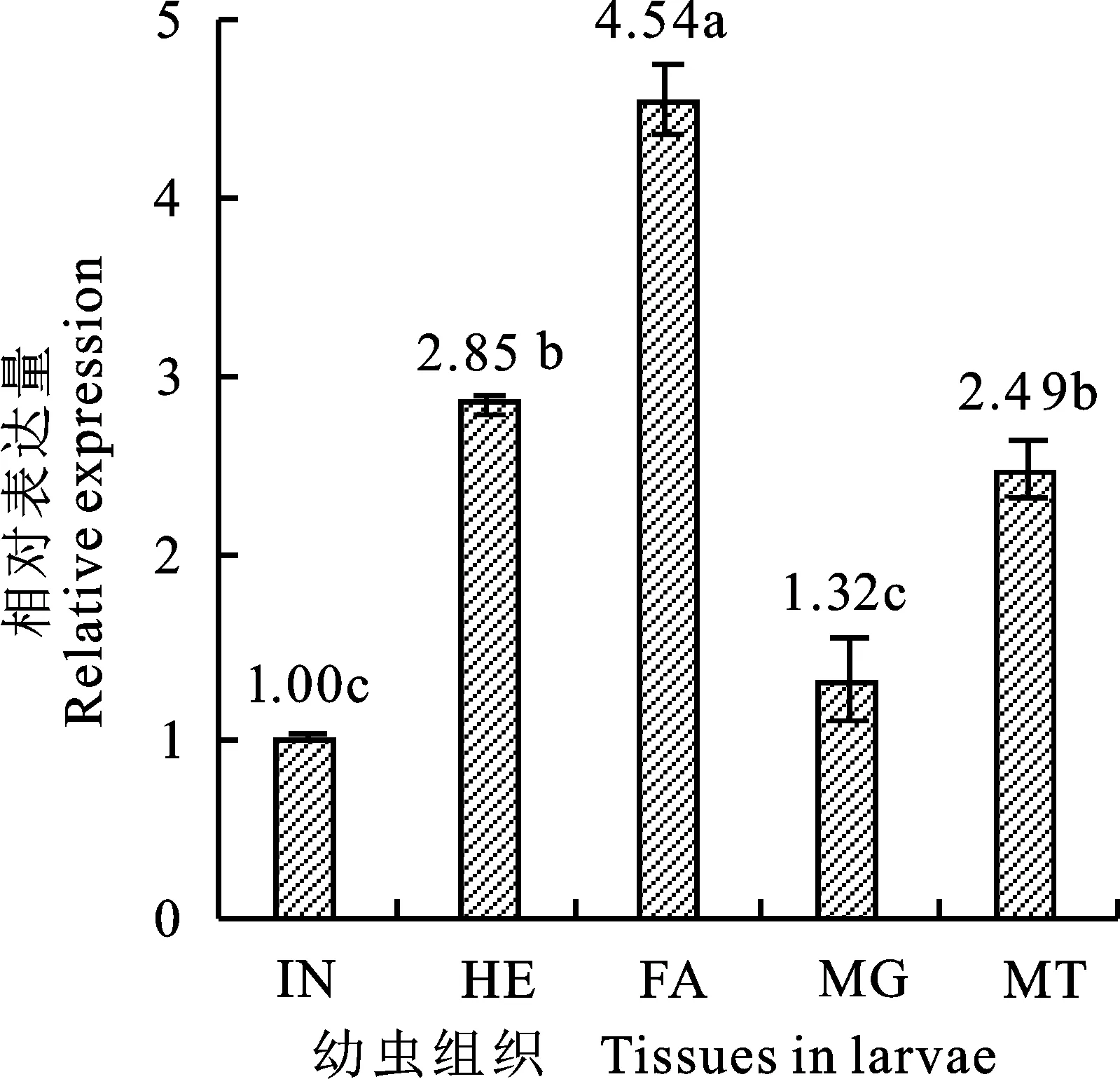
IN.体壁 Integument;HE.头 Head;FA.脂肪体 Fat body;MG.中肠 Midgut;MT.马氏管 Malpighian tubule
图7HvEcR基因在黄野螟幼虫中的表达
Fig.7 The expression level ofHvEcRin the larvae ofH.vitessoides
研究昆虫EcR的分子机制,不仅有助于深入理解昆虫的蜕皮行为,还能为害虫防控提供新思路。基于本试验对黄野螟HvEcR功能的初步研究,后续还将通过RNA干扰技术及相关生理生化试验,进一步研究该基因的功能,为开发环境友好型的EcR靶标杀虫剂提供更多的分子生物学参考。
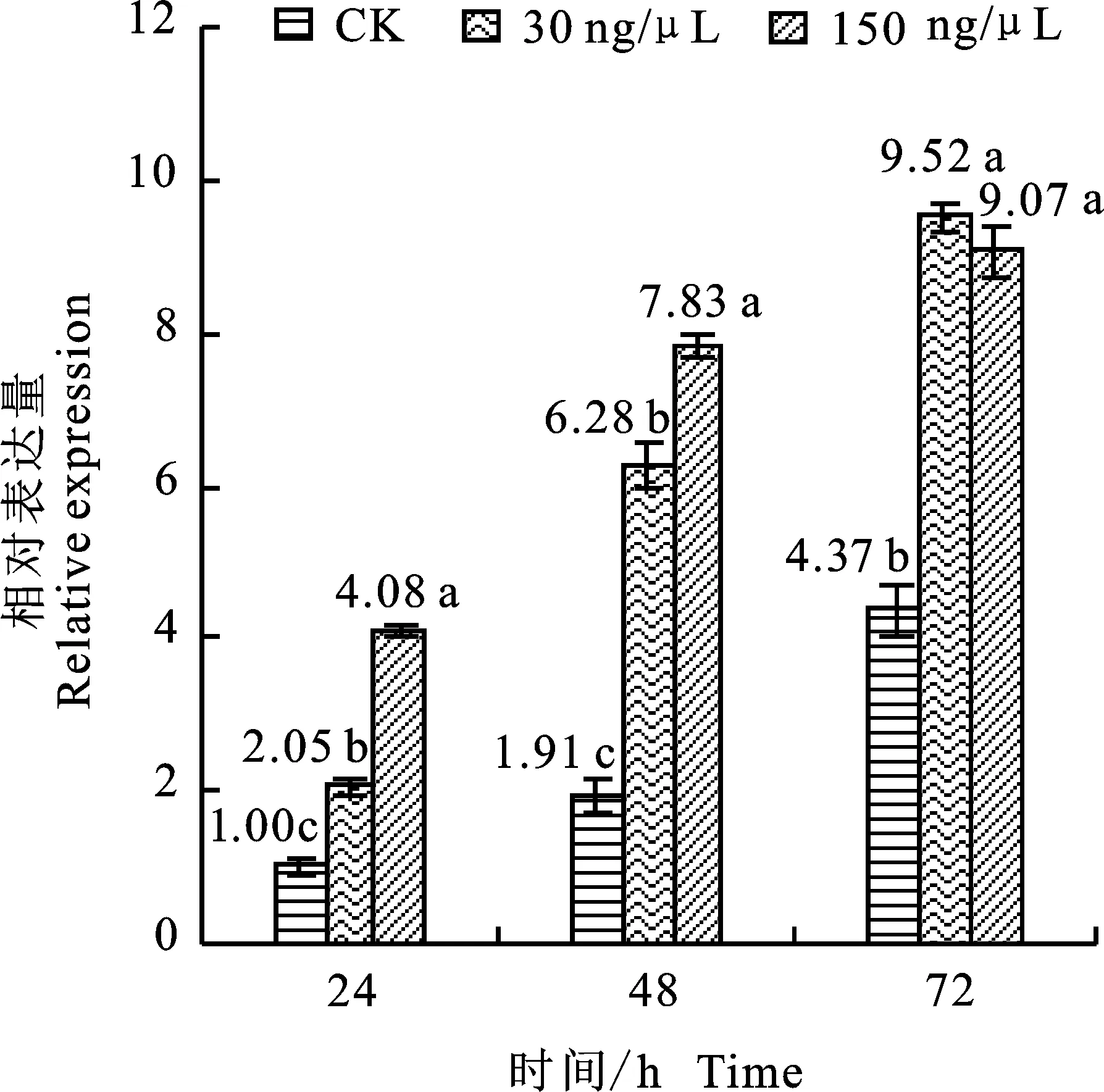
图8 不同时间不同质量浓度的20E处理对HvEcR基因表达的影响Fig.8 Expression profile of HvEcR at differrent time and different 20E mass concentration
