PMA-qPCR技术在发酵食品活菌计数中的应用
2019-07-04陈卓君魏铭林果陈昱锜梁杉张柏林朱保庆
陈卓君,魏铭,林果,陈昱锜,梁杉,张柏林,朱保庆*
1(林业食品加工与安全北京市重点实验室(北京林业大学),北京,100083) 2(中粮营养健康研究院,北京,102209) 3(北京工商大学,北京食品营养与人类健康高精尖创新中心,北京,100048)
发酵食品是以农产品、林产品和畜产品等为原料,利用微生物(不包含食品中直接加入的有益微生物)发酵而成的一类特殊食品。发酵食品最初是以延长食品保质期、拓展食品在不同季节的可食性为目的;因其具有独特的风味和丰富的营养价值,近年越来越受到消费者的欢迎[1]。微生物作为发酵食品的“灵魂”,与发酵食品的品质直接相关,在提高产品贮藏期、富集功能因子和保障安全等方面都发挥着不可替代的作用[2]。因此,准确计数发酵食品中的不同微生物,是研究发酵食品品质形成机理以及工艺改进和产品质量控制的基础。
传统的微生物计数方法为菌落计数法,该方法易受主观影响,繁琐复杂,耗时耗力,存在较大误差,既无法区分“可生存但不可培养”(viable but non-culturable,VBNC)状态下细胞也无法对复杂菌系中的单一菌种进行有效定量。随着现代分子生物技术的发展,高灵敏度和特异性的实时荧光定量PCR (quantitative real-time PCR,qPCR)也被开发用于发酵食品的微生物计数[3],该方法的主要缺陷在于难以区分死菌和活菌,使得死菌中存留的大量DNA也可被扩增,导致计数结果出现偏差[4]。近年来出现的叠氮溴化丙锭(propidium monoazide,PMA)偶联qPCR技术,可以有效避免常规qPCR的缺陷,也可检测VBNC状态的微生物。目前,该技术在发酵食品中已被用于多种发酵常见微生物的计数。本文系统地综述了PMA-qPCR技术的原理、影响因素及在常见发酵食品中应用现状,同时提出了该技术进一步发展和拓展应用的展望。
1 PMA-qPCR方法介绍
1.1 实时荧光定量聚合酶链式反应(qPCR)原理及特点
qPCR是在标准PCR技术基础上,使用荧光报告基团(包括双链DNA结合染料或在扩增过程中掺入PCR产物的、与PCR引物或探针结合的染料分子)进行DNA扩增,利用具有热循环功能及荧光染料筛查能力的仪器,每次循环结束后通过荧光染料检测DNA含量,荧光染料产生的荧光信号与生成的PCR产物分子(扩增片段) 数成正比。利用反应指数期采集的数据,生成有关扩增靶点起始量的精度极高的定量信息。与传统PCR相比,荧光定量PCR具有特异性更强、灵敏度更高、检测周期更短、自动化程度高等特点。近年来,随着分子生物学技术的不断发展,实时荧光定量PCR技术在微生物计数方面得到了广泛的应用[5]。然而微生物在自然环境中以多种生理状态存在:可培养的活菌,活的非可培养状态,具有完整结构但无生物活性的死菌以及膜损伤死菌。qPCR技术虽能克服传统计数方法无法检测VBNC状态下细胞的缺陷,但无法区分死活菌DNA,这对活菌计数带来巨大的挑战[6]。
1.2 PMA-qPCR检测活菌的机理
膜损伤细胞没有代谢活性,适宜条件下培养难以恢复生长,因此将膜完整性作为评判活菌标准已被多数学者接受[7]。基于活菌定量条件,叠氮溴化丙锭(propidiummonoazide,PMA)常被用于qPCR的前处理步骤[8],待测菌与适宜浓度PMA避光条件下孵育一定时间,经光钝化后即可提取活菌DNA。
PMA是一种具有高亲和力的光敏性DNA结合染料,如图1所示,该染料自身荧光弱,与核酸结合后荧光信号大大增强。在DNA提取之前将PMA与待测样品混合,PMA会选择性的进入膜损伤细胞并嵌入双链DNA结构中,每分子染料可与4~5个核苷酸相结合。在可见光(最大吸收峰为464 nm)作用下,PMA分子中叠氮基团分解产生高活性的氮烯类化合物,与DNA分子发生共价交联反应生成沉淀,进而抑制膜损伤细胞qPCR过程中DNA的扩增。溶液中残留的过量染料与水分子经光照反应生成羟胺化合物,进而失去交联活性,不会影响后续活细胞DNA扩增[8]。利用PMA选择渗透性和qPCR特异性,能够显著提高检测速度和灵敏度。
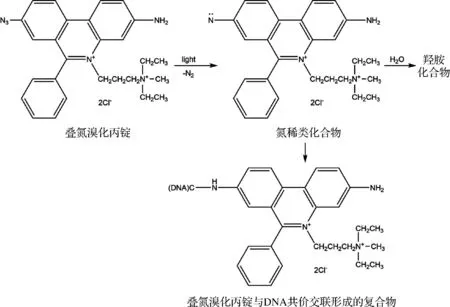
图1 PMA与DNA反应原理示意图[9]Fig.1 Schematic diagram for the reaction bewteen PMA and DNA
2 影响PMA-qPCR检测效率的因素
2.1 目标微生物
表1汇总了利用qPCR对常见发酵微生物计数的目的基因、引物序列等信息,诸多学者针对实验菌株会选择不同目的基因,细菌通常采用16S rDNA编码基因,而26S rDNA及ITS编码基因常用于酵母的靶点设计。

表1 不同微生物qPCR检测目的基因的选取及引物列表Table 1 Selected genes and related primers used in qPCR for different microorganisms
续表1

微生物 引物 目的基因 扩增长度/bp引物序列(5’-3’) 参考文献Tet_R107AATCAACACCAACCGAGAATCC[38]真菌ITS1qITS2ITS1-5.8SrDNA-ITS2290TCCGTAGGTGAACCTGCGGTTYGCTGYGTTCTTCATCG[16] [17]酵母YEAST-F26S rDNA124GAGTCGAGTTGTTTGGGAATGC[18]YEAST-RTCTCTTTCCAAAGTTCTTTTCATCTTT[18]酿酒酵母CESP-FSCER-RITS2 和5.8S rDNA175ATCGAATTTTTGAACGCACATTGCGCAGAGAAACCTCTCTTTGGA[18][18]布鲁氏酒香酵母DekITSITS308GACACGTGGAATAAGCAAGG[24]BruxITRATTATCCCCTCACTCCCCTC[24]葡萄有孢汉生酵母CESP-FHUV-RITS2和5.8S rDNA121ATCGAATTTTTGAACGCACATTGAACCCTGAGTATCGCCCACA[24][24]酿酒酵母SC1dSC1rRAPD bands301ACATATGAAGTATGTTTCTATATA-ACGGTGTGGTGCTGGTGCGGATCTA[18][18]
研究表明[10],对不同微生物进行PMA处理时应进行优化时,目标菌种不同(特别是革兰氏阴性菌和革兰氏阳性菌的细胞膜结构差异)可能对DNA活性染料效果有很大影响。革兰氏阴性菌细胞壁含有外膜,而革兰氏阳性菌主要为肽聚糖层[4],这种差异使PMA可以更快渗透入膜损伤的革兰氏阴性菌,如果两种细菌存在于同一环境样本中,最终可能产生群落定量偏差[11]。值得指出的是,现阶段研究多针对于细菌,对真菌和病毒的描述较少。同时为了正确地描述食品生态系统中的微生物种群,需要考虑的重要因素之一是待扩区域的选择。目标基因必须有2个基本特征: (1)存在于待检测的微生物组的所有成员中;(2)具有可设计通用引物的保守区域和可分化的可变区域[12]。针对目的基因进行正确的引物设计也至关重要,LAI等[13]基于内转录间隔基因和23S rDNA基因,设计了格氏乳杆菌(Lactobacillusgasseri)和唾液乳杆菌(Lactobacillussalivarius)特异性引物,从而可以有效计数益生菌产品中两种乳酸菌活细胞值。
2.2 PMA处理条件
除待测菌自身差异外,PMA处理条件也是影响扩增效果的重要因素,关键点包括:PMA浓度、暗反应时间、光反应时间[14-19],三者需要针对反应体系进行平衡优化(表2)。
PMA浓度应以能够最大程度抑制死菌,同时不影响活菌的后续定量为先决条件。TANTIKACHORNKIAT等[20]在研究中推测PMA的最佳浓度可能与细胞密度有关:细胞密度越高,则需要更高的PMA浓度才能有效抑制死菌DNA的扩增。有研究表明密度处于106~107CFU/mL的酵母和密度近似108CFU/mL的细菌得到准确定量结果的条件均为6 μmol/L PMA。
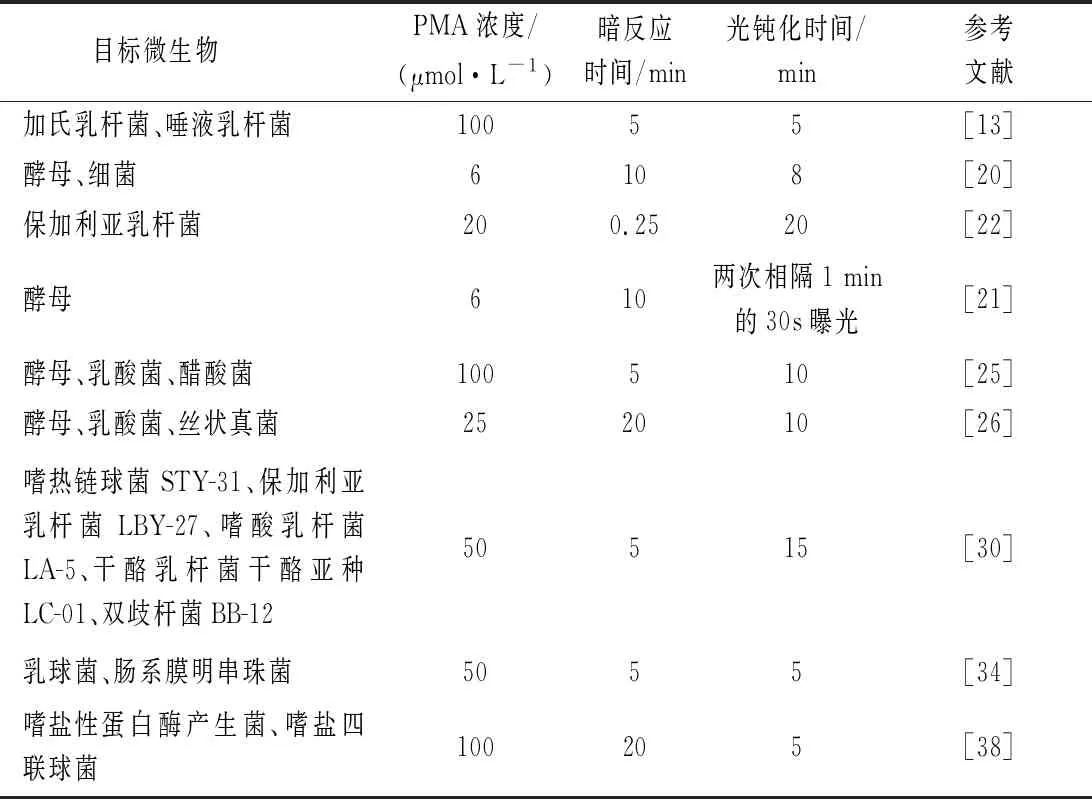
表2 针对不同微生物PMA处理条件优化Table 2 The optimal conditions of the PMA treatments for different microorganisms
暗反应时间的长短决定了PMA嵌入DNA的充分程度,嵌入DNA的PMA在强光照射下与DNA共价交联,未反应PMA则被钝化,光反应时间决定后续活菌DNA扩增效率。ANDORR等[21]测定酿酒酵母时发现使用6 μmol/L PMA处理样品,进行10 min暗反应后采用两次相隔1 min的30秒曝光方法定量结果更为准确。SHAO等[22]使用了20 μmol/L PMA进行20 min光钝化,可有效检测德氏乳杆菌(Lactobacillusdelbrueckiissp.)是否进入VBNC状态,这与NOCKER等[8]针对纯净环境培养细菌优化后的处理条件有所不同。
3 PMA-qPCR计数在发酵食品中的应用
3.1 酒
酿酒是一种复杂的生物化学反应过程,受到多种微生物和环境因素的影响,如何对酒样中微生物定性定量,对工艺控制及成品酒贮存至关重要。现阶段以此方法进行酒类研究较少,主要集中于葡萄酒。酿造中,随着主要发酵产物乙醇浓度的增加,酒样中细胞的膜流动性受到影响,对膜蛋白产生毒性,从而抑制细胞生长甚至导致死亡[23]。ANDORR[21]推测,发酵过程中高浓度酒精导致死菌大规模的出现,可能会对活菌定量产生影响;在实验中添加106个/mL膜损伤菌进行检测后发现,过量死亡细胞的存在并未干扰到菌群密度为103~107CFU/mL酵母菌的计数结果。将优化后的PMA处理条件用于发酵酒及陈酿酒中总酵母、酿酒酵母、非酿酒酵母和腐败酵母的检测,结果发现不同生理状态下酵母均可被快速定量。同时在不同葡萄酒酒样中利用Ct值和标准曲线对微生物进行计数时,应考虑基质的影响。RIZZOTTI等[24]在检测10种葡萄酒中酵母、乳酸菌及醋酸菌密度时,针对不同基质制作了相应标准曲线,大幅降低环境因素对结果的影响。
PMA-qPCR技术在其他酒类中也有应用,VENDRAME等[25]以布鲁氏酒香酵母(Brettanomycesbruxellensis)为目标菌株,建立起了一套能够在9 h快速检测酒样中是否存在腐败酵母的方法,检测限在红酒、白酒、啤酒中分别为0.83、0.63、0.23 lg CFU/mL。中国传统真菌发酵米酒中,LV[26]优化了对于酿酒酵母(S.cerevisiae),植物乳杆菌(L.plantarum)和紫红曲霉((M.purpureus)的PMA处理条件,同时对qPCR、PMA-qPCR、RT-qPCR处理结果进行了对比,发现PMA-qPCR与传统计数方法最为拟合,同时检测限较低,据此建立了监控米酒发酵过程中整体菌群动态变化的高效途径。
3.2 发酵乳
发酵乳是乳和乳制品在发酵菌的作用下发酵而成的酸性凝乳状制品,主要包括酸奶、奶酪及开菲尔等。发酵乳常被用于引入除发酵剂外益生菌的载体,益生菌是一种通过改善肠道微生物平衡从而对宿主施加有益影响的微生物添加物,乳杆菌和双歧杆菌是发酵乳生产中常使用的两种益生菌,用于改进发酵乳的口感、质地等感官特性,增强发酵乳的健康性能[27]。国际食品法典标准发酵乳卷[28](CODEX STAN 243—2003) 中规定在最低保质期内发酵乳中发酵微生物应是大量存活的,在除了用于发酵的特定发酵剂之外,发酵乳中微生物的计数至少要有106CFU /g。所以在发酵乳的生产和保存中微生物的存活率有直接影响。
3.2.1 酸奶
酸奶主要是以新鲜牛乳为原料,经乳酸菌发酵形成的风味独特、营养丰富的功能性乳制品。酸奶是益生菌最常见且经济的载体,在传统发酵剂基础上,发展益生菌酸奶可有效解决耐药菌株问题,同时满足人民对健康食品的不断需求[29]。以混合菌株作为发酵剂是增强酸奶益生性的较优方法,GARCA-CAYUELA等[30]利用PMA-qPCR检测了货架期内(28 d)及超出货架期30和60 d的酸奶样品中种水平上嗜热链球菌(Streptococcusthermophilus)STY-31、德氏乳杆菌保加利亚亚种(Lactobacillusdelbrueckiisubsp. bulgaricus)LBY-27、嗜酸乳杆菌(Lactobacillusacidophilus)LA-5、干酪乳杆菌干酪亚种(Lactobacilluscaseisubsp.casei)LC-01 和双歧杆菌(Bifidobacteriumlactis)BB-12的动态变化;结果发现, PMA-qPCR可以在设计了种间特异性引物的基础上,在复杂微生物环境条件中做到105CFU/mL菌浓度检测范围内的简单快速计数,实验中不会出现交叉污染现象,菌株的生存能力在货架期后才会减弱。目的基因的选择与特异性引物的设计在定量微生物方法中的重要性也被SCARIOT等[31]进一步证实,利用PMA-qPCR检测副干酪乳杆菌(L.paracasei)ATCC 10746的tuf基因,观察其在纯净环境和酸奶发酵过程中的生存状况,得到不同基质中标准曲线平均效率为94%和96% (R2> 0.98),两个样本的检测限(LOD)均为104CFU/mL。将发酵1和30 d的酸奶进行对比,发现目标菌株仍保持在108CFU/mL,qPCR方法得到的计数值高于PMA-qPCR。这些研究证明PMA-qPCR技术可用于在复杂酸奶发酵环境中快速定量益生菌,帮助生产者选取生存能力更强的优势菌株。
3.2.2 奶酪
奶酪同样是一种以混合发酵剂发酵的食品,作为益生菌的优良载体,具有独特的风味和质地。PMA-qPCR技术已被用于监控切打干酪(chadder cheese)发酵过程中益生菌生长情况,ÉMILIE[32]探究了添加3种益生菌对车打干酪发酵及成熟期的影响,样品以乳酸乳球菌为发酵剂,分别添加双歧杆菌(B.animalissubsp. lactis)BB-12、鼠李糖乳杆菌(L.rhamnosus)RO011、瑞士乳杆菌(L.helveticus)RO052和3种益生菌混合物,结果表明,益生菌添加会影响发酵菌株的生长,混合菌株之间虽存在抑制作用,但在成熟期奶酪中仍能保持较高活性(lg 9 CFU/g)。除此之外,也有学者使用PMA-qPCR技术与体外模拟实验相结合从而得出比培养依赖型定量更为高效的方法。VILLARREAL等[33]使用qPCR、PMA-qPCR结合平板计数法评价了嗜酸乳杆菌La-5、双歧杆菌BB-12和清酒乳杆菌(L.sakeisubsp. sakei)2a在小瑞士奶酪货架期间的存活率,发现3种方法在测定初期没有较大差异,然而随着体外诱导试验的变化,不同方法之间差异增大,电镜结果显示qPCR计数了大量膜损伤细胞而PMA-qPCR克服了这个问题。在检测中显示BB-12菌株有较好的生存能力,能够维持在6 lgCFU/g及以上。基于这些研究,ERKUS等[34]发掘了PMA-qPCR更大的应用潜力,以8种混合菌株为发酵剂的GUODA干酪在成熟过程中会出现大量膜不完整细胞,这给宏基因组带来了巨大挑战,用PMA处理发酵期和成熟期样品后选择性剔除了膜损伤细胞,从而可以对奶酪微生物群落中活细胞进行更高效的宏基因组鉴别分析。
3.2.3 开菲尔
开菲尔(Kefir)是一种利用开菲尔粒(Kefir grain),对牛奶、羊奶等发酵,得到的含醇、酸及少量CO2的发酵乳。开菲尔粒是一种天然存在的混菌发酵体系,内含多种微生物,分析其菌相组成是探讨发酵过程、代谢机制及确保发酵乳安全性的前提[35]。PORCELLATO等[36]将PMA-qPCR技术与变性梯度凝胶电泳(denaturing gradient gel electrophoresis,DGGE)及自动核糖体间隔基因分析(automated Ribosomal intergenic spacer analysis,ARISA)联用,探究了3个生产批次的开菲尔产品在货架期及超过货架期两周内的菌群动态变化。结果表明在储存过程中,PMA处理后的Kefir样品与原始样品中菌群存在显著差异,作为发酵剂的主要构成菌株链球菌属(Streptococcus)、乳球菌属(Lactococcus)和乳杆菌属(Lactobacillus)浓度明显下降,DGGE及ARISA检测不同批次样品中均出现了污染微生物。这种直接从食品中提取DNA进行分析的方法为开菲尔类发酵产品的研究提供了新的思路。
3.3 鱼露
鱼露因富含谷氨酸而被作为食品中天然的增鲜剂。它以低值鱼虾或水产品加工下脚料为原料,利用环境、鱼自身的酶系及耐盐酵母菌、乳酸菌、醋酸菌等微生物在一定条件下发酵而成[37]。因其自然发酵周期较长(12~18个月),生产中通常采用添加发酵剂的形式加速发酵和提高产品品质,所以监测添加菌种的生长变化对于发酵过程的质控是必要的。为了快速检测出富含28株原生菌株的复杂初始发酵环境中添加发酵剂的生长情况,UDOMSIL等[38]使用基于目标菌株碱性丝氨酸蛋白酶X基因(aprX)及内转录间隔区(ITS)设计的特异性引物进行精确定量,在优化后的PMA处理条件下,对嗜盐性蛋白酶产生菌(Virgibacillussp.)SK37和嗜盐四生球菌(Tetragenococcus.halophilus) MS33的最低限可达到103、102CFU/mL。实验结果证实PMA-qPCR检测的稳定性未受到复杂基质的影响,所以可被用于监测发酵剂发酵过程的实用工具(表3)。

表3 不同发酵食品活菌检测线性范围及最低检测限Table 3 The detection limits of PMA-qPCR technique for enumeration of living microbes in different fermentation foods
4 展望
PMA-qPCR技术是近年来较为新型的微生物计数技术,以其较高的工作效率和准确的定量结果受到很多学者的青睐。本文综述了近年来PMA-qPCR技术在发酵食品科研领域的应用情况;值得注意的是,由于特定发酵食品内环境的复杂性和特异性,在实际应用中需针对相应的食品基质进行PMA反应条件优化。
