皱皮香猪3 个基因组拷贝数变异的多态性分布
2019-06-18冉雪琴黄世会王嘉福
王 州,冉雪琴*,牛 熙,李 升,黄世会,王嘉福,2*
(1.贵州大学动物科学学院,山地植物资源保护与保护种质创新教育部重点实验室,贵州贵阳 550025;2. 铜仁学院,贵州铜仁 554300)
香猪体躯短圆,脂肪沉积能力强,属于脂肪型猪品种[1]。香猪主产区位于贵州省从江县月亮山区。皱皮香猪是从正常香猪群体分离而来,存在于不同猪场,在香猪群体中呈散发性存在,皱皮香猪作为亲本产生的后代中大约三分之一有皱皮性状,其主要特征是皱皮香猪躯干部皮肤表皮层和真皮层明显增厚,皮肤弹性差,背部以后皮下脂肪层变薄,后躯体侧特别是臀部皮肤褶皱明显。皮肤是动物机体最大的器官,从外到内分为表皮层、真皮层和皮下组织,具有保护机体内环境稳定、物质吸收、感受外界刺激、分泌、排泄、调节体温、参与新陈代谢和免疫反应等生理功能[2]。对中国沙皮犬的皮肤表型和周期性发热综合症进行的研究证明,透明质酸合成酶2(Hyaluronan Synthase 2,HAS2)基因上游约350 kb 处一段16.1 kb 的不稳定重复是导致沙皮犬周期性发热、透明质酸沉积、皮肤大量褶皱的主要原因[3]。基因中的重复变异属于基因组拷贝数变异范畴。拷贝数变异(Copy Number Variation,CNV)是与表型变异和疾病易感性相关的遗传变异的重要来源[4]。CNV 包括1 kb 到若干Mb 范围内的重复、插入、缺失和复杂多位点的微观和亚微观结构变异[5]。与单核苷酸多态性(Single Nucleotide Polymorphisms,SNP) 相 比,CNV 对基因组结构和序列区域的影响更大,通过改变基因的结构和剂量、影响基因调控元件和暴露隐性等位基因等方式影响基因的表达量,并引起表型差异和表型适应性变化[6]。过去几年中,在人、猪、鸡、牛和羊等多种生物中进行了大量的全基因组CNV 研究[7-13],多数CNV 出现在富含基因的区域,主要与一些复杂性状、疾病易感性及物种进化相关。如Agouti 信号蛋白(Agouti Signaling Protein,ASIP)基因编码区重复可能与绵羊的色素沉着有关[14]。涉及3 个成纤维细胞生长因子FGF3、FGF4 和FGF19 基因的133 kb 重复和ORAOV1 的复制使犬毛发脊和皮样窦的易感性增加[15]。SOX5 内含子1 的拷贝数增加导致鸡的豌豆冠[16]。在相同遗传背景的人群中,CCL3L1(MIP-1alphaP)基因拷贝数下降与人免疫缺陷病毒(HIV)/ 获得性免疫缺陷综合征(AIDS)易感性增强相关[17],还与类风湿性关节炎(RA)有显著关联[18]。KIT 基因拷贝数增加导致欧洲猪白毛色和白斑表型[19-20],但荣昌猪的白毛色可能与KIT 基因的CNV 没有直接联系[21]。这些研究提示,基因中的CNV 可能影响动物的生长发育、疾病易感性,以及饲料利用率、生育力和产奶量等多种表型,但可能具有品种特异性。
为探究香猪皱皮个体产生皮肤褶皱的原因,本文利用香猪皱皮个体全基因组测序数据,与猪参考基因组序列、大白猪、长白猪全基因组重测序数据相比较,得到5 538 个CNVs,从中选取3 个候选CNVs,研究皱皮香猪个体与正常香猪之间的群体变异规律。
1 材料与方法
1.1 CNV 信息 基于重测序数据预测基因组拷贝数变异,去重得到非冗余的CNVs,从中选取3 个CNVs 进行群体比较研究。CNVs 基本信息见表1。
1.2 材料 30 头正常香猪和30 头皱皮香猪(图1)的耳组织采于贵州大山地生态养殖有限责任公司猪场,30头大白猪血样采于贵州毕节。

图1 皱皮香猪(上)和正常香猪(下)皮肤褶皱比较
1.3 主要试剂 血液/组织基因组提取试剂盒、胶回收试剂盒、质粒提取试剂盒和荧光染料SYBR 均购自北京天根生化科技有限公司;大肠杆菌DH5α(实验室保存);pGEM-T Easy Vector 购自普洛麦格(北京)生物技术有限公司。酶标仪购自美国Bio Tech 公司;定量PCR仪(CFX96)为美国Bio-Rad 公司设备。
1.4 DNA 提取 参照基因组提取试剂盒说明书提取样品基因组DNA,质检合格存于-20℃备用。
1.5 引物设计 根据猪参考基因组序列(Scrofa 11.1),使用Primer Premier 5.0 设计检测CNVs 的特异性引物,以单拷贝的GCG(Glucagon)为内参,以DNAstar 分析引物的二级结构,mFold 在线预测(http://unafold.rna.albany.edu/?q=mfold/DNA-Folding-Form)目的片段的发卡结构,NCBI 网站的Blastn(https://www.ncbi.nlm.nih.gov/tools/primer-blast/)程序分析引物的特异性。引物由英潍捷基贸易有限公司合成(表2)。
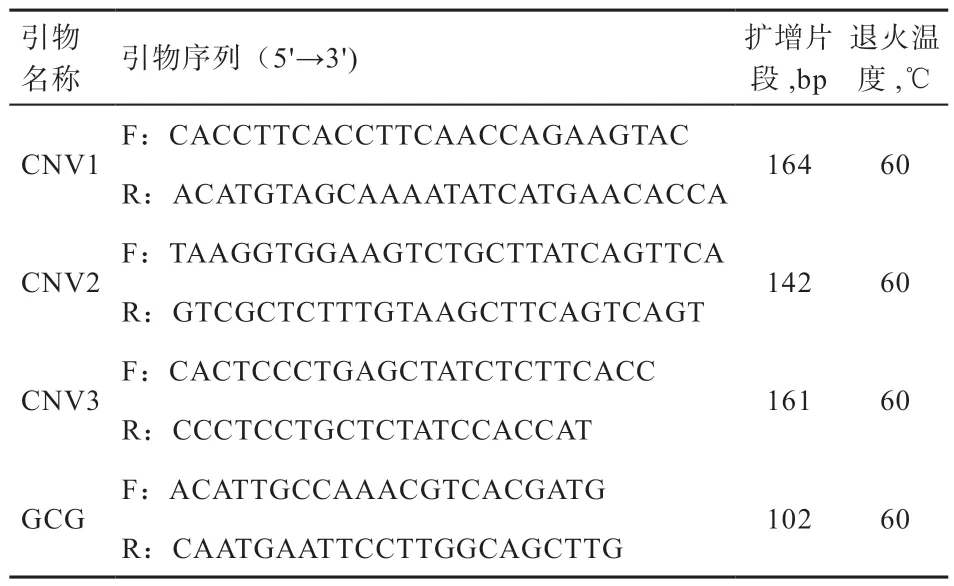
表2 引物序列
1.6 基因片段扩增与测序 以基因组DNA 为模板,使用20 µL PCR 反应体系:基因组DNA 1 µL,10 pmol/L 的上、下 游 引 物 各0.5 µL,2×PCR Mix 10 µL, 三 馏 水8 µL。PCR 反应条件:95℃预变性5 min;95℃变性30 s,58℃退火30 s,72℃延伸40 s,35 个循环;72℃延伸10 min。取60 µL PCR 产物进行琼脂糖凝胶电泳检测,回收目的片段,连接pGEM-T Easy Vector 载体,转化DH5α 菌,克隆得到阳性菌株,由贵州明涵生物科技有限公司测定核苷酸序列,提取重组质粒DNA。

表1 CNVs 基本信息
1.7 qPCR 检测拷贝数变异 将重组质粒DNA 按10 倍浓度梯度稀释(10-2~10-7),作为qPCR 扩增的模板,建立标准曲线。qPCR 反应体系10 µL:2 × SYBR Green Supper mix 5 µL,模板1 µL,浓度为10 pmol/L 的上、下游引物各0.3 µL,三蒸水3.4 µL。PCR 反应条件:95℃预变性10 min;95℃变性10 s,60℃退火30 s,共40 个循环 。熔解曲线55℃至95℃,递增速度为0.5℃/5s。采用相对定量的方法测定样本基因组中CNVs 的拷贝数,每个反应设3 次技术重复,以GCG 基因作为内参基因,利用2-ΔCt进行定量分析,依据文献[22]方法确定样品的拷贝数类型。
1.8 CNV 基因注释及功能分析 使用Ensembl 的VEP(http://asia.ensembl.org/Tools/VEP)工具注释CNV 中的基因,使用Kobas3.0(http://kobas.cbi.pku.edu.cn/anno_iden.php)作KEGG 和GO 富集分析,使用Panther(http://www.pantherdb.org/)分析基因的功能,使用Animal QTL Database(https://www.animalgenome.org/cgi-bin/QTLdb/SS/search)分析CNVs中的QTL 类型。
1.9 统计分析 数据按CNV 类型进行统计,计算各组CNV 类型的分布频率,包括拷贝数正常、拷贝数缺失、拷贝数增加3 种类型,通过SPSS 20.0 设置交叉表采用卡方检验分析CNVs 类型间的差异显著性。
2 结 果
2.1 基因片段克隆与测序 以样品基因组DNA 为模板,采用特异性引物扩增目的片段,经克隆测序,3 个CNVs 扩增片段与猪参考基因组(Scrofa11.1)序列的相似性均为99%,GCG 基因为100%。说明获得的扩增片段确实为猪基因组目的序列。
2.2 猪群基因组CNV 检测
2.2.1 标准曲线建立 以10 倍梯度稀释的重组质粒为模板,经qPCR 扩增绘制标准曲线。CNV1、CNV2 和CNV3 和GCG 基因的标准曲线呈线性,相关系数R2均在大于0.990,扩增效率在100%±10%(表3)。3 个CNVs 基因及内参GCG 基因定量引物的熔解曲线峰型单一,无杂峰;扩增曲线均呈S 形,拐点清楚,曲线整体平行性良好,曲线指数期的斜率与扩增效率呈正比,基线平直(图2~5)。表明扩增产物为单一的特异性产物,无引物二聚体等非特异性扩增产生,可以用2-ΔCt方法对基因进行相对定量。

表3 标准曲线和扩增效率
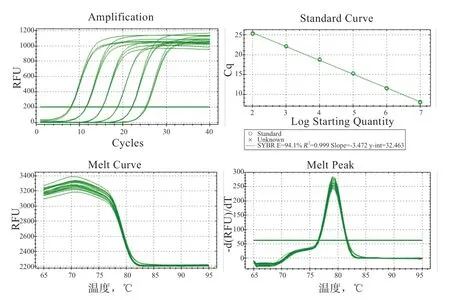
图2 CNV1 标准曲线

图3 CNV2 标准曲线

图4 CNV3 标准曲线

图5 GCG 基因标准曲线
2.2.2 3 个CNVs 的群体拷贝数变异规律 以基因组DNA为模板,采用荧光定量PCR 对90 头猪进行拷贝数检测表明, 3 个CNVs 真实存在,组间CNVs 分布频率不同,NR 值于0.01~7.4 范围[22]。3 个群体中,拷贝数缺失型变异主要以纯合缺失为主,拷贝数增加型变异主要以双拷贝增加和多拷贝增加为主,单拷贝的缺失或增加在群体中出现频率较低。
CNV1 在3 个群体中的优势变异类型频率为(表4),大白猪以拷贝数缺失为主(72.41%),皱皮香猪群体以拷贝数缺失为主(83%),正常香猪群体则以拷贝数正常占优势(63.33%)。对于CNV2(表5),大白猪、正常香猪群体和皱皮香猪群体均以拷贝数缺失为主,变异频率达93%、97%和37%。3 个群体中,大白猪的CNV3 以拷贝数缺失型变异占主要优势(90%),皱皮香猪群体以拷贝数增加型变异为主(66.67%),正常香猪拷贝数增加与拷贝数正常型较多,变异频率均为36.67%(表6)。
2.3 CNVs 基因注释及功能分析使用VEP 注释3 个CNVs 中的基因(表7),CNV1 内有14个基因,分别是SFT2D1、PRR18、RPS6KA2、FGFR1OP、CCR6、UNC93A、MPC1、GPR31、RNASET2、TTLL2、TCP10L2 和3 个Novel基因(ENSSSCG00000034196、ENSSSCG00000036759、ENSSSCG00000040654);CNV2 包含13个基因:KATNB1、ADGRG3、ADGRG1、DRC7、CCL17、RF00017、CX3CL1、COQ9、POLR2C、ADGRG5、CCDC102A、DOK4 和CIAPIN1;CNV3 有4个基因,其中LOC100513601 的3´ 端不在该CNV3 内,SLA-5、RF00001 和ENSSSCG00000001231 处于CNV3 之中。
与正常香猪和大白猪相比,CNV1 在皱皮香猪中以拷贝数缺失为主,KEGG 富集分析提示CNV1 中的基因参与调节多条信号通路(表8),包括多个疾病相关的通路(如I 型糖尿病、病毒性心肌炎和自身免疫性甲状腺疾病)、孕酮介导的卵母细胞成熟、卵母细胞减数分裂、胰岛素抵抗、抗原加工和递呈等。皱皮香猪的CNV2 和CNV3 以拷贝数增加占优势,KEGG 富集分析得出(表9),二者参与趋化因子信号通路以及多条与CNV1 相同的疾病相关通路、RNA 聚合酶、抗原加工和递呈等。GO 富集分析表明,3 个CNVs 主要参与生物调节、发育、免疫、代谢和刺激反应等生物学过程,且具有多种分子功能,主要涉及抗原结合、受体结合、酶活性(水解酶活性、连接酶活性和蛋白激酶活性)、G 蛋白偶联受体活性、细胞因子受体活性以及细胞骨架的结构成分等。
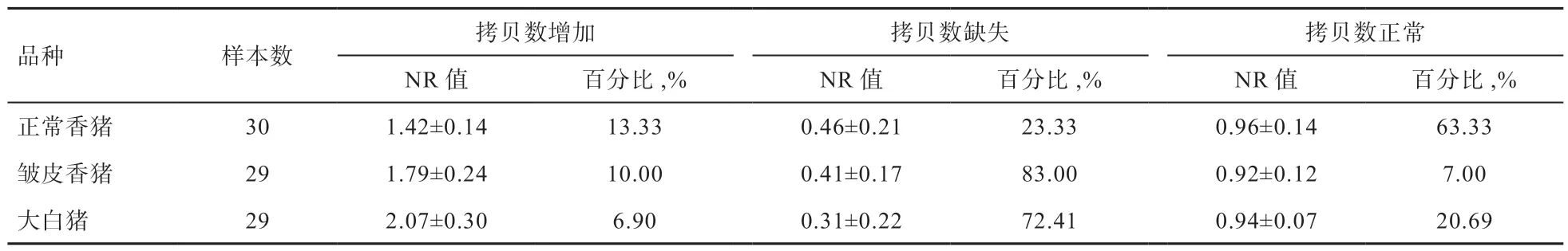
表4 组间CNV1 拷贝数变异分析

表5 组间CNV2 拷贝数变异分析

表6 组间CNV3 拷贝数变异分析
将3 个CNVs 与猪QTL Database 进行重叠分析,CNV1 区内未发现任何QTL 重叠;CNV2 和CNV3 2个区与125 个QTL 重叠,包括免疫能力、肉质和肉色、生长和繁殖、肩部皮下脂肪厚度和肌内脂肪含量等性状。
3 讨 论
3.1 贵州地方猪品种的皮肤比欧洲猪品种厚 皮肤是哺乳动物最大的器官,在保护机体内环境稳定和抵御外界病原物入侵等方面起着重要的屏障作用。研究表明,皮肤厚度会直接影响皮肤的感觉、防御、分泌、排泄等功能[23]。本实验结果表明,贵州地方猪品种的皮肤比欧洲猪品种厚。大白猪背部皮下脂肪平均厚度为14 mm,其表皮和真皮层平均厚度为2.54 mm[24];本实验中经实际测定,2 头正常香猪背部皮下脂肪平均厚60 mm,表皮和真皮层平均厚5 mm;2 头皱皮香猪背部皮下脂肪平均厚24 mm,表皮和真皮层平均厚8 mm。
3.2 CNV1 中的基因拷贝数缺失可能影响猪的脂肪代谢皱皮香猪具有皮下脂肪薄的优良特性,其脂肪代谢可能与CNV1 中的基因拷贝数缺失有关。群体检测结果表明,CNV1 在皱皮香猪群体和大白猪群体主要以拷贝数缺失(Deletion)型变异为主,变异频率分别为83.00% 和72.41%,而正常香猪群体的拷贝数缺失型变异频率为23.33%。卡方检验表明,皱皮香猪和大白猪的CNV1缺失型频率极显著高于正常香猪,皱皮香猪和大白猪之间差异不显著。线粒体丙酮酸载体1(Mitochondrial Pyruvate Carrier 1,MPC1,ENSSSCG00000032916)基因存在于CNV1 中,是控制丙酮酸通过线粒体内膜转运的关键因子,对碳水化合物、脂肪和氨基酸分解代谢以及合成代谢途径起重要作用。MPC1 基因被敲除或活性受抑时,线粒体丙酮酸代谢受阻,ATP 合成量下降,促进脂肪分解代谢[25]。Zou 等[26]研究证明,抑制MPC1 的活性可以促进脂肪酸氧化。MPC1±小鼠表现出脂解代谢加强、脂肪合成代谢下降[27]。MPC1 基因发生拷贝数缺失可能抑制其活性,继而促进脂解代谢和降低脂肪合成代谢。对CNV1 进行KEGG 富集分析表明,核糖体蛋白S6 激酶2(Ribosomal protein S6 Kinase A2,RPS6KA2/RSK3)富集到胰岛素抵抗信号通路,在该通路中RSK3 由胰岛素前体激活,然后磷酸化蛋白磷酸酶1,蛋白磷酸酶1 去磷酸化糖原合成酶,促进糖原合成。RSK3 基因的拷贝数缺失可能导致RSK3 酶活性下降,继而影响糖原的合成与存储,机体的能量需求转而由脂肪酸氧化提供。CNV1 中MPC1 和RSK3 基因发生拷贝数缺失,可能是影响皱皮香猪皮下脂肪变薄的原因之一。
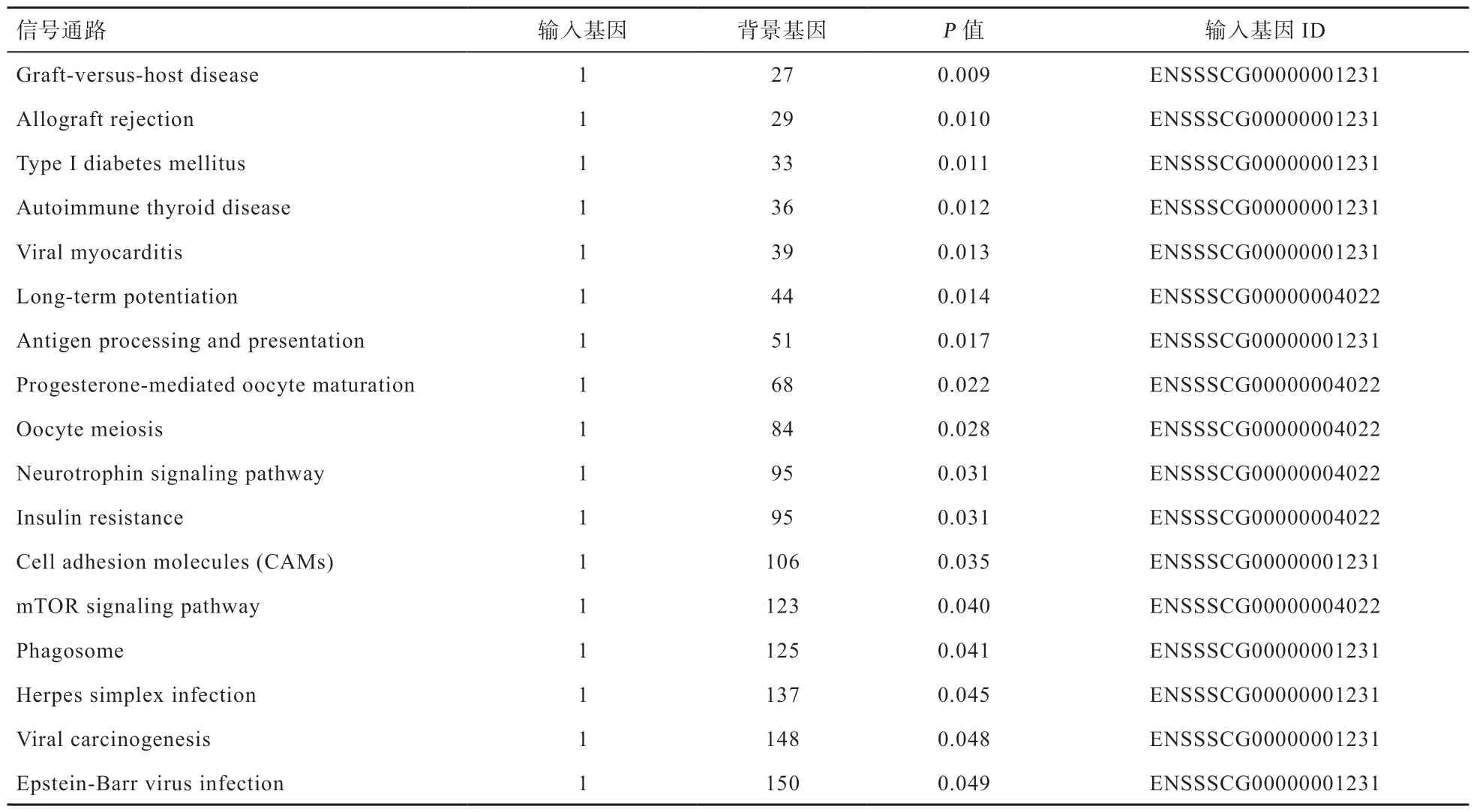
表8 CNV1 内基因参与的信号通路

表9 CNV2 和CNV3 内基因参与的信号通路
3.3 CCL17 和CX3CL1 基因拷贝数增加可能通过剂量效应引起猪皮肤产生褶皱 皱皮香猪具有皮肤表皮和真皮层增厚、褶皱明显和发红等特征,其可能与CNV2 和CNV3 中的CCL17 和CX3CL1 基因拷贝数增加有关。CNV2 和CNV3 在皱皮香猪群体中拷贝数增加型变异所占的比率显著高于正常香猪和大白猪群,正常香猪与大白猪群的构成比差异不显著。趋化因子CX3CL1(C-X3-C Motif Chemokine Ligand 1,CX3CL1/fractalkine)和胸腺和活化调节趋化因子(Thymus and Activation Regulated Che-mokine,TARC/CCL17)存在于CNV2 中,其参与调节趋化因子信号通路,参与各种皮肤炎症的发生[28-29],促进炎症部位白细胞的募集过程,可能与香猪皱皮性状的发生有关。Fractalkine 和CX3CR1 基因在系统性硬化症(SSc)患者受影响的皮肤中表达量上调,促进CX3CR1 阳性细胞向受影响组织的募集,导致皮肤炎症和血管损伤[30];Fractalkine 和CX3CR1 mRNA 表达在注射TGF-β 的WT 小鼠中增加,导致小鼠皮肤胶原蛋白过量、引发皮肤纤维化增生[31]。CCL17 由树突细胞(DC)、内皮细胞、角质形成细胞(KC)和成纤维细胞产生,参与炎症浸润,被指定为Th2 型趋化因子,诱导Th-2 型淋巴细胞向皮肤募集,启动Th2 细胞相关皮肤炎症的发展[32]。CX3CL1 和CCL17 基因的表达量在银屑病患者的受损部位皮肤中升高[33-34],CCL17 基因在急性过敏性接触性皮炎(ACD)患者的表达水平增加90 倍,CCL17 蛋白增加50 倍[35]。这些研究表明,趋化因子的表达水平与皮肤病的发生发展有着密切的关系,提示皱皮猪CX3CL1 和CCL17 基因发生拷贝数增加,其可能通过剂量效应引起猪皮肤产生褶皱。
4 结 论
本研究结果提示,3 个CNVs在猪群中呈现出多态性变化。CNV1 在皱皮香猪群中以拷贝数缺失型为主,其中的MPC1 和RSK3 基因拷贝数缺失可能与皱皮猪脂肪代谢有关;CNV2 和CNV3 在皱皮香猪群中以拷贝数增加型占优势,其中的趋化因子CCL17 和CX3CL1 基因可能通过剂量效应引起猪皮肤产生褶皱。
