木薯MeNAC29、MeNAC30基因的克隆及表达分析
2019-06-11林兆威李超萍时涛王国芬李博勋蔡吉苗黄贵修
林兆威 李超萍 时涛 王国芬 李博勋 蔡吉苗 黄贵修

摘 要 NAC(NAM/ATAF/CUC)基因家族是广泛分布于陆生植物的一类转录因子,在植物生长发育和胁迫应答的调控中发挥重要作用。本文对该基因家族在木薯抗细菌性萎蔫病方面的作用机制进行了初步研究。采用RT-PCR技术从木薯cDNA中克隆了MeNAC29和MeNAC30基因,并采用qRT-PCR技术对抗、感种质受木薯萎蔫病菌(Xanthomonas axonopodis pv. manihotis,Xam)侵染后2个基因的表达变化进行了定量分析。结果表明,2个基因均含有3个外显子和2个内含子,且均有保守的NAM结构域。MeNAC29基因的开放阅读框全长888 nt,编码295 aa。MeNAC30基因的开放阅读框全长870 nt,编码289 aa。接种木薯萎蔫病菌后,抗病种质中MeNAC29和MeNAC30基因的表达量显著高于感病品种,表明这2个基因参与了木薯对细菌性萎蔫病菌的抗性反应。
关键词 木薯;MeNAC29基因;MeNAC30基因;表达分析中图分类号 S533 文献标识码 A
Abstract The NAC (NAM/ATAF/CUC) transcription factor gene family was widely distributed in terrestrial plants and plays an important role in the regulation of growth and stress responses. The preliminary research about the role of this gene family on the cassava against bacterial blight was carried out in our lab. Two genes, MeNAC29 and MeNAC30, were cloned from cassava cDNA by RT-PCR, the different expression changes between resistant and susceptible cassava germplasms infected by Xanthomonas axonopodis pv. manihotis (Xam) were also quantified by qRT-PCR. The results showed both MeNAC29 and MeNAC30 contain three exons, two introns, and conserved NAM domain. The length of MeNAC29 genes ORF was 888 nt, which encode 295 amino acids, while the MeNAC30 genes ORF was 870 nt and encode 289 amino acids. Compared with susceptible cassava germplasms, both two genes expression was significantly increased in the resistant cassava germplasms - Xanthomonas axonopodis pv. manihotis interaction, which showed they are involved in resistance response of cassava to bacterial blight.
Keywords cassava; MeNAC29 gene; MeNAC30 gene; expression analysis
DOI 10.3969/j.issn.1000-2561.2019.04.012
由地毯草黄单胞木薯萎蔫致病变种(Xan tho monas axonopodis pv. manihotis,简称Xam)引起的木薯细菌性萎蔫病(也称细菌性枯萎病或细菌性疫病)是一种世界性病害,可造成木薯产量严重损失,甚至毁种绝收[1]。近年来,该病在广西、广东、海南等地区频发流行,在广西贵港、广东湛江等地区为害严重。培育和种植抗病(或耐病)品种是防治该病最有效的方法之一,但由于对木薯细菌性萎蔫病的抗病机制缺少研究,严重限制了相关工作的开展。
NAC基因家族是广泛分布于陆生植物的一类转录因子,在植物生长发育和胁迫应答的调控中发挥重要作用[2]。1996年Souer等[3]首次从矮牵牛中发现NAC基因家族。目前已确定拟南芥中有117个[4]、烟草中有152个[5]、水稻中有151个[6]、葡萄中有74個[7]、棉花中有145个NAC成员[8]。功能分析证实NAC家族部分基因参与植物抗病性调控作用,其中的ATAF亚类基因不仅在结构上相似,在胁迫响应上也具有保守性[9]。拟南芥ATAF类基因ATAF1的过表达能增强拟南芥对丁香假单胞杆菌番茄致病变种的敏感性[10],而过表达ATAF2能抑制尖胞镰刀菌相关蛋白的合成[11]。辣椒受细菌性斑点病病原侵染时,ATAF类基因CaNAC1的表达量快速增加,通过作用于病原侵染信号转导途径提高抗病性[12]。孙利军[13]研究发现,水稻中ATAF类基因OsNAC4在水稻与白叶枯病菌的非亲和性互作过程中上调表达。
目前有关NAC家族在木薯抗病反应中的作用研究还很缺乏。Hu等[14]通过转录组学分析,鉴定了木薯基因组中有96个NAC基因并将其分成16类,其中属于ATAF类(MeNAC29和MeNAC30)的MeNAC29在渗透、盐、冷、H2O2及脱落酸等处理中被诱导表达,分析MeNAC29和MeNAC30有可能参与木薯的抗病反应。本研究开展了这2个基因的克隆及其在木薯抗、感病种质受细菌性萎蔫病菌侵染过程中的差异表达分析,以期为进一步的调控作用机理研究和抗病育种工作提供理论依据。
1 材料与方法
1.1 材料
供试木薯抗病种质为‘优抗9号(RXC9)、‘优抗10号(RXC10)和‘优抗11号(RXC11),感病品种为‘华南205(SC205)、‘桂热4号(GR4)和‘华南8号(SC8),木薯种茎、细菌性萎蔫病菌菌株XamGX11均由中国热带农业科学院环境与植物保护研究所提供。
RNAprep Pure多酚多糖植物总RNA提取试剂盒、反转录合成第一链试剂盒FastQuant RT kit(With gDNase)、SuperReal PreMix Plus (SYBR Green)实时荧光定量PCR试剂盒购自天根生化科技(北京)有限公司。PCR产物回收纯化试剂盒E.Z.N.A.TM Gel Extraction Kit购自Omega Bio-Tek公司。pMD-18T载体、Taq DNA聚合酶、dNTP等购自宝生物工程(大连)有限公司。DNA Marker、Trans5α大肠杆菌感受态细胞购自北京全式金生物技术有限公司。引物合成与测序由北京六合华大基因股份有限公司完成。
1.2 方法
1.2.1 病原菌接种 病原菌菌株活化后,用YGP培养基(参照《植病研究方法》[15]的配方制备)培养并制备3×108 cfu/mL的菌液。参照卢昕等[16]的方法,将木薯种茎种植于直径30 cm的塑料花盆中,常规管理2个月后,在日平均气温30 ℃条件下,选择完全展开、完全转绿、无病虫危害的健康叶片,用注射法进行人工活体接种,分别在接种后0、12、24、48、72 h取叶片样品。
1.2.2 木薯叶片RNA提取及合成第一链cDNA 木薯叶片RNA提取参照RNAprep Pure多酚多糖植物总RNA提取试剂盒说明书,合成第一链cDNA参照FastQuant cDNA试剂盒明书。
1.2.3 MeNAC29、MeNAC30的克隆 以获得的第一链cDNA为模板,根据NCBI上登录的木薯种质为AM560-2的NAC29(GenBank登录号:XM_021773675.1)和NAC30(GenBank 登录号:XM_021748980.1)设计特异性引物(表1)进行RT-PCR扩增。PCR产物回收、连接、转化后,选取3个阳性克隆送至深圳华大基因股份有限公司进行测序。
1.2.4 MeNAC29、MeNAC30生物信息学分析 利用NCBI在线工具、ProtParam、TMHMM、SignalP、Promoter Scan、SOPMA、SWISS-MODEL等软件分析目的基因的结构、同源性、编码蛋白理化性质、二级结构、三级结构、聚类分析等。
1.2.5 MeNAC29、MeNAC30在木薯-细菌性萎蔫病菌互作过程中的差异表达分析 根据克隆的
MeNAC29、MeNAC30的cDNA序列设计特异性引物(表1),以各样品的第一链cDNA为模版,参照SuperReal PreMix Plus (SYBR Green)说明书进行实时荧光定量PCR。扩增程序:95 ℃预变性15 min,95 ℃变性10 s,60 ℃退火/延伸30 s(荧光信号采集),共40个循环。基因的相对表达量用2-ΔΔCT法计算。实验数据用SAS 9.1软件进行数据差异显著性分析[17]。
2 结果与分析
2.1 MeNAC29与MeNAC30的克隆与序列分析
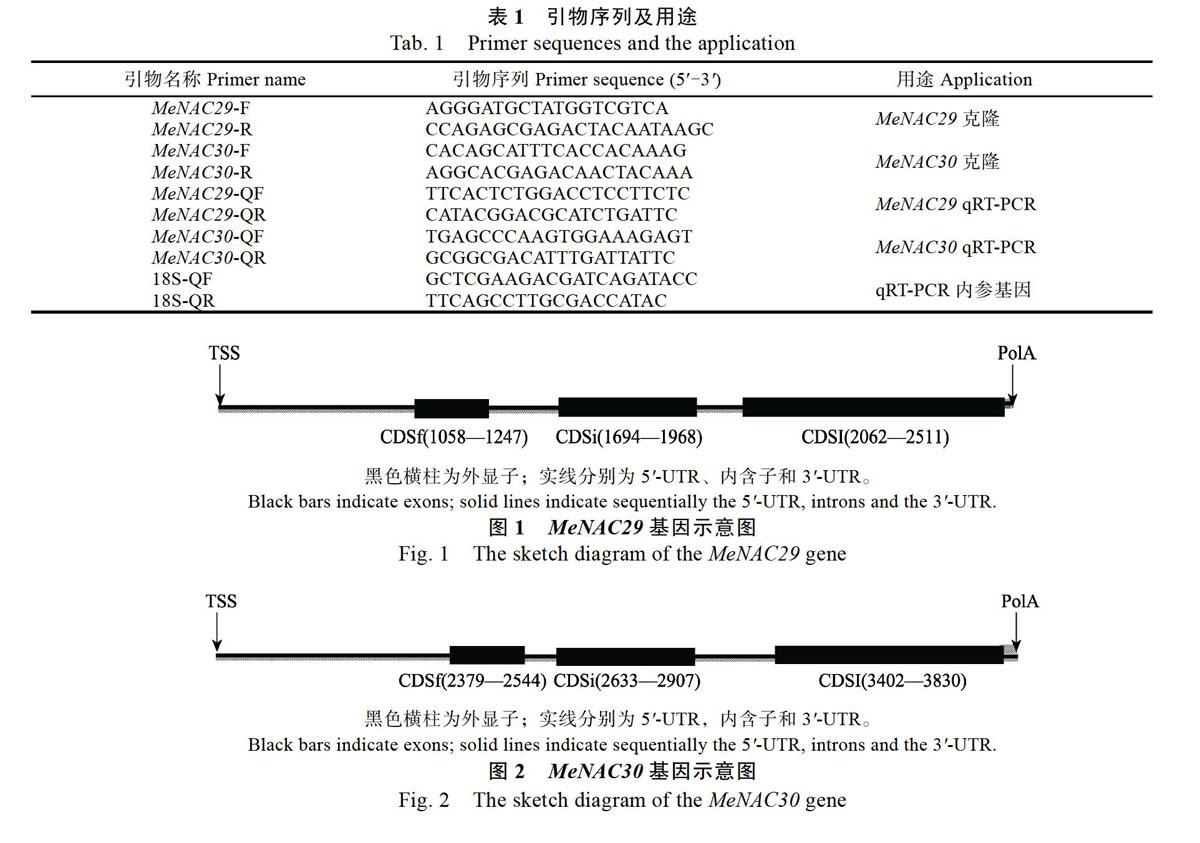
2.1.1 MeNAC29与MeNAC30的克隆及核苷酸序列分析 提取木薯种质RXC9、RXC11及GR4样品RNA,并合成第一链cDNA。以cDNA作为模板,克隆得到的MeNAC29基因片段长度分别为1020、1019、1010 nt,3个核苷酸序列之间的同源性在97.36%以上,均有一个全长为888 nt的ORF,预测其编码295 aa,且3个氨基酸序列之间同源性为99.77%;与木薯基因组(品种AM560-2)比对发现[18],MeNAC29含3个外显子和2个内含子,转录起始位点(TSS)位于起始密码子上游1084 nt处(图1)。木薯种质RXC9、RXC11及GR4的MeNAC30克隆片段长度分别为1213、1163、1184 nt,序列之间同源性均在94.77%以上,均有一个全长为870 nt的ORF,预测其编码289 aa,3个氨基酸序列之间同源性为99.65%;与木薯基因组(品种AM560-2)本地Blast比对发现,MeNAC30含3个外显子和2个内含子。转录起始位点(TSS)位于起始密码子上游2376 nt处(图2)。分析MeNAC29基因编码蛋白含有TFIID元件和JunB-US2元件,MeNAC30基因含TFIID元件、CTF/NF-1和CTF元件;2個基因起始密码子上游均无CpG island 。
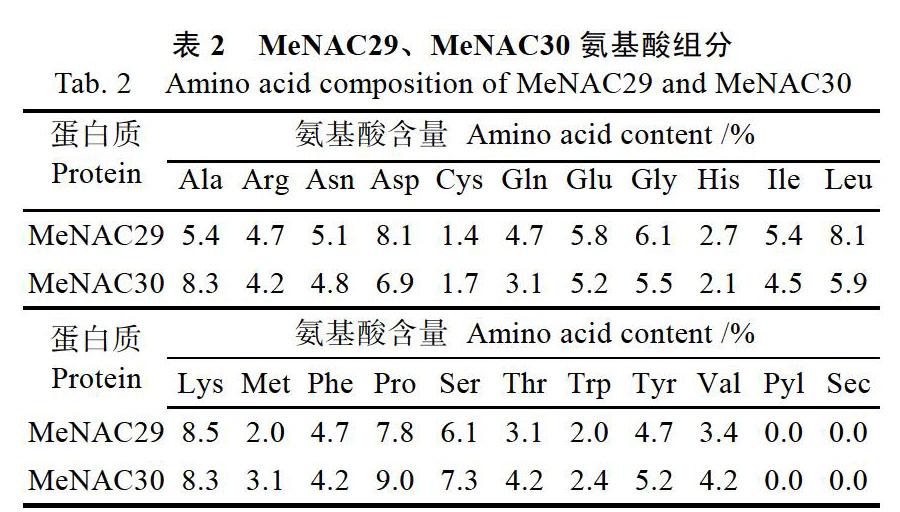
2.1.2 MeNAC29与MeNAC30氨基酸序列分析 分析结果表明,MeNAC29编码蛋白的分子质量为34.02 ku,等电点为6.46,含正电荷氨基酸残基数(Arg+Lys)为39,含负电荷氨基酸残基数(Asp+Glu)为41,在组成肽链的20种氨基酸中,赖氨酸(Lys)含量最高,占8.5%(表2)。MeNAC30编码蛋白的分子质量为32.76 ku,等电点为7.61,
含正电荷氨基酸残基数(Arg+Lys)为36,含负电荷氨基酸残基数(Asp+Glu)为35,组成肽链的20中氨基酸中,脯氨酸(Pro)含量最高,占9.0%(表2)。 2个基因均不含吡咯赖氨酸(Pyl)和硒代半胱氨酸(Sec)密码子,预测结果显明二者表达的蛋白质均不稳定且无信号肽和跨膜区域。蛋白保守结构域分析表明,MeNAC29与MeNAC30预测蛋白均含有NAC家族保守的NAM结构域,并在N端具有高度的相似性,均含α-螺旋、β-转角和无规则卷曲3种结构(图3)。以水稻NAC家族的NAC1为模型,获得了2个蛋白的三维结构模型(图4、图5),且与预测的二级结构基本一致。
2.2 聚类分析
选取与MeNAC29蛋白同源的14个NAC蛋白进行聚类分析。结果表明,MeNAC29与MeNAC30位于2个完全不同的分枝,MeNAC30与同属于大戟科的麻风树亲缘关系最近,与十字花科的拟南芥、萝卜较近;MeNAC29与同属于大戟科的蓖麻、橡胶亲缘关系最近,与豆科的大豆、鹰嘴豆较近;葫芦科的黄瓜、南瓜和茄科的辣椒、西红柿,均与MeNAC29与MeNAC30亲缘关系较远;单子叶植物玉米、粳稻、小麦另聚为一类(图6)。
2.3 MeNAC29、MeNAC30基因在木薯-细菌性萎蔫病菌互作过程中的差异表达分析
qRT-PCR结果发现,抗病种质(RXC9、RXC10、RXC11)在接种XamGX11 48 h时,MeNAC29 与MeNAC30的表达量高于对照(0 h),说明抗病种质接种病原菌后2个基因的表達被诱导;而感病种质(SC205、GR4、SC8)在接菌后所有取样点的MeNAC29 与MeNAC30表达量均低于对照(0 h),说明感病种质接病原菌后2个基因表达受到抑制。抗、感种质之间比较发现,抗病种质MeNAC29 与MeNAC30基因表达量在侵染过程中均显著高于感病种质(P< 0.05)。MeNAC29在抗病种质中的表达模式相似,均呈先下降后上升再下降的规律;MeNAC29 与MeNAC30在RXC9中表达模式均呈先下降后上升再下降的规律(图7)。总之,抗病种质被病原菌侵染后MeNAC29 与MeNAC30能被诱导表达,而感病种质接种病原菌后基因表达受到抑制,抗病种质MeNAC29和MeNAC30的表达量显著高于感病种质。故认为MeNAC29 与MeNAC30参与木薯和Xam之间的互作反应。
3 讨论
本研究从木薯种质RXC9、RXC11、GR4中克隆了MeNAC29和MeNAC30基因,其预测蛋白结构与绝大部分NAC家族成员相似[19-20],N端均含有一个高度保守的NAM结构域,核苷酸序列比对同源性达到97%以上,氨基酸序列比对仅存在1个aa的差异,表明这些蛋白的功能具有很强的保守性。聚类分析显示MeNAC29与MeNAC30位于不同的分支上,可能与NAC家族成员C端多样性较高、保守性较低有关,由于C端作为转录激活结构域,主要起转录激活的作用[21-23],故对其功能的发挥影响不大。本研究发现受Xam侵染后,抗病种质中MeNAC29和MeNAC30的表达量均显著升高,而感病种质中的表达量则下调,分析MeNAC29和MeNAC30参与木薯抗Xam侵染过程,与该基因家族成员的功能研究结果基本相似[13-14]。
相关研究表明,木薯对细菌性萎蔫病菌的抗性是一种数量遗传关系。Jorge等[24]研究发现木薯有8个数量遗传性状基因和抗性相关,能够解释9%~20%的表型差异。Wydradeng 等[25]利用木薯种质CM2177-2和TMS30572的杂交子代及其回交代进行了4个病菌分离物的接种试验,结果表明这些子代中有16种基因型呈现不同水平的抗性反应。Wang等[26]比较了野生木薯(W14)和栽培种木薯(KU50)基因组,发现其具有高度杂合性和数百万个单核苷酸变异。而本研究所选用的木薯抗性种质和主栽品种均为人工选育而来,遗传背景复杂。MeNAC29和MeNAC30基因在抗、感病种质受病菌侵染时的表达量变化及变化趋势并不相同,分析不同种质的抗病性反应调控机制之间的差异将是后续研究的重点。
NAC基因家族成员之间存在功能冗余现象,如拟南芥的NAC019/ANAC055、ATAF2/At5g63790为功能冗余基因对,这些基因对的单个基因插入突变或沉默不会引起表型改变,只有2个基因同时突变时才有表型改变[27-28]。由于MeNAC29与MeNAC30在RXC9中表达模式相似,二者之间是否存在基因功能冗余现象还有待进一步研究。
参考文献
Lozano J C. Cassava bacterial blight: a manageable disease[J]. Plant Disease, 1986, 70(12): 1089-1093.
Olsen A N, Ernst H A, Leggio L L, et al. NAC transcription factors: structurally distinct, functionally diverse[J]. Trends in Plant Science, 2005, 10(2): 79-87.
Souer E. The no apical meristem gene of Petunia is required for pattern formation in embryos and flowers and is expressed at meristem and primordia boundaries[J]. Cell, 1996, 85(2): 159-170.
Riechmann J L, Heard J, Martin G, et al. Arabidopsis transcription factors: genome-wide comparative analysis among eukaryotes[J]. Science, 2000, 290(5499): 2105-2110.
Rushton P J, Bokowiec M T, Han S, et al. Tobacco transcription factors: novel insights into transcriptional regulation in the Solanaceae[J]. Plant Physiology, 2008, 147(1): 280-295.
Nuruzzaman M, Manimekalai R, Sharoni A M, et al. Genome-wide analysis of NAC transcription factor family in rice[J]. Gene, 2010, 465(1): 30-44.
Wang N, Zheng Y, Xin H P, et al. Comprehensive analysis of NAC domain transcription factor gene family inVitis vinifera[J]. Plant Cell Reports, 2013, 32(1): 61-75.
Shang H H, Li W, Zou C S, et al. Analyses of the NAC Transcription Factor Gene Family in Gossypium raimondii Ulbr.: Chromosomal Location, Structure, Phylogeny, and Expression Patterns[J]. Journal of Integrative Plant Biology, 2013, 55(7): 663-676.
李 偉, 韩 蕾, 钱永强, 等. 植物NAC转录因子的种类、特征及功能[J]. 应用与环境生物学报, 2011, 17(4): 596-606.
Wang X, Basnayake B, Zhang H, et al. The Arabidopsis ATAF1, a NAC transcription factor, is a negative regulator of defense responses against necrotrophic fungal and bacterial pathogens[J]. Molecular Plant-Microbe Interactions, 2009, 22(10): 1227-1238.
Delessert C, Kazan K, Wilson I W, et al. The transcription factor ATAF2 represses the expression of pathogenesis –rela ted genes in Arabidopsis[J]. Plant Journal, 2010, 43(5): 745-757.
Oh S K, Lee S, Yu S H, et al. Expression of a novel NAC domain-containing transcription factor (CaNAC1) is preferentially associated with incompatible interactions between chili pepper and pathogens[J]. Planta, 2005, 222(5): 876-887.
孙利军. 水稻ONAC家族基因重叠表达特性及其在抗病抗逆中的功能研究[D]. 杭州: 浙江大学, 2012.
Hu W, Wei Y, Xia Z, et al. Genome-wide identification and expression analysis of the NAC transcription factor family in Cassava[J]. Plos One, 2015, 10(8): e0136993.
方中达. 植病研究方法(第3版) [M]. 北京: 中国农业出版社, 1998: 47.
卢 昕, 李超萍, 时 涛, 等. 国内603份木薯种质对细菌性枯萎病抗性评价[J]. 热带农业科学, 2013, 33(4): 57-70, 90
阮 敬. SAS统计分析从入门到精通[M]. 北京: 人民邮电出版社, 2009: 96-118.
Prochnik S, Marri P R, Desany B, et al. The cassava genome: current progress, future directions[J]. Tropical Plant Biology, 2012, 5(1): 88-94.
Nuruzzaman M, Manimekalai R, Sharoni A M, et al. Genome-wide analysis of NAC transcription factor family in rice[J]. Gene, 2010, 465(1-2): 30-44.
Cenci A, Guignon V, Roux N, et al. Genomic analysis of NAC transcription factors in banana (Musa acuminata) and definition of NAC orthologous groups for monocots and dicots[J]. Plant Molecular Biology, 2014, 85(1-2): 63-80.
Xie Q, Frugis G, Colgan D, et al. Arabidopsis NAC1 transduces auxin signal downstream of TIR1 to promote lateral root development[J]. Genes & Development, 2000, 14(23): 3024-3036.
Hegedus D, Min Y, Baldwin D, et al. Molecular characterization of Brassica napus NAC domain transcriptional activators induced in response to biotic and abiotic stress[J]. Plant Molecular Biology, 2003, 53(3): 383-397.
Tran L S, Nakashima K, Sakuma Y, et al. Isolation and functional analysis of Arabidopsis stress-inducible NAC transcription factors that bind to a drought-responsive cis- element in the early responsive to dehydration stress 1 promoter[J]. Plant Cell, 2004, 16(9): 2481-2498.
Jorge V, Fregene M A, Duque M C, et al. Genetic mapping of resistance to bacterial blight disease in cassava (Manihot esculenta Crantz) [J]. Theoretical and Applied Genetics, 2000, 101(5-6): 865–872.
Wydra K, Zinsou V, Jorge V, et al. Identification of pathotypes of Xanthomonas axonopodis pv. Manihotis in africa and detection of quantitative trait loci and markers for resistance to bacterial blight of cassava[J]. Phytopathology, 2004, 94: 1084-1093.
Wang W, Feng B, Xiao J, et al. Cassava genome from a wild ancestor to cultivated varieties[J]. Nature Communications, 2014, 10(5): 5110.
Cuperus J T, Fahlgren N, Carrington J C. Evolution and functional diversification of MIRNA genes[J]. Plant Cell, 2011, 23(2): 431-442.
Rhoades M W, Reinhart B J, Lim L P, et al. Prediction of plant microRNA targets[J]. Cell, 2002, 110(4): 513-520.
