亚硝态氮胁迫下菲律宾蛤仔实时定量PCR内参基因的筛选
2019-06-10王爱云曹滕飞吕家森丛明
王爱云,曹滕飞,吕家森, #,丛明
1. 鲁东大学生命科学学院,烟台 264025 2. 烟台大学生命学院,烟台 264003 3. 中国科学院烟台海岸带研究所,烟台 264003
近岸海域是海水增养殖的重要区域,其水质状况直接制约着该区域水产养殖业的健康发展。《中国近岸海域环境质量公报》显示,我国近岸海域的局部海域水质污染程度较重,无机氮是主要的污染因子之一[1-3],而氨氮和亚硝态氮是水环境中2种主要的有毒含氮化合物。环境中的氨氮在氨氧化细菌的作用下发生化学反应,转化成亚硝态氮,后者在硝化细菌的作用下转化成硝酸氮。由于近岸海洋环境中含氮有机污染物的浓度远远超过环境微生物的硝化能力,因此亚硝态氮成为近海海域一种常见的含氮污染物。我国浅海滩涂为贝类的黄金养殖地带,近岸的亚硝态氮也就成为贝类养殖环境中不可忽视的污染物之一,严重威胁贝类养殖业的健康发展。国内外关于亚硝态氮对贝类的生态毒理研究,主要集中在致死效应和生理生化反应等方面[4-7],毒性相关基因的研究比较少。
为了更好地研究亚硝态氮对贝类的毒性效应,尤其是从基因角度评价亚硝态氮毒性效应,需要一个合适的内参基因作为评价标准。据文献报道,0.1~0.5 mg·L-1的亚硝酸盐(以氮计)能对水生生物造成中毒症状,超过0.1 mg·L-1时会造成鱼虾等慢性中毒,而当亚硝酸氮浓度达到0.5 mg·L-1时,则会导致养殖生物大面积死亡[8-9]。为了研究低浓度亚硝态氮对贝类的毒性影响情况,本实验采用0.2 mg·L-1亚硝酸氮作为胁迫浓度。贝类的鳃组织是重要的摄食、呼吸器官,也是与外界海水环境进行接触的主要器官,因此被认为是最易受到外界环境胁迫的靶器官。本研究以菲律宾蛤仔的鳃组织为研究对象,采用0.2 mg·L-1亚硝酸氮作为胁迫浓度对菲律宾蛤仔进行不同时间(1 d、7 d)的胁迫实验,利用内参基因分析软件geNorm和NormFinder筛选稳定表达的内参基因。本研究的顺利开展,将为研究菲律宾蛤仔鳃组织的毒理机制,筛选毒性关键基因提供基础数据;为从基因的角度系统研究亚硝态氮对贝类的致毒机理,防治亚硝态氮引致的危害奠定基础。
1 材料与方法(Materials and methods)
1.1 材料
实验用菲律宾蛤仔购于山东省烟台市大润发超市,选取足部伸缩有力、健康的菲律宾蛤仔(11±2.0) g作为实验对象,暂养10 d。暂养和胁迫期间,全天连续充气,水温(20±2) ℃,盐度30.0,pH值8±0.1,实验用海水经过3级沙滤,每日喂食螺旋藻液1次(终浓度为5×103cells·mL-1左右),在喂食前和喂食2 h后100%换水各1次。每天喂食换水后,在胁迫组水体中加入新鲜配制的亚硝酸钠母液,使其氮浓度达到0.2 mg·L-1。亚硝酸氮浓度每天于喂食换水前和喂食2 h换水后各检测1次(检测方法参照GB/T 7493—1987),亚硝酸氮浓度范围为(0.24±0.04) mg·L-1。实验设置胁迫时间为7 d,分别在实验的第1和7天取样,作为急性和亚急性胁迫样品。
实验用亚硝态氮源为亚硝酸钠(国药集团化学试剂有限公司生产, 分析纯),烘干至恒重,用0.22 μm滤膜过滤除菌的海水配成1 mol·L-1的母液,避光冷藏保存,备用。本实验设2组:对照组(0 mol·L-1),胁迫组(0.2 mol·L-1),每组设3个重复,每个重复用菲律宾蛤仔30只。每个重复随机选取6只菲律宾蛤仔,取其鳃组织,将其完全浸入TRIzol液中,-20 ℃保存。
1.2 总RNA的提取及cDNA制备
用Trizol (TaKaRa)试剂盒提取菲律宾蛤仔鳃组织的总RNA,用琼脂糖凝胶电泳(12.0 g·L-1)检测其完整性和纯度,用紫外分光光度计NanoDrop 2000 (Thermo Scientific, America)确定RNA浓度,-80 ℃保存样品总RNA。用试剂盒EraserPrimeScriptTMRT reagent Kit with gDNA Eraser (TaKaRa, 大连)去除基因组DNA (gDNA)后,再进行反转录实验,获得菲律宾蛤仔鳃组织的cDNA。采用SYBR Green qPCR (染料法),使用20.0 μL的反转录体系,加入总RNA 1.0 μg、去除gDNA的反应液10 μL,其他成分和用量参照试剂盒说明;反转录程序:37 ℃、15 min,85 ℃、5 s,4 ℃储存,最后于-20 ℃保存反转录产物。
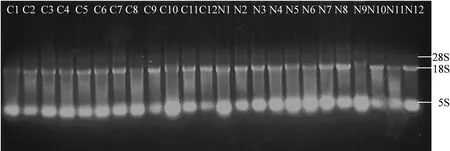
图1 对照组和亚硝态氮胁迫组菲律宾蛤仔鳃组织总RNA凝胶电泳图谱注:C1~12,Control组样品1~12;N1~12,亚硝态氮胁迫组样品1~12。Fig. 1 Gel electrophoresis of total RNA samples from the gill tissues of R. philippinarum in the control and nitrite-exposed groupsNote: C1-12 represented samples from the control groups; N1-12 represented samples from the nitrite-exposed groups.
1.3 引物特异性检测
本研究选取微管蛋白基因(beta-tubulin,Tubu)、肌动蛋白基因(β-actin,Actin)、转录延伸因子基因(elongation factor 1-alpha,EF1α)、核糖体(18SrRNA, 18S)、泛素蛋白基因(Ubiquitin,Ubi)和亲环素基因(cyclophilin A,CyPA)等6个基因作为候选内参基因。根据课题组前期实验结果[10],选用6对候选内参基因的引物并交由生工生物技术(上海)有限公司合成。利用普通PCR扩增、琼脂糖凝胶电泳(12.0 g·L-1)验证6对引物的可靠性。20.0 μL的PCR反应体系包括10 × PCR Buffer 2.0 μL,dNTP Mixture 1.5 μL,MgCl21.2 μL,Taq 0.2 μL,上、下游引物各1.0 μL,以等量混合的胁迫组和对照组鳃组织RNA反转录合成的cDNA做模板,用量1.0 μL,RNAase-Free Water 10.1 μL。反应程序为94 ℃预变性5 min,94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸30 s,循环36次,最后,72 ℃保温10 min,产物4 ℃储存。
1.4 实时荧光定量PCR的扩增
将亚硝态氮胁迫的实验组及对照组的cDNA各取2.0 μL混匀,将其浓度分别稀释20、50、100、200、500倍作cDNA模板。按照SYBR Select Master qRT-PCR试剂盒 (MixApplied Biosystems, America)使用说明,在7500 Fast Real-Time PCR System (Applied Biosystems, America)上进行扩增反应。20.0 μL的反应体系包括SYBR Select Master Mix 10.0 μL,上、下游引物(10 μmol·L-1)各0.6 μL,cDNA(50×)模板5 μL,ddH2O 3.8 μL。qRT-PCR反应程序为94 ℃预变性7 min;94 ℃变性10 s;60 ℃退火延伸30 s;循环40次。扩增实验结束后,进行熔解曲线分析。
1.5 数据处理
根据7500 System Software获取的qRT-PCR扩增曲线,用-2-ΔΔCt法[11]计算样本的相对表达量;分别利用软件geMorm和NormFinder对6个内参基因的表达稳定性值(expression stability value,M)进行评价,再采用加权赋值法[12]综合比较基因的表达稳定性,从而确定最适合的内参基因。
2 结果(Results)
2.1 总RNA提取
随机选取2个处理组的24个样品进行琼脂糖凝胶(1.2 g·L-1)电泳检测,发现样品总RNA 28S、18S、5S的条带清晰(图1)。NanoDrop 2000检测其A260/A280在1.8~2.0范围内,A260/A230均大于2.0,说明RNA的提取质量符合后续的qRT-PCR要求。
2.2 cDNA质量和引物特异性检测
以鳃组织的混合cDNA为模板进行普通PCR扩增后,用12.0 g·L-1的琼脂糖凝胶电泳检测,发现扩增产物均为单一清晰条带,且产物大小与目标产物大小匹配。荧光定量PCR熔解曲线分析显示,6对引物的扩增产物均呈现单一峰且重复性好(图2)。由此说明,所采用的6对引物不存在非特异性扩增,也没有形成引物二聚体,均适用于qRT-PCR分析。将模板进行不同浓度梯度稀释后用引物扩增,结果表明,cDNA模板稀释50倍时,内参基因的Ct值均在12.0~25.0范围内,适合作为检测的模板浓度。
2.3 内参基因表达的稳定性评估
2.3.1 geNorm软件评估
将对照组和亚硝态氮胁迫(1 d、7 d)的菲律宾蛤仔的鳃组织cDNA分别进行qRT-PCR扩增,用-2-ΔΔCt法计算出各候选基因的相对表达量,将其值输入geNorm软件进行分析。根据分析结果,获得6个候选内参基因的平均表达稳定值M:18S(1.066)、Actin(0.829)、Tubu(1.195)、EF1α(0.938)、Ubi(0.948)和CyPA(0.882) (表1)。依据geNorm软件的分析原则,M值越小,则说明该基因表达越稳定。因此,候选内参基因的表达稳定性由高到低依次排序为Actin>CyPA>EF1α>Ubi>18S>Tubu,即Actin和CyPA的表达相对稳定。
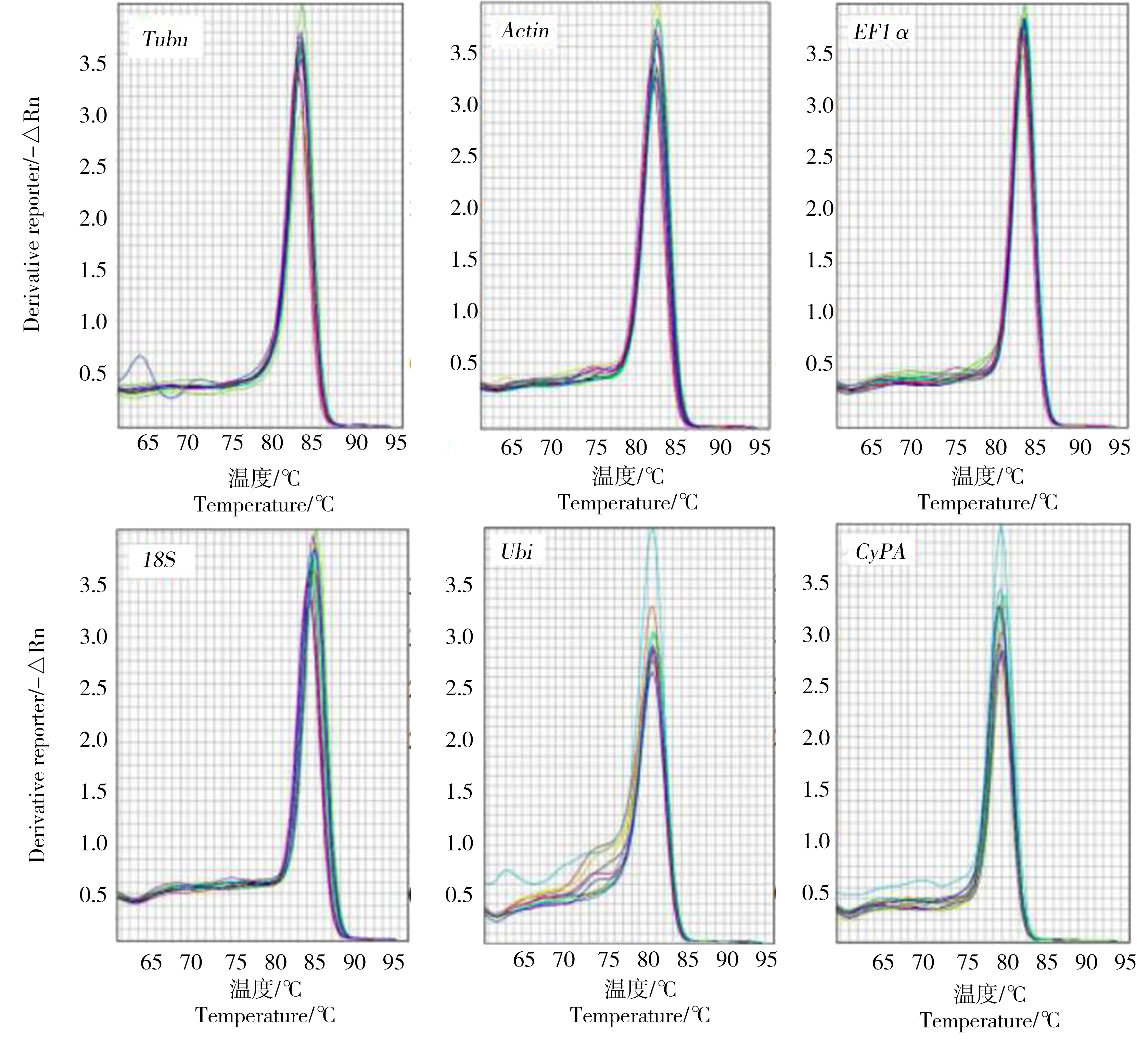
图2 菲律宾蛤仔6个候选内参基因扩增产物的熔解曲线Fig. 2 Melting curves of 6 candidate reference genes from R. philippinarum
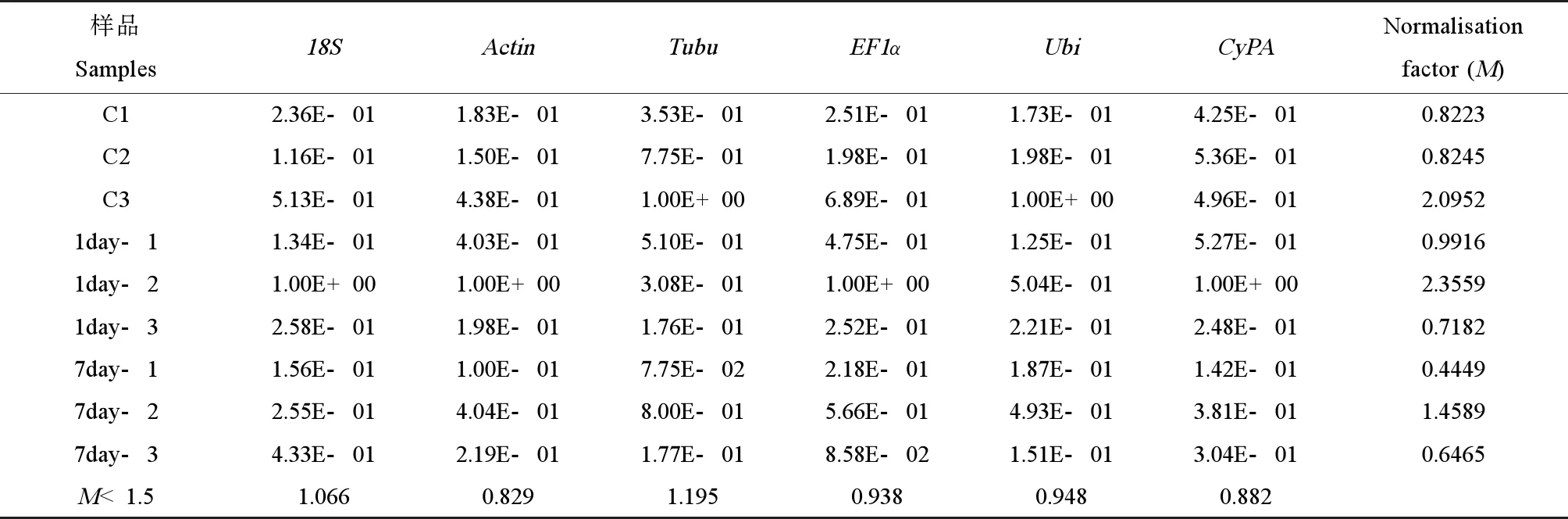
表1 geNorm评估6个候选内参基因的稳定性情况Table 1 Stability values of 6 candidate reference genes calculated by geNorm
注:C1、C2、C3表示对照组1、2、3号样品;1day-1、1day-2、1day-3表示亚硝态氮胁迫1天的样品1、2、3号;7day-1、7day-2、7day-3表示亚硝态氮胁迫7天的样品1、2、3号。
Note: C1, C2, C3 stand for control 1, control 2, control 3; 1day-1, 1day-2, 1day-3 stand for sample 1, sample 2, sample 3 exposed to nitrite for 1 day; 7day-1, 7day-2, 7day-3 stand for sample1, sample 2, sample 3 exposed to nitrite for 7 days.
为了校正系统偏差,geNorm软件一般选出2个以上的候选基因作为内参基因。内参基因数目的多少主要通过计算内参基因的配对差异值Vn/Vn+1来确定,其配对差异值以0.15为默认取舍值。当配对差异值小于0.15时,候选内参基因中适合作qRT-PCR内参基因的数目确定为n个[13]。本研究中配对差异值均大于0.15(图3)。
2.3.2 NormFinder软件评估
NormFinder软件通过程序运行生成基因表达的稳定值,稳定值越低表明内参基因表达越稳定[14]。由软件分析结果可知(表2),基因的稳定性由高到低依次为Actin>Ubi>CyPA>EF1α>18S>Tubu,即Actin的表达稳定性最好。
2.3.3 内参基因表达稳定性综合分析
根据软件geNorm和NormFinder的分析结果,采用加权赋值法[12],对每个基因的表达稳定性分别赋值后再求和,依据分数越小稳定性越好的原则,对结果重新排序,分数最小者排在第1位,其他依次排名为2、3、4、5、6位。统计结果显示(表3),得分最低的是Actin(2分),排名第1,说明6个候选内参基因中Actin的表达稳定性最高,其他依次为CyPA、Ubi、EF1α、18S、Tubu。由此说明,菲律宾蛤仔受到亚硝态氮胁迫后,鳃组织中Actin基因的表达比较稳定,适合作为内参基因对其他基因的表达水平进行标准化分析。
3 讨论(Discussion)
使用qRT-PCR技术分析目标基因的表达趋势时,往往把内参基因作为数据标准化的对照进行校正[15]。在贝类生物中,开展了很多筛选相关内参基因的工作,为进一步研究贝类分子毒理学提供了参考依据。Bai等[16]筛选出了三角帆蚌(Hyriopsiscumingii)在生物矿化过程中的内参基因Ubi、Rpl18和EF1α;不同组织和发育时期的合浦珠母贝(Pinctadafucata)[17]、不同发育阶段和雌激素暴露后的栉孔扇贝(Chlamysfarrer)[18]、以及多氯联苯胁迫下红树蚬(Polymesodaerosa)[19]体内最稳定的内参基因均为Actin。鲍相渤等[20]分别探讨了饥饿、致病菌感染和环境水温胁迫条件下虾夷扇贝(Patinopectenyessoensis)成体的鳃、肾、闭壳肌组织、外套膜、血淋巴和肝胰腺等不同组织的适宜内参基因。本研究通过qRT-PCR比较了菲律宾蛤仔在亚硝态氮胁迫下鳃组织的6个候选内参基因的表达稳定性,确定了Actin为其最适内参基因。
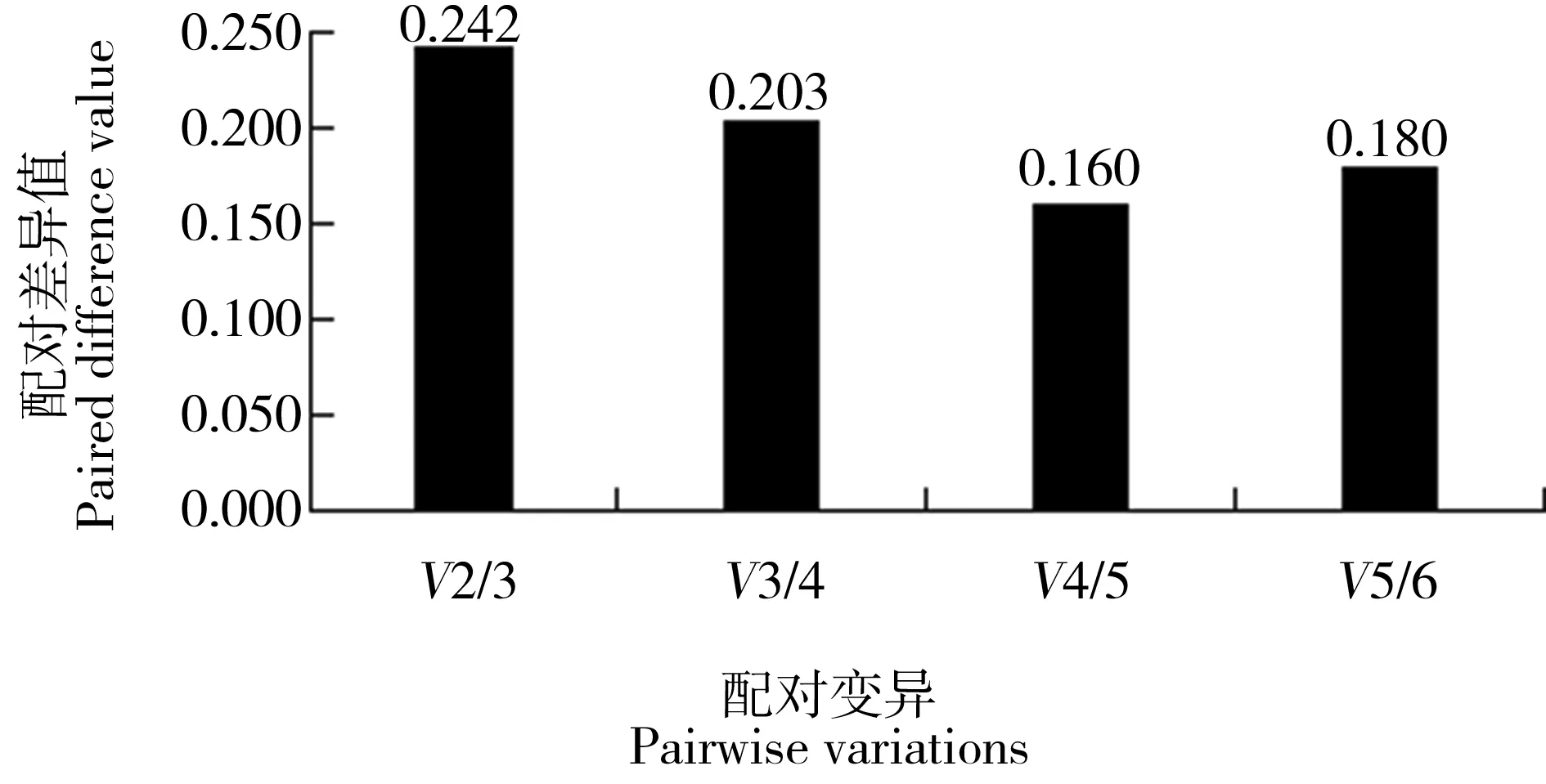
图3 geNorm评估6个内参基因的稳定性Fig. 3 Stability values of 6 reference genes calculated by geNorm

表2 NormFinder评估6个内参基因的稳定性Table 2 Stability values of 6 reference genes calculated by NormFinder
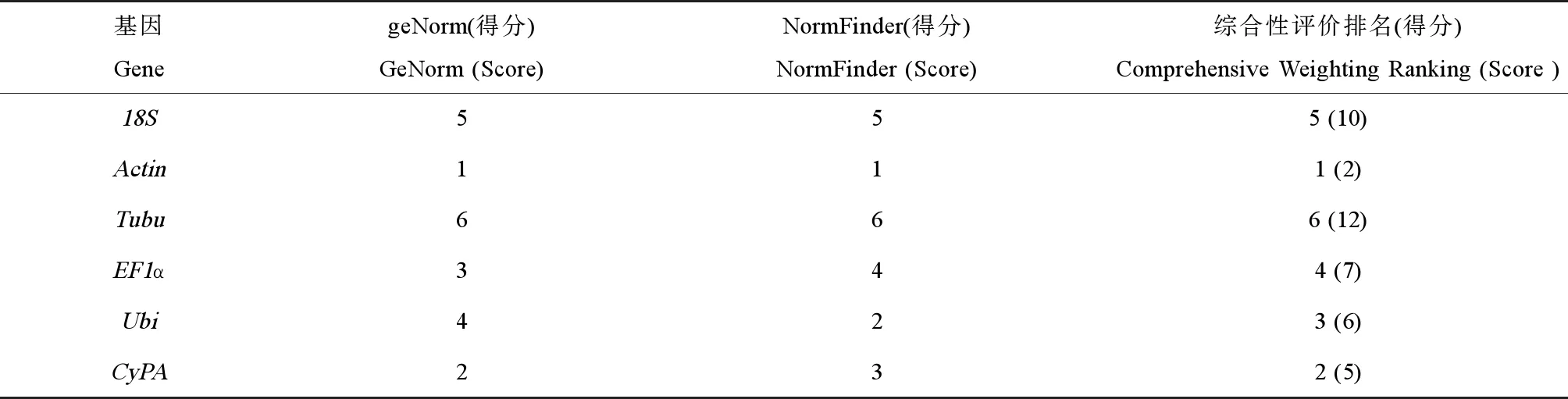
表3 内参基因稳定性的综合分析Table 3 Comprehensive weighting comparison of reference genes’ stability
有大量研究认为,同一管家基因在不同细胞类型和不同生理状态下的表达并不是恒定不变的[19,21-22],但Actin基因的表达量在超过90%研究条件下都处于稳定状态,适合单独作为qRT-PCR的内参基因[23]。本研究所测试的6个表达量相对稳定的基因中,Actin基因同样是表达水平最为稳定的一个。曹滕飞等[10]确定了氨氮胁迫后菲律宾蛤仔鳃组织中最适宜的内参基因为EF1α。由此可见,利用qRT-PCR研究基因表达的稳定性时,即便是同一物种的同一组织处于不同胁迫条件下,内参基因的选择也会不同[19],需要具体情况具体分析。
目前,用于评价内参基因表达稳定性的常用软件有geNorm、NormFinder、BestKeeper等,但是采用不同软件分别分析得到的排序结果之间往往存在差异,导致排序不一致,而综合多个软件的分析结果相互验证则可提高研究的准确性[19,24-25]。geNorm是Vandesompele等[26]2002年开发的一款用于微软Excell平台的VBA宏程序,将计算平均表达稳定值M作为基因稳定性的评价指标,据M值分析得出配对差异值(Vn/Vn+1),其配对差异值一般以0.15为默认取舍值,若Vn/Vn+1<0.15,则最适内参基因的数量为n个;若Vn/Vn+1>0.15,则最适内参基因的数量为(n+1)个。NormFinder是Andersen等[14]2004年研发的筛选稳定性内参基因的另一款程序,其程序计算原理和geNorm相似,也是先获得内参基因的表达稳定值M,再根据M值的大小来筛选最适合的内参基因,M值越小越稳定。前者可用于筛选任何样品的任何数量的内参基因,通过程序分析后确定适合的内参基因和最适宜的内参基因数目;后者则不仅能比较内参基因的表达差异,还可以计算样品组间的差异,但只能筛选出一个最适合的内参基因[27]。考虑到两者程序计算原理的相似性,本实验采用了geNorm和NormFinder两个筛选软件的评价结果相互验证,但两个软件所获得的基因表达稳定性顺序出现了不一致,故利用Su等[12]的加权赋值法,综合比较了geNorm和NormFinder的评价结果,从而获得了这6个内参基因表达稳定性,表达较为稳定的基因为Actin和CyPA,Actin为最稳定的内参基因。
使用geNorm软件分析内参基因稳定性时,可根据Vn/Vn+1(<0.15时)来确定内参基因的适宜数目,但geNorm使用指南中也明确地指出0.15并非严格标准值。多项实验研究中也出现了差异分析值大于0.15的情况,鲍相渤等[20]认为不同组织间的Vn/Vn+1大于0.15,是不同组织的细胞类型和比例不同以及实验中的候选基因有限所致。Li等[28]认为,在人类卵巢肿瘤的内参基因选择中,Vn/Vn+1大于0.15是由内参基因的表达受组织类型及患者年龄、肿瘤的所处阶段等生理因素和病理因素的影响不同而造成的。Penning等[29]比较猫不同组织中的10个候选内参基因的表达稳定性时发现,需同时选用6个内参基因才能保证对所有组织进行最佳标准化,而此时的Vn/Vn+1亦大于0.15。蒋婷婷等[30]对石蒜属(Lycoris)植物内参基因的筛选、曹腾飞等[10]在筛选氨氮胁迫下菲律宾蛤仔的内参基因时均遇到了Vn/Vn+1大于0.15的情况,但结果并未拘泥于Vn/Vn+1≤0.15。本实验中差异分析值Vn/Vn+1大于取舍值0.15,可能是在亚硝态氮胁迫后的不同时间段取材所导致的。
