ROCK1基因敲除对乳腺癌细胞系迁移及侵袭的影响
2019-06-03王晓杰王子宇赵晓婷岳文涛
王晓杰,滕 宇,顾 勐,王子宇,赵晓婷,岳文涛*
(1.首都医科大学附属北京胸科医院/北京市结核病胸部肿瘤研究所细胞生物学研究室,北京 101149;2.首都医科大学附属北京妇产医院中心实验室,北京 100026)
乳腺癌(breast cancer)是全球最常见的女性肿瘤[1],是女性癌症死亡的主要原因,约11%的乳腺癌发生在中国,而且近几十年来发病率及死亡率迅速上升[2],其发生发展机制涉及诸多肿瘤相关基因的异常表达。事实上,在临床表现之前,肿瘤细胞转移性播散的最主要事件是细胞迁移,这也是肿瘤细胞转移过程的核心[3]。大多数乳腺癌患者死于多器官转移的晚期,是临床治疗失败的主要原因。Rho相关蛋白激酶(Rho-associated protein kinase,ROCK)大约在20年前首次被发现在细胞迁移中起作用[4],它参与了几乎所有的迁移模式[5-6]。ROCK通过增强肿瘤细胞的侵袭性和运动性而显著促进转移[6-7]。因此Rho/ROCK通路通过调节肌动蛋白细胞骨架重组参与肿瘤发展进程。已有研究[8-9]报道,特定的ROCK抑制剂可以抑制肿瘤的生长和转移。ROCK1是小型G蛋白GTPase RhoA的下游效应物,通过肌球蛋白轻链(myosin light chain,MLC)的磷酸化调节细胞的运动,从而促进应力纤维的形成,增强细胞的运动和迁移能力[10]。由此可见,ROCK1可能在肿瘤细胞迁移、侵袭中发挥作用。但在各种肿瘤中的作用和相关机制还有待明确。
本研究中,我们通过成簇规律间隔的短回文重复序列(clustered regularly interspaced short palindromic repeats/Cas9,CRISPR/Cas9)[11]技术在MCF-7乳腺癌细胞系中构建稳定的ROCK1基因敲除细胞模型,为后续鉴定ROCK1直接调控的靶基因,研究其参与乳腺癌细胞侵袭转移的分子机制奠定基础。
1 材料与方法
1.1 主要试剂及仪器
Oligo引物(Generay Biotech),2×Taq Plus Master Mix(Vazyme P212-03),BsmB I(赛默飞),T4 DNA 连接酶(Fermentas EL0016),琼脂糖凝胶DNA回收试剂盒和无内毒素质粒小提中量试剂盒均购自TIANGEN公司,胰蛋白胨和琼脂粉均购自OXOID公司,嘌呤霉素和琼脂糖购自Sigma公司,DTT二硫苏糖醇(Sangon D0281-5g),胎牛血清和RPMI-1640培养基购自Gibco,引物、TOP10感受态、ROCK基因敲除慢病毒、Polybrene感染增强剂均购自Genechem公司,人乳腺癌细胞系MCF-7源自医学科学院基础所细胞资源中心,ROCK1兔源单抗和HRP标记的GAPDH单抗购自Abcam公司。
主要仪器包括:电泳仪(Tanon EPS-600),Alpha凝胶成像分析系统(FluorChemTMSP),摇床(华利达实验设备公司HI-9211K),GHP-9080隔水式恒温培养箱(上海一恒科学仪器有限公司),PCR仪(Applied Biosystems 2720 thermal),高速离心机(Thermo cycler 17),微型掌式离心机(GeneSci mini-6k)。
1.2 质粒构建
针对目的基因靶基因序列,设计向导RNA(small guide RNA,sgRNA)靶点序列,靶点序列分别为LVROCK1-sgRNA-1(ATCCGAATTCACTTCCGATT)、LVROCK1-sgRNA-2(CTTCATAGCATATACCTTCC)、LVROCK1-sgRNA-3(TTAGGATGGATTGGATGCTT)及对照病毒LV-NC的随机序列(CGCTTCCGCGGCCCGT TCAA)。载体两端BsmB I酶切位点序列如下:CACCGGAGACGCGTCTCT/GTTT,GTGGCCTCTGCG CAGAGACAAA/(斜线表示酶切产生的黏端,方向相同的斜线对应双链的互补碱基,下划线表示互补序列),设计原理为在sgRNA两端加入BsmB I位点切割后产生的互补黏端,对GV392载体进行酶切,使其线性化,反应体系:GV392质粒(1 μg/μL)2 μL,10×酶切缓冲液 2 μL,DTT(20 mmol/μL)1 μL,BsmB I酶 1 μL,ddH2O补至20 μL;反应条件:37℃酶切3 h。由引物合成公司合成单DNA oligo(PAGE纯化),线性化的载体DNA与退火的DNA双链进行连接,连入Lenti-CAS9-sgRNA-tag载体(Puromycin耐药标签)。反应体系及条件如下:线性化的载体DNA(50 ng/μL)2 μL,退火的双链DNA 0.5 μL,10×T4缓冲液1 μL,pEG4000 1 μL,T4 DNA 连接酶1 μL,ddH2O补足至10 μL;16℃反应3 h。将连接好的产物冰浴30 min后转化TOP10感受态细胞,吸取适量菌液均匀涂布在Amp抗性的LB固体培养基上,37℃恒温箱中倒置培养过夜,次日进行菌落PCR鉴定。菌落PCR得到阳性克隆后测序,从而得到序列正确的表达sgRNA的过表达慢病毒质粒。
1.3 重组质粒的鉴定
在超净工作台中,以无菌吸头挑取单个菌落至微型管中进行PCR鉴定,反应体系为:上游引物(10 μmol/L)0.5 μL,下游引物(10 μmol/L)0.5 μL,2×Taq Plus Master Mix 10 μL,模板菌落,ddH2O补足至20 μL。扩增循环反应条件为94℃、30 s,55℃、30 s,72℃、90 s,共22个循环。上游引物序列采用质粒共有序列:5'-CCATGATTCCTTCATA TTTGC-3',即 Primer(+);下游引物序列采用sgRNA的oligo序列(5'-CGCGTGGATAACCGTATTAC-3')为反义链,即Primer(-),扩增产物经1%琼脂糖凝胶电泳鉴定阳性克隆,将阳性菌落接种于200 μL含氨苄抗性的LB培养基中,37℃振荡培养2 h,取适量菌液进行测序,测序结果与设计的靶点序列进行比对分析。
1.4 慢病毒感染及细胞模型的筛选
人乳腺癌细胞系MCF-7培养于含10%胎牛血清的DMEM培养基中。感染前一天,消化MCF-7细胞,调整细胞浓度至3×104个/mL,每孔200 μL接种至96孔板。次日根据预实验确定合适的病毒复数(multiplicity of infection,MOI)值,用含有感染增强剂聚凝胺的细胞培养基稀释病毒,每孔加入100 μL病毒稀释液感染上述乳腺癌细胞,感染8 h后更换常规培养基,培养72 h后,换成含嘌呤霉素(3 μg/mL)的培养基继续培养48 h,对病毒感染细胞进行筛选,最终获得慢病毒感染且稳定敲除ROCK1基因的目的乳腺癌细胞株。
1.5 Western blot检测
将细胞株扩大培养,并分别收集稳定敲除ROCK1基因的乳腺癌细胞株、MCF-7亲本细胞以及阴性对照组细胞(LV-NC),加入适量混匀的蛋白裂解液。蛋白裂解液的配制比例为每100万细胞内加入100 μL RIPA裂解液、1 μL cooktail、1 μL胆碱脂酶抑制剂PMSF以及二硫苏糖醇(DL-dithiothreitol,DTT,稀释1 000倍使用),充分震荡,12 000 r/min 4℃离心15 min,取上清液经BCA蛋白定量检测法测定样品浓度,按照与上样蛋白体积1∶4的比例加入含β-巯基乙醇的5×上样缓冲液,混匀后进行聚丙烯酰胺凝胶电泳(SDS-PAGE)。电泳结束,经恒流电泳转移、5%脱脂牛奶封闭、兔源一抗孵育、辣根过氧化物酶标记的二抗孵育、0.1%TBST洗膜、ECL化学发光等步骤,在Alpha凝胶成像分析系统(FluorChemTMSP)中曝光图像并分析结果。
1.6 划痕实验
将处于对数生长期的稳定敲除ROCK1基因的乳腺癌细胞株及对照组细胞株用胰酶消化后计数,接种于6孔板,接种数量以次日铺满6孔板一层为宜。接种24 h后,在6孔板中划痕,生理盐水冲洗3次,更换含有0.2%牛血清白蛋白(albumin from bovine serum,BSA)的DMEM培养基,分别于划痕后0、12、24 h观察细胞的迁移情况并拍照记录,实验重复3次。
1.7 Transwell实验
不铺胶:将处于对数期的稳定敲除ROCK1基因的乳腺癌细胞株及对照组细胞株接种于25 cm2的细胞培养瓶,24 h后换成含有0.2%BSA的DMEM培养基培养过夜,另将小室置于24孔板中,上室中加入100 μL含有0.2%BSA的DMEM培养基,下室加入含10%胎牛血清的DMEM培养基平衡过夜。次日消化,计数细胞,调整细胞浓度为5×105个/mL,取100 μL加入上室。24 h后用2%龙胆紫将小室染色30 min,洗净,将小室的膜取下,封片拍照。
铺胶:将小室置于24孔板中,上室中加入50 μL含有0.2%BSA的DMEM培养基经1∶5稀释的基质胶,37℃温箱过夜。次日,在上室中加入100 μL含有0.2%BSA的DMEM培养基,下室加入含10%胎牛血清的DMEM培养基,水化30 min后加入100 μL细胞稀释液(5×105个/mL),48 h后用2%龙胆紫将小室染色30 min,洗净,将小室的膜取下,封片拍照,其余步骤同上。
1.8 统计学分析
每项实验均至少独立重复3次;应用SPSS 16.0统计学软件对数据进行分析,图像分析所得相对灰度值以xˉ±s表示,两样本均数之间采用配对t检验进行比较。以α=0.05为检验水准。
2 结果
2.1 ROCK1质粒的构建与鉴定
针对目的基因靶基因序列,设计sgRNA靶点序列,单链DNA oligo两端含酶切位点黏端,经退火处理形成双链DNA,连入Lenti-CAS9-sgRNA-tag载体(GV392、Puromycin药物筛选标记,图1A)。将连接好的产物使用TOP10感受态转化,菌落PCR得到阳性克隆后测序,从而得到序列正确、表达sgRNA的过表达慢病毒质粒,然后进行慢病毒的包装。LV-ROCK1-sgRNA-1(图 1B)、LV-ROCK1-sgRNA-2(图 1C)和 LVROCK1-sgRNA-3(图1D)的质粒测序结果正确。
2.2 稳定敲除乳腺癌细胞株中ROCK1的鉴定
为检验ROCK1基因敲除后,细胞株中ROCK1蛋白的表达情况,对LV-ROCK1-sgRNA-1/2/3和阴性对照(LV-NC)及未经感染的MCF-7细胞进行Western blot检测(图2)。在被LV-NC感染的对照组细胞及亲本细胞MCF-7中检测到特异性ROCK1蛋白条带,分子量约165 kD,在LV-ROCK1-sgRNA-1、LV-ROCK1-sgRNA-2感染的MCF-7细胞的对应位置可见到明显减弱的蛋白条带,而在LV-ROCK1-sgRNA-3感染的MCF-7细胞对应位置未见到明显蛋白条带,表明ROCK1敲除乳腺癌细胞模型构建成功。后续研究使用LV-ROCK1-sgRNA-3敲除ROCK1基因的细胞模型作为实验组;LV-NC感染的细胞作为对照组。
2.3 ROCK1敲除对MCF-7细胞迁移和侵袭能力的影响
划痕实验结果见图3。ROCK1敲除细胞株24 h划痕愈合度为(60.600±0.047)%,对照组细胞24 h划痕愈合度为(80.404±0.018)%,提示ROCK1敲除细胞株在24 h内划痕愈合面积较对照组明显降低,差异有统计学意义(P=0.003)。
图1 ROCK1质粒的构建与鉴定

图2 MCF-7细胞中敲除ROCK1的表达情况
Transwell结果见图4。结果显示在不铺胶情况下,实验组ROCK1敲除细胞株在24 h的迁移细胞数为(448.3±5.5)个,对照组的迁移细胞数为(271.3±5.0)个,提示ROCK1敲除细胞株在24 h内穿过小室底膜的细胞数与对照组相比明显降低,差异有统计学意义(P=0.001)。提示ROCK1敲除可明显抑制乳腺癌细胞MCF-7的迁移能力。

图3 划痕实验结果及分析
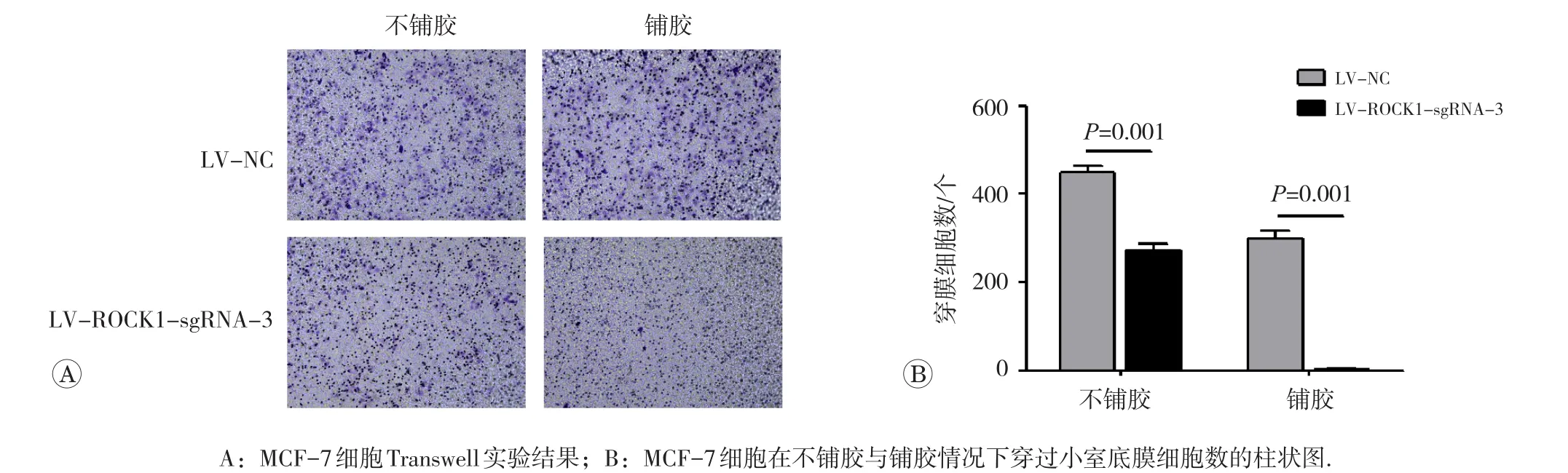
图4 Transwell实验结果及统计分析
在铺胶情况下,实验组中ROCK1敲除细胞株48 h内的迁移细胞数为(1.7±2.9)个,对照组的迁移细胞数为(298.3±5.7)个,提示ROCK1敲除细胞株在48 h时内穿过基质胶的细胞数较对照组明显降低,差异有统计学意义(P=0.001)。说明ROCK1敲除可明显抑制乳腺癌MCF-7细胞的侵袭能力。
3 讨论
乳腺癌患者的死亡率呈逐年上升的态势,肿瘤转移是大多数癌症患者死亡的主要原因[12]。肿瘤细胞的侵袭与转移是肿瘤转移过程的核心环节。小GTPase Rho家族通过调节细胞形态、生长和运动的功能在肿瘤的侵袭与转移过程中起着关键作用。ROCK1作为小型GTPase Rho家族主要的下游效应物,是激活Rho的主要介体,也是促进乳腺癌进展过程的重要因素[13-17]。Bottino等[14]用免疫组化的方法对乳腺癌肿瘤碎片中的ROCK1进行染色,结果发现有淋巴结转移的肿瘤组织中ROCK1蛋白含量远高于无淋巴结转移者,ROCK1蛋白的表达量与临床分期及有无远处转移也密切相关。除乳腺癌外,Vigil等[15]的研究表明,ROCK1作为非小细胞肺癌侵袭及转移的重要因素,可以作为治疗非小细胞肺癌的重要靶点。Sahai等[16]研究表明Rho GTPases调节细胞骨架和细胞迁移,在肿瘤中过表达,ROCK1也是其他癌症[17]细胞运动/侵袭的关键因素。
本研究利用CRISPR/Cas9技术,应用乳腺癌细胞株MCF-7构建ROCK1敲除细胞模型,通过DNA测序和Western blot证实ROCK1基因敲除细胞模型构建成功,进一步通过细胞表型实验研究ROCK1敲除细胞模型对乳腺癌细胞侵袭和迁移能力的影响。在用Western blot验证细胞模型是否构建成功的结果中(图2),被LV-ROCK1-sgRNA-1或LV-ROCK1-sgRNA-2感染的MCF-7细胞仍存在减弱的蛋白条带,这表示插入的干扰序列未能完全抑制ROCK1蛋白的表达。在LV-ROCK1-sgRNA-3感染的MCF-7细胞的对应位置未见到明显蛋白条带,表明ROCK1敲除乳腺癌细胞模型构建成功。在划痕实验以及Transwell实验中,与LV-NC对照组相比,被LV-ROCK1-sgRNA-3感染的细胞在侵袭及转移方面的能力显著降低。
综上所述,区别于其他应用抑制剂抑制ROCK1作用的研究实验,本研究用Cas9的方法敲除了ROCK1基因,探究ROCK1基因的敲除对乳腺癌细胞侵袭转移能力的影响。通过Transwell实验和划痕实验,本研究发现相较于对照组,ROCK1基因敲除的细胞株迁移和侵袭能力明显减弱,ROCK1基因的失活突变可以抑制乳腺癌细胞的侵袭和迁移能力,实验结果与其他研究的观点相符,说明ROCK1在乳腺癌的侵袭和转移中发挥重要作用,为后续鉴定ROCK1直接调控的靶基因,研究其参与乳腺癌细胞侵袭转移的分子机制建立了稳定的细胞模型。
