双重RT-RPA法快速检测沙门氏菌及其1类整合酶基因
2019-04-18俞圣韬叶仲杜
程 辉,梁 奕,俞圣韬,叶仲杜,蒋 晗,朱 诚,*
(1.浙江省海洋食品品质及危害物控制技术重点实验室,浙江 杭州 310018; 2.中国计量大学 生命科学学院,浙江 杭州 310018)
沙门氏菌(Salmonella)是肠杆菌科的一类革兰氏阴性菌,它是全球最重要的食源性致病菌之一,且被世界卫生组织 (World Health Organization, WHO)列为具有中等乃至严重危害的食源性病原菌[1]。据文献报道,越来越多的沙门氏菌在致病的同时,具有多重耐药性[2-4],其携带的耐药基因可以通过水平基因转移(horizontal gene transmission, HGT)等方式迁移扩散[5],严重危害人类健康,其中整合酶基因(integrase gene,intI)尤其是1类整合酶基因(intI1)在此扩散中发挥关键作用[6]。因此,建立一种快速、准确、高效且可同时检测沙门氏菌及其耐药相关整合酶基因的方法,对于食品安全保障和细菌耐药性防控具有重要意义。
针对沙门氏菌和intI1基因,研究人员已开展了常规PCR、荧光定量PCR、环介导等温扩增法(loop-mediated isothermal amplification, LAMP)等研究[7-10],其中PCR方法虽灵敏度高,但检测效率较低;LAMP方法虽用时短且摆脱了对仪器的依赖,但假阳性高,极易污染[11]。重组酶聚合酶扩增技术(recombinase polymerase amplification,RPA)是一种在常温下可以使核酸快速扩增的方法。在25~42 ℃等温条件下,重组酶uvsX与引物DNA紧密结合,形成酶和引物的聚合体,当引物在模板DNA上搜索到与之完全互补的序列时,在单链DNA结合蛋白的作用下,使模板DNA解链,并在链置换DNA聚合酶Bsu的作用下,形成新的DNA互补链,被替换的DNA链与SSB结合,防止进一步替换。在这个体系中,由两条引物起始一个合成事件,整个过程进行得非常快,一般在30 min内即能获得可检出水平的扩增产物[12-16]。本文所采用的荧光定量重组酶聚合酶扩增(duplex real-time RPA, RT-RPA)方法,可在常温下高速扩增沙门氏菌的特异基因fimY和1类耐药相关整合酶基因intI1,其关键技术在于exo探针的设计,该方法特异性强、灵敏度高、速度快(检测时间20 min),在实际样品检测试验中准确率高。这对沙门氏菌及其耐药性相关整合子基因的快速检测、早期预警、切断传播途径从而保障人类安全健康具有重要意义。
1 材料与方法
1.1 试剂与菌株
肠炎沙门氏菌SalmonellaenteritidisGIMCC1.345、金黄色葡萄球菌StaphylococcusaureusGIMCC1.142、阪崎肠杆菌CronobacterSakazakiiGIMCC1.296、志贺氏菌Shigellaspp. GIMCC1.424均购自广东省微生物菌种保藏中心。β溶血性链球菌StreptococcushemolyticusATCC21059、副溶血性弧菌VibrioparahaemolyticusATCC17802、单增李斯特菌ListeriamonocytogenesATCC19115均购自美国典型培养物保藏中心。实际样品检测试验中,2株携带intI1基因的沙门氏菌,555株携带intI1基因的大肠埃希菌从生猪养殖场、屠宰场、超市和农贸市场筛选得到[17]。所有微生物实验均在Ⅱ级生物安全实验室完成。培养基均购自杭州微生物试剂有限公司。分子相关试剂均购自北京百泰克生物技术有限公司。其他化学试剂均购自Sigma。
1.2 仪器与设备
美国Bio-Rad CFX 96荧光定量PCR仪、美国Thermal离心机、美国Bio-Rad PCR仪、美国Bio-Rad电泳仪、美国Bio-Rad凝胶成像分析系统等。
1.3 引物与探针
根据大肠埃希菌uidA基因(GenBank登录号:S69414),沙门氏菌fimY基因(GenBank登录号:JQ665438)、intI1基因(GenBank登录号:HM569736),设计PCR引物、荧光定量PCR引物、RPA引物和exo探针(表1),exo探针中间标记荧光基团和荧光淬灭基团,插入四氢呋喃(tetrahydrofuran,THF)基团(图1)。
1.4 细菌培养与基因组提取
在-80 ℃ 冰箱中取出实验室冻存的沙门氏菌菌种进行复苏培养,随后划线接种于亚硫酸铋培养基,于37 ℃过夜培养。挑取单菌落于亚硫酸铋培养基中,于37 ℃,200 r·min-1培养至菌液D600值为1.0。其余菌株在各自最适条件下培养。需检测的革兰氏阴性菌菌液采用煮沸法制备菌液模板DNA。取1 μL待测菌液加入30 μL Tris缓冲液,煮沸5 min,冰上冷却2 min,12 000×g离心2 min,取上清液用作DNA模板。革兰氏阳性菌模板DNA采用试剂盒法,操作按试剂盒操作说明进行。
表1引物和探针序列
Table1Sequences of primers and exo probes

图1 RPA exo探针设计图Fig.1 Exo probe of RPA
1.5 浓度梯度样品稀释
取D600为1.0的肠炎沙门氏菌培养液、实验室筛选到的携带intI1的大肠埃希菌培养液各1 mL,按照10倍梯度进行稀释并进行平板活菌计数,每组浓度3个平行,取平均值计算原液中的细菌浓度。将经活菌计数的肠炎沙门氏菌培养液、携带intI1基因的大肠埃希菌培养液,分别进行10倍梯度稀释,分别提取DNA,获得肠炎沙门氏菌、携带intI1基因的大肠埃希菌的系列浓度梯度标准品(108~100CFU·mL-1)。
1.6 RPA方法的建立与优化
50 μL RPA反应体系包含2× RE缓冲液25 μL,250 mmol·L-1醋酸镁溶液2.5 μL,2 μL的primerF和primerR,1 μL的exo探针和2 μL的DNA模板和2 μL酶工作液。反应程序为37 ℃ 反应20 min (30 s一个循环,每个循环收集1次荧光),反应过程借助荧光定量PCR仪实时监测。为获得较优的结果,对双重RT-RPA的引物和探针浓度、反应温度进行了优化,反应温度从30 ℃到41 ℃进行梯度优化,引物和探针浓度按表2所示浓度进行优化。
1.7 特异性实验
使用双蒸水作为阴性对照,分别使用沙门氏菌、副溶血性弧菌、志贺氏菌、金黄色葡萄球菌、β溶血性链球菌、单增李斯特菌、阪崎肠杆菌、携带intI1基因的大肠埃希菌、大肠埃希菌DH5α、大肠埃希菌BL21、携带intI1基因的沙门氏菌等11种菌种基因组DNA作为模板,反应体系采用前期实验优化后的RPA体系,进行RT-RPA特异性验证。
1.8 灵敏度实验
以1.5节制备的标准品为模板,在最佳条件下进行RT-RPA反应,以检验反应灵敏度,并与常规RT-PCR灵敏度进行比较分析。
1.9 双重RT-RPA方法在实际菌株鉴定中的应用
参照GB 4789.38—2012《食品微生物学检验 大肠埃希氏菌计数》、GB 4789.4—2010《食品微生物学检验沙门氏菌检验》中的方法,从养猪场、屠宰场、农贸市场和超市环境样品与猪肉样品分离耐药性大肠埃希菌和沙门氏菌,并参照Zhu等[18]的方法对intI1基因进行鉴定。对比选择性平板培养、FDA推荐的微生物分析法[19]、PCR扩增和RT-RPA扩增等方法进行菌株结果鉴定。
2 结果与分析
2.1 RPA检测方法的建立与双重RT-RPA的优化
分别以阳性沙门氏菌基因组DNA和携带intI1基因的耐药大肠埃希菌为模板,筛选RT-RPA引物探针组合、优化单重RT-RPA的反应温度、探针浓度,建立最优的单重RT-RPA反应体系。结果显示:沙门氏菌RT-RPA引物探针组合1优于组合2,intI1引物探针组合1优于组合2(图2-A),选择引物探针组合1进行后续的RT-RPA实验。
设置30~41 ℃的温度梯度 (30.0 ℃、30.7 ℃、32.1 ℃、34.3 ℃、36.9 ℃、39.1 ℃、40.3 ℃、41 ℃),优化RT-RPA反应温度,结果显示,反应温度为36.9 ℃时,两组RT-RPA反应均有较好的扩增(图2-B和图2-C)。优化exo探针浓度优化试验结果显示,fimY探针浓度为60 nmol·L-1时,检测fimY效率较高,intI1探针浓度为100 nmol·L-1时,检测intI1效率较高。在单重RT-RPA优化条件的基础上,进行双重RT-RPA引物和探针浓度优化,结果显示,当fimY引物终浓度为320 nmol·L-1,intI1引物400 nmol·L-1,fimY探针终浓度为60 nmol·L-1,intI1探针终浓度为100 nmol·L-1时,fimY和intI1同步检测效果最优(表2)。
2.2 特异性实验
按照1.7节特异性实验的方法进行检测,如图3-A所示,经过双重RT-RPA检测,在添加有沙门氏菌、携带intI1基因的沙门氏菌、携带intI1基因的大肠埃希菌样品中均出现明显的扩增曲线,且携带intI1基因的沙门氏菌出现2条扩增曲线,显示为阳性。添加其他干扰菌的样品均无明显扩增,结果为阴性。实验结果表明:本研究建立的多重荧光定量检测方法对沙门氏菌及其耐药相关intI1基因有较好的特异性,与其他相关细菌无交叉反应。
2.3 灵敏度实验
按照1.8节灵敏度实验的方法进行检测,对浓度为1.29×105、1.29×104、1.29×103、1.29×102、1.29×101、1.29×100CFU·mL-1的沙门氏菌进行RT-RPA和荧光定量PCR检测,对浓度为1.60×105、1.60×104、1.60×103、1.60×102、1.60×101、1.60×100CFU·mL-1的intI1基因进行RT-RPA和荧光定量PCR检测。结果表明,RT-RPA方法对沙门氏菌检测下限为1.29×101CFU·mL-1,对intI1基因检测下限为1.60×101CFU·mL-1(图3-B和图3-C),与常规RT-PCR的检测灵敏度一致(图3-D和图3-E)。
2.4 实验样品的检测
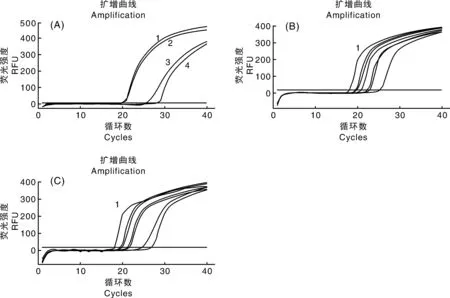
A: 1,fimY引物探针组合1;2,intI1引物探针组合1;3,fimY引物探针组合2;4,intI1引物探针组合2。B、C: 1,温度36.9 ℃。A: 1, The first combination of fimY primer and probe; 2, The first combination of intI1 primer and probe; 3, The second combination of fimY primer and probe; 4, The second combination of intI1 primer and probe. B and C: 1, The temperature is 36.9 ℃.图2 RT-RPA反应条件优化Fig.2 Optimization of reaction for RT-RPA
表2双重RT-RPA引物与探针浓度优化
Table2Optimization of primers and probes concentration for duplex RT-RPA
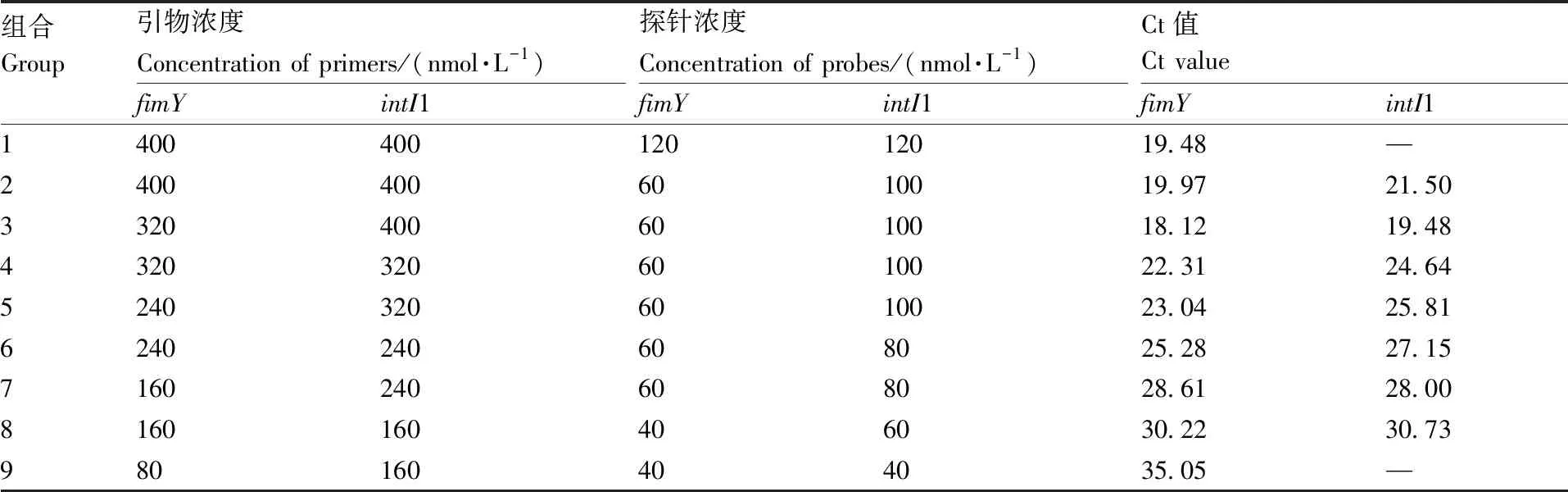
组合Group引物浓度Concentration of primers/(nmol·L-1)fimYintI1探针浓度Concentration of probes/(nmol·L-1)fimYintI1Ct值Ct valuefimYintI1140040012012019.48—24004006010019.9721.5033204006010018.1219.4843203206010022.3124.6452403206010023.0425.816240240608025.2827.157160240608028.6128.008160160406030.2230.73980160404035.05—
如表3所示,双重RT-RPA方法对在猪肉生产链中筛选到的沙门氏菌、大肠埃希菌及其携带intI1基因的检测结果与FDA推荐的微生物分析法[18]、PCR法检测结果一致,但双重RT-RPA方法的检出时间较微生物分析法、PCR法有明显缩短。微生物分析法需过夜培养,PCR法鉴定需要2 h以上,RT-RPA可以在20 min完成检测。此外,RT-RPA结果直接通过扩增曲线判定,不需要开盖电泳,故与PCR产物电泳鉴定方法相比,降低了扩增产物对环境的污染,这对降低因环境污染导致的假阳性率大有裨益。
3 结论
本研究以RPA方法为技术基础,建立了一种能单管同时快速鉴定沙门氏菌及其耐药相关intI1基因的双重RT-RPA方法。该方法以沙门氏菌特异毒力基因fimY和细菌耐药相关1类整合酶基因intI1为靶标序列,建立了基于exo探针的双重RT-RPA方法,当fimY引物终浓度320 nmol·L-1,intI1引物终浓度400 nmol·L-1,fimY探针终浓度60 nmol·L-1,intI1探针终浓度100 nmol·L-1,反应温度37 ℃,反应20 min 时,双重RT-RPA扩增效率最高,且特异性好,灵敏度高。实际应用中,该方法检测准确率高。
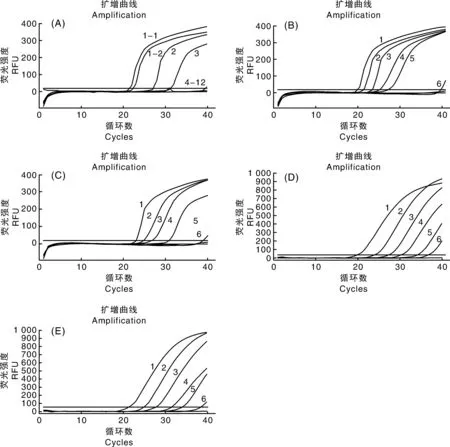
A: 1-1,携带intI1基因的沙门氏菌fimY基因扩增曲线;1-2,携带intI1基因的沙门氏菌intI1基因扩增曲线;2,沙门氏菌;3,携带intI1基因的大肠埃希菌;4-11,副溶血弧菌、志贺氏菌、金黄色葡萄球菌、β溶血性链球菌、单增李斯特菌、阪崎肠杆菌、大肠埃希菌DH5α、大肠埃希菌BL21;12,阴性对照。B: 1,1.29×105 CFU·mL-1;2,1.29×104 CFU·mL-1;3,1.29×103 CFU·mL-1;4,1.29×102 CFU·mL-1;5,1.29×101 CFU·mL-1;6,1.29×100 v·mL-1。 C: 1,1.60×105 CFU·mL-1;2,1.60×104 CFU·mL-1;3,1.60×103 CFU·mL-1;4,1.60×102 CFU·mL-1;5,1.60×101 CFU·mL-1;6,1.60×100 CFU·mL-1。D: fimY基因常规RT-PCR灵敏度试验,1,1.29×105 CFU·mL-1;2,1.29×104 CFU·mL-1;3,1.29×103 CFU·mL-1;4,1.29×102 CFU·mL-1;5,1.29×101 CFU·mL-1;6,1.29×100 CFU·mL-1。E: intI1基因常规RT-PCR灵敏度试验,1,1.29×105 CFU·mL-1;2,1.29×104 CFU·mL-1;3,1.29×103 CFU·mL-1;4,1.29×102 CFU·mL-1;5,1.29×101 CFU·mL-1;6,1.29×100 CFU·mL-1。A: 1, Amplification of fimY(intI1-positive salmonella); 1-2, Amplification of intI1 (intI1-positive salmonella); 2, Salmonella; 3, IntI1-positive E.coli; 4-11, Vibrio parahaemolyticus, Shigella, Staphylococcus aureus, β-hemolytic streptococcus, Listeria monocytogenes, Enterobacter sakazakii, E. coli DH5α, E. coli BL21; 12, Negative sample. B: 1, 1.29×105 CFU·mL-1; 2, 1.29×104 CFU·mL-1; 3, 1.29×103 CFU·mL-1; 4, 1.29×102 CFU·mL-1; 5, 1.29×101 CFU·mL-1; 6, 1.29×100 CFU·mL-1. C: 1, 1.60×105 CFU·mL-1; 2, 1.60×104 CFU·mL-1; 3, 1.60×103 CFU·mL-1; 4, 1.60×102 CFU·mL-1; 5, 1.60×101 CFU·mL-1; 6, 1.60×100 CFU·mL-1. D: Sensitivity of RT-PCR for fimY, 1, 1.29×105 CFU·mL-1; 2, 1.29×104 CFU·mL-1; 3, 1.29×103 CFU·mL-1; 4, 1.29×102 CFU·mL-1; 5, 1.29×101 CFU·mL-1; 6, 1.29×100 CFU·mL-1. E: Sensitivity of RT-PCR for intI1, 1, 1.29×105 CFU·mL-1; 2, 1.29×104 CFU·mL-1; 3, 1.29×103 CFU·mL-1; 4, 1.29×102 CFU·mL-1; 5, 1.29×101 CFU·mL-1; 6, 1.29×100 CFU·mL-1.图3 RT-RPA特异性与常规RT-PCR灵敏度Fig.3 Specificity and sensitivity of RT-RPA and RT-PCR
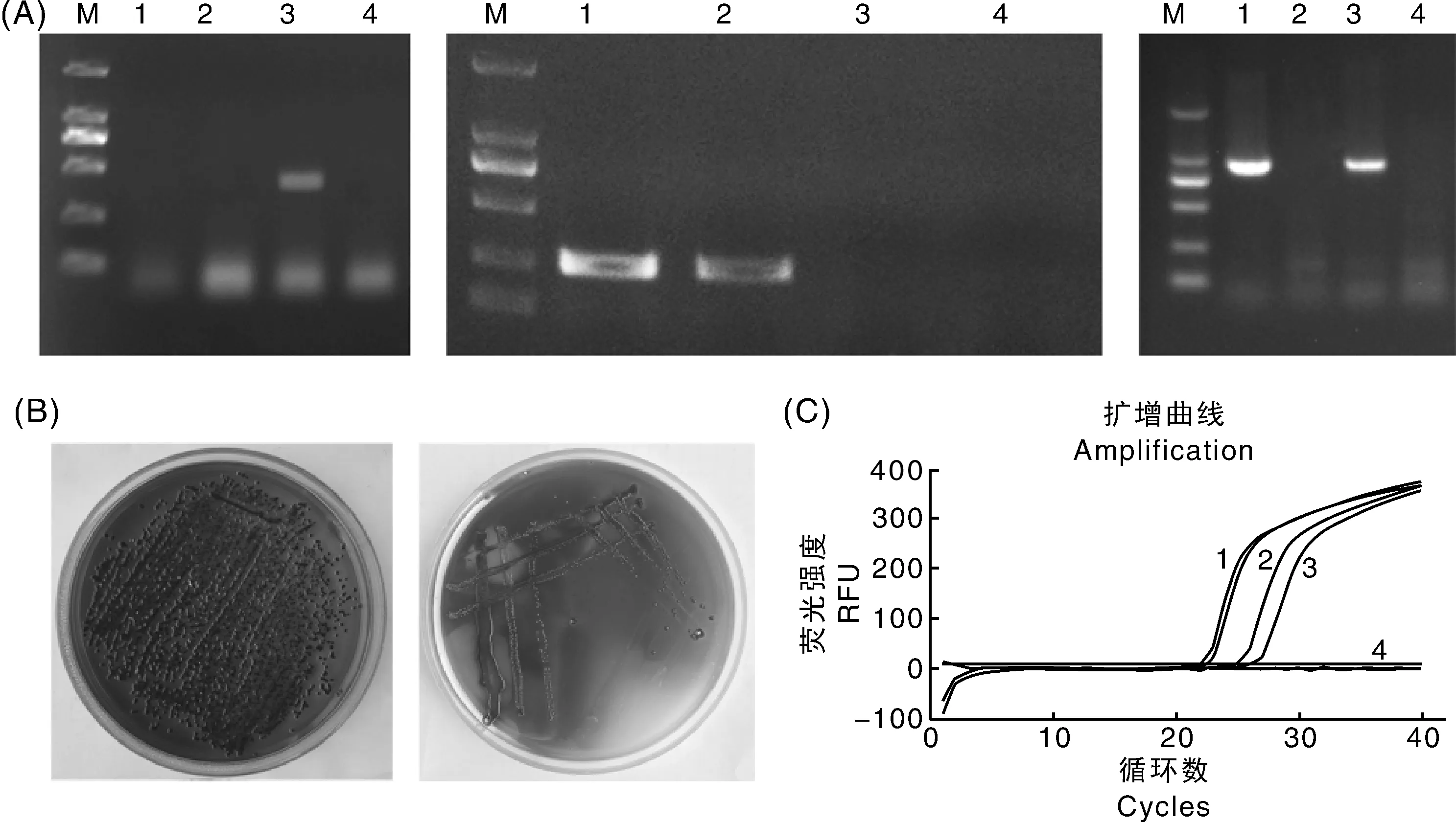
A: uidA基因、fimY基因、intI1基因PCR电泳结果,M为DL 2 000 marker;1为携带有intI1基因的沙门氏菌;2为不携带intI1基因的沙门氏菌;3为携带有intI1基因的大肠埃希菌;4为阴性对照。B:大肠埃希菌和沙门氏菌选择性培养结果,大肠埃希菌在伊红美蓝选择性平板上为绿色金属光泽菌落,沙门氏菌在亚硫酸铋选择性平板上为灰色金属光泽菌落。C: RT-RPA鉴定结果,1为携带有intI1基因的沙门氏菌;2为不携带intI1基因的沙门氏菌;3为携带有intI1基因的大肠埃希菌;4为阴性对照。A: uidA, fimY and intI1 of PCR, M represented DL 2 000 marker; Lane 1 represented intI1-positive Salmonella; Lane 2 represented intI1-negative Salmonella; Lane 3 represented intI1-positive E.coli; Lane 4 represented negative control. B: Selective plate for E. coli and Salmonella, E. coli was green metallic luster colony on the EMB plate, and Salmonella was gray metallic luster colony on the BS plate. C: Lane 1 represented intI1-positive Salmonella; Lane 2 represented intI1-negative Salmonella; Lane 3 represented intI1-positive E.coli; Lane 4 represented negative control.图4 不同方法在菌株和intI1基因鉴定中的应用Fig.4 Application of different methods in identification of strains and intI1 gene
表3RT-RPA方法在菌株及intI1基因鉴定中的应用
Table3Application of RT-RPA method in identification of strains andintI1 gene

目标菌株Target isolates鉴定结果Identification results/strain选择性培养法Selective culturePCRRT-RPA细菌培养法Bacterium culture大肠埃希菌Escherichia coli14601038—1038沙门氏菌Salmonella168616161携带intI1大肠埃希菌intI1-positive Escherichia coli—555555—携带intI1沙门氏菌intI1-positive Salmonella—22—
本研究将RT-RPA方法应用到耐药性相关1类整合酶基因的检测,并实现了对沙门氏菌和1类整合酶基因的双重同步检测。该方法检测时间只需20 min,较传统的方法大大缩短。该方法的建立对沙门氏菌及其耐药性相关整合子基因的快速检测、早期预警、切断传播途径进而保障人们安全健康具有重要意义。也可为食品中其他致病微生物及其耐药性的快速检测研究提供借鉴,使食品中致病微生物及其耐药性检测向高通量、高灵敏、简便经济的方向发展。
