创新生物科技对猪营养和猪肉生产效率改进的推动作用
2019-04-08伍国耀
■伍国耀
(美国德克萨斯州农工大学动物科学系,大学城 77843)
2.2.3 基因(基因组)编辑生产基因敲除或敲入的转基因动物
最初用于生产转基因家畜的方法会导致随机转基因的插入,因此需要新技术来提高基因靶向效率。通过原核注射将DNA递送到受精卵中的方法在概念上是简单的,但该方法在技术上具有挑战性,并且注射的DNA构建体会随机整合到基因组中,产生不可预测的转基因表达谱。此外,显微注射会损伤受精卵,并且还需要昂贵的设备。这些缺点能够通过基因(基因组)编辑方法的发展部分性克服,该方法使用序列特异核酸酶(作为一对分子剪刀)在所需的基因组位点产生DNA双链断裂(图5)。随后,两种内源性修复机制之一可以修复DNA双链断裂:非同源末端连接(non-homologous end joining,NHEJ)和同源定向修复(homology-directed re⁃pair,HDR)。在容易出错的非同源末端连接途径中,DNA双链断裂的两个末端汇聚并连接在一起,然而没有同源模板用于修复,其经常插入或删除核苷酸(插入缺失)以引起基因破坏。如果插入缺失标记导致移码突变,则靶基因可能失去功能(敲除)。同源定向修复途径需要提供外源DNA模板以及位点特异性基因组编辑核酸酶以修复DNA双链断裂,从而导致所需的DNA序列插入胚胎或动物细胞的基因组中。在实践中,靶基因的修饰通常通过显微注射来实现,体外受精或胞质内转移获得的胚胎,通过基因编辑系统(由内源核酸酶的DNA序列、指导RNA和DNA模板组成)修饰。
早期的序列特异核酸酶是锌指蛋白核酸酶(ZFN,第一个基因编辑工具),随后发现的序列特异核酸酶是转录激活因子样效应核酸酶(TALEN,第二个基因编辑工具),两者都含有适应性强的DNA结合域的模块化蛋白质。ZFN方法涉及产生一种包含能与DNA结合的锌指结构域和有限制性内切核酸酶活性的结构域的蛋白质。TALEN方法利用含有能与DNA结合的结构域和单独的能切割DNA的结构域的工程酶。近年来,成簇规律间隔的短回文重复-核酸酶-9(CRISPR/Cas9)被用作核酸酶,为基因组工程提供更高效、更准确、更通用、更强大、更简单的设计工具(Tan等,2016;Niu等,2017)。ZFN、TALEN或CRISPR/Cas9组分通过转染(基于脂质的试剂,电穿孔,核转染或显微注射)或噬菌体递送到靶细胞中,这取决于细胞类型和质粒(Bamford等2014;Bikard等,2014;Kim等,2010)。
在过去的5年中,CRISPR/Cas9作为家畜物种的首选基因编辑工具迅速获得了应用。CRISPR/Cas9系统于2007年在细菌(如革兰氏阳性球菌或球形细菌属)和古细菌中被发现,被用于抵御入侵的病毒(噬菌体)。细菌受病毒感染后,CRISPR/Cas9将被短RNA片段(被称为指导RNA)引导剪切一段病毒DNA,在其靶基因座中产生双链断裂。指导RNA与靶生物的基因组区段互补,因此Cas9核酸酶能够高度特异性地切割DNA。值得注意的是,Cas9对靶DNA的识别取决于直接位于非靶向DNA链下游的前间隔序列邻近基序(PAM)的存在(Ryu等,2018)。因此,作为核糖核蛋白的CRISPR系统由两种组分组成,Cas9内切核酸酶和指导RNA。在实验上,可以使用实验室中的分子生物学工具设计指导RNA,将Cas9导向几乎任何基因组位点的DNA,将其进行切割。表2中列出了使用CRIS⁃PR/Cas9生产基因编辑猪的里程碑。
2.2.3.1 优势
传统的畜牧业受到诸如繁殖周期长和遗传资源限制等问题的困扰。相比之下,基因组编辑工具通过提供更精确、更具体、更可预测和更快速的解决方案,能够以相对可承受的成本来解决这些问题(Ryu等,2018)。CRISPR除敲除基因功能外,还能够从动物基因组中删除大的DNA片段。此外,基因编辑技术与先前的动物转基因方法相比,步骤少并且更效率。例如,Tan等(2016)报道,他们在对家畜受精卵的基因编辑中能够通过ZFN、TALEN和CRISPR/Cas9技术实现30%的编辑频率。与其它基因沉默技术(如RNAi)相比,CRISPR/Cas9具有更高的效率、更易于设计和更大的灵活性。据报道,几个实验室已经成功地生产了基因编辑猪,这些猪可以用作器官供体、疾病模型、生物反应器、猪内源性逆转录病毒的灭活,或生产具有提高生产力(如肌肉生长)或抗病性状的转基因动物(Fischer等,2018;Tan等,2016)。因此,基因编辑已经成功应用到家畜基因组的单基因和多等位基因的修饰,以及在胚胎发育过程中外源基因的位点特异性的引入。
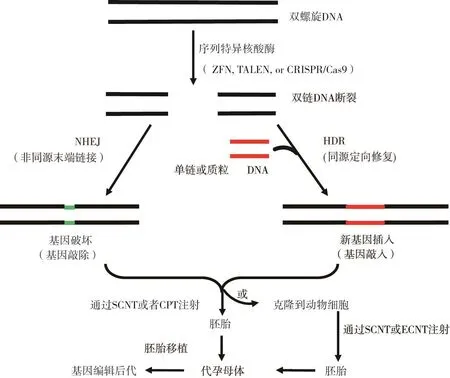
图5 使用ZFN、TALEN或CRISPR/Cas9技术对动物进行基因(基因组)编辑
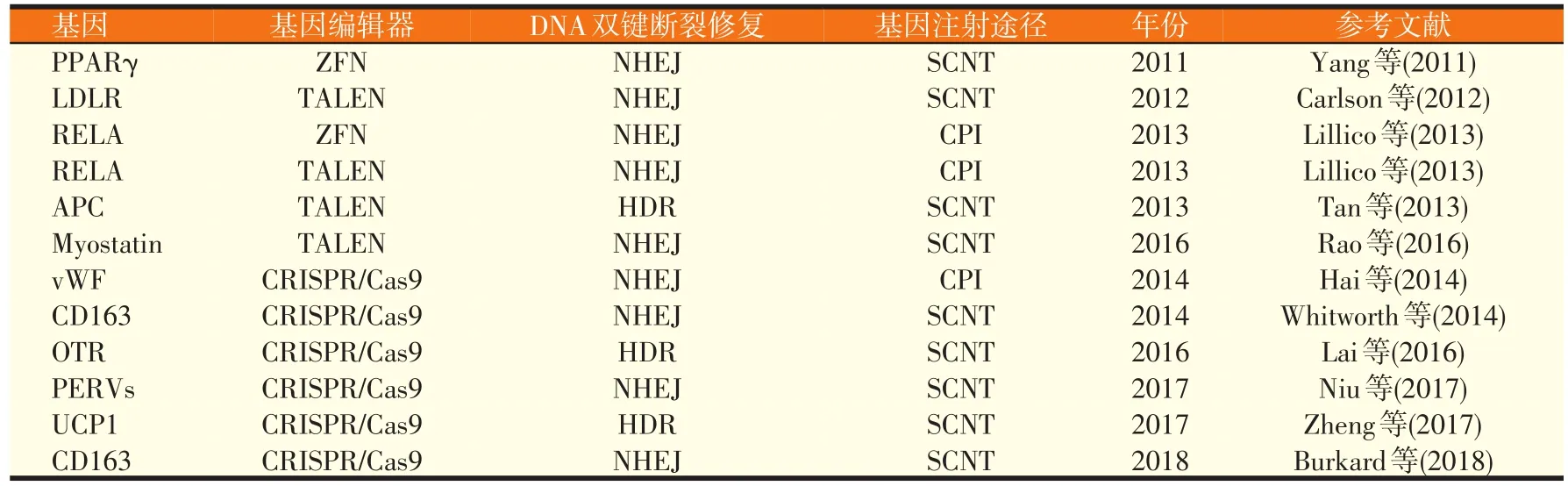
表2 使用CRISPR/Cas9生产基因编辑猪的里程碑
表3中总结了具有重要生产性状的转基因猪的实例。首先,使用TALEN作为基因编辑器破坏肌肉生长抑制素(MSTN,肌细胞生成的负调节因子),成功地创造了MSTN敲除猪,其表现出双肌肉表型、更高的体重和背最长肌肌肉质量以及多于野生型猪100%的肌纤维数量(Rao等,2016)。第二,Zheng等(2018)利用CRISPER/Cas9技术生产出具有功能性解偶联蛋白1(UCP1)的猪。UCP1在许多动物物种的棕色脂肪组织中表达,发挥非寒战产热的功能,从而在防寒和调节能量稳态中起关键作用。然而,现代猪缺乏功能性UCP1,因此很容易受到冷应激,导致新生儿死亡率高,并且在体内自发积累大量白色脂肪组织,导致生产性能下降(Ji等,2017)。值得注意的是,通过CRIS⁃PR/Cas9作为编辑器将小鼠脂联素-UCP1基因插入到猪内源性UCP1基因座中生产UCP1-敲入猪,其表现出体温维持能力增加、白色脂肪沉积减少和组织中胴体瘦肉增加(Zheng等,2017)。第三,CRISPR/Cas9基因靶向和SCNT技术已被用于产生没有CD163基因的猪,此基因编码猪生殖和呼吸综合征病毒(PRRSV,也称为“蓝耳病”)的细胞受体。例如,Burkard等(2018)报道,具有CD163敲除的猪对PRRSV具有完全抗性。Yang等(2011)也观察到类似的结果。雄性和雌性个体可以用作种畜生产PRRSV抗性的后代。
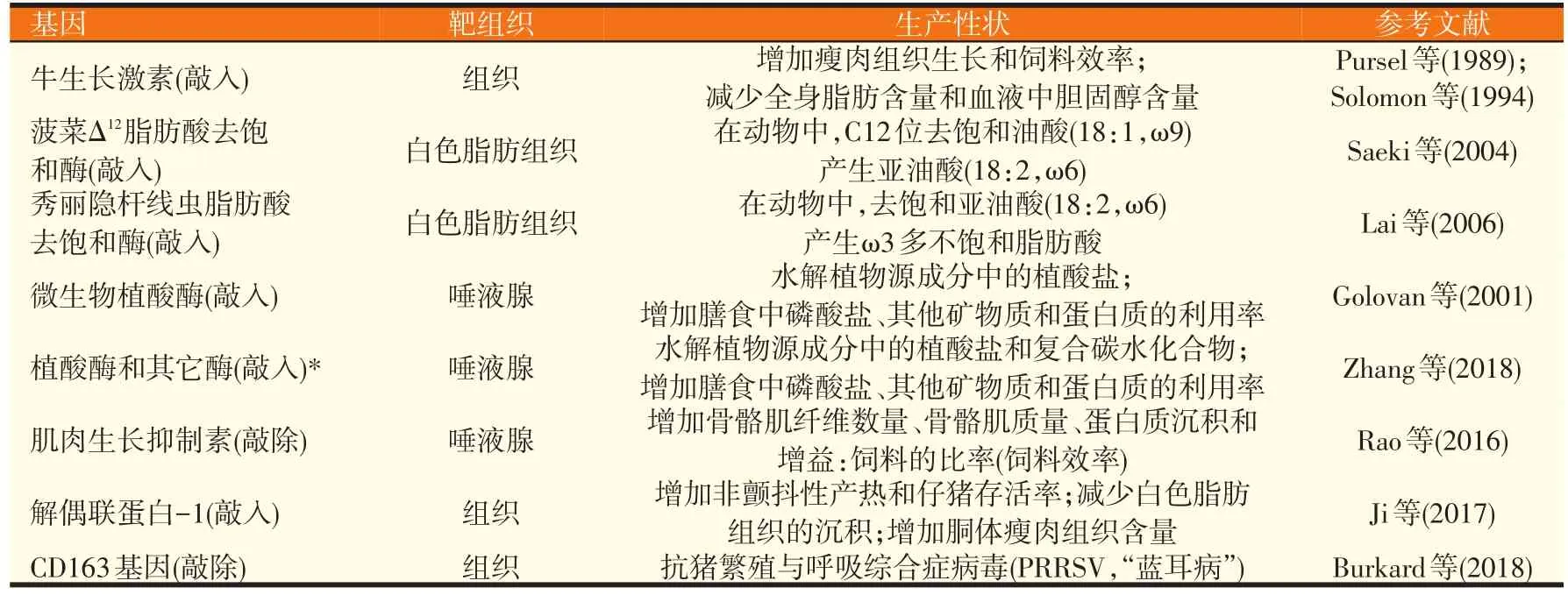
表3 通过秀丽隐杆线虫生产具有重要生产性状的转基因猪
2.2.3.2 缺点
尽管ZFN方法在位点特异性基因编辑方面实现了首次突破,但它仍有一些局限性,例如DNA的脱靶切割、细胞毒性、昂贵、耗时和效率低,因此一次只能进行一次基因组编辑,并且在准备有效的ZFN工具上也面临着技术挑战(Ryu等,2018)。与ZFN编辑器相比,TALEN技术在基因工程中更加灵活,因为其DNA结合域可以靶向更广泛的DNA序列。虽然TALEN比ZFN更容易设计,但TALEN方法价格昂贵且在技术上难以同时对基因组进行多次编辑。除此之外,通过显微注射将基因编辑Cas9直接递送至胚胎仍然是一个具有挑战性的过程,并且显微注射本身可能损害胚胎。目前来说,通过现用的基因编辑方法所产生胎儿的产前死亡率远高于常规胎儿。迄今为止,包括猪在内的家畜基因编辑效率仍然不理想。基因编辑的程序应该更容易和更廉价,这样更多的生产者才能够在他们自己的农场利用这种创新技术来改善动物繁殖。
3 通过生物技术理解动物和人类的抗生素耐药性
自从1928年青霉素被发现以来,抗生素被普遍用于治疗人类和动物的细菌感染。19世纪60年代以来,人们开始在传统日粮内添加亚治疗剂量的抗生素用以提高猪和禽类的生产性能。然而,由于耐药菌的产生和传播,许多国家已经禁止饲用抗生素的使用(如:欧盟各国);并且一些主要的猪肉输出大国,如美国和中国等国家已经在逐步淘汰饲用抗生素。一些耐药菌仅耐受一类抗生素,但有些耐药菌可耐受多种抗生素,因此导致了严重的全球健康问题(Koch等,2017)。为了确保抗生素在治疗人类和动物的细菌感染上发挥最好效果,全世界对于抗生素耐药性(antimicrobial resis⁃tance,AMR)的担忧日益加剧,抗生素耐药性被定义为细菌能耐受抗菌药物(如:抗生素)的能力。
微生物的抗生素耐药基因可能会通过分裂从母细胞遗传到子细胞,也可以通过质粒从一株细菌转移至另一株细菌。有趣的是,细菌体内的质粒(独立于染色体DNA的小DNA分子)通常会携带一些信息,这些信息可以通过耐受自身或环境中其他微生物产生的抗生素,进而利于自身生存(Kim等,2016)。2007年,人们在分析中国分离的黏杆菌素耐受大肠杆菌时,发现了一个带有19种抗生素耐药基因的质粒。在那以后,这令人烦恼的抗生素很长一段时间无人使用,这导致细菌的抗性水平降低;但当再次使用后,细菌抗性水平会再次升高(Dutil等,2010)。因此,迫切需要确定新的方法,来替代在全球猪生产中使用的饲用抗生素。通过生物技术来了解抗生素耐药性的发生原理,将极大促进这一进程。
大量证据表明,细菌自然会获得新的基因(包括抗生素耐药基因),以便在新宿主体内存活(Greene,2018)。抗生素耐药基因会产生多种酶(例如,大肠杆菌内的超广谱β-内酰胺酶)以破坏或灭活抗生素(图6)。例如,青霉素耐受细菌(如:金黄色葡萄球菌和大肠杆菌)通过合成的β-内酰胺酶,将青霉素的内酰胺环分解为一种非活性物质。通过这种机制,细菌能不被青霉素杀死,从而产生抗生素耐药性。现在,正在开发基于CRISPR技术的方法,用于杀死抗生素耐受细菌。因为CRISPR/Cas具有选择性地靶向特定DNA序列的能力,因此可以很容易地区分致病菌或共生菌种类(Choi等,2016;Kim等,2016)。特别值得注意的是,噬菌体(对动物和人类来说通常是安全的)已经被用来运送CRISPR/Cas系统至细菌体内(Greene,2018)。例如,没有自身DNA的噬菌体会得到一种编码的DNA,这种DNA可以编码一个向导RNA和Cas9(Bikard等,2014)。噬菌体然后被转入抗药性细菌(例如:艰难梭状芽孢杆菌),Cas9被向导RNA引导去切割在特定位点的细菌DNA,诱发细菌自毁(图7)。类似地,CRISPR/Cas3系统已经通过噬菌体运送到革兰氏阳性和阴性细菌,在多个位点切割DNA分子,从而诱导细胞程序性死亡(Reardon,2017)。此外,Kim等(2016)利用CRISPR/Cas9系统敲除了产生抗生素耐药性的基因,恢复多重耐药菌的敏感性,使其能够被抗生素杀死。最后,CRISPR/Cas9系统被构造成CRISPR干扰(CRISPRi)质粒载体,携带灭活的Cas9的DNA序列和向导RNA,已被用于消除膜结合毒性蛋白(如:凝血酶A和肠毒素C)和金黄色葡萄球菌(革兰氏阳性细菌,Sato’o等,2018)的抗生素耐药基因(如:β-内酰胺酶)。在这种方法中,灭活的Cas9中有两个域发生了突变,并且这种蛋白质只有DNA结合活性而不能分裂DNA。灭活的Cas9(dCas9)的结合,阻止细菌的转录机制调控目标基因,从而使其表达沉默,干扰细菌基因表达。因此,包含噬菌体或质粒在内的CRISPR/Cas9技术,有望杀灭和清除动物胃肠道中包括抗生素耐药细菌在内的细菌以及细菌产生的酶(表4)。该技术的实际应用是减轻抗菌素抗性并开发猪饲料中抗生素的替代物。这种基因工程方法,以及饲料发酵和利用饲料蛋白制备抗菌肽(Hou等,2017),有望最大限度地提高养分利用效率,并维持全球猪肉行业的发展。
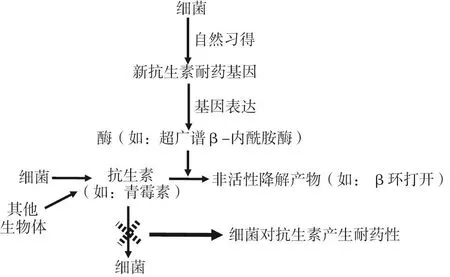
图6 细菌抗生素耐药性发展的机制
总而言之,对高品质肉类蛋白质的需求,促使全球猪肉业提高生产率,同时减少碳排放和废物排泄。为了实现这一目标,在过去的35年里,动物生物技术取得了革命性的进步,生产了重组蛋白质(包括酶)、有机营养(包括氨基酸和维生素)、克隆猪和用于生物医学和农业用途的转基因猪。近年来,基于锌指核酸酶(ZFN)、TALEN和CRISPR/Cas9的基因(基因组)编辑技术已经可以用于在DNA序列的特定位点上删除、插入,或修饰动物和细菌的基因组。与ZFN和TALEN基因编辑相比,CRISPR/Cas9的效率更高、脱靶效应更低、更易于设计,CRISPR/Cas9为基因工程学提供了更高效、更易于设计,并且更灵活的思路。因此,这种生物技术在保护猪的品种多样性、提高饲料效率和猪肉产量,以及在未来开发抗生素替代品方面均具有广阔前景。
致谢:
作者实验室的研究工作得到了美国德州农工大学AgriLife Research的支持(H-8200)。

图7 利用CRISPR系统作为新的抗生素替代品
