羊种布鲁氏菌OMP16蛋白结构的生物信息学分析
2019-02-19,,,,,,,
,,, , ,,,
布鲁氏菌常导致家畜感染,进而感染人类引起布鲁氏菌病,以羊种布鲁菌为主[1]。全球每年新发布病约50万例[2],布病临床症状不典型,慢性化率高,长期不愈并累多关节脏器,使患者反复遭受疾病折磨[3-4],给很多国家的人类健康和经济发展造成巨大的伤害[5-6]。研发针对布鲁氏菌的疫苗可以有效减少布病的发病,但目前的灭活疫苗和减毒活疫苗还存在效果不佳和不良反应多等不足[7]。OMP16蛋白是布鲁氏菌的一种外膜蛋白,具有很好的免疫原性,本研究利用生物信息学分析手段,对OMP16蛋白进行理化性质及结构进行分析,预测可能的B和T细胞的优势抗原表位,为布鲁氏菌诊断和疫苗研究的提供基础。
1 材料与方法
1.1 材 料
1.1.1氨基酸序列 从NCBI网站中的protein(https://www.ncbi.nlm.nih.gov/protein/)中获取B.melitensis的OMP16蛋白的氨基酸序列(GenBank: AAD33089.2),序列如下:Mgyfpswrltsnavvlpderlpaaaavpmgiqhllamsgstivapllmgfdpnvavffsgig-tllfflitagrvpsylgssfafiavviaatgysgsgpnpniaialggiiac-galysliglivmflgtgwveklmppavtgaigvsiglnlapvaigqlk-gtgahtsialltvlcmamisaygprgtrrlsvlfallfgyllvliagngl-givpgidftavknaawfglphfttpafnwqavtliapvaivlvaenlg-hikalgsitgrnmdgyigrgffsdglatmisgagggtgvttyaenig-vmamtkvystlifviaaimalilgmspkfgailqtipapvlaglavsv-fgliasamariwivnkvdfadsrnlftvgvalifgagdftlnignfalg-giatstlaalvlyqllgigrsds
1.1.2生物信息学分析软件 蛋白理化分析:ProtParam(http://web.expasy.org/protparam/);二级结构分析:DNAstar 7.2软件的Protein模块,SOMPA在线分析软件(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html);三级结构分析:Phyre2在线分析(http://www.sbg.bio.ic.ac.uk/phyre2/html); Rasmol 2.7.2.1;T和B表位分析:IEDB(http://tools.immuneepitope.org/mhci/)、(http://tools.immuneepitope.org/mhcii/)、(http://tools.immuneepitope.org/main/bcell/)、SYFPEITHI (http://www.syfpeithi.de/bin/mhcserver.dll/epitopeprediction.htm)、ABCpred Prediction(http://www.imtech.res.in/raghava/abcpred/)、BepiPred 1.0 Server(http://www.cbs.dtu.dk/services/BepiPred/)、DNAstar 7.2。
1.2 方 法
1.2.1利用ProtParam进行OMP16蛋白理化性质预测 分析蛋白的氨基酸组成、分子量、理论等电点、不稳定系数和疏水性等理化性质。
1.2.2利用DNAstar软件Protein模块和SOMPA分析OMP16蛋白质二级结构 采用Gramier-Robson和Chou-Fasman方法预测OMP16蛋白α螺旋,β折叠,β转角和无规卷曲的二级结构特点。
1.2.3利用Phyre2和Rasmol蛋白质三级结构分析 将OMP16氨基酸序列录入Phyre2在线服务器,分析蛋白氨基酸序列并组合模板,构建OMP16蛋白的三级结构并生成pdb数据文件,用Rasmol软件读取数据文件并对三级结构进行分析。
1.2.4B和T表位预测分析 利用IEDB、DNAStar、ABCpred和BepiPred预测OMP16蛋白的B细胞抗原表位;利用SYFPEITHI和IEDB进行OMP16蛋白的T抗原表位预测。MHC-I(The major histocompatibility complex class-I)分子选择HLA-A*1101,MHC-II分子选择HLA-DRB1*0701进行预测。通过比较分析最终OMP16蛋白的B细胞及T细胞优势抗原表位。
2 结 果
2.1理化性质 ProtParam显示OMP16蛋白有426个氨基酸序列,蛋白分子质量为43678.81 Daltons;理论等电点为9.72;原子组成为C2026H3209 N499O536S17;不稳定系数为26.57,归类为稳定蛋白;平均亲水系数GRAVY:0.981,归类为疏水性蛋白。
2.2二级结构 经DNAstar软件分析OMP16蛋白的二级结构结果见图1。OMP16蛋白的α-螺旋,β-折叠,β-转角和无规则卷曲的具体区域位置见表1。
SOPMA服务器在线分析OMP16二级结构检测显示:α-螺旋40.61%,β-折叠19.01%,β-转角8.45%,无规则卷曲31.92%。结构见图2。
2.3三级结构 为了直观的分析蛋白质的构象结构,利用Phyre2服务器在线分析OMP16蛋白的三级结构,分析发现构建的蛋白序列覆盖率为92%,可信度达到100.0%。建模后,通过RasMol软件分析预测OMP16蛋白的三级结构。结果显示OMP16蛋白结构成环状,结构稳定,有较多的β-折叠位于蛋白质的中部。上下部有很多易于形成构象抗原表位的结构。结果见图3。
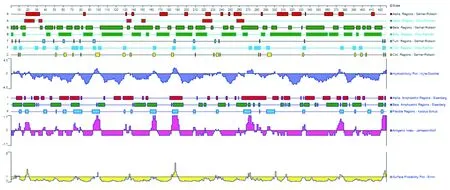
图1 DNAStar分析OMP16蛋白的二级结构结果Fig.1 Secondary structure prediction results for the OMP16 protein by DNAStar
表1 DNAStar分析OMP16蛋白的二级结构结果
Tab.1 Secondary structure prediction results for the OMP16 protein by DNAStar
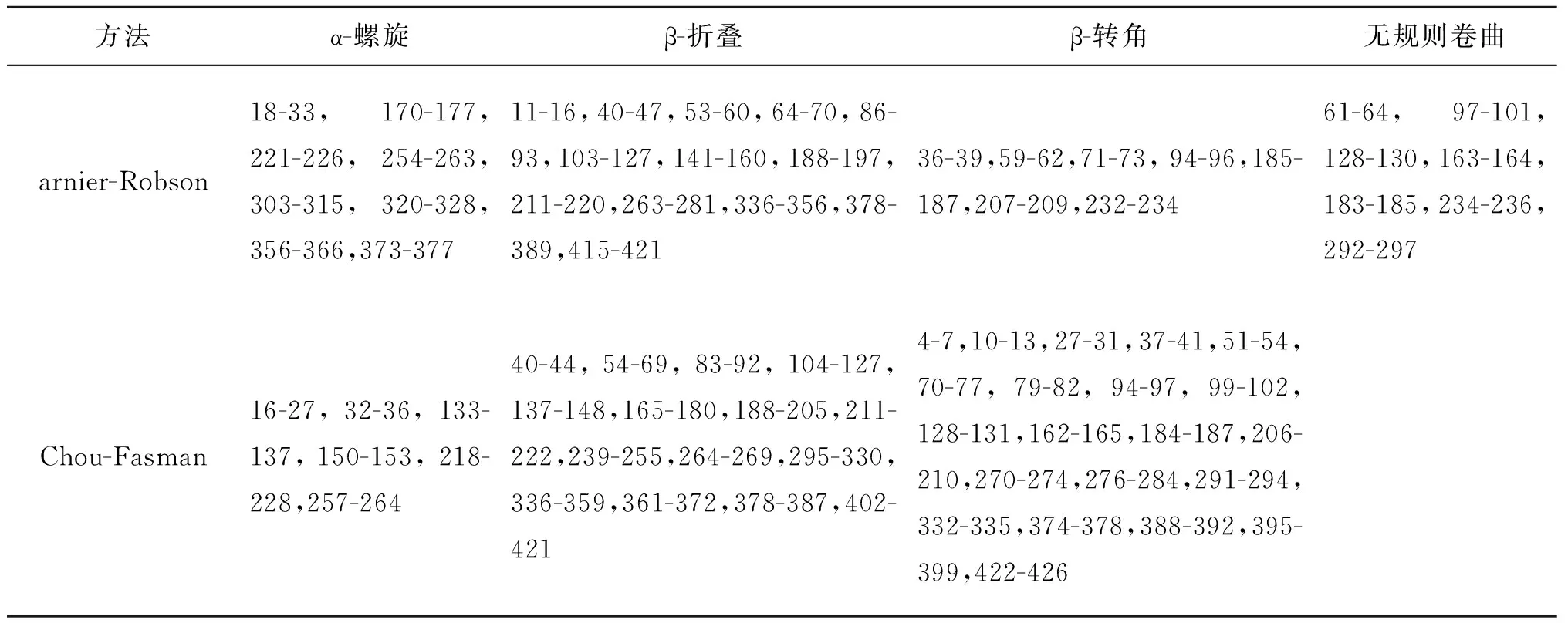
方法α-螺旋β-折叠β-转角无规则卷曲arnier-Robson18-33,170-177,221-226,254-263,303-315,320-328,356-366,373-37711-16,40-47,53-60,64-70,86-93,103-127,141-160,188-197,211-220,263-281,336-356,378-389,415-42136-39,59-62,71-73, 94-96,185-187,207-209,232-23461-64,97-101,128-130,163-164,183-185,234-236,292-297Chou-Fasman16-27,32-36,133-137,150-153,218-228,257-26440-44,54-69,83-92,104-127,137-148,165-180,188-205,211-222,239-255,264-269,295-330,336-359,361-372,378-387,402-4214-7,10-13,27-31,37-41,51-54,70-77,79-82,94-97,99-102,128-131,162-165,184-187,206-210,270-274,276-284,291-294,332-335,374-378,388-392,395-399,422-426
2.4 B和T细胞表位
2.4.1B细胞表位的预测 IEDB在线表位预测软件结果(图4)显示:线性表位由高到低分别是263-307,1-30,210-228,332-348,91-105,130-145(图4A)。β-转角由高到低分别是92-103,290-299,72-82,265-285,181-190,159-167,182-190(图4B)。表面可及性由高到低分别,182-191,16-22,297-303,92-102,267-273,309-314,369-379,3-11(图4C)。可塑性由高到低分别为92-102,290-305,183-191,158-168,37-42,372-379,16-21(图4D)。抗原性由高到低依次为243-256,191-206,410-419,151-158,83-91,339-358,314-327,106-126(图4E)。亲水性由高到低依次是91-103,284-306,265-277,159-168,181-191,369-380,72-80,8-23(图4F)。
利用BepiPred 1.0b Server和ABCpred Prediction Server预测分析B细胞表位,寻找共同预测的表位,结果见表2。然后结合IEDB和DNAStar显示的OMP16外模蛋白结构的线性表位,β-转角,亲水性,可塑性和表面可及性的特点,最终确定B细胞表位。
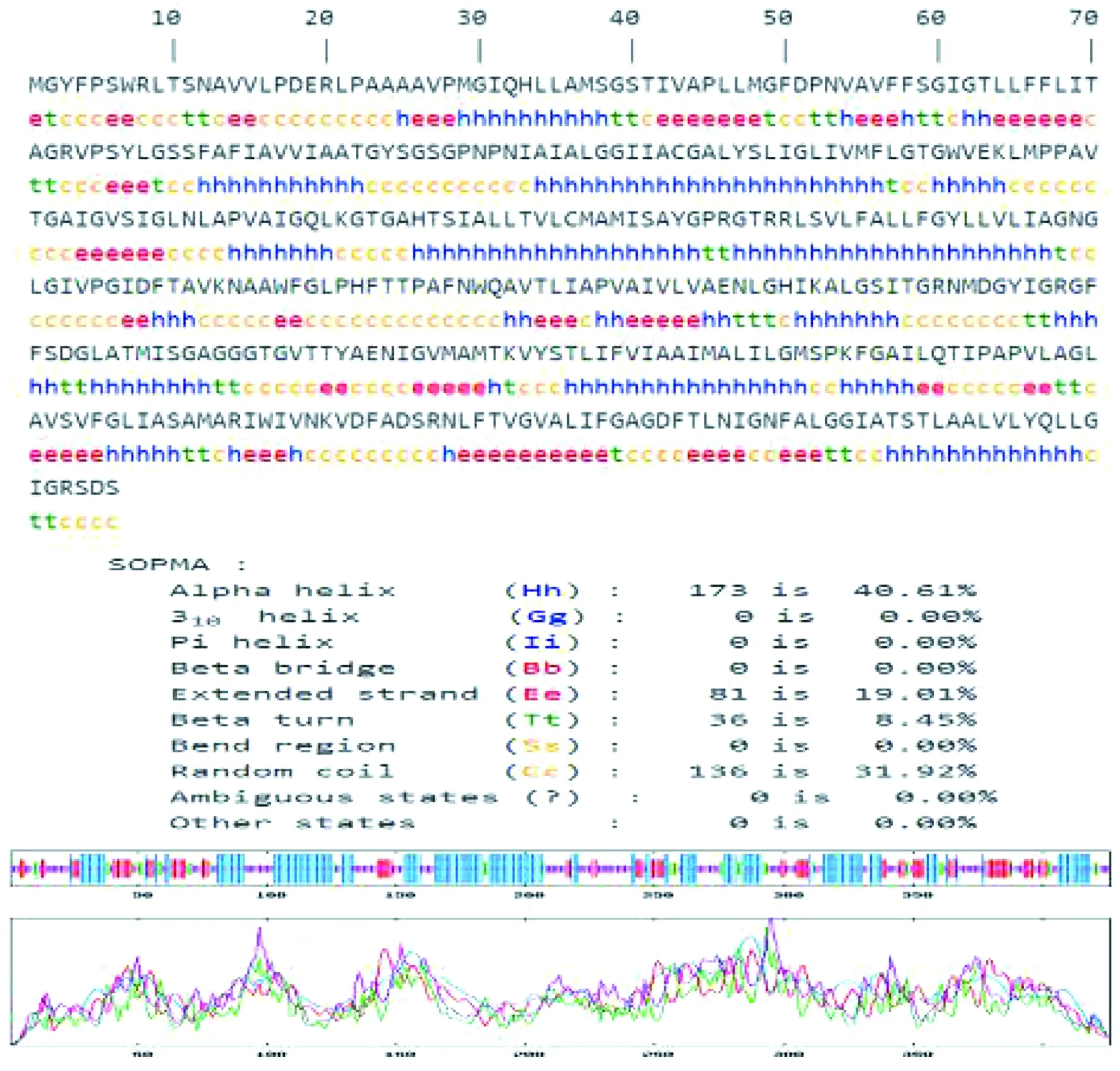
图2 SOPMA分析OMP16蛋白的二级结构结果Fig.2 Secondary structure of OMP16 protein predicted by SOPMA
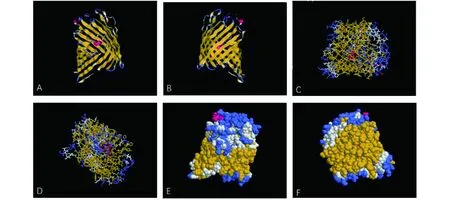
注:OMP16蛋白的三级结构由Phyre构建,其中红色为α-螺旋,黄色为β-折叠,蓝色为无规则卷曲,其他为白色。Cartoons模式(A)图为正面,(B)图为背面;Stick模式(C)图为正面,(D)图为背面;Spacefill模式(E)图为正面,(F)图为背面。图3 OMP16三级结构预测结果Fig.3 Tertiary structure prediction results for the OMP16 protein
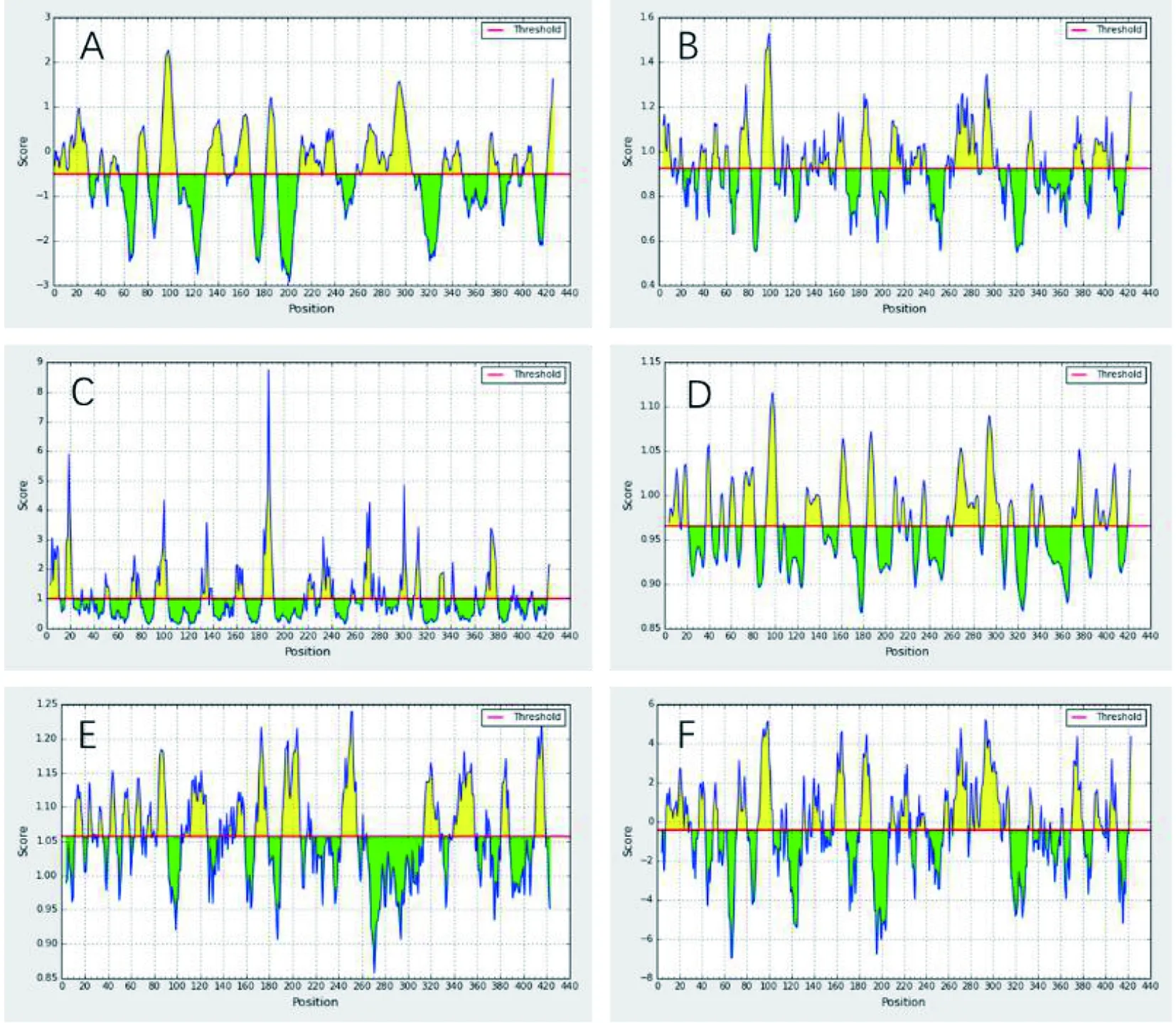
A:线性表位;B:β-转角 ;C:表面可及性;D:可塑性;E:抗原性;F:亲水性图4 IEDB分析预测结果Fig.4 Result of IEDB prediction
表2 BepiPred和ABCpred预测的B细胞表位结果
Tab.2 Results of B cell epitopes predicted by BepiPred and ABCpred
BepiPred 1.0b ServerABCpred Prediction Server 位置序列得分位置序列得分19-24ERLPAA0.9639-55GSTIVAPLLMGFDPNV0.9693-102GYSGSGPNPN2.2786-102AVVIAATGYSGSGPNP0.95137-143PPAVTGA0.72326-342ALILGMSPKFGAILQT0.9160-166LKGTGAH0.84273-289DGYIGRGFFSDGLATM0.9183-187YGPRGT1.20177-193MAMISAYGPRGTRRLS0.88269-275GRNMDGY0.44139-155AVTGAIGVSIGLNLAP0.88290-301SGAGGGTGVTT1.58294-310GGTGVTTYAENIGVMA0.85
2.4.2T细胞表位预测 利用SYFPEITHI和IEDB在线表位预测软件,HLA等位基因选择HLA-A*1101预测分析CTL细胞表位,选择HLA-DRB1*0701挑选重叠预测的Th表位。结果见表3。
2.4.3经过筛选和比较最终确定了4个B细胞的优势表位,5个CTL和5个Th细胞优势表位,结果见表4。
表3 SYFPEITHI和IEDB预测的T细胞表位结果
Tab.3 Results of T cell epitopes predicted by SYFPEITHI and IEDB

表位SYFPEITHIIEDB位置序列分数位置序列百分位等级CTL表位408STLAALVLY23281FSDGLATMI0.369ITAGRVPSY2258FSGIGTLLF0.45267ITGRNMDGY21408STLAALVLY0.65193VLFALLFGY18267ITGRNMDGY0.65293GGGTGVTTY17373FADSRNLFT0.786AVVIAATGY16191LSVLFALLF0.75109GIIACGALY1669ITAGRVPSY0.8175LCMAMISAY16380FTVGVALIF1.25373FADSRNLFT13175LCMAMISAY1.3Th表位4FPSWRLTSNAVVLPD32316TLIFVIAAIMALILG0.2232QHLLAMSGSTIVAPL3061IGTLLFFLITAGRVP0.25352VSVFGLIASAMARIW28310MTKVYSTLIFVIAAI0.26180ISAYGPRGTRRLSVL2662GTLLFFLITAGRVPS0.463TLLFFLITAGRVPSY2429MGIQHLLAMSGSTIV0.5974VPSYLGSSFAFIAVV24311TKVYSTLIFVIAAIM0.61121GLIVMFLGTGWVEKL2463TLLFFLITAGRVPSY0.66307VMAMTKVYSTLIFVI243YFPSWRLTSNAVVLP0.76316TLIFVIAAIMALILG2430GIQHLLAMSGSTIVA0.81
表4 B和T细胞优势表位
Tab. 4 B and T cell dominance epitopes

表位方法位置序列B细胞表位ABCpred93-102GYSGSGPNPNBepiPred183-187YGPRGTIEDB294-301GGTGVTTYDNAstar273-289DGYIGRGFFSDGLATMCTL细胞表位69-77ITAGRVPSY175-183LCMAMISAYSYFPEITHI267-275ITGRNMDGYIEDB373-381FADSRNLFT408-416STLAALVLYTh细胞表位4-20FPSWRLTSNAVVLPD32-46QHLLAMSGSTIVAPLSYFPEITHI63-77 TLLFFLITAGRVPSYIEDB316-330TLIFVIAAIMALILG352-366VSVFGLIASAMARIW
3 讨 论
布鲁氏菌病是全球很常见的人兽共患病,有160多个国家和地区受到布病的威胁。布鲁氏菌有10个种,造成人感染的主要是羊种、牛种和猪种布鲁氏菌,而羊种引起布鲁氏菌病最为常见[1,8]。感染布鲁氏菌后可引起波浪形发热并造成各种关节脏器损害,包括关节炎,脊椎炎,骶髂关节炎,心内膜炎和神经系统并发症。由于布鲁氏菌缺乏外毒素、菌毛等良好抗原表位,LPS也表现为低反应性。这些特点使布鲁氏菌发生了免疫逃避,并在宿主细胞中存活以建立持久感染[9]。布鲁氏菌疫苗这一很好的控制手段由于存在安全性等问题,还需要进行深入的研究[10]。
布鲁氏菌细胞膜有3层,OMP16位于细胞膜外侧,是外膜结构中必不可少的成分。与OMP10和OMP19属于第一组外膜蛋白[11]。根据之前的研究认为OMP16具有很强的免疫原性,可以激活TLR4-MyD88信息转导通路,刺激机体产生足够强度的免疫应答[12-13]。与传统的实验方法进行表位预测相比,生物信息学技术使蛋白结构分析和表位的预测变得更加高效和准确[14],目前已成为很多表位和疫苗研究首选方法[15-16],也常用于布鲁氏菌表位预测和疫苗的研制[17]。本研究使用多个软件对OMP16蛋白进行多角度的分析,分析结果显示OMP16蛋白总体是一个结构稳定的疏水性蛋白,二级结构中α-螺旋占40.61%,β-折叠占19.01%,β-转角占8.45%,无规则卷曲占31.92%。Phyre构建的三级结构显示OMP16蛋白具有多个可以形成表位的空间结构。α-螺旋和β-折叠是位于蛋白内部的刚性结构,较难形成表位,β-转角区域和无规则卷曲区域是很好的形成抗原表位区域。亲水性强、可及性和可塑性较好的区域也是形成表位的优势区域[18]。OMP16的蛋白结构显示其具有很好的表位预测价值。
本研究预测的4个B细胞优势表位均是在BepiPred和ABCpred共同预测表位分数较高的基础上,结合IEDB和DNAStar预测的亲水性等参数,除外在α-螺旋和β-折叠区域的表位,通过选取两种预测软件重合的区域最终确定93-102,183-187,273-289,294-301为B细胞优势表位。其中3个位于无规则卷曲区域,1个位于β-转角区域。BepiPred软件通过隐马尔可夫模型和倾向量表法来预测预测B细胞表位[19],而ABCpred软件是通过基于神经网络的一种预测方法[20],都具有很较高的预测精度。本次研究着重于线性B细胞表位的预测,因为它们更适用于开发基于肽的疫苗和诊断试剂[21]。
T细胞包括细胞毒性T淋巴细胞(CTL)和辅助性T细胞(Th)。CTL识别内源性抗原,可以与MHC Ⅰ类分子结合,Th细胞识别MHC II类分子。Th细胞通过分化为不同的Th亚群来抵御布鲁氏菌,Th1细胞激活并产生IFN-r等细胞因子发挥着关键的杀菌作用。同时布鲁氏菌是一种胞内感染菌,预测CTL和Th细胞表位均对布鲁氏菌清除具有重要意义。本研究使用SYFPEITHI和IEDB进行了分析和比较[22-23],HLA等位基因选择新疆地区人群出现频率较高的HLA-A*1101和HLA-DRB1*0701[24]进行预测,最终筛选出CTL和Th细胞优势表位各5个。
本研究预测的二级结构和T、B表位均采用多种方式在多个角度进行了分析和预测。T、B表位通过比较分析寻找两种软件的重叠区域,有效的规避了单一软件预测正确率不是很高的缺陷,可以提高预测结果的正确率。总之,我们认为筛选出的OMP16蛋白的B和T细胞优势表位具有很好的免疫原型,能够为进一步疫苗开发和布病诊断提供一定的理论基础。
利益冲突:无
