百子莲2个ARF基因与2个Aux/IAA基因的全长克隆与序列分析
2019-01-24王俊杰
杨 舟,吕 可,吕 珊,王俊杰,张 荻
(上海交通大学 农业与生物学院 园林科学与工程系,上海 200240)
百子莲(Agapanthuspraecox)又称蓝百合,是百子莲科百子莲属多年生观赏花卉,原产于非洲南部,花量大而美丽,在欧美地区常作为庭院观赏和高档鲜切花应用,具有良好的观赏价值和应用前景,我国于2002年将其引入上海地区,引种栽培研究中发现夏季高温高湿气候引起百子莲结实率与种子萌发率下降,严重限制了品种生产与推广。2010年后建立百子莲体细胞胚胎发生体系、组织培养扩繁与种质资源保存等体系,为该品种在园林景观的应用奠定了重要技术保障[1-2]。目前,体细胞胚胎发生是百子莲快速繁殖和优质种质资源保存的主要方法[1]。
生长素作为唯一具有极性运输特征的植物内源激素,是大多数植物体细胞胚胎发生必不可少的启动因子,对花芽分化、根茎生长发育、器官发育与衰老、维持顶端优势、胚胎极性结构建立等诸多植物生长过程起到重要的调节作用[3-4]。近年研究表明,生长素对于百子莲花芽分化、胚胎发育和胚性愈伤组织诱导等生理过程具有重要的调控作用[2]。生长素信号通过作用于下游复杂的调控网络调控植物多方面的生物过程,其主要元件(TIR/AFB、Aux/IAA与ARF)都具有各自的基因家族[5]。其中,生长素响应因子(auxin response factors,ARF)与生长素信号负转录调控因子生长素/吲哚乙酸(auxin/indole acetic acid proteins,Aux/IAA)是参与生长素信号调控下游基因表达的主要转录因子[6-7]。Aux/IAA蛋白通过蛋白互作控制ARF蛋白的活性,进而调节生长素相关基因的表达[8-9]。当植物体内的生长素含量较低时,Aux/IAA蛋白与ARF蛋白相结合,抑制生长素相关基因的转录;当生长素含量上升时,生长素会结合自身受体蛋白TIR1,促进Aux/IAA蛋白的降解,减少其对ARF转录因子的抑制作用,使得ARFs能够调控生长素信号应答基因的表达[5, 9]。
前期有关百子莲体胚体系构建的研究发现,适宜浓度的外源生长素类调控物质毒莠定PIC(4-氨基-3,5,6-三氯吡啶羧酸,picloram)对于百子莲愈伤组织的胚性启动与胚性保持具有良好的诱导及调控作用[10],而浓度偏高或偏低都会导致细胞胚性丧失或退化(图1-A),并通过影响内源生长素信号调控体细胞胚性发育与形态建成(图1-B)。应用二代测序技术对不同浓度PIC继代的胚性愈伤组织(embryogenic callus,EC)进行比较转录组学研究,发现在胚性稳定维持的细胞中ARF家族基因与IAA20表达增强;与非胚性细胞相比,EC中的Aux/IAA家族的IAA20、IAA26、IAA30与IAA4均呈现下调表达,而ARF家族基因明显上调表达。因此推断生长素信号在百子莲愈伤组织的胚性诱导与保持过程中具有重要调控作用,且IAA4、IAA26、IAA20、IAA30、ARF1与ARF2等基因可能在此过程中发挥重要的信号传导调控作用(图2)。
本文通过百子莲体胚发生差异表达文库,获得与生长素信号转导途径密切相关的ARF和Aux/IAA基因的核心序列,应用RACE(rapid amplification of cDNA ends)技术克隆其中2个Aux/IAA与2个ARF家族基因全长,并分析蛋白结构功能,为揭示其在体胚诱导调控过程中的分子作用机制奠定基础。
1 材料与方法
1.1 实验材料

A,百子莲愈伤组织胚性保持的细胞形态学差异;B,百子莲胚性愈伤组织细胞内源IAA含量差异;不同小写字母代表生长素含量在不同样品间的显著性差异,LSD P<0.05。A, PIC plays an important role in Agapanthus praecox embryogenic induction and keeping; B, IAA contents of callus and embryogenic callus in Agapanthus praecox. The bars with different lowercase letters showed the significant difference (P<0.05).图1 不同浓度PIC继代的百子莲胚性愈伤组织Fig.1 Embryogenic callus of Agapanthus praecox treated with different concentrations of PIC

图2 百子莲胚性愈伤组织间生长素信号转导途径中的基因差异表达变化Fig.2 Auxin signal transduction pathway related-differentially expressed genes between EC subcultured by different concentrations of PIC in Agapanthus praecox
百子莲实生苗幼叶取自上海交通大学农业与生物学院试验田,液氮速冻后存于-80 ℃冰箱;Trizol试剂购于Invitrogen公司;SMARTerTMRACE cDNA Amplification Kit购于Clonetech公司;DH5-α感受态细胞购自Tiangen公司;SanPrep柱式DNA 胶回收试剂盒购自上海生工有限公司;pHB质粒购于Biovector中国质粒保存中心;Prime Script Reverse Transcriptase,EXTaqenzyme,LATaqenzyme,Prime Script Reverse Transcriptase,Oligo dT18,RNase inhibitor,DNaseⅠ,pMD18-T vector,SYBR Premix ExTaq等购于TaKaRa公司;引物由上海生工有限公司合成;测序由上海Invitrogen生物技术有限公司完成。
1.2 实验方法
1.2.1ApARF与ApAux/IAA基因全长克隆
以百子莲叶片为材料,利用Trizol试剂提取并纯化总RNA[11]后采用Prime Script Reverse Transcriptase试剂盒进行反转录。以ApARF与ApAux/IAA基因核心片段序列为参考设计特异性引物(表1)进行PCR扩增,反应程序为:94 ℃ 3 min;94 ℃ 30 s,55 ℃ 30 s,72 ℃ 1 min,35个循环;72 ℃ 3 min。产物经琼脂糖电泳检测回收后连接pMD18-T,转化大肠埃希菌测序验证核心序列的准确性,以其为依据设计3′-RACE与5′-RACE的特异性引物(表1)。按照购于TaKaRa公司的OligotexTM-dT30
按照SMARTerTMRACE用户手册分别配置ApARF与ApAux/IAA基因的3′, 5′-RACE第1轮PCR扩增体系,以RACE cDNA文库为模板,采用通用引物(UPM)和特异性引物(3′, 5′-GSP1)扩增。程序为94 ℃ 30 s,72 ℃ 3 min,5个循环;94 ℃ 30 s,70 ℃ 30 s,72 ℃ 3 min,5个循环;94 ℃ 30 s,68 ℃ 30 s,72 ℃ 3 min,25个循环。3′,5′-RACE第2轮巢式PCR扩增体系以稀释后的第一轮扩增产物为模板,利用NUP和3′, 5′-GSP2为引物,扩增程序为94 ℃ 2 min;94 ℃ 30 s,68 ℃ 30 s,72 ℃ 1 min,30个循环;72 ℃ 3 min。
表1ApARF与ApAux/IAA基因扩增与RACE引物列表
Table1Sequences of primers for PCR ofApARFandApAux/IAA
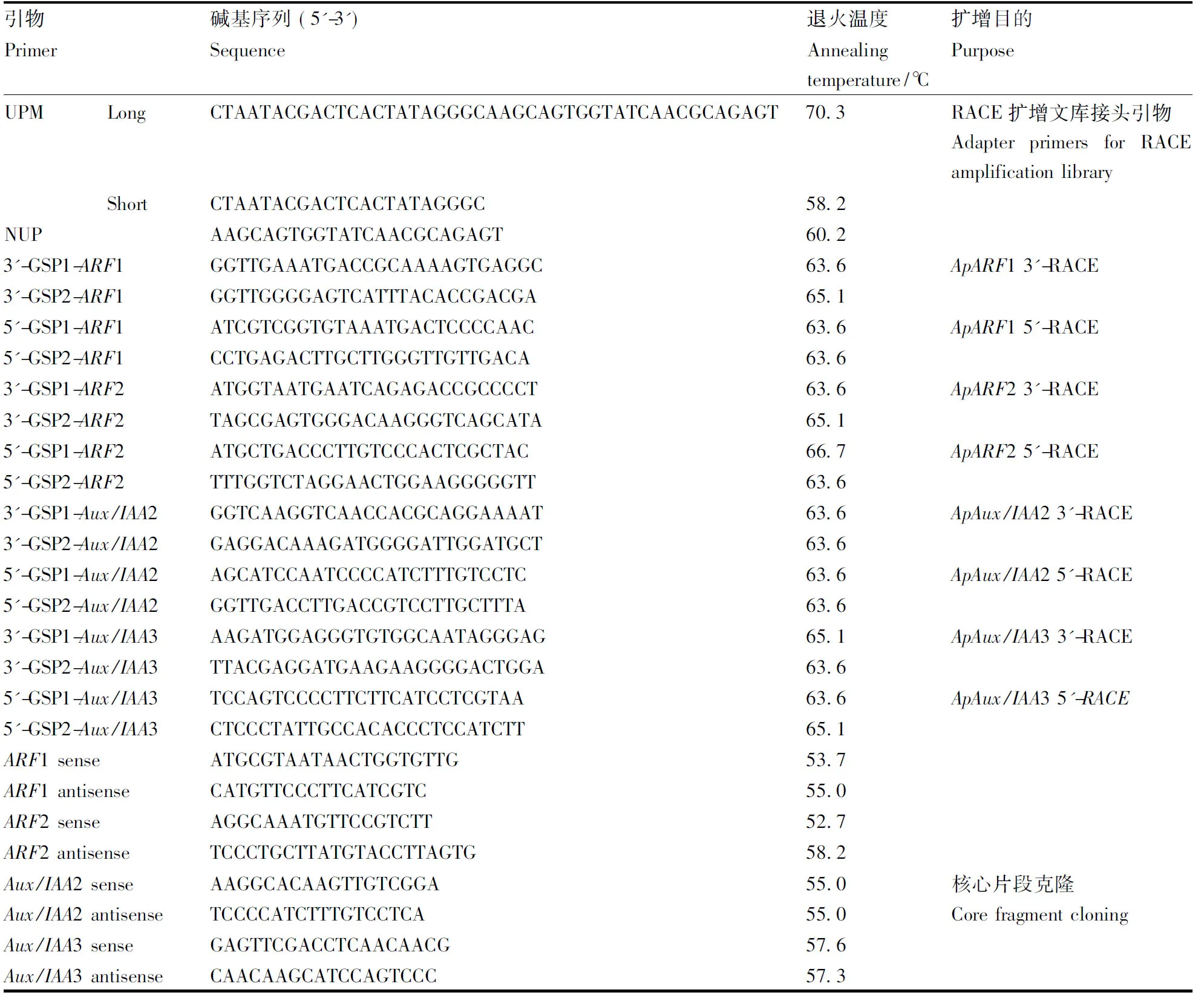
引物碱基序列 (5'-3')退火温度扩增目的PrimerSequenceAnnealingtemperature/℃PurposeUPMLongCTAATACGACTCACTATAGGGCAAGCAGT-GGTATCAACGCAGAGT70.3RACE扩增文库接头引物Adapter primers for RACE amplification libraryShortCTAATACGACTCACTATAGGGC58.2NUPAAGCAGTGGTATCAACGCAGAGT60.23'-GSP1-ARF1GGTTGAAATGACCGCAAAAGTGAGGC63.6ApARF1 3'-RACE3'-GSP2-ARF1GGTTGGGGAGTCATTTACACCGACGA65.15'-GSP1-ARF1ATCGTCGGTGTAAATGACTCCCCAAC63.6ApARF1 5'-RACE5'-GSP2-ARF1CCTGAGACTTGCTTGGGTTGTTGACA63.63'-GSP1-ARF2ATGGTAATGAATCAGAGACCGCCCCT63.6ApARF2 3'-RACE3'-GSP2-ARF2TAGCGAGTGGGACAAGGGTCAGCATA65.15'-GSP1-ARF2ATGCTGACCCTTGTCCCACTCGCTAC66.7ApARF2 5'-RACE5'-GSP2-ARF2TTTGGTCTAGGAACTGGAAGGGGGTT63.63'-GSP1-Aux/IAA2GGTCAAGGTCAACCACGCAGGAAAAT63.6ApAux/IAA2 3'-RACE3'-GSP2-Aux/IAA2GAGGACAAAGATGGGGATTGGATGCT63.65'-GSP1-Aux/IAA2AGCATCCAATCCCCATCTTTGTCCTC63.6ApAux/IAA2 5'-RACE5'-GSP2-Aux/IAA2GGTTGACCTTGACCGTCCTTGCTTTA63.63'-GSP1-Aux/IAA3AAGATGGAGGGTGTGGCAATAGGGAG65.1ApAux/IAA3 3'-RACE3'-GSP2-Aux/IAA3TTACGAGGATGAAGAAGGGGACTGGA63.65'-GSP1-Aux/IAA3TCCAGTCCCCTTCTTCATCCTCGTAA63.6ApAux/IAA3 5'-RACE5'-GSP2-Aux/IAA3CTCCCTATTGCCACACCCTCCATCTT65.1ARF1 senseATGCGTAATAACTGGTGTTG53.7ARF1 antisenseCATGTTCCCTTCATCGTC55.0ARF2 senseAGGCAAATGTTCCGTCTT52.7ARF2 antisenseTCCCTGCTTATGTACCTTAGTG58.2Aux/IAA2 senseAAGGCACAAGTTGTCGGA55.0核心片段克隆Aux/IAA2 antisenseTCCCCATCTTTGTCCTCA55.0Core fragment cloningAux/IAA3 senseGAGTTCGACCTCAACAACG57.6Aux/IAA3 antisenseCAACAAGCATCCAGTCCC57.3
琼脂糖凝胶电泳检测回收产物,连接pMD18-T vector载体进行测序,获得4个目的基因的3′-和5′-端序列。
1.2.2 生物信息学分析
采用DNAMAN软件拼接5′-RACE、3′-RACE与核心序列,获得目的基因cDNA全长序列;NCBI ORF Finder(http://www.ncbi.nlm.nih.gov/gorf/orfig.cgi)分析序列的CDS-ORF区,DNAMAN软件对ORF区编码的氨基酸序列进行翻译。NCBI网站BlastP(https://blast.ncbi.nlm.nih.gov/Blast.cgi)比对氨基酸序列,采用ClustalX软件分析序列一致性和同源性。TAIR网站(http://www.arabidopsis.org/)下载拟南芥(Arabidopsisthaliana)中已知的23个ARF家族和29个AUX/IAA家族蛋白序列,运用MEGA 5.0软件构建系统进化树(neighbor-joining tree,Bootstrap=1 000)。应用SOPMA软件对氨基酸二级结构进行预测分析。通过NCBI 的Conserved Domain Database数据库(http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi),EMBI-EBI的Pfam 数据库(http://pfam.xfam.org/)和PROSITE(http://prosite.expasy.org/)在线软件对蛋白保守域进行分析[12-13]。利用ProtParam在线工具(http://expasy.org/tools/protparam.html)分析氨基酸的构成,MEME在线工具(http://meme-suite.org/tools/meme)对Aux/IAA蛋白的基序进行预测,输入百子莲与BLAST获得的其他物种中的共12个Aux/IAA蛋白的序列,设定基序最大发现数目为4[14-15]。
1.2.3 基因过表达载体构建并转化拟南芥
采用pHB质粒并选定PstⅠ和BamHⅠ为限制性酶切位点。拼接测序结果获得基因全长序列后设计扩增引物(表2),以百子莲cDNA文库为模板,在基因ORF片段的上下游分别引入BamHⅠ和PstⅠ酶切位点。反应程序为94 ℃ 3 min;94 ℃ 30 s,55 ℃ 30 s,72 ℃ 1 min,35个循环;72 ℃ 3 min。随后采用酶切连接法构建载体,测序验证载体构建成功后转入农杆菌GV3101感受态,挑取农杆菌阳性单克隆经PCR检测后,以花序浸染法侵染拟南芥。
1.2.4 转基因拟南芥筛选鉴定及观察
收获的T1代种子播种后采用0.05%(V/V)草甘膦进行抗性筛选从而确定转化成功后,选择5~6个株系进行培育并收获T2代种子。将T2代拟南芥种子与野生型(Columbia)种子经4 ℃春化后播种于穴盘中。在22 ℃,16 h/8 h(光照/黑暗)的人工气候箱中培养,2周后对植株生长情况进行表型观察。取表型差异明显且稳定的转基因株系叶片组织提取RNA并进行反转录(方法同前),随后采用qRT-PCR测定4个基因型T2代中目的基因的表达量。使用Beacon Design 7软件设计qRT-PCR引物(表2)。反应程序为94 ℃ 10 min;94 ℃ 20 s,55 ℃ 20 s,72 ℃ 20 s,40个循环。每个样品进行3个重复,反应完成后采用2-ΔΔCt法进行数据分析,采用UBQ5作为内参基因(表2)。
2 结果与分析
2.1 ApARF与ApAux/IAA家族基因的全长序列获得
获得ApAux/IAA2、ApAux/IAA3、ApARF1、ApARF2的基因核心片段序列分别为330、474、921、1 617 bp;3′-端克隆序列长度分别为501、320、453、785 bp。5′-端第2轮巢式PCR扩增后,ApARF2基因出现了3个较明显的条带(图3-D),对各个条带都进行回收和测序比对分析后,确定长度在1 000 bp以上的ApARF2扩增条带为正确结果。最终获得ApAux/IAA2,ApAux/IAA3,ApARF1,ApARF2的5′-端克隆序列长度分别为375、399、1 576、1 356 bp。
将5′-RACE与3′-RACE的测序结果与核心序列进行拼接后,获得ApARF1、ApARF2、ApAux/IAA2、ApAux/IAA3基因的cDNA全长序列分别为2 343、2 888、1 034、821 bp;起始密码子和终止密码子分别位于256 nt/2 023 nt、200 nt/2 546 nt、105 nt/583 nt、77 nt/623 nt;各基因的5′非编码区和3′非编码区长度分别为255 bp/318 bp、199 bp/340 bp、104 bp/450 bp、76 bp/196 bp;CDS-ORF全长分别为1 770、2 349、480和549 bp;分别编码589、782、159和182个氨基酸残基,分子量分别为65.99、87.78、18.06、20.72 ku,pI分别为6.36、6.36、6.54和6.92(图4)。
2.2 蛋白二级结构分析
蛋白二级结构分析表明(图5),ApARF1蛋白由31.24%的α-螺旋(alpha helix),39.56%的随机卷曲(random coil),22.24%的延伸链(extended strand)和6.96%的β-转角(beta turn)组成,其中α-螺旋与随机卷曲占比最大;ApARF2蛋白由53.84%的随机卷曲,20.84%的延伸链,19.44%的α-螺旋和5.88%的β-转角组成,其中随机卷曲占比最大。ApAux/IAA2蛋白由33.33%的延伸链,28.93%的α-螺旋,25.16%的随机卷曲和12.58%的β-转角组成,其中α-螺旋和延伸链占比最大;ApAux/IAA3蛋白由39.56%的随机卷曲,25.82%的α-螺旋,22.53%的延伸链和12.09%的β-转角组成,其中随机卷曲和α-螺旋占比最大。四个蛋白中的α-螺旋、延伸链、β-转角和随机卷曲分布规律性不明显。
表2ARF与Aux/IAA过表达载体构建与qRT-PCR检测引物列表
Table2Sequences of primers for construction of over-expression vector and qRT-PCR ofApARFandApAux/IAA

引物名称碱基序列(5'-3')引物名称碱基序列(5'-3')PrimersSequencePrimersSequenceApARF1-RsATGGATCCATGGTGTTAGATGGTAAApARF1-RaTTACTGCAGTCAAGGTTGCAACTTCTApARF2-RsATGGATCC ATGGCGTCTCCTGAGGTApARF2-RaTTACTGCAGTCATGACTGGCTGTTGTApAux/IAA2-RsATGGATCCATGAGAGATCCAATGGAAGApAux/IAA2-RaTTACTGCAGTTACCCCCATGGAACATCApAux/IAA3-RsATGGATCCATGGAGCTAGAGTTAGGCCTApAux/IAA3-RaTTACTGCAGCTATGCAG-GTCTCTCTCGTTqT-F-ARF1TTCAGAGTCCATAGTTCCTTATqT-R-ARF1AACCTTGAGATGCTTCCAqT-F-ARF2ACTCAGATGGATTACTCACAAqT-R-ARF2GGCTCATAGGTAGGATTCAAqT-F-AUX2GACGATTGTGGTGTTGATqT-R-AUX2CCATTGGAGCAAGTTCTTqT-F-AUX3CAATAACCATATCGCCTATCCqT-R-AUX3ATCGTCCTCCTTGTCATCUBQ5-FGACGCTTCATCTCGTCCUBQ5-RCCACAGGTTGCGTTAG

A,3′-RACE第一轮扩增产物;B,3′-RACE第二轮巢式扩增产物;C,5′-RACE第一轮扩增产物;D,5′-RACE第二轮巢式扩增产物。M,DL2000 DNA marker。A, The first amplification of 3′-RACE; B, The second amplification of 3′-RACE; C, The first amplification of 5′-RACE; D, The second amplification of 5′-RACE. M, DL2000 DNA marker.图3 3′,5′-RACE扩增产物电泳检测图谱Fig.3 Agarose gel electrophoresis of RACE products

图5 百子莲ApARF与ApAux/IAA蛋白二级结构特征Fig.5 Secondary structure prediction of ARF and Aux/IAA proteins in Agapanthus praecox
2.3 蛋白结构域分析
蛋白保守域分析结果表明,百子莲ApARF1具有B3-DNA结合域(pfam02362)与Auxin_resp结构域(pfam06507,也称ARF结构域),位于氨基酸序列的113~212位与239~321位;ApARF2具有结构域B3-DNA,Auxin_resp与AUX_IAA(pfam02309)分别位于序列的141~242位,267~349位与665~737位。ApAux/IAA2与ApAux/IAA3均具有AUX_IAA 结构域(pfam02309),位于氨基酸序列的21~159位和69~171位。
同时,ApAux/IAA与ApARF氨基酸序列都在C-端含有60~90 aa的PB1保守域(Phox and Bem1,PS51745),且与ApAux/IAA2、ApAux/IAA3中的Aux/IAA结构域部分重合,与ApARF2中的Aux/IAA结构域基本完全重合(图6)。
氨基酸数目统计结果表明,ApARF1与ApARF2中间区域均富含丝氨酸和脯氨酸(ApARF1: Pro7.9%,Ser 13.1%;ApARF2: Pro 10.6%,Ser 12.8%)。

图6 百子莲ARF 与AUX/IAA家族蛋白保守结构域Fig.6 Conserved protein domain of ARF and Aux/IAA protein in Agapanthus praecox
基序分析获得4个基序,长度分别为21,41,50,29个氨基酸,对应Aux/IAA蛋白中的Ⅰ、Ⅱ、Ⅲ和Ⅳ结构域(图7)。分析结果表明,百子莲Aux/IAA 2蛋白具有4个结构域,其中Ⅳ结构不完整;百子莲Aux/IAA 3蛋白具有Ⅰ、Ⅲ和Ⅳ结构域,而结构域Ⅱ保守性低(图8)。
2.4 氨基酸序列同源性分析
BlastP分析表明,ApARF1与ApARF2基因编码的氨基酸序列与多种其他植物中的ARF蛋白具有较高的同源性,为50%以上(图8)。其中,与ApARF1氨基酸序列同源性最高的是油棕(ElaeisguineensisARF 24-like),同源性为65%;与海枣(PhoenixdactyliferaARF 24-like isoform X2)的同源性为64%。与ApARF2氨基酸序列同源性最高的是油棕(ElaeisguineensisARF 4-like),同源性为65%;其次是荷花(NelumbonuciferasARF 2-like),同源性为62%。
ApAux/IAA2与ApAux/IAA3编码的氨基酸序列同样与其他植物中的Aux/IAA家族蛋白具有较高的同源性,为50%以上(图9)。与百子莲ApAux/IAA2氨基酸序列同源性最高的是海枣(PhoenixdactyliferaAUX 22D-like 8132),同源性为61%,其次是油棕(ElaeisguineensisAUX 22D-like 3480),同源性为60%。与ApAux/IAA3氨基酸序列同源性最高的是凤梨(AnanascomosusAux IAA20),同源性为60%,其次是小果野蕉(Musaacuminatasubsp.MalaccensisAux IAA9-like X1),同源性为55%。

方框圈定的区域为图9中标注的保守结构域的序列。Positions of conserved domains displayed in Fig.9 were boxed.图7 Aux/IAA家族蛋白4个基序Fig.7 Four motifs distribution of 12 Aux/IAA proteins

Asparagus officinalis ARF 2-LIKE isoform X2(XP_020274070.1),Asparagus officinalis ARF 2-LIKE isoform X1(XP_020274069.1),Asparagus officinalis ARF 23-LIKE isoform X3(XP_020274071.1),Phoenix dactylifera ARF 24-like isoform X2(XP_008813740.1),Elaeis guineensis ARF 24-like isoform X2(XP_010914409.1);Nelumbo nucifera ARF 2-like(XP_010249209.1),Elaeis guineensis ARF4-like(XP_010926858.1),Asparagus officinalis ARF 2-like X1(XP_020261175.1),Asparagus officinalis ARF 2-like X2 (XP_020261177.1),Asparagus officinalis ARF 2-like(XP_020248955.1).图8 ApARF1与ApARF2氨基酸序列同源性比对Fig.8 Multiple sequence alignment of ARF in Agapanthus praecox and other plants

Phoenix dactylifera AUX 22D-like 8132(XP_008788132.1),Elaeis guineensis AUX 22D-like 3480(XP_010923480.1),Elaeis guineensis AUX 22D 6161(XP_010936161.1),Asparagus officinalis AUX 22D-like 4650(XP_020274650.1),Asparagus officinalis A4U43 4425(ONK64425.1),Elaeis guineensis Aux IAA20 X2(XP_010904976.1),Phoenix dactylifera Aux IAA9-like X5 (XP_008809248.1),Musa acuminata subsp. malaccensis Aux IAA9 X1(XP_009419087.1),Asparagus officinalis AuxIAA20-like(XP_020244002.1),Ananas comosus Aux IAA20 (OAY64285.1).图9 ApAux/IAA2与ApAux/IAA3氨基酸序列同源性比对Fig.9 Multiple sequence alignment of Aux/IAA in Agapanthus praecox and other plants
2.5 系统进化树分析
植物ARF与Aux/IAA家族蛋白成员众多,已经在多种草本和木本植物中被分析研究[4],其表达特征也比较复杂[7,16]。因此采用拟南芥(Arabidopsisthaliana)中研究较为深入的23个ARF和29个Aux/IAA家族蛋白[17]分别与ApARF1、ApARF2及ApAux/IAA2、ApAux/IAA3蛋白构建系统进化树,分析蛋白在生长素调控途径中的作用和潜在的相互作用机制。
系统进化树分析结果(图10)表明,ARF家族蛋白在系统进化树中聚合成4类,其中ApARF1与AtARF1(AT1G59750)聚合,ApARF2与AtARF2(AT5G62000)聚合,4个蛋白均在同一聚类中。ApAux/IAA2与AtIAA1(AT4G14560)、AtIAA2(AT3G23030)、AtIAA3(AT1G04240)、AtIAA4(AT5G43700.1)聚合成一簇,与AtIAA4的亲缘关系最近;ApAux/IAA3与AtIAA29(AT4G32280)、AtIAA20(AT2G46990)、AtIAA30(AT3G62100)、AtIAA31(AT3G17600)聚合为一簇。
2.6 转基因植株功能验证
基因荧光定量实验表明,在ApARF1、ApARF2、ApAux/IAA2和ApAux/IAA3过表达拟南芥中,目的基因的表达量分别是内参基因UBQ5的2.6、3.6、3.4和3.2倍,而在野生型植株中均未见表达(图11)。
表型比对结果表明,ApARF1与ApARF2转基因植株的生长速度均明显快于野生型(图11-C、D),可见ApARF1与ApAPF2正向调控植株的开花成熟及生长过程。ApAux/IAA2和ApAux/IAA3转基因拟南芥植株矮小,生长速度则明显慢于野生型,发育受到阻遏,说明ApAux/IAA2和ApAux/IAA3基因对于植物的生长存在负调控作用(图11-G、H)。
3 讨论
目前已在植物中发现众多ARF与Aux/IAA家族蛋白成员。Aux/IAA在大豆(Glycinemax)、玉米(Zeamays)、水稻(Oryzasativa)[16]、马尾松(Pinusmassoniana)[18];ARF在水稻(Oryzasativa)、玉米(Zeamays)、番茄(Solanumlycopersicum)、大豆(Glycinemax)、油菜(Brassicanapus)、葡萄(Vitisvinifera)[4]等植物中被发现和分析。

左为Aux/IAA家族,右为ARF家族。Bootstrap=1 000。Neighbor-joining bootstrap phylogenetic trees of ARFs (left) and Aux/IAAs (right) from Arabidopsis thaliana and Agapanthus praecox. The bootstrap was 1 000.图10 ApAux/IAA与ApARF家族蛋白系统进化树Fig.10 Phylogenetic analysis of Aux/IAA and ARF proteins

A,ApARF1基因相对表达量;B,ApARF1转基因拟南芥表型;C,ApARF2基因相对表达量;D,ApARF2转基因拟南芥表型;E,ApAux/IAA2基因相对表达量;F,ApAux/IAA2转基因拟南芥表型;G,ApAux/IAA3基因相对表达量;H,ApAux/IAA3转基因拟南芥表型;虚线表示内参基因UBQ5表达量。A, Relative gene expression of ApARF1; B, Phenotype of ApARF1 over-expression Arabidopsis thaliana; C, Relative gene expression of ApARF2; D, Phenotype of ApARF2 over-expression Arabidopsis thaliana; E, Relative gene expression of ApAux/IAA2; F, Phenotype of ApAux/IAA2 over-expression Arabidopsis thaliana; G, Relative gene expression of ApAux/IAA3; H, Phenotype of ApAux/IAA3 over-expression Arabidopsis thaliana. The dotted line indicated the expression of reference gene UBQ5.图11 ApARF和ApAux/IAA转基因拟南芥植株qRT-PCR检测结果与生长表型Fig.11 qRT-PCR detection and phenotype observation of ApARF and ApAux/IAA genes in wild type and transgenic over-expression Arabidopsis thaliana
本研究通过RACE技术获得百子莲中2个Aux/IAA基因和2个ARF基因的全长,并对各个基因所编码的蛋白进行分析。在蛋白结构域组成上,ApARF2符合典型的ARF家族蛋白构造,具有N-端的B3-like DNA结合域,C-端Aux_IAA结构域和中间区域[4,7];ApARF1缺失C-端负责形成ARF、Aux/IAA间同源/异源二聚体的AUX_IAA结构域,但具有PB1保守域(图6)。PB1结构域能够介导蛋白间异源或同源聚化[19],如拟南芥AtARF17蛋白,虽缺失C端的保守域,但仍能够与Aux/IAA蛋白互作[9,13],序列分析表明其具有PB1结构域,可见其起到了代替C端的AUX_IAA结构域的作用。系统进化树的聚合结果(图10)表明,ApARF1、ApARF2与转录抑制因子AtARF1、AtARF2聚合。百子莲ApARF1、ApARF2的中间结构域都富含丝氨酸与脯氨酸,也表明其应具有转录抑制功能,在植物中功能类似AtARF1与AtARF2,通过抑制植物细胞分裂相关基因的转录来正向调节叶片的衰老、花脱落、角果成熟等相关生理过程[8,12,20]。转基因表型实验也初步验证了二者与植株生长成熟过程相关的推测。
百子莲ApAux/IAA2蛋白具有完整的Ⅰ、Ⅱ、Ⅲ结构域,虽蛋白互作相关结构Ⅳ结构域不完整,但因具有PB1保守域,应仍具有形成蛋白二聚体的功能。系统进化树中(图10),ApAux/IAA2与具有完整结构的AtAux/IAA1和AtAux/IAA3蛋白功能相近,应能与ARF家族蛋白互作并响应生长素信号,负调节生长素响应因子,抑制植株的地上部分或根系的生长[21-22]。ApAux/IAA2基因过表达植株出现的生长延缓表型特征初步验证了这一推测。百子莲ApAux/IAA3蛋白具有Ⅰ、Ⅲ、Ⅳ结构域,结构域Ⅱ不完整(图8)。系统进化树中与其聚合的蛋白中,AtIAA31、AtIAA20、AtIAA30、AtIAA29也缺失结构域Ⅱ[15-16]。结构域Ⅱ的缺失会使IAA蛋白更加稳定且难以被降解,进而抑制ARF转录活性从而影响相关生长素介导的发育过程[23],如AtIAA31蛋白,稳定性高于典型Aux/IAA蛋白,基因过表达可能造成植株矮化等生长素缺陷表型[24],与ApAux/IAA3基因过表达植株的表型相一致。此外,AtIAA30蛋白具有促进体细胞胚胎的发生的功能[24],ApAux/IAA3也可能具有类似的调控作用。
本研究对百子莲Aux/IAA家族和ARF家族蛋白在体胚诱导和生长素信号转导中的作用进行分析,并通过基因克隆、蛋白结构和功能验证,为进一步探究植物细胞胚性诱导和构建体胚体系提供理论指导。目前对于ARF与Aux/IAA家族蛋白在植物体内的功能研究表明,ARF与Aux/IAA之间的同源或异源二聚体化与它们在生长素信号调控过程中发挥的生理功能紧密相关[9]。为进一步验证百子莲ARF与Aux/IAA蛋白之间的互作和相互调节,在后续实验中可通过酵母双杂实验检验百子莲ApARF与ApAux/IAA蛋白之间是否存在直接对应的相互作用,并通过酵母单杂交方式研究ARF蛋白是否对通过结合Aux/IAA上游的元件激活并调节基因的表达,从而进一步揭示ARF与Aux/IAA在观赏植物生长素信号传导过程中的调控作用。
