基于竞争性等位基因特异性PCR技术的麦芽品种纯度定性和定量检测
2019-01-07蒋培基王德良徐东东黄承雪郭刚刚栾春光
蒋培基,王德良,徐东东,黄承雪,郭刚刚,栾春光,*,蒲 彪*
(1.四川农业大学食品学院,四川 雅安 625014;2.中国食品发酵工业研究院,北京 100015;3.中国农业科学院作物科学研究所,北京 100081)
大麦属于小麦族大麦属,是古老的禾谷物作物之一,是我国继水稻、小麦、玉米之后的第4大禾本科作物[1]。麦芽是由大麦浸泡吸水发芽后烘干而成,是啤酒酿造的主要原料[2]。大麦麦芽一直以来都是啤酒厂把控产品质量的关键[3]。麦芽质量的好坏不仅影响啤酒品质以及口感,而且不同麦芽品种之间的淀粉、蛋白质等成分存在的差异也会造成啤酒厂在酿造工艺上存在一定的差别[4]。目前,大麦种植过程中会出现由于机械、生物等途径造成的混杂,制麦和销售中也会出现不法商贩的人为掺假,导致市面上的大麦麦芽均一性不高,因此给酿造工艺的选择上带来很多问题,啤酒质量得不到保障[5-8]。所以,对于啤酒行业开展大麦麦芽的品种和纯度检测势在必行。
近些年来,快速发展的分子标记技术以其遗传稳定、重复性好及易操作等优点已经开始替代同工酶电泳和简单理化指标分析等传统方法,成为农作物品种鉴定的主要方法[9]。基于第1、2代DNA分子标记技术建立起来的农作物品种鉴定方法,比如限制性片段长度多态性[10-14]、随机扩增片段长度多态性[15-16]和简单重复序列[17-18],相对于蛋白水平的鉴定具有一定优势,但这些方法都是基于核酸片段的多少和片段大小完成物种的鉴别。对于亲缘关系相近的品种之间存在判断上的误差,且无法完成混杂或人为掺杂的样品进行检测[19]。为克服以上缺点,人们开发出了第3代DNA分子标记,即单核苷酸多态性(single nucleotide polymorphisms,SNP)。该分子标记的出现为农作物的品种鉴定提供了更为高效、准确、灵敏的检测手段。杨润婷等[20]对比了两种常用的SNP分型方法,等位基因特异性聚合酶链式反应(allele-specific polymerase chain reaction,ASPCR)和高分辨率熔解曲线分析(high resolution melting analysis,HRMA),对16 个柚栽培品种和8 个柚杂种后代材料进行7 个SNP位点的分型研究时发现AS-PCR和HRMA均适用于柚类品种的区分和鉴定。 而AS-PCR方法是一种准确、低成本的SNP分型方法,适合普通实验室使用。Bungartz等[21]用SNP基因芯片对20 个大麦品种进行品种鉴定研究,共发现6 334 个SNP标记。高丽峰等[22]分析来自Illumina小麦9K Infmium SNP芯片数据中6 254 个位点,从中筛选出43 个足以将193 份名优特和359 份实验材料分开的SNP位点。除在基因组水平分析寻找相应的SNP标记物外,Pattemore等[23]还通过两个大麦品种的转录组进行序列分析并确定了两个典型大麦品种SNP的数量和分布,生成一组能区分两个品种的特异性SNPs。以上研究表明,大麦基因组中含有丰富的多态性良好的SNP位点,可以用来对品种进行快速有效的鉴定。但以上研究中并未提出基于SNP检测技术鉴定大麦麦芽品种的具体方法,而利用SNP分子标记技术对大麦麦芽纯度的检测也鲜有研究报道,张利莎等[24]利用30 个SNP位点成功对大麦麦芽盲样进行检测,但是实验中并没有提供大麦品种在各个SNP位点的基因型,无法直接用于实际检测。
目前,SNP检测方法虽然有所发展,但通过直接测序法确认的SNP结果往往受测序深度的影响,无法从测序误差中鉴别出杂合子,所以需要更多的重复实验提高准确性[25]。随着测序技术的改进和发展,测序成本越来越低,直接测序法得到越来越多人的青睐[26-27]。Taq man探针与竞争性等位基因特异性PCR(kompetitive allele specific PCR,KASP)技术的出现,不仅使得SNP的检测更为高效、准确,而且操作简单、可重复性高[28-30]。从而也使得基于SNP对品种鉴定的方法得以应用与推广。本研究拟通过测序的方法,筛选并确认出能够区分7 个大麦麦芽品种的SNP位点,进而运用KASP技术对麦芽样品进行定性定量的检测,从而建立了可以用来对麦芽进行定性和定量检测的方法。
1 材料与方法
1.1 材料与试剂
花30、风啤麦4号、风啤麦3号、Sebastian、麦特卡夫、卡普兰德、海德曼斯,由中国食品发酵工业研究院收集。预混样品由麦特卡夫与卡普兰德混合,混合比例为1∶1,并标注为“麦特卡夫”,由中国食品发酵工业研究院提供。
Taq酶、dNTP、10×Loading buffer 大连宝生物技术有限公司;PCR引物合成由生工生物工程(上海)股份有限公司完成;KASP引物 上海艾吉析科技有限公司;植物基因组DNA高效提取试剂盒 北京天根生化科技有限公司。
1.2 仪器与设备
Veriti 96 Well Thermal Cycler、Qubit3.0荧光定量PCR仪北京Thermo Fisher Scientific有限公司;7500实时荧光定量PCR仪 美国ABI公司。
1.3 方法
1.3.1 大麦麦芽基因组的提取
准确称取20 mg液氮研磨的麦芽粉末放入2 mL EP(Eppendorf)管中,按天根高效植物DNA提取试剂盒步骤提取大麦麦芽基因组DNA。提取的基因组DNA采用0.8%琼脂糖TAE凝胶电泳进行质量检测。经0.8%凝胶电泳检测后,条带完整,没有降解。且条带大小处于所需目的片段范围之内。经Qubit3.0荧光定量PCR仪检测后,DNA质量浓度均大于120 ng/μL,完全符合实验所需的DNA质量浓度要求。
1.3.2 SNP位点的筛选
从NCBI上下载大麦基因组序列,通过Visual Genomics软件筛选所需的SNP位点,参数选择默认值。然后,在筛选到的多态性较好的108 个位点上下游300~500 bp位置设计引物,作为扩增的目的片段,扩增片段的引物采用premier 5.0进行设计。然后,以7 种大麦麦芽的基因组DNA为模板,用PCR仪扩增目的片段,PCR体系为20 μL:10×loading buffer 2 μL;上游引物0.5 μL;下游引物0.5 μL;基因组DNA 1 μL(120 ng);Taq酶0.2 μL;ddH2O 13.8 μL。PCR条件:95 ℃预变性5 min;95 ℃变性30 s,52~60 ℃退火30 s,72 ℃延伸30 s,35 个循环;72 ℃延伸7 min。扩增之后的产物送生工生物工程(上海)股份有限公司北京测序部进行测序。测序结果使用MegAlign软件进行比对,确认筛选出的SNP位点,并建立对应7 个品种的SNP位点序列表与品种区分路线图。
1.3.3 KASP引物的验证
根据SNP位点前后的序列信息设计合成KASP引物(表1)。以7 种大麦麦芽品种基因组DNA为模板,利用ABI7500实时PCR仪进行KASP检测。KASP体系:DNA(50 ng/μL)1 μL ,2×master mix 5 μL,双蒸水4 μL,primer mix 0.14 μL。KASP条件:94 ℃预变性15 min;94 ℃变性20 s,61~55 ℃退火、延伸60 s,10 个循环(每个循环降低0.6 ℃);94 ℃变性20 s,55 ℃退火、延伸60 s,26个循环。反应条件的设置以及结果的读取均在7500 Software v2.3软件上进行,对比确认基因型检测的结果是否与一代测序统计结果SNP序列表中数据一致,验证KASP引物的准确性。

表1 KASP引物信息Table 1 Information about KASP primers
1.3.4 预混麦芽样品的定性检测
从预混样“麦特卡夫”中称取100 mg大麦麦芽样品,在研钵中加液氮研磨,称取50 mg放入EP管中。按照1.3.1节的方法提取DNA以及对DNA质量进行检测。以上实验重复2 次,提取3 份样品DNA,以确认实验的准确性。以检测合格的DNA样品为模板,选择验证过的具有较好多态性的SNP位点设计对应的KASP引物进行检测,检测在实时荧光定量PCR仪上进行。此外,每个位点加入对应两种不同基因型的大麦麦芽DNA作为阳性对照,阳性对照样品如表2所示。反应体系与反应条件见1.3.3节,反应条件的设置以及结果的读取均在7500 Software v2.3软件上进行。该KASP实验结果用于对比各SNP位点实测基因型是否与表2中麦特卡夫的基因型的一致性。

表2 定性检测所使用阳性对照Table 2 Positive controls used for qualitative detection in this study
1.3.5 预混麦芽样品定量检测
根据1.3.4节的检测结果,选取杂合基因型的SNP作为定量检测的位点。从“麦特卡夫”样品中随机挑取100 粒大麦麦芽,每粒麦芽按照1.3.1节的方法提取基因组DNA作为定量检测的DNA模板,用杂合基因位点的KASP引物在ABI7500实时PCR仪器上进行检测。反应体系与反应条件如1.3.3节所示,反应条件的设置以及结果的读取均在ABI7500 Software v2.3软件上进行。定量检测的结果以杂合位点上出现的麦特卡夫个数占总反应数的比值计算“麦特卡夫”样品的检测结果。
2 结果与分析
2.1 SNP位点的筛选
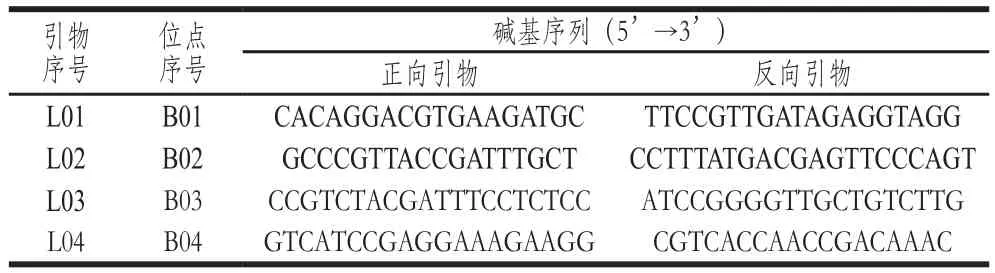
表3 普通PCR引物信息Table 3 Information about the common PCR primers

表4 SNP标记信息Table 4 Information about SNP markers
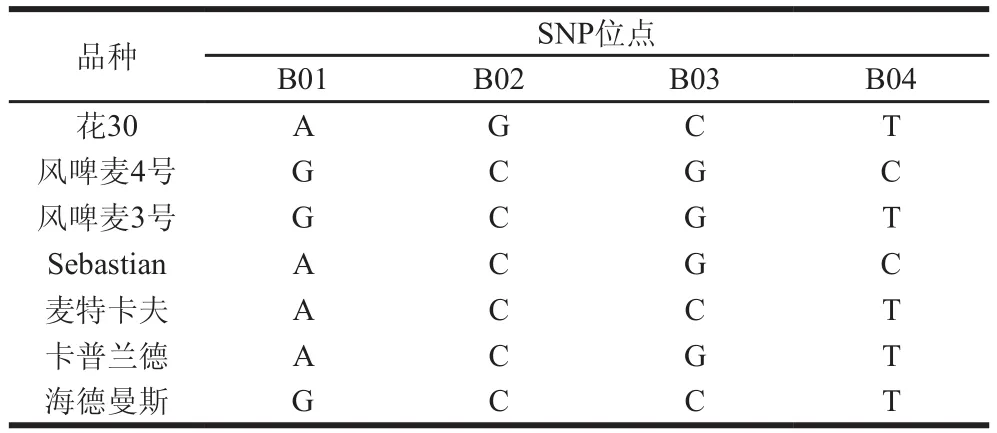
表5 大麦麦芽品种的SNP信息Table 5 SNP data for 7 barley cultivars
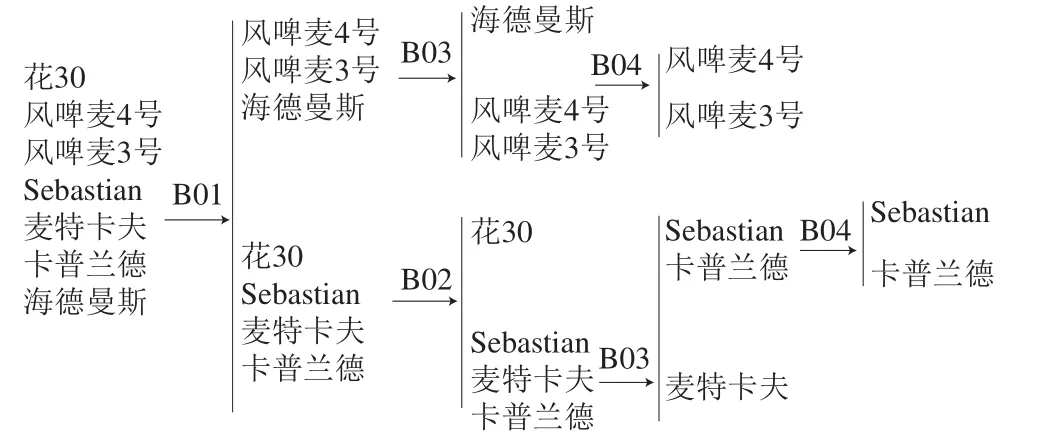
图1 7 个品种基于SNP区分路线图Fig. 1 SNP-based discrimination of 7 varieties
本研究成功筛选4 个多态性良好的SNP位点。用于扩增4 个SNP位点的普通PCR引物信息如表3所示,对应的SNP位点信息如表4所示。同时,并根据不同麦芽品种在各位点的碱基差异建立7个麦芽品种的SNP位点数据表(表5)及样品纯度鉴定的图谱(图1)。
2.2 KASP引物的验证
以7 个大麦麦芽品种的基因组DNA为模板,利用每个位点对应的KASP引物在7500实时荧光定量PCR仪上进行基因分型的检测,以此确认位点以及KASP引物的准确性。SNP基因型检测结果如图2所示。B01位点的引物验证结果(图2a)表明:风啤麦4号、风啤麦3号、海德曼斯的检测的位点结果为GG;花30、Sebastian、麦特卡夫、卡普兰德的检测的位点结果为AA。B02位点引物验证结果(图2b)表明:花30检测结果为GG;风啤麦4号、风啤麦3号、Sebastian、麦特卡夫、卡普兰德以及海德曼斯的检测结果为CC。B03的引物验证结果(图2c)表明:花30、麦特卡夫、海德曼斯检测结果为CC;风啤麦4号、风啤麦3号、Sebastian、卡普兰德检测结果为GG。B04号位点的引物验证结果(图2d)表明:风啤麦4号、Sebastian检测结果为CC;花30、风啤麦3号、麦特卡夫、卡普兰德、海德曼斯检测结果为TT。实验结果表明,4 个位点的KASP引物对7 个品种进行SNP检测结果与一代测序结果一致,表明SNP位点和KASP引物的正确性,位点和引物可用于后续的检测。
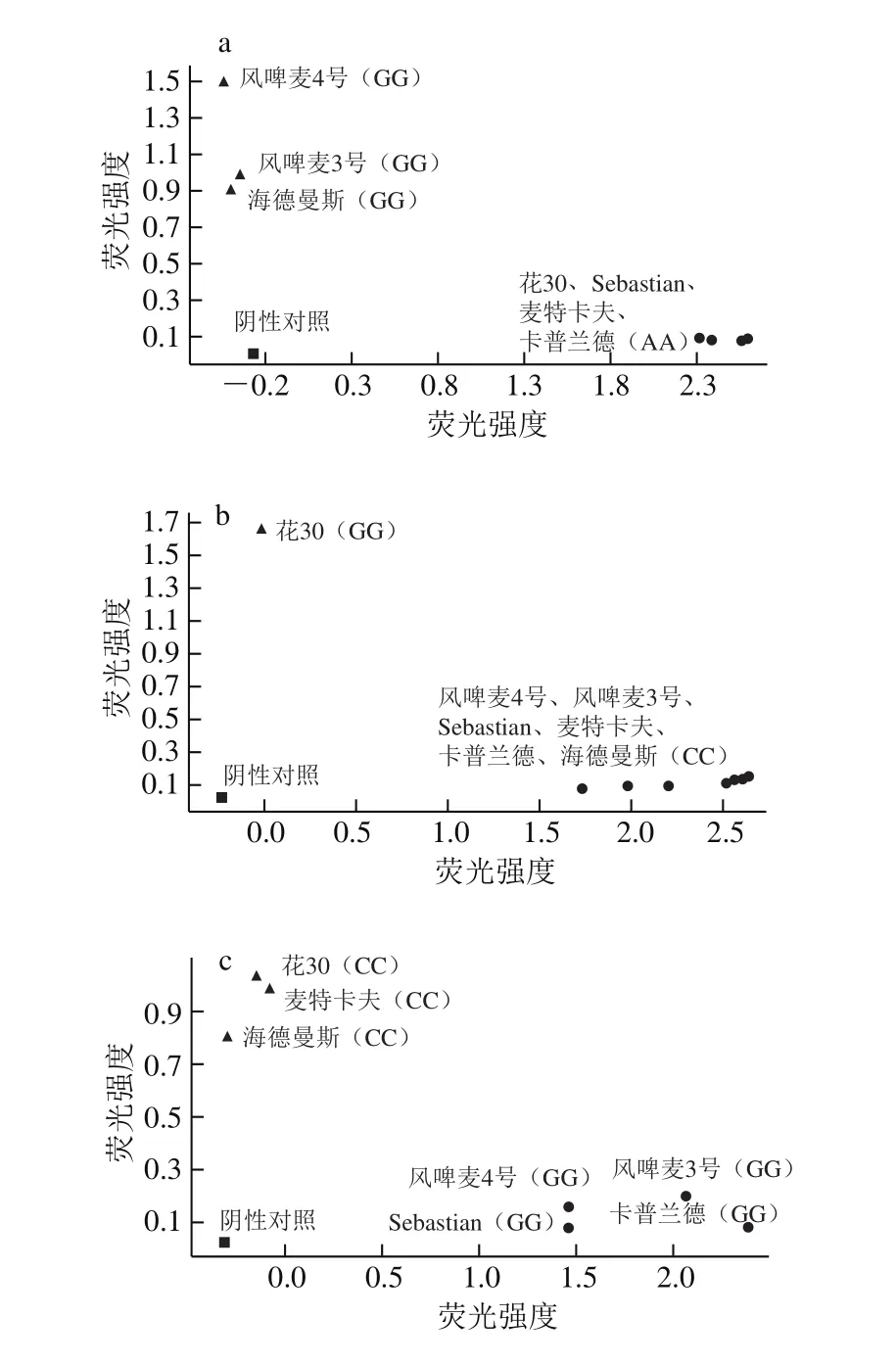
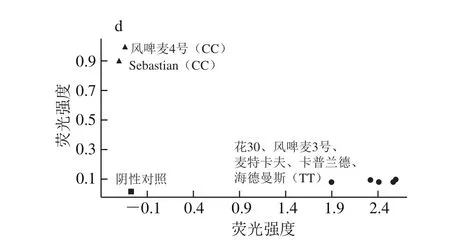
图2 B01(a)、B02(b)、B03(c)、B04(d)位点KASP引物验证结果图Fig. 2 Results of KASP primer validation at B01 (a), B02 (b), B03 (c), and B04 (d)
2.3 预混麦芽样品的定性检测结果
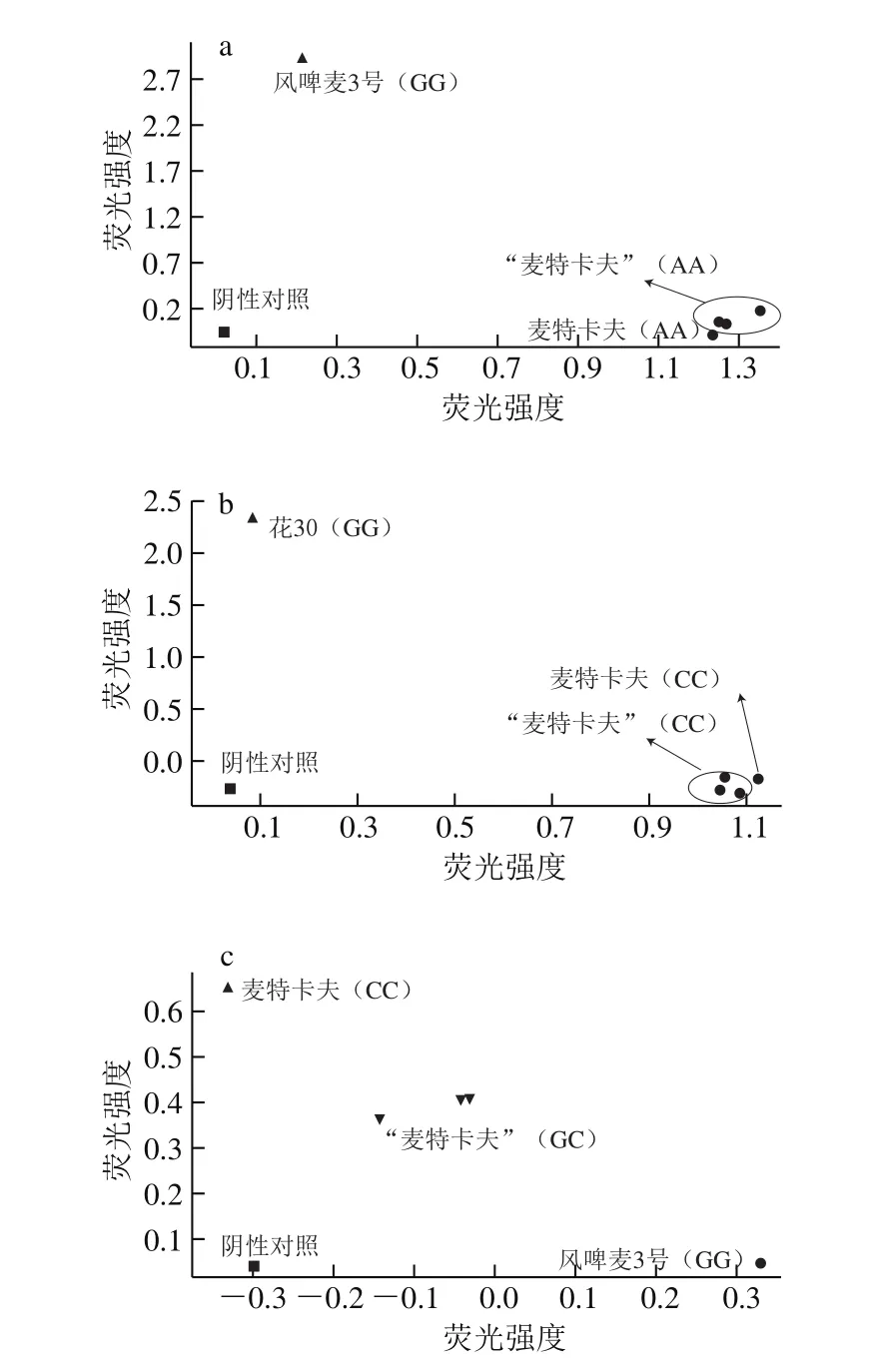
图3 B01(a)、B02(b)、B03(c)位点样品基因型检测结果Fig. 3 Qualitative detection of B01 (a), B02 (b), and B03 (c)SNP markers
图3 a表明,3份“麦特卡夫”样品DNA的检测结果为AA。图3b表明,阳性对照麦特卡夫与3份“麦特卡夫”样品DNA检测结果相同,为CC。图3c表明,3份“麦特卡夫”样品检测结果为GC。统计3 个位点的检测结果与麦特卡夫标准品在这3 个SNP位点基因型做比较,见表6。B01、B02位点检测结果与麦特卡夫一致,B03位点出现了杂合,说明该SNP标记在“麦特卡夫”混合样中具有多态性,能够用于后续麦芽混合样的定量检测。
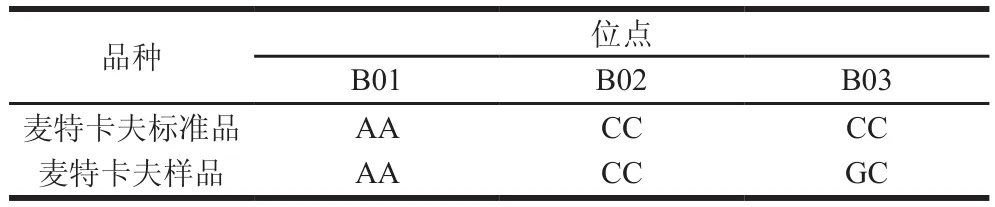
表6 麦特卡夫样品与麦特卡夫标准品在SNP位点的检测结果比较Table 6 Comparison of the results for Metcalf and Metcalf standards with three SNP markers
2.4 预混麦芽样品的定量检测结果
由定性检测结果可知B03位点为杂合位点,可用于定量检测。在检测的100 粒大麦麦芽DNA样品中,48.6%的样品基因型在B03位点上是G(舍去少量未成功检测的DNA样品),即 “麦特卡夫”麦芽样品纯度为48.6%(图4),与预混样真实值50%之间的误差小于3%。
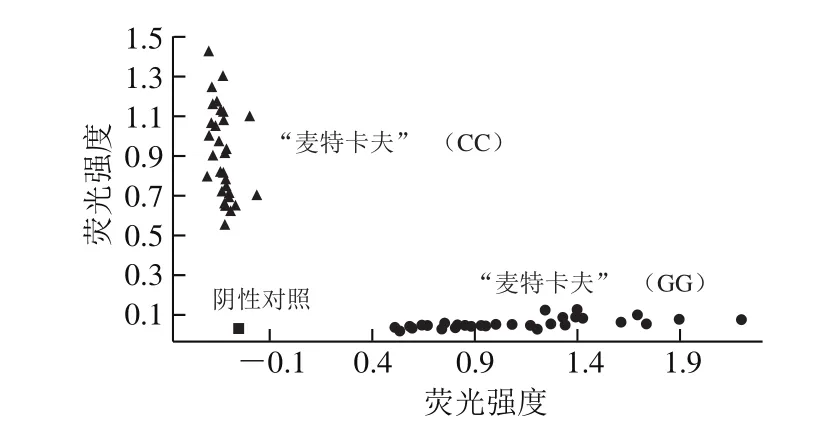
图4 B03位点“麦特卡夫”定量检测结果Fig. 4 Quantitative detection of Metcalfe using B03 SNP marker
3 结 论
本研究利用生物信息学方法,筛选并鉴定出4 个能够区分7 个大麦麦芽品种的SNP位点。同时,合成了KASP引物,并验证了KASP引物的正确性。结合KASP基因分型技术,对预混麦芽样品“麦特卡夫”样品进行检测。检测结果表明该样品为混合样品,“麦特卡夫”的纯度为48.6%。检测的效率与准确度能够满足啤酒企业的需求,该方法为后续大麦麦芽品种的SNP分子标记鉴定方法的建立提供了新的实验思路以及数据库信息,为啤酒企业对原料质量的控制提供了可实际应用的方法。
