用单分子技术研究Sso7d与DNA的相互作用∗
2018-10-29滕翠娟陆越马建兵李明陆颖徐春华
滕翠娟陆越马建兵李明陆颖 徐春华†
1)(中国科学院物理研究所,北京凝聚态物理国家研究中心,软物质物理重点实验室,北京 100190)2)(中国科学院大学物理科学学院,北京 100049)
(2018年4月9日收到;2018年4月21日收到修改稿)
为了维持基因的稳定性,每种生物体都含有一套独特的染色质蛋白来保护脱氧核糖核酸(DNA)的结构,观察染色质蛋白对DNA结构的作用过程和结果,可以帮助人们了解这些蛋白的具体功能和作用机理.硫化叶菌是一种能在高温下存活的古细菌,Sso7d是硫化叶菌的一种染色质蛋白.深入地了解Sso7d和DNA链的相互作用,有助于解释硫化叶菌的DNA为何能在高温环境下保持活性,本文通过原子力显微镜(AFM)和磁镊两种单分子操作手段,研究了Sso7d与DNA的相互作用.AFM的实验结果给出了Sso7d与DNA的作用过程:结合Sso7d后,DNA首先发生弯折,然后出现loop结构,最终DNA会团聚为致密的核结构.利用磁镊装置测量了Sso7d的结合对打开DNA双链的影响,实验结果表明Sso7d的结合导致打开DNA双链的力的增大,经过数据分析,计算出Sso7d与DNA结合的结合能∆G=3.1kBT,平均每5.5个碱基对(bp)结合一个Sso7d,较高的结合密度和较大的结合能,两方面的作用结果,解释了Sso7d能够稳定DNA结构的原因.
1 引 言
在生物体内,脱氧核糖核酸(DNA)的解旋、转录和复制等一切活动都是在各种相关蛋白质的调控下进行的.其中染色质蛋白在一切生物体内都是必不可少的,其在基因的结构和功能的调节中具有重要作用,尤其是能够有效地折叠DNA,保证基因结构的稳定[1].根据16S核糖体核糖核酸(RNA)的不同,将生物分为三个域[2],分别为细菌(bacteria)、真核生物(eukarya)和古细菌(archaea).在真核生物中,DNA在组蛋白(histone)的作用下缠绕成DNA的基本结构单元——核小体[3].在原核生物体内,存在很多DNA结合蛋白,如HU,IHF,H-NS,Fis和Lrp等[4,5],在这些蛋白的作用下,染色质DNA被折叠加工为一个致密的结构,称之为拟核.在古细菌中,染色质蛋白的情况也是相当复杂的[6].古细菌两个最大的门:广古菌(Euryarchaeota)和泉古菌(Crenarchaeota),其中的广古菌,存在类似histone的染色质蛋白[7,8];而泉古菌中,没有这种染色质蛋白,取而代之的是一系列分子量比较小的染色质蛋白,如Alba,Sul7d,CC1和Cren7等[9,10].其中的Sul7d存在于硫化叶菌目(Sulfolobales)中,是一系列分子量为7 kDa的染色质蛋白,本文研究的是其中的Sso7d.
在硫化叶菌(Sulfolobus solfataricus)中,Sso7d是一种大量存在的染色质蛋白,其占细胞中蛋白质总量的5%[11],近年来对Sso7d的研究已逐步深入.Sso7d由63个氨基酸组成[12],分子量为7 kDa[13].在溶液中,Sso7d以单体形式结合在双链DNA(dsDNA)上[12,14],每个Sso7d分子可以结合4—6个碱基对[15].Sso7d的结合可以增加DNA的负超螺旋[16,17],并且能够使DNA的熔解温度提高30.6◦C[18],其人工突变体能够承受更高的熔解温度和pH值范围[19].Sso7d现在经常作为拼接蛋白连接DNA和其他有机分子[20],还可以作为支架蛋白把多个蛋白连接为一个功能性的复合体,并促进其互相作用[19,21].更加深入地掌握Sso7d和DNA的作用机理,可以更好地发挥Sso7d在这些系统构建中的功能.Sso7d与小片段DNA(8 bp,bp为碱基对)结合后的复合物的晶体衍射结果显示,Sso7d结合在DNA小沟上,通过嵌入DNA双螺旋使DNA产生60◦的弯折[22,23].目前,对于较长片段的DNA与Sso7d的结合状态,有电子显微镜和原子力显微镜(atomic force microscope,AFM)的结果[12,24],但并没有直观地给出Sso7d与DNA的作用过程.本文采用AFM研究Sso7d的结合对DNA形态的影响,观察不同浓度Sso7d条件下较长片段DNA与Sso7d结合后的结构图像,分析得出DNA的团聚过程;用单分子磁镊研究Sso7d的结合对打开DNA双链的影响,测定了不同Sso7d浓度下打开DNA双链的力的大小,计算得到了Sso7d和DNA结合的结合能.
2 实验材料与方法
2.1 蛋白质及反应溶液
染色质蛋白Sso7d,由中国科学院微生物研究所的黄力研究员提供,制备方法参考文献[25,26].Sso7d溶于保存溶液中,具体成分为10 mmol/L Hepes(pH值7.6,25◦C),0.2% 牛血清白蛋白(BSA),0.1%Tween-20,25 mmol/L NaCl,−20◦C冰箱保存.磁镊实验中,反应溶液和Sso7d的保存溶液相同.AFM实验中,为避免BSA对成像的影响,在制作样品过程中所用的DNA与Sso7d反应的溶液中不含有BSA.
2.2 DNA的制备
实验中使用了三种不同的DNA以实现不同的实验目的:一是总长度为3000 bp的直链DNA(DNA1);二是含有1200 bp的直链DNA(DNA2);三是含有发卡(hairpin)结构的总长度为3000 bp的DNA(DNA3).DNA1和DNA2直接由聚合酶链式反应(PCR)制得.DNA3的制备可参考文献[27],DNA3含有一个hairpin结构和两个dsDNA手柄.其中,hairpin结构含有120 bp,两个手柄DNA长度分别为2300和700 bp.
2.3 DNA-Sso7d结合物的制备
在反应溶液(10 mmol/L Hepes,pH值 7.6,25 mmol/L NaCl)中,终浓度为0.5 nmol/L的DNA1分别与终浓度为0.5,1.0,3.0µmol/L的染色质蛋白Sso7d混合均匀,室温下反应5 min,分别得到DNA:Sso7d质量比为18:7,9:7,3:7的DNA1-Sso7d反应产物;之后,加入与各产物溶液等体积的戊二醛固定液(10 mmol/L Hepes,pH值7.6,25 mmol/L NaCl,0.8%戊二醛),固定20 min;最后,用AFM制片溶液(10 mmol/L Tris-HCl,pH值 7.4,10 mmol/L MgCl2)稀释上述所得各溶液到合适的浓度,以备滴至云母片制作样品.DNA2-Sso7d复合物的制备方法同上.
2.4 AFM及其样品的制备
实验所用的AFM是Bruker公司的高分辨AFM Multimode 8.扫描模式为scanasyst,扫描探针型号为scanasyst-air,扫描速率为0.977 Hz.待扫描样品的准备过程为:取12µL待测已稀释到合适浓度的DNA与Sso7d的反应溶液,滴到干净的云母片(1 cm×1 cm)上,孵育5 min;之后用4 mL的超纯水冲洗云母片以去除盐离子和未吸附到云母片上的DNA-Sso7d复合物;最后,用氮气缓慢吹干云母表面.
2.5 磁 镊
实验所用磁镊为实验室搭建的纵向磁镊装置,采样频率为20 Hz.实验中所用DNA为DNA3.首先,DNA和顺磁性小球(Invitrogen,Dynabeads M-280,表面Streptavidin修饰)按1:10的摩尔比混合,室温缓慢旋转混匀5 min;然后,向反应体系中加入磷酸缓冲液稀释至适当浓度,注入磁镊样品槽,孵育5 min;最后,通过向样品槽中缓慢加入磷酸缓冲液,冲去游离的顺磁性小球和未连接的DNA,即完成目标单分子连接的构建.
3 实验结果与讨论
3.1 Sso7d结合导致的DNA链凝聚的作用过程
DNA1分别与不同浓度的Sso7d溶液反应5 min后,用戊二醛固定液固定20 min,然后用AFM 制片溶液稀释,得到终溶液,分别滴到云母片上制得待扫描样品.从AFM图像上可以看出,反应时间设定为5 min时,当Sso7d:DNA的质量比(7:18)比较低时,如图1(a)所示,DNA上出现很多弯折点,弯折点处结合了Sso7d.随着Sso7d:DNA质量比(7:9)的提高,DNA在Sso7d的作用下,凝聚出一个比较紧密的小核,如图1(b)所示.当Sso7d:DNA质量比(7:3)进一步提高,如图1(c),整条DNA凝聚为致密的核结构,此反应条件下,剩余的自由游离的DNA链变得很少.
为了进一步研究Sso7d与DNA的结合状态是否随时间发生变化,本文做了另一组对照实验:其他反应条件同上,只改变反应时间,将Sso7d和DNA1的反应时间设定为60 min.图1(d)显示Sso7d:DNA的质量比为7:18时,反应60 min后,DNA1-Sso7d复合物的构象图,从图中可以看出DNA出现loop结构.当Sso7d:DNA的质量比为7:9时,反应60 min后,DNA1-Sso7d复合物的构象图如图1(e)所示,可以看到更多的loop结构,而且DNA进一步团结在小的凝聚核周围.当Sso7d与DNA的质量比达到7:3时,反应60 min后,如图1(f)所示,DNA完全凝聚为致密的核结构,无游离的自由DNA链.从实验结果可以看出,整体而言,在Sso7d:DNA质量比相同的条件下,和5 min反应时间的结果相比,反应时间延长之后,在染色质蛋白Sso7d的作用下,DNA变得更加致密.
综合以上结果,可以看出,DNA的凝聚成核过程始于Sso7d的作用导致的DNA的弯折,随着反应时间的延长,loop出现;之后DNA在Sso7d的作用下凝聚出一个致密的小核,其余游离的DNA链分布在小核周围,直至整条DNA凝结为较为致密的核结构.而且,高浓度的Sso7d才能够使DNA高度凝聚,这说明硫化叶菌中Sso7d的含量很高[11]是很有必要的.
Loop结构的出现是DNA的凝聚过程中十分重要的环节.由于DNA1-Sso7d复合物的结构较复杂,若DNA太长则不易看清细节.因此,本文选用一条较短的DNA链(DNA2,1200 bp)做了Sso7d与DNA相结合的实验,其AFM图像如图2所示,更加清楚地展示了DNA loop的形成过程.实验中,Sso7d和DNA的质量比为7:18,反应时间分别设定为5 min和60 min.反应5 min后,DNA构象如图2(b)和图2(c)所示,DNA链出现不同程度的弯折;反应60 min后,DNA构象如图2(d)所示,弯折更加明显,并出现loop结构.整个反应过程解释为:Sso7d结合在DNA上,使DNA发生弯折;又由于DNA带负电,Sso7d带正电,Sso7d的结合中和了DNA的部分电荷,DNA的静电排斥变弱,DNA变得更容易弯曲;弯曲的DNA链相互靠近,结合在DNA上的Sso7d之间可能发生相互作用导致了loop结构的出现,从而促进DNA的进一步凝聚.
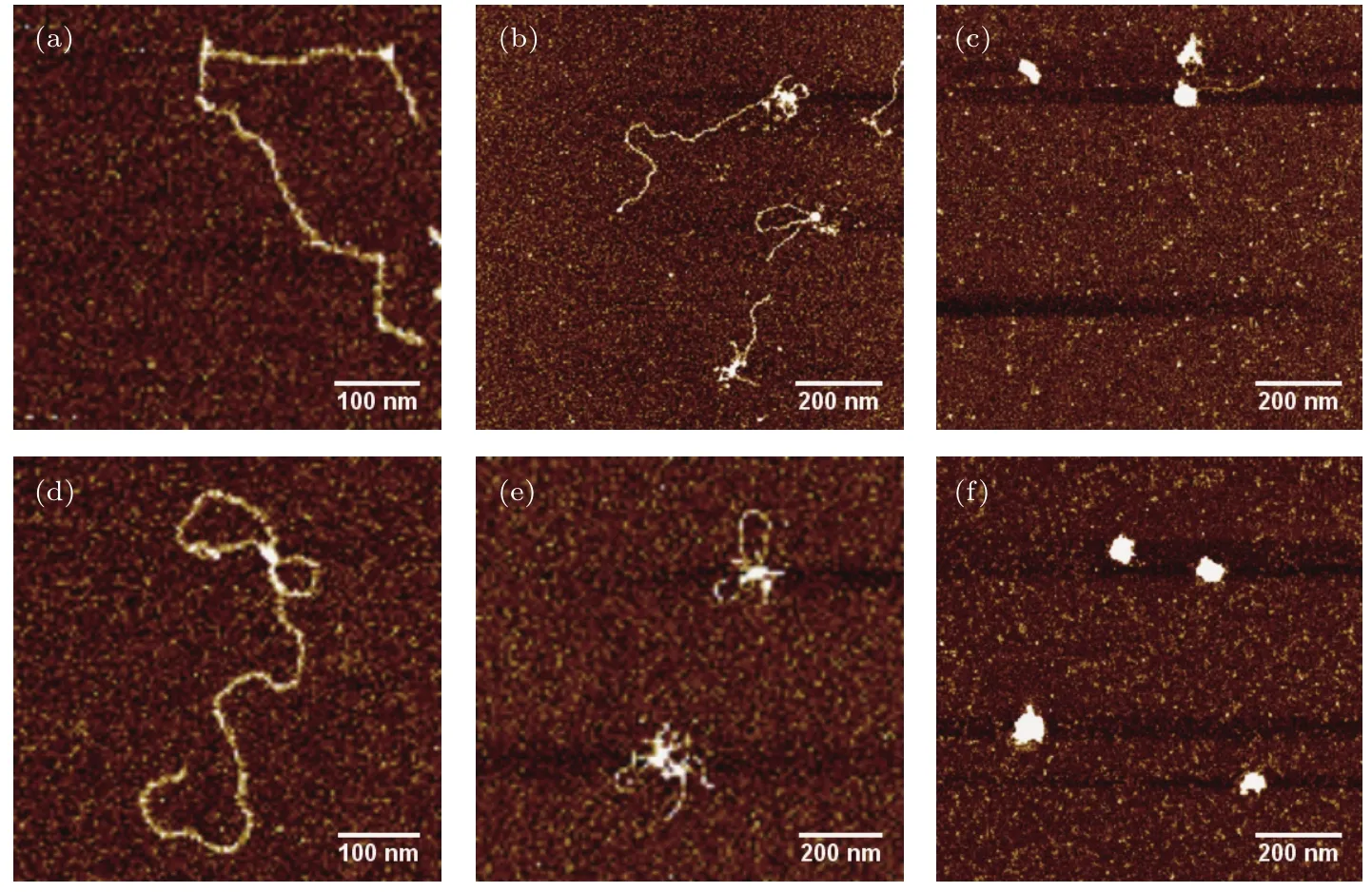
图1 3000 bp DNA1与不同浓度的Sso7d溶液作用后的构象图 (a)0.5µmol/L Sso7d,5 min;(b)1µmol/L Sso7d,5 min;(c)3 µmol/L Sso7d,5 min;(d)0.5 µmol/L Sso7d,60 min;(e)1 µmol/L Sso7d,60 min;(f)3µmol/L Sso7d,60 minFig.1.Conformations of 3000 bp DNA1 reacted with Different Sso7d concentration:(a)0.5µmol/L Sso7d,5 min;(b)1µmol/L Sso7d,5 min;(c)3µmol/L Sso7d,5 min;(d)0.5µmol/L Sso7d,60 min;(e)1µmol/L Sso7d,60 min;(f)3µmol/L Sso7d,60 min.

图2 1200 bp的DNA2及其与0.5µmol/L的Sso7d溶液作用不同时间后的构象图 (a)1200 bp DNA2;(b),(c)5 min;(d)60 minFig.2.Conformations of 1200 bp DNA2 with 0.5µmol/L Sso7d reacted at Different time:(a)1200 bp DNA2;(b),(c)5 min;(d)60 min.
3.2 Sso7d使DNA弯折的角度测量
在图1(a)和图2(b)中,DNA被Sso7d结合后发生弯折,关于Sso7d引起的DNA的弯折角度的大小,有相关文献报道.有核磁共振实验得到了Sso7d引起的DNA弯折角度为30◦[28],但是在核磁共振实验中,溶液环境下DNA在结合态和自由态之间的切换很容易导致弯折角度测量结果出现偏差;另外有文献得到了由Sso7d与DNA相互作用后的结晶结构,测得Sso7d使DNA弯折60◦[22,23],仍有研究对此表示质疑,其认为在晶体衍射实验中,Sso7d-DNA复合物只能被固定为一种趋向于形成晶体结构的构象,并不能反映其在溶液中的真实状态[24].在AFM实验中,Sso7d和DNA在液体环境中相互作用,之后其复合物构象被戊二醛固定,因此通过AFM图像能够准确地反映其在液体环境中的状态,更加真实地反映其生理状态.本文对AFM结果中的DNA的弯折角度Φ进行了测量统计,如图3所示,通过高斯拟合,得到弯折角度值为58.5◦.
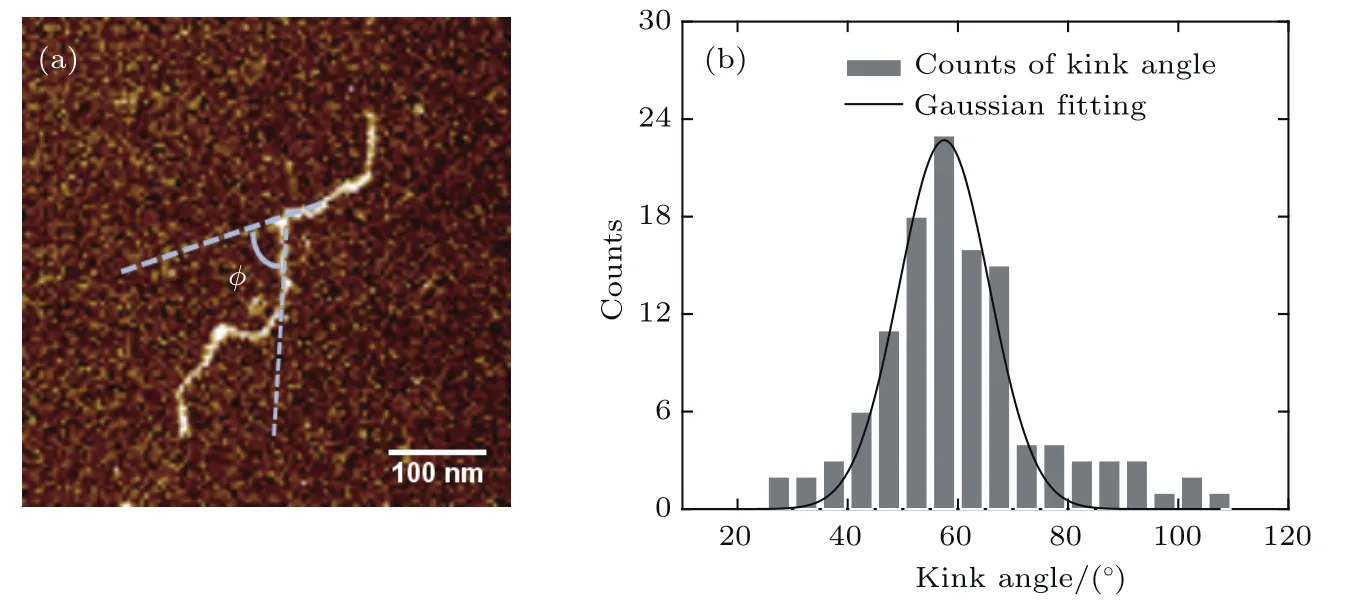
图3 DNA弯折角度测量 (a)DNA弯折角度测量示意图;(b)弯折角度统计分布图和高斯拟合曲线Fig.3.Measurement of the DNA kink angle induced:(a)Schematic diagram of DNA kink angle measurement;(b)histogram of the counts of kink angle and Gaussian fitting curve.
3.3 Sso7d的结合对打开DNA双链的影响
硫化叶菌中的Sso7d含量极高,占其细胞内蛋白质总重量的5%[11],如此高浓度的Sso7d,足以将DNA团聚为如图1(f)所示的紧致结构,使DNA变得相当稳定.有DNA热熔解实验显示,在DNA溶液中加入一定量的Sso7d,可以使DNA的熔解温度提高30.6◦C[18].可见,染色质蛋白Sso7d可以提高dsDNA的热稳定性.因此,本文预测结合染色质蛋白Sso7d后,会提高DNA双链的结合能,此结合能的提高可以直观地表现为打开DNA双链所需要的力的变化.而打开DNA双链的力可以通过设计磁镊实验进行测量.
本文所用磁镊装置示意图如图4(a)所示,所用DNA底物是DNA3,其含有一段120 bp的hairpin结构.图4(b)举例说明了实验过程中所施加磁力的变化过程以及DNA的拉伸曲线.
对于所用的120 bp hairpin DNA,在本文实验条件下,不含有染色质蛋白的情况下,当施加在磁性小球上的拉力增大到约14 pN时,hairpin被完全打开.随着加入Sso7d浓度的提高,打开hairpin所需要的拉力逐渐增大.如图4(c)所示,当Sso7d浓度达到1µmol/L时,打开hairpin的力的大小为19 pN时,之后再提高Sso7d的浓度,打开hairpin的力不再变化.
实验测量了hairpin被打开成为单链的长度,此长度随Sso7d浓度的变化而发生变化,如图4(d)所示.当Sso7d的浓度从0开始逐渐增大时,hairpin的打开长度逐渐增大,其原因是随着Sso7d浓度的增大,打开hairpin的力在增大,碱基之间的距离随力的增大而增大,从而导致整个hairpin打开成单链后的长度增大.但是,当Sso7d的浓度进一步增大时,hairpin打开后的长度反而缩小,此时可能是因为hairpin变为单链后仍有Sso7d结合在上边导致其长度缩短.
结合能是表征结合强度的物理参量,平衡常数能表征化学反应进行的最大程度,对实验所得的120 bp的hairpin在不同浓度的Sso7d的条件下打开hairpin的力进行分析,可以得到Sso7d与DNA反应的结合能和平衡常数两个表征量.
Sso7d以单体的形式结合在DNA上,尚未发现Sso7d的结合具有协同效应,因此假设每个Sso7d的结合之间不存在协同作用,将Sso7d与DNA的逐个结合反应表示为

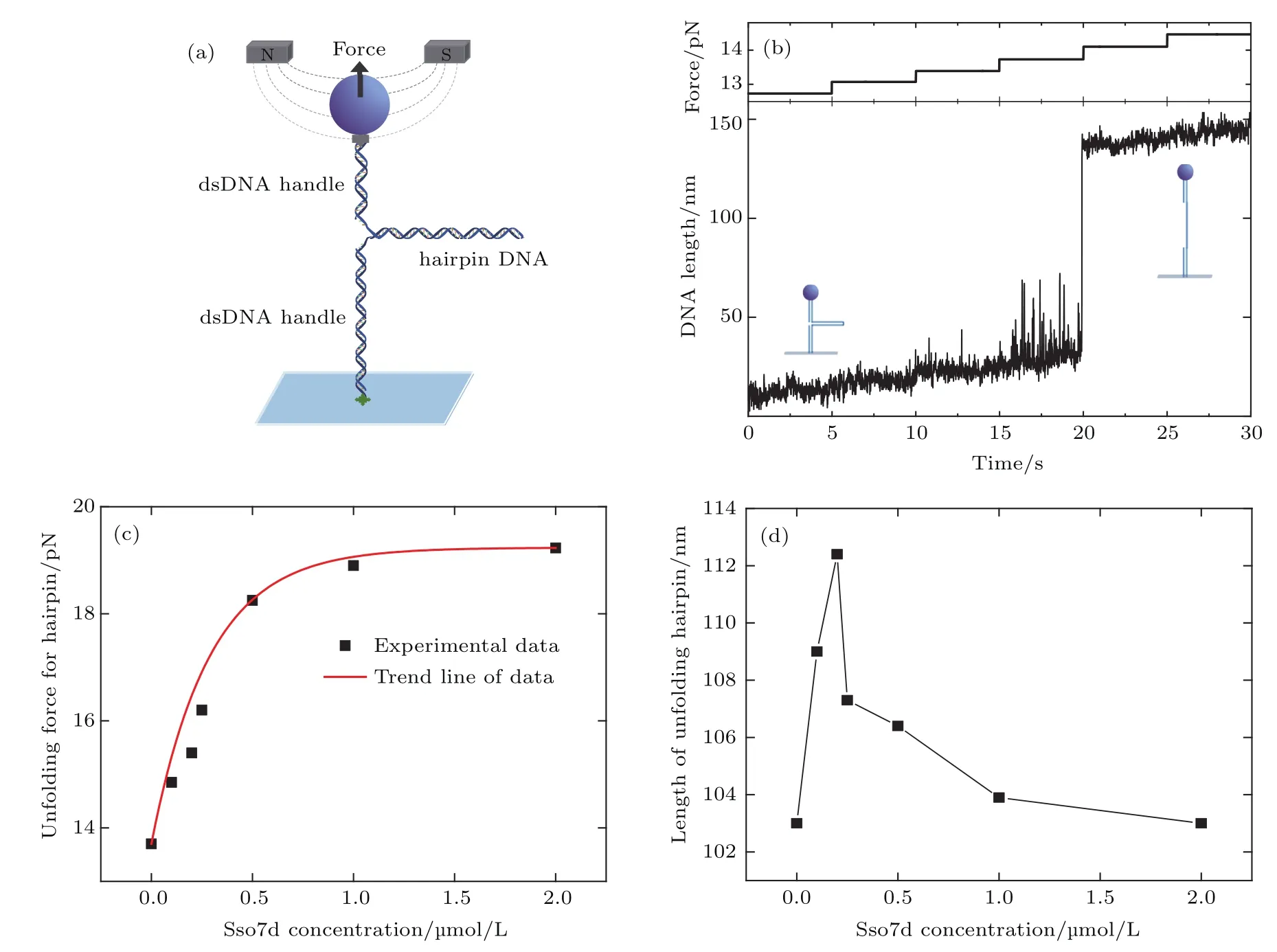
图4 Sso7d对打开dsDNA的影响 (a)实验示意图;(b)hairpin被打开的实验曲线;(c)打开hairpin的力随Sso7d浓度的变化;(d)不同浓度Sso7d条件下,hairpin被打开为单链的长度Fig.4.Influence of Sso7d on dsDNA:(a)Schematic diagram of the experiment;(b)example curves of hairpin unfolding;(c)variation of hairpin unfolding force with Sso7d concentrations;(d)length of unfolded hairpin DNA at Different Sso7d concentrations.

Sso7d与hairpin的反应体系达到稳态时,正逆反应速率相等,反应的每个组分所占的比例不变,形成动态平衡过程.从DNA与不同浓度Sso7d结合后的电泳实验数据[16,29]以及本文实验数据都可以看出,对于不同的Sso7d浓度,hairpin上结合的Sso7d数量是不一样的,这个现象可以用达到平衡时各DNA-Sso7d复合物之间的浓度比例来描述.对于反应方程式(1)存在如下关系:
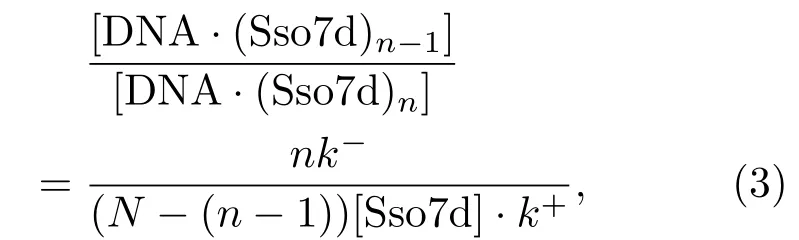
[DNA·(Sso7d)n−1]和[DNA·(Sso7d)n]分别对应达到动态平衡时结合n−1个Sso7d和结合n个Sso7d的DNA-Sso7d复合物浓度.从(3)式中可以发现整个DNA-Sso7d复合物体系的浓度变化规律:对于某个特定的Sso7d浓度,随着Sso7d结合个数n的增加,在某个n值,也就是对应着某种特定的DNA-Sso7d复合物时,其平衡浓度所占的比例最大,在实验中就表现出结合了相应的蛋白数量.这就是为什么在不同Sso7d浓度下,可以在hairpin上观察到不同的Sso7d结合数量的原因.
对于结合第N个Sso7d(饱和)的状况,存在如下关系:
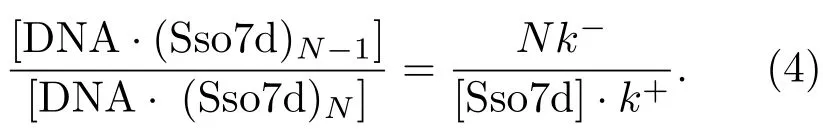
随着Sso7d浓度的增大,结合在hairpin上的Sso7d数量增多,当Sso7d浓度大于某一临界值时,结合在DNA上的Sso7d的数量达到饱和,此时
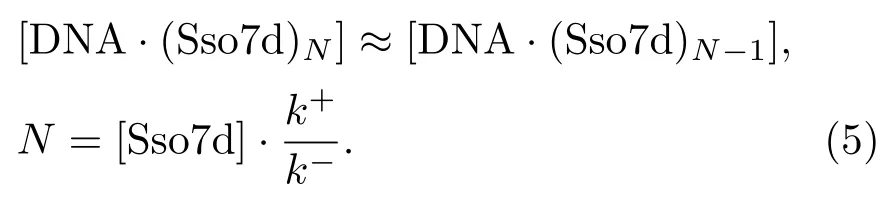
从打开hairpin的力随Sso7d浓度的变化的实验曲线图4(c)可以看出,当Sso7d浓度高于1µmol/L时,打开hairpin的力几乎不再增大.这也就是说,当Sso7d浓度为1µmol/L时,Sso7d的结合基本达到饱和,因此,(5)式成立时[Sso7d]=1µmol/L.
由于Sso7d的结合导致打开hairpin的力变大,Sso7d与DNA的结合能与打开hairpin的能量的变化相等,根据贝尔公式[30],可以得到如下关系:
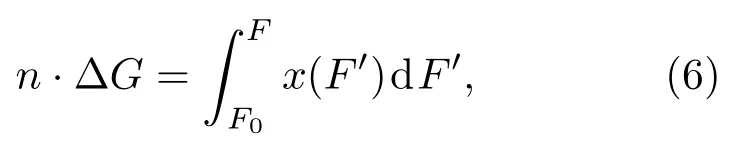
式中F0为未加入Sso7d时打开hairpin的力的大小,在本文实验条件下为13.7 pN;F为加入Sso7d后打开hairpin的力的大小;x(F′)表示拉力为F′时打开hairpin所需的反应长度变化;n为结合在hairpin上的Sso7d个数;∆G为单个Sso7d结合的能量.
图1(e)和图2(d)所用DNA:Sso7d质量比相同,对比两图可知,DNA越长,凝聚越明显,因此推测,短DNA不易凝聚.由于hairpin的长度较短,故假设hairpin没有凝聚,不考虑除单个Sso7d与hairpin结合之外的其他能量.由此可以得出特定Sso7d浓度下的结合总能量关系.
由前文分析可知,当Sso7d浓度为1µmol/L时,Sso7d的结合达到饱和,此后的hairpin打开拉力也几乎不变,大小为19 pN.因此可以计算出对于饱和Sso7d浓度,N·∆G=68.1kBT.将这一结果与前面得到的蛋白浓度和化学反应系数的关系(5)式联立,根据阿伦尼乌斯关系可以得到

Keq为化学反应的平衡常数;ω0为由尝试频率推导得出的参数,与化学反应类型有关[31−33],此处可以取值为106(mol/L)−1.利用实验所得的打开hairpin的力的值以及结合达到饱和时Sso7d的浓度值,可以计算出一个Sso7d与hairpin的结合所产生的结合能∆G=3.1kBT,平衡常数Keq=2.2×107(mol/L)−1,还能计算出120 bp hairpin可以结合Sso7d的个数N=22,平均每个Sso7d占据DNA5.5个碱基对的位置,这一结果与DNA-Sso7d复合物的结晶结构解析结果相一致[22],验证了本文的推导和假设是成立的.从Sso7d与hairpin的结合所产生的结合能可以看出,每一个Sso7d的结合能都很可观,而生物体内大量的Sso7d足以将DNA完全包被,每一个Sso7d与DNA的结合都会引入同等大小的结合能,这从结合能的角度解释了Sso7d为何能够提高DNA的熔解温度以及硫化叶菌如何在高温下维持其基因的稳定.
4 结 论
1)通过对比不同反应条件下生成的DNASso7d复合物的AFM图像,看到DNA在Sso7d作用下逐步凝聚的过程:Sso7d和DNA的相互作用,首先表现为DNA的弯折,然后会产生loop结构,以此为基础进一步高度凝聚.其中loop结构的形成过程也被清晰地观测到.
2)测量了结合Sso7d后的DNA链的弯折角度,高斯拟合结果为58.5◦,与核磁共振成像和晶体衍射成像方法相比,更加真实地反映了生理状态下Sso7d和DNA的结合状态.
3)用磁镊方法得到了Sso7d的结合对打开DNA双链的力的影响,数据分析得到一个Sso7d结合在DNA双链上所产生的结合能∆G=3.1kBT,解释了Sso7d能够提高DNA的熔解温度的原因以及硫化叶菌如何在高温下维持基因的稳定.
以上实验结论,解释了Sso7d对DNA的保护机理,这给硫化叶菌生活在高温环境下而核酸却能够保持稳定提供了可能的条件.近年来,Sso7d的突变体经常以脚手架的形式出现在大分子系统中,深入地研究Sso7d与DNA的相互作用过程,为精准地设计和构建Sso7d的功能提供了理论基础.
感谢中国科学院微生物研究所黄力研究员提供实验所用蛋白Sso7d.
