基于COⅠ基因的中国广头叶蝉亚科系统发育初探
2018-10-29李虎戴仁怀
李虎, 戴仁怀
(1. 陕西理工大学,陕西省资源生物重点实验室,陕西汉中723000; 2.贵州大学昆虫研究所,贵州山地农业病虫害重点实验室,贵阳550025)
广头叶蝉亚科Macropsinae隶属于半翅目Hemiptera头喙亚目Auchenorrhyncha角蝉总科Membracoidea叶蝉科Cicadellidae,广泛分布于世界各地。目前全世界已报道该亚科约19属750余种,我国记录7属108种。广头叶蝉大多取食木本植物,为重要的生态、经济林木害虫。目前已知的寄主主要有蔷薇科Rosaceae、胡颓子科Elaeagnaceae、榆科Ulmaceae、壳斗科Fagaceae、小檗科Berberidaceae(Tishechkin,1994,1999,2002)、杨柳科Salicaceae和桦木科Betulaceae等植物(Lietal.,2012,2013,2014);也有报道广头叶蝉取食桑科Moraceae植物(魏重生,蔡平,1998)。
广头叶蝉亚科分类系统较混乱,部分族属级分类地位一直存在争议。Evans(1937)最先提出广头叶蝉科Macropsidae概念,随后又降为亚科(Evans,1946),包含Macropsini和Nioniini两族;Linnavuori(1978)将Nioniini提升为亚科Nioniinae,同时将其中的Neopsis属提升为亚科级Neopsinae,但Hamilton(1983)仍以族级水平对待;Dietrich(2005)根据形态特征恢复Neopsinae。在属级水平上,横皱叶蝉属Oncopsis和斜皱叶蝉属Pediopsis初为Bythoscopus的亚属,后分别被提升为属,但包括广头叶蝉属Macopsis、暗纹叶蝉属Pediopsoides、Galboa、Stenoscopus在内早期均属于短头叶蝉科Bythoscopidae,后被移入广头叶蝉亚科(Evans,1937)。Hamilton(1980)依据外形及雄虫外生殖器特征将古北区特有属Macropsidius降为亚属,但Tishetshkin(2006,2007,2014)、Yang和Zhang(2015)均以属级水平对待;在Hamilton(1980)的分类系统中,将Parasitades、Nanopsis、Kiamoncopsis也降为亚属,但也存在争议。
为了进一步掌握广头叶蝉亚科属种间的分子差异程度和系统发育关系,本文选择昆虫分子系统学研究中常用的线粒体细胞色素c氧化酶亚基Ⅰ(COⅠ)为标记基因,对中国常见的广头叶蝉亚科5属22种进行COⅠ基因序列扩增,研究目标序列的序列组成和遗传距离特征,并构建中国广头叶蝉亚科系统发育框架,研究结果可为整个亚科的系统发育构建及物种的快速鉴定提供一定的参考依据。
1 材料与方法
1.1 实验材料
选取中国广头叶蝉亚科5属22种和片角叶蝉亚科Idiocerinae 1种进行实验。样本主要来源于无水乙醇浸泡标本和针插保存的干燥标本,详细信息见表1。
1.2 实验方法
1.2.1DNA提取使用ddH2O清洗样本,去ddH2O后加入STE缓冲液(朴美花等,2002)浸泡过夜,弃去STE缓冲液,用冲洗缓冲液清洗2~3次,吸水纸吸去样本残余液体、晾干。然后取虫体头胸部研磨碎,使用血液/细胞/组织基因DNA提取试剂盒[tgDP304-02,天根生化科技(北京)有限公司]提取总DNA测定浓度后调整为1~2 ng·L-1保存于-20 ℃冰箱备用。
1.2.2PCR扩增引物及反应体系使用通用引物(Folmeretal.,1994)进行COⅠ基因序列片段的PCR扩增:上游引物LCO1490:5’-GGTCAACAAATCATAAAGATATTGG-3’;下游引物HCO2198:5’-TAAAC-TTCAGGGTGACCAAAAAATCA-3’。PCR反应体系参考PCR试剂盒Taq PCR Master Mix,选用30 μL反应体系,针对无水乙醇浸泡标本和针插干标本进行一定的调整,模板DNA、上下游引物(10 μM)、Mix、ddH2O分别为:1 μL、2 μL、15 μL、12 μL(无水乙醇浸泡标本);4 μL、2 μL、15 μL、9 μL(针插干标本)。PCR反应条件为:95 ℃预变性5 min;95 ℃变性1 min,40~43 ℃退火1 min,72 ℃延伸1 min,35个循环;最后72 ℃总延伸5 min。PCR产物用1%琼脂糖凝胶电泳检测。PCR产物原液送北京华大基因组测序部进行常规纯化双向测序。
1.2.3DNA数据处理与分析使用SeqMan对所得序列进行拼接和编辑,使用MEGA 6.06、Clustal W进行序列比对。序列组成分析和遗传距离的计算使用MEGA 6.06,系统发育建树使用MrBayes 3.2和PAUP*4.0。
1.2.4遗传距离计算遗传距离的计算基于MEGA 6.06的K2参数模型。利用Excel的数据分析功能统计平均值(Means)和标准差(SE),使用SPSS 22.0基于最小显著差异法(LSD)对遗传距离进行差异显著性分析。
1.2.5系统发育树构建选用最优模型GTR+I+G(参数:-lnL=5 421.611 8,K=10,AIC=10 863.223 6),使用PAUP*4.0 [参数:Lset Base=(0.340 3,0.166 2,0.109 6),Nst=6,Rmat=(0.883 4,6.885 8,2.787 8,0.547 6,17.361 7),Rates=gamma,Shape=0.814 6,Pinvar=0.416 0],最大似然(ML)法构建系统发育树;使用MrBayes 3.2[参数:lset nst=6,rates=gamma,prset statefreqpr=fixed (0.340 3,0.166 2,0.109 6,0.383 8),shapepr=fixed(0.8146),pinvarpr=fixed(0.416 0);prset revmatpr=fixed (0.883 4,6.885 8,2.787 8,0.547 6,17.361 7,1.000 0)],贝叶斯(BI)法构建系统发育树。
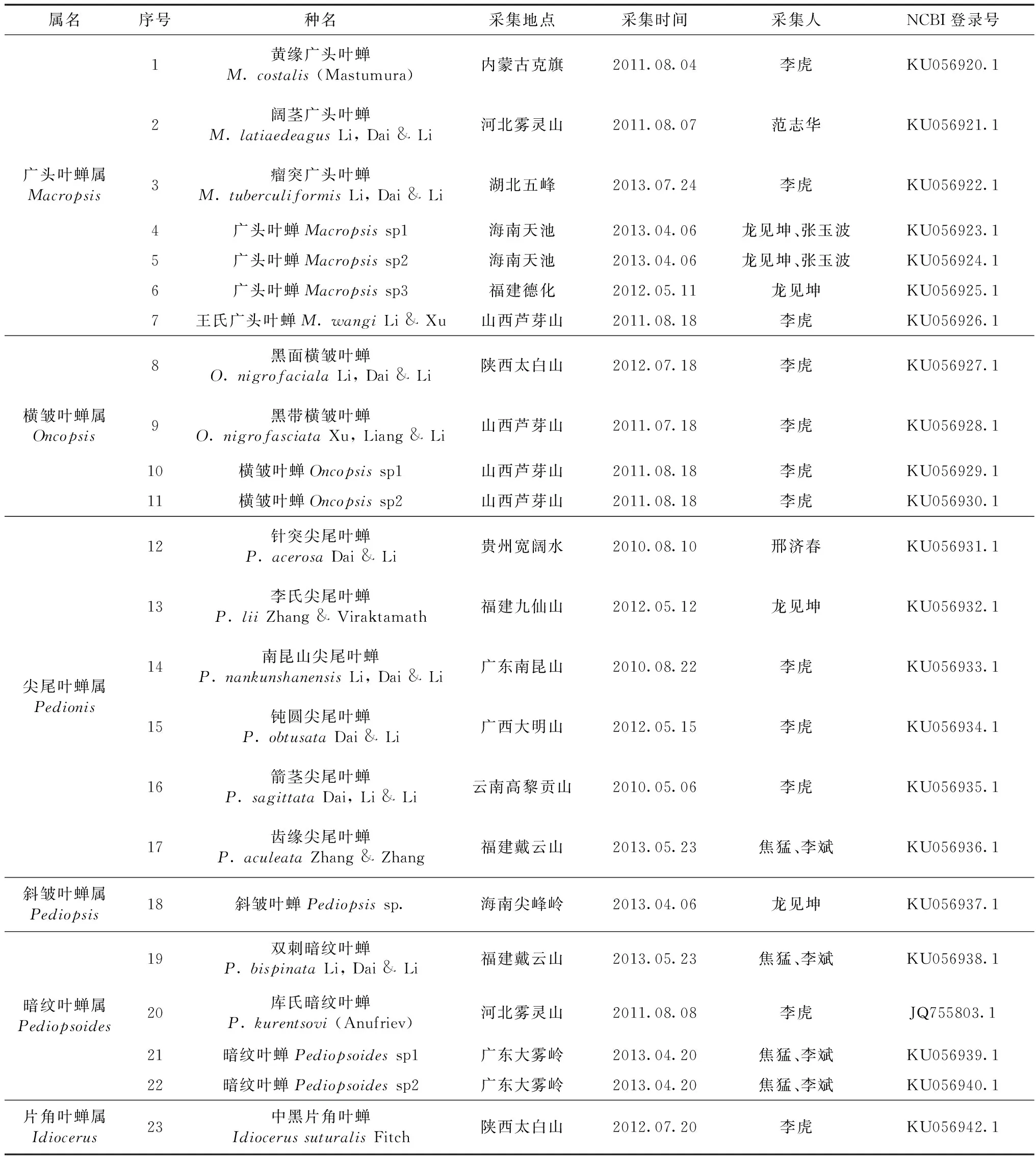
表1 广头叶蝉亚科系统发育分析选择的代表种Table 1 Representative species used for phylogeny analysis of Macropsinae
2 结果
2.1 序列组成
序列拼接和比对删除两端参差不齐的碱基序列后为642 bp,序列组成分析结果见表2。内群数据组各成分与含外群数据组之间除简约信息位点有所下降、自裔位点轻微上升外没有明显的差异。23条序列中,保守位点304个,变异位点338个,简约信息位点289个,自裔位点49个,T、C、A、G含量分别为35.7%、17.4%、32.6%和14.3%,A+T含量为68.3%,明显高于G+C含量(31.7%)。

表2 COⅠ基因序列数据组的序列组成统计值Table 2 Statistics of nucleotide composition of each data set for COⅠ gene sequences
2.2 遗传距离
经统计,COⅠ基因序列片段在种间、属间和亚科间的遗传距离的差异具有统计学意义(表3)。
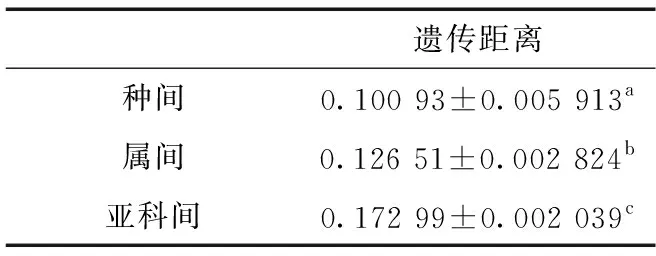
表3 COⅠ基因序列数据组不同阶元间P距离的差异统计Table 3 Statistics of P-distance of each data set for COⅠ gene sequences in different taxa
注: 不同字母表示两者之间差异有统计学意义(LSD:P<0.05)
Notes: different letters indicate there is a significant difference (LSD:P<0.05)
2.3 系统发育分析
ML树(图1:左)和BI树(图1:右)的聚类结果基本一致,仅针突尖尾叶蝉Pedionisacerosa和齿缘尖尾叶蝉P.aculeata的聚类稍有差异。最先分出广头叶蝉属的4种,有较高的置信度;随后斜皱叶蝉属种类Pediopsissp.分出;接着南昆山尖尾叶蝉Pedionisnankunshanensis与广头叶蝉属的2种聚在一起;暗纹叶蝉属2种与尖尾叶蝉属Pedionis4种聚成一大支;横皱叶蝉属4种聚在一起形成一支,并与尖尾叶蝉属、广头叶蝉属和暗纹叶蝉属剩余种聚合在一起。

图1 基于COⅠ基因最大似然法(左)和贝叶斯法(右)构建的广头叶蝉亚科系统发育树Fig. 1 Phylogeny trees based on COⅠ gene sequences for Macropsinae using Maximum Likelihood (left) and Bayesian Inference (right)
3 讨论
本文获得了广头叶蝉亚科22种和片角叶蝉亚科1种共23条COⅠ基因的部分片段序列,其中,保守位点304个,变异位点338个,简约信息位点289个,简约信息位点占总位点数的45.0%,能够较好地提供系统发育所需信息;T、C、A、G含量分别为35.7%、17.4%、32.6%、14.3%,AT偏倚明显。不同种类间的遗传距离平均值约为0.10,而不同阶元间(种-属-亚科)遗传距离差异具有统计学差异。ML树和BI树较好地反映了中国广头叶蝉亚科属间系统发育关系,支持了横皱叶蝉属和尖尾叶蝉属的单系性,但广头叶蝉属和暗纹叶蝉属较为混乱。
3.1 COⅠ基因在广头叶蝉亚科物种鉴定中的应用
广头叶蝉亚科22种的种间遗传距离大于Hebert等(2003a,2003b)推荐的区分物种的最小种间遗传距离(0.02),一定程度上说明了基于COⅠ基因的DNA条形码对于鉴定广头叶蝉亚科种类是比较适用的。本文提供了22种广头叶蝉的DNA条形码序列,为广头叶蝉亚科的分子鉴定提供了基础数据。
3.2 广头叶蝉亚科的系统发育关系
广头叶蝉属是广头叶蝉亚科中物种数最多的类群,其形态特别是外生殖器特征高度相似(Lietal.,2012,2013,2014),所以从形态学上看,广头叶蝉属的分类毋庸置疑,但从系统发育结果来看,该属物种并未全部聚合成一支,其原因可能是存在趋同进化。钝圆尖尾叶蝉Pedionisobtusata与横皱叶蝉属聚合支构成姊妹关系,对比其形态学特征发现,钝圆尖尾叶蝉的阳茎、连索等特征完全符合尖尾叶蝉属特点,说明尖尾叶蝉属与横皱叶蝉属有较近的亲缘关系。尖尾叶蝉属除钝圆尖尾叶蝉和南昆山尖尾叶蝉外,都能聚在一起,基本支持其单系性;钝圆尖尾叶蝉如前所述,与横皱叶蝉属关系较近,而南昆山尖尾叶蝉和瘤突广头叶蝉Macropsistuberculiformis聚为一支,其原因尚需进一步研究。暗纹叶蝉属最为混乱,而该属也是目前广头叶蝉中最有争议的一个属,其分类地位可能存在问题;依Hamilton(1980)的系统,该属包含5个亚属,作者与俄罗斯学者Tishechkin博士(Department of Entomology,Faculty of Biology,M.V. Lomonosov Moscow State University,Vorobyevy Gory,Moscow,Russia)和日本学者Okudera博士(Entomological Laboratory,Faculty of Agriculture,Kyushu University,Fukuoka,Japan)交流,均表示该属目前存在一些问题,但由于标本掌握不全,有些亚属仅分布在新北区,有些仅分布在非洲区,加之该属的形态特征高度近似,且存在交叉,因而无法提出合适的修订意见;该属的演化关系需在补充标本后进一步研究。
本文为广头叶蝉亚科系统发育关系提供了重要的基础信息。昆虫线粒体COⅠ基因被广泛用于物种鉴定,因其序列片段较短,能够提供用于系统发育构建的信息可能并不完整。本文中广头叶蝉属、尖尾叶蝉属和暗纹叶蝉属的单系性并不理想,这样的结果或许并不是分类本身的原因,也可能是由于当前的数据不足以支撑其构成单系,后续可获取多基因的序列数据来进一步探讨和完善中国广头叶蝉亚科的系统发育关系。
