C1GALT1基因变异与IgA肾病遗传易感性的相关性研究
2018-10-24列才华梁兰青郭志勇
黄 海,列才华,梁兰青,屈 姗,郭志勇
IgA肾病(IgA nephropathy,IgAN)是一种多基因疾病,遗传因素的确切作用仍然难以捉摸。越来越多的证据表明IgAN发病机制中IgA1分子的异常半乳糖化[1],半乳糖基转移酶核心1β3-Gal-T及其分子伴侣Cosmc在IgA1分子的β1,3糖基化中发挥重要作用[2],核心 1结构 Galβ1→3GalNAcα1-R 由GalNAcα1-R通过核心1尿苷二磷酸-半乳糖:Gal-NAc-α-Rβ1,3-半乳糖基转移酶(核心 1β3-Gal-T)的作用合成,编码该酶的基因是C1GALT1(GeneID:56913)[3]。进一步研究发现核心1β3-Gal-T活性需要表达命名为Cosmc(核心1β3-Gal-T特异性分子伴侣)的分子伴侣[4],其编码基因是 C1GALT1C1(GeneID:29071),C1GALT1C1基因的突变能够显著改变C1β3Gal-T的酶活性[5]。本研究探索C1GALT1和C1GALT1C1基因的单核苷酸多态性(single nucleotide polymorphisms,SNPs)对新疆地区维吾尔族人群IgAN易感性的影响。
1 资料和方法
1.1 临床资料 本研究纳入1 014名新疆地区维吾尔族人,其中595例经肾活检证实为IgAN,419例为民族和地理分布相同血尿分析正常的健康对照者。纳入标准:IgAN的诊断通过免疫荧光检测观察IgA在肾小球系膜中的颗粒沉积,以及在系膜超微结构检查中电子致密物质的沉积来证实。排除标准:患有过敏性紫癜,系统性红斑狼疮和慢性肝病的患者。肾活检时患者的年龄:12~76(30.6±10.7)岁;性别比例:(男∶女)=(1.29∶1)。 IgAN 患者的肾小球滤过率通过肾脏疾病饮食改良缩写方程估算。
该基因研究方案经新疆军区总医院医学伦理委员会批准,并获得所有参与者的基因研究书面同意书。
1.2 基因研究方案 首先,扫描C1GALT1基因和C1GALT1C1基因来鉴定子样本中的所有多态性,然后选择单体型标签SNPs。其次,在IgAN患者组和对照组中进一步对htSNP进行基因分型。然后使用基于单倍型的关联分析来检测其多态性可能对IgAN的影响。
1.3 SNPs检测 通过盐析程序从乙二胺四乙酸-抗凝全血样品中提取患者的基因组DNA。参考序列C1GALT1基因和C1GALT1C1基因获自美国国家生物技术信息中心基因数据库(http://www.ncbi.nlm.nih.gov/Entrez)。对于C1GALT1基因,通过聚合酶链式反应(polymerase chain reaction,PCR)扩增来自24个样品(包括12个不相关的IgAN患者和12个不相关的健康对照)的基因组DNA,并直接测序以筛选SNP。PCR扩增区域包括具有50~100 bp的侧翼内含子序列,5′和3′非翻译区以及转录起始位点上游1 kb的每个外显子。为了筛选C1GALT1C1基因内的变异,招募了46名个体,包括27名患有IgAN(6名女性患者)患者和19名正常对照者(6名女性对照)。C1GALT1C1基因的外显子从46个个体的58个染色体中扩增出来。通过Primer3程序设计PCR引物。在20 μL最终反应体积中,通过PCR从50 ng基因组DNA中扩增靶序列。使用ABI PRISM 3700自动测序仪(美国PE ABI公司)对产物进行测序,并用Phred/Phrap/Consed软件包进行分析。使用Polyphred29检测多态性位点并手动确认。
1.4 基因分型 通过htSNPer1.0选择常见SNP(具有次要等位基因频率>10%的SNP)作为htSNP。根据单倍型多样性进行软件应用,在一个模块中,这个定义要求htSNPs应该解释多样性不低于0.8的阈值。在C1AGLT1基因中,5个标记SNP,被命名为SNP1(-734C/T),SNP4(-465A/G),SNP6(-330G/T),SNP7(-292C/-)和 SNP8(1365G/A)进行进一步分析。通过定向测序对SNP8进行基因分型,并且通过标准PCR-限制性片段长度多态性方法在所有1 160个受试者中对另外4个SNP进行基因分型。表1列出了引物和限制性核酸内切酶。每个SNP位点的40个PCR产物被重新测序以准确确认PCR限制性片段长度多态性分析。
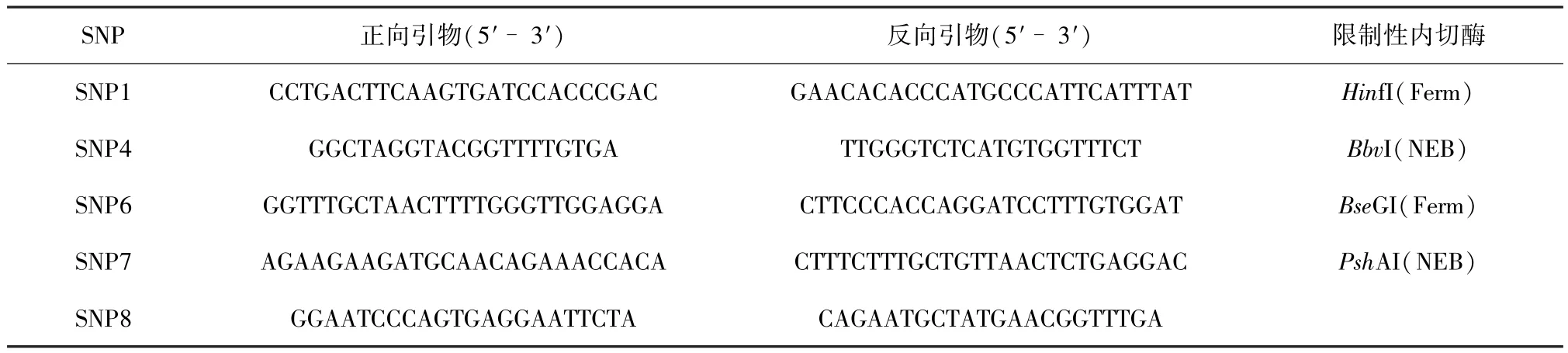
表1 C1GALT1基因SNP的引物和限制性内切酶
1.5 统计学处理 应用SPSS 10.0软件进行描述性统计分析。Fisher精确检验用于确定基因型的分布是否符合Hardy-Weinberg平衡。将每个SNP之间的配对连锁不平衡量化为由GLOD软件包测量的D′和Δ2。使用Haplo.Stats1.2.0软件测试统计推断的单倍型与IgAN的关联。通过Bonferroni校正来调整P值的水平。由于本研究中的单倍型是通过估计构建的,所以综合P值是通过使用软件置换测试经验性地评估的。以P<0.05为差异有统计学意义。
2 结果
2.1 多态性检测 C1GALT1基因内的9个SNPs在24个个体中被鉴定。7个SNP(SNP1-SNP7)分别位于第一个转录位点上游的位置-734,-722,-552,-465,-449,-330和-292的 5′侧翼区,2个 SNP(SNP8,SNP9)分别定位于3′非翻译区1365和1484位。在C1GALT1基因启动子区检测到一个插入/缺失SNP(-292C/-)。3个SNP,-722G/T,-552A/G,-465A/G,是dbSNP数据库的新型SNP(http://www.ncbi.nlm.nih.gov/projects/SNP/)。表2列出了所有SNP的详细信息以及次要等位基因频率。
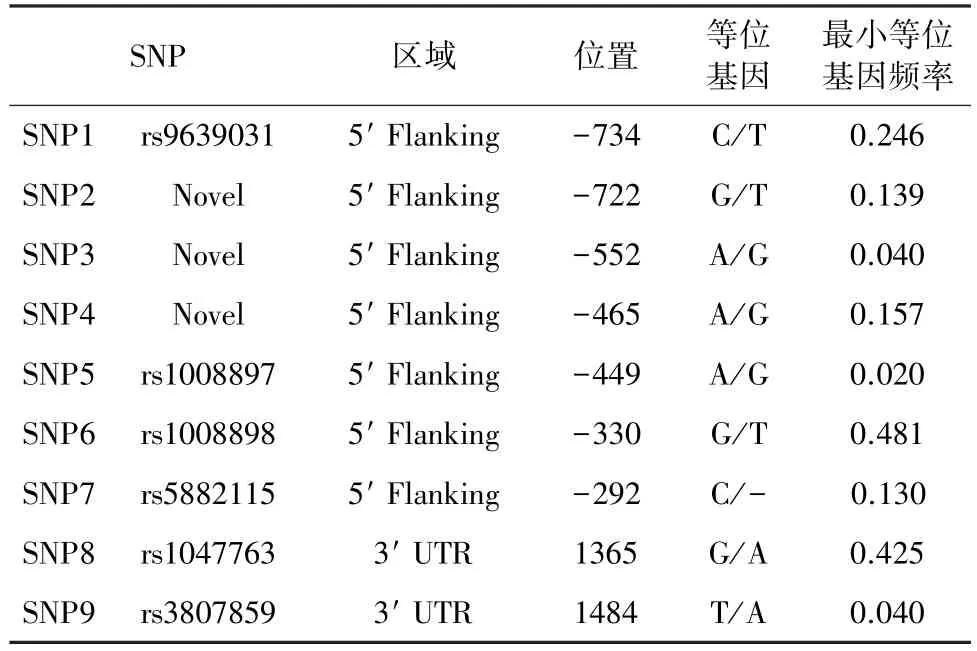
表2 C1GALT1基因多态性检测结果
2.2 连锁不平衡分析 C1GALT1基因中9个SNP的成对连锁不平衡系数(D′)和相应的P值(表3)。通过Bonferroni校正(P<0.05/=0.0014)。字体加粗区域表明C1GALT1基因内存在显着的连锁不平衡(P<0.0014)。通过htSNPer 1.0软件选择SNP1(-734C/T),SNP4(-465A/G),SNP6(-330G/T),SNP7(-292C/-)和SNP8(1365G/A)5个多态性位点作为关联研究中进一步调查的SNP标记。

表3 C1GALT1基因连锁不平衡分析
在46名招募个体(包括12个女性个体总共58个X染色体)的C1GALT1C1基因的外显子中仅鉴定到1个SNP T393A(rs17261572)。A等位基因在1名IgAN患者和1名健康对照者中被鉴定,并且AA基因型在1名女性患者中被鉴定。因此,SNP的A等位基因频率是0.069(4/58)。由于标签SNP的次要等位基因频率应大于0.10,C1GALT1C1基因中的T393A SNP不包括在关联研究的进一步分析中。
2.3 IgAN患者和正常对照者的多态性 关联分析分别针对每个SNP进行,然后进行基于单倍型的分析。比较了IgAN患者和正常对照组之间的等位基因和基因型频率。SNP分析结果显示(表4)SNP7位点I等位基因频率显着低于对照组(0.091 vs 0.129,χ2=9.827,P=0.004);SNP7 II/DI基因型在患者中明显较少(0.179 vs 0.243,χ2=9.130,P=0.007);其他4个SNPs的等位基因和基因型在2组之间都无显著差异(P>0.05)。
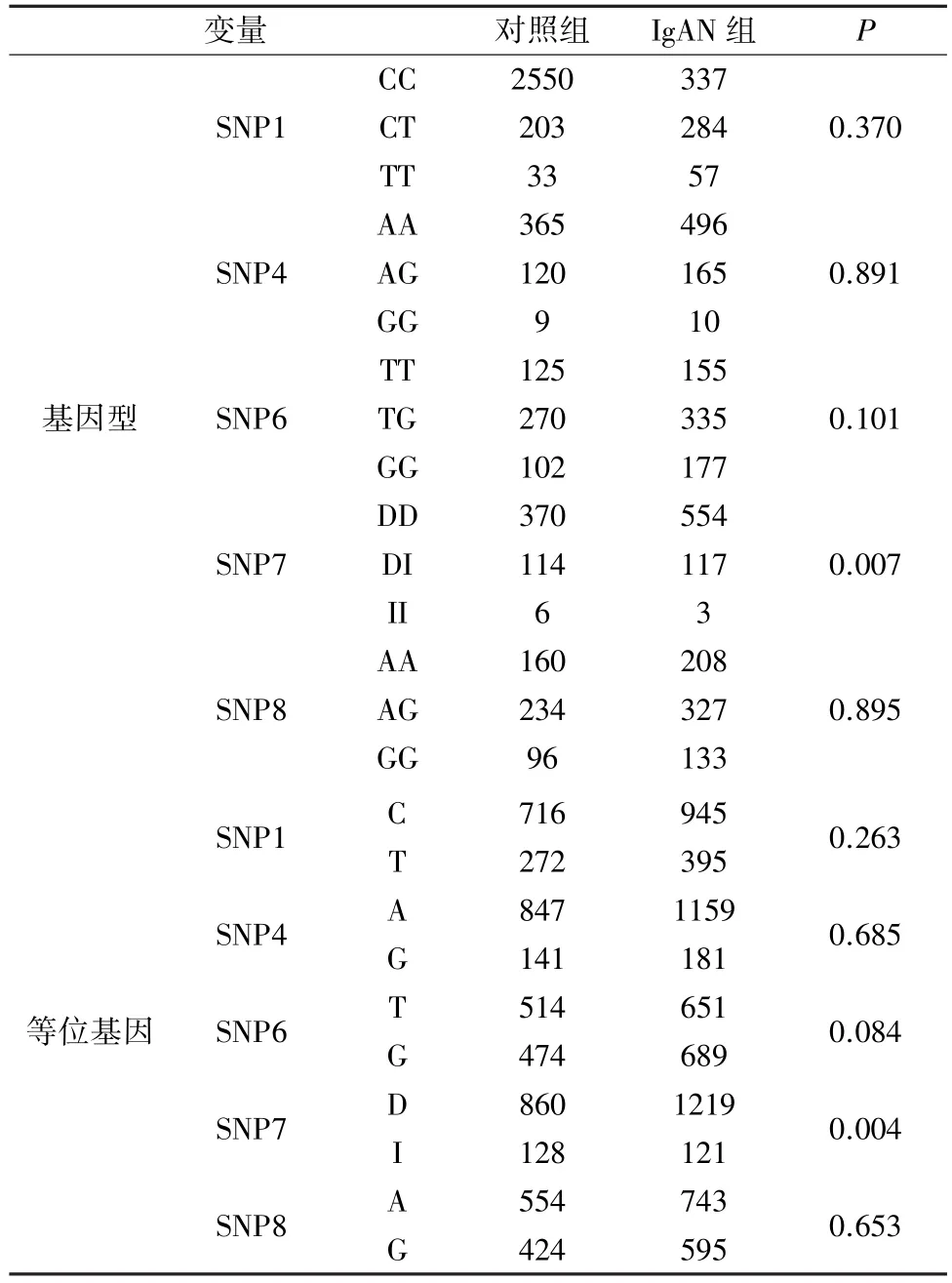
表4 IgAN患者和正常对照者的多态性
2.4 IgAN患者和正常对照者的单体型 通过haplo.cc(在haplo.stats程序中实施)分析,已经计算了IgAN易感性与由5个 SNP的不同数目(2~5个SNP范围)组成的单倍型的不同意义。除第1个SNP外,其余4个SNP得分最大,调整后总体得分(50.122)和最小全局P值(P=5.950×10-7)。通过置换测试(2万个置换)测试了IgAN患者与对照组之间单体型频率分布的全球差异。发现2组间单体型频率有显着的总体差异(P=0)。单倍型YATIG(Y=C或T)频率明显低于对照组(0.0726 vs 0.1202,t=8.692,P=2.684×10-4)。 其比值比(odds ratio,OR)为0.68[95%置信区间(confidence interval,CI):0.50~0.95](表5)。其余2种单体型YAGDA(0.1187 vs 0.0684,t=7.801,P=3.671×10-3)和YATDG(0.0827 vs 0.0275,t=9.463,P=1.197×10-5)的频率显著高于对照组。其OR分别为1.72(95%CI:1.28~2.46)和 2.87(95%CI:1.87~4.90)(表 5)。
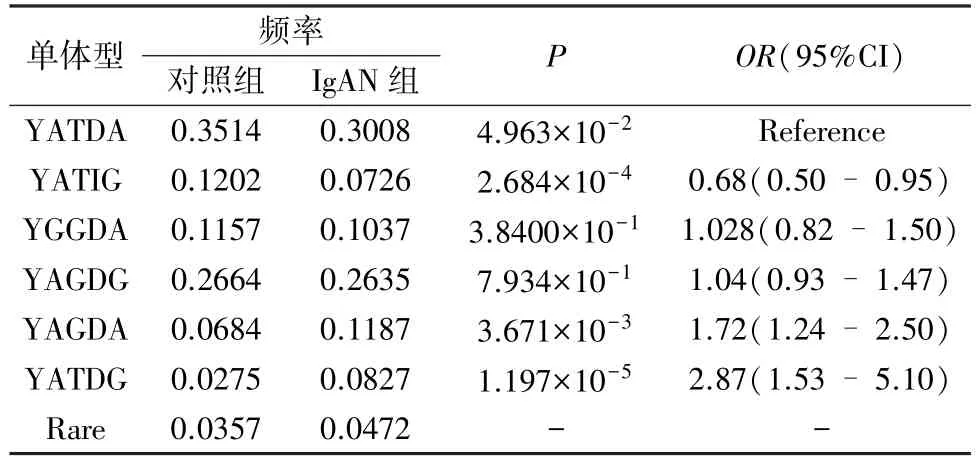
表5 IgAN患者和正常对照者的单体型
3 讨论
IgAN被认为是一种多基因和多因素疾病[6-7]。广泛的证据表明遗传因素参与了IgAN的易感性和进展[8-9]。基于家族性IgAN数据的连锁分析显示,家族性IgAN可以在显性模型具有不完全的外显率[10]。然而,在这些关联的区间内没有鉴定出IgAN的候选基因。因此,散发性IgAN患者的SNP分析将有助于鉴定可能最初牵连于该多基因疾病的其他基因。在过去的20年中,许多研究进行了遗传关联研究,他们发现许多候选基因与IgAN的易感性和进展有关,这些基因大多集中于IgAN的进展而不是IgAN的发病[11-12]。另一方面,大多数关于IgAN的关联性研究是在相对较少的病例对照人群内进行的,并用单一SNP进行分析[13]。可能导致大多数IgAN遗传学关联研究缺乏可重复性。在进行基于人群的遗传关联研究时,大样本(超过1 000人)的分析趋向于产生更多的可复制关联,而不是小样本[14]。否则,htSNPs选择和单体型分析的最新发展提供了更多强大的遗传关联研究。基于上述考虑,本研究设计了基于单体型分析的新疆地区维吾尔族IgAN患者和地理民族匹配的健康对照的大样本人群中的遗传关联研究。结果表明,C1GALT1基因的变异,尤其是单体型 YATIG、YAGDA、YATDG与IgAN易感性有关。
本研究首先筛选了一个亚样品中C1GALT1和C1GALT1C1基因的多态性,并分别检测到9个SNP和1个SNP。然后选择C1GALT1基因内的5个htSNP进行进一步的关联分析。IgAN患者-292C/D等位基因或DD基因型频率显着高于对照组。单倍型YATIG频率明显低于对照组,而单倍型YAGDA或YATDG频率明显高于对照组。这些结果表明C1GALT1基因的变异会影响IgAN的易感性。正如我们在开始时提到的,核心1β3-Gal-T活性需要分子伴侣Cosmc的表达。然而,我们只在C1GALT1C1基因的编码区内检测到1个SNP(T393A),其次要等位基因的频率仅为0.069,它不能与进一步关联分析的标准相匹配。另一方面,尽管在天冬氨酸至谷氨酸(Asp131→Glu)的氨基酸取代中报道了SNP,T393A(rs17261572),但是在之前的研究中显示正常的核心1β3-Gal-T活性[15]。这表明C1GALT1C1基因编码区内的变异对于IgA1的异常半乳糖基化不是必需的。
Pirulli等[16]研究发现C1GALT1基因的变异与意大利IgAN患者对IgAN的易感性相关,确定了C1GALT1基因的1365G/G基因型,在健康对照者中启动子CGATW单体型显着较少。虽然1365G/A多态性与IgAN易感性之间的关系不能在较大的人群中复制,但本研究发现单倍型YATIG的频率显着降低,YAGDA和YATDG频率显着高于对照组。本研究结果表明,1365G/A多态性可能影响IgAN与C1GALT1基因中其他SNPs结合的易感性。在本研究中,我们鉴定了C1GALT1基因启动子区域的7个SNPs;而在意大利人群中有2个SNPs[16]。这表明筛选不同人群特别是来自不同地域不同种族的人群的多态性是很重要的。除样本量外,其他因素如种族差异,未知的环境因素以及IgAN的遗传异质性可能会带来不同的结果。
