一株湖北HP-PRRSV分离株nsp2基因遗传变异分析
2018-10-11陈盛楠陈奎蓉赵雯娟杨永磊赵一苹曹素芳
陈盛楠,陈奎蓉,赵雯娟,杨永磊,赵一苹,曹素芳
(河南牧业经济学院 动物医学院,河南 郑州450046)
猪繁殖与呼吸综合征( PRRS)俗称猪“蓝耳病”,其病原为猪繁殖与呼吸综合征病毒(PRRSV)。PRRS最早发生于美国,随后蔓延于世界。我国1996年出现该病。研究发现PRRSV易变异,2006年我国就出现了由经典PRRSV演化而来的高致病性PRRSV[1],其毒力大大增强,主要表现是体温高烧不下、呼吸障碍、死亡率高。目前,猪场内除了高致病PRRSV,经典PRRSV、疫苗株外,还有PRRSV类NADC30株、重组毒株等,不同毒株引发的临床症状严重程度不同,各毒株间也可发生重组导致新的毒株。2014年以来,PRRSV类NADC30株已成为我国猪场主要流行毒株[2],其提高母猪流产率,加重保育猪及育肥猪的呼吸道症状,还常引发其它病原微生物混合或继发感染,死亡率大大增加,严重影响了养猪业的发展。
PRRSV为单股正链 RNA有囊膜病毒,基因全长约为15~15.5kb,至少含有ORF1a、ORF1a′、ORF2a、ORF2b、ORF3、ORF4、ORF5a、ORF5、ORF6及ORF7等11个开放阅读框 (open readingframe,ORF)[3]。最容易变异的存在于ORF1a中的nsp2基因,它编码最大的非结构蛋白Nsp2[4]。研究显示,在高致病性PRRSV中的Nsp2蛋白含有30个氨基酸的不连续缺失[5],但在PRRSV类NADC30株中,Nsp2则以“111+1+19”模式缺失131个氨基酸[6]。为了了解湖北地区某猪场PRRSV毒株流行情况,掌握该毒株的nsp2遗传变异特点,本试验采用RT-PCR法扩增湖北分离株(命名为HDZZ1株)的nsp2基因,并对获得的基因序列进行遗传变异分析,为设计和开发更有效的PRRSV 诊断制剂提供技术支持,为早期预警 PRRS 的发生和流行提供依据。
1 材料和方法
1.1 材料
PRRSV阳性病料(湖北某猪场);TRIzolRegment(Invitrogen公司);AMV反转录酶、RNasin(Promega公司);EX taq DNA聚合酶等PCR试剂、DL2000 Maker、MiniBEST Agarose GelDNA Extraction Kit Ver 4.0胶回收试剂盒(上海生工)。
1.2 引物的设计与合成
参照Genbank中登录的HP-PRRSV基因序列及文献[8],设计nsp2引物:nsp2-F:5′-CCTCCGTGGTGCAACAAATCTTG-3′;nsp2-R:5′-CGATGATGGCTTGAGCTGAGTAT-3′,扩增片段约为1065bp。引物由英潍捷基(上海)贸易有限公司合成。
1.3 RT-PCR扩增
1.3.1 病毒培养 将采集的病料进行处理后,接种Marc-145细胞,六次传代后,待细胞出现典型的CPE后收获细胞,反复冻融三次,离心,取上清备用。
1.3.2 反转录 根据TRIzolRegment试剂盒说明书进行提取病毒总RNA,进行反转录,体系20μL:模板RNA 12.5μL,AMV RT 5X Buffer 4μL,4dNTP 1μL,RNasin 0.5μL,上下游引物各1μL,AMV反转录酶1μL, 42℃水浴60 min,冰浴5min,取出即得cDNA。
1.3.3 PCR扩增 PCR扩增体系为25μL:10×Ex TaqBuffer2.5μL,上下游引物各0.5μL,4dNTP 0.5μL,cDNA模板1μL,Ex Taq0.5μL,ddH2O 19.5μL。nsp2基因扩增条件为:94℃ 5min;94℃30s、56℃ 30s、72℃ 45s,30个循环,72℃延伸10min,电泳检测扩增结果。
1.3.4 序列测定及分析 用MiniBEST Agarose GelDNA Extraction Kit Ver 4.0胶回收试剂盒,将目的条带回收后送至上海生工进行测序。对获得的序列与Genbank中的序列进行同源性分析,其中选取13株高致病性毒株(07BJ、BJ0708、GD、GDYC、HEB1、Henan-1、HN-HW、HPBEDV、HUB1、HUN4、JXA1、SX-1、SX2009)、美洲代表株VR-2332、4株美洲经典型(CH-1a、CC-1、BJ-4、S1)、疫苗毒株(MLV)、美国NADC30株(JN654459.1)、国内NADC30-like型HNjz15株(KT945017.1)及欧洲代表株Lv共22株(表1),应用Lasergene R7.0 (DNAStar) 和DNAMan软件对毒株序列进行多重比对,并绘制系统进化树。

表1 同源比较的22参考株PRRSV信息
2 结果
2.1 目的基因RT-PCR结果
应用RT-PCR获得了HDZZ1株的nsp2部分基因,结果显示,与预期大小一致的片段(图1)序列大小约为1065bp。

图1 HDZZ1株Nsp2基因RT-PCR扩增结果M:DL2000;Nsp2:nsp2片段
2.2 PRRSV HDZZ1株Nsp2部分基因核苷酸及其编码氨基酸的序列分析
2.2.1 核苷酸同源性分析 将HDZZ1株nsp2与PRRSV欧洲型毒株Lv的Nsp2部分基因的核苷酸序列对比,发现同源性为39.5%,与PRRSV疫苗株MLV同源性为77.5%,与美洲原型毒株VR-2332的同源性为77.7%,与4株经典毒株CH-1a、CC-1、BJ-4、S1的同源性分别为90.8%、77.6%、77.4%、77.1%,与美国NADC30株的同源性为54.4%,与国内NADC30-like型HNjz15株的同源性为55.1%,而与13株高致病毒株07BJ、BJ0708、GD、GDYC、HEB1、Hennan-1、HN-HW、HPBEDV、HUB1、HUN4、JXA1、SX-1、SX2009的同源性分别为98.5%、98.3%、98.8%、98.6%、97.6%、97.3%、98.3%、98.3%、97.7%、98.3%、98.2%、96.4%、96.5%。分析结果显示:HDZZ1毒株为高致病性PRRSV(图2)。

图2 分离毒株与参考毒株Nsp2核苷酸序列同源性对比
2.2.2 核苷酸序列差异分析 HDZZ1株与美洲原型毒株VR-2332和经典毒株CH-1a进行比较,发现2处不同位置的连续缺失,缺失基因位于694bp~696bp和850bp~936bp,共90个碱基。与美国NADC30株、国内NADC30 HNjz15株比较,发现14处不同位置的基因缺失和6处不同位置的基因添加,基因缺失分别位于37bp~40bp、51bp、86bp~87bp、103bp~106bp、187bp~194bp、210bp~214bp、281bp、396bp、408bp、427bp~428bp、694bp、783bp~785bp、850bp、908bp~936bp,共60bp,基因添加分别位于245bp~246bp、337bp~340bp、499bp~500bp、540bp、796bp、843bp~845bp,共13bp。与以JXA1为代表的高致病性PRRSV基因缺失或基因添加的位置一致(如图3)。
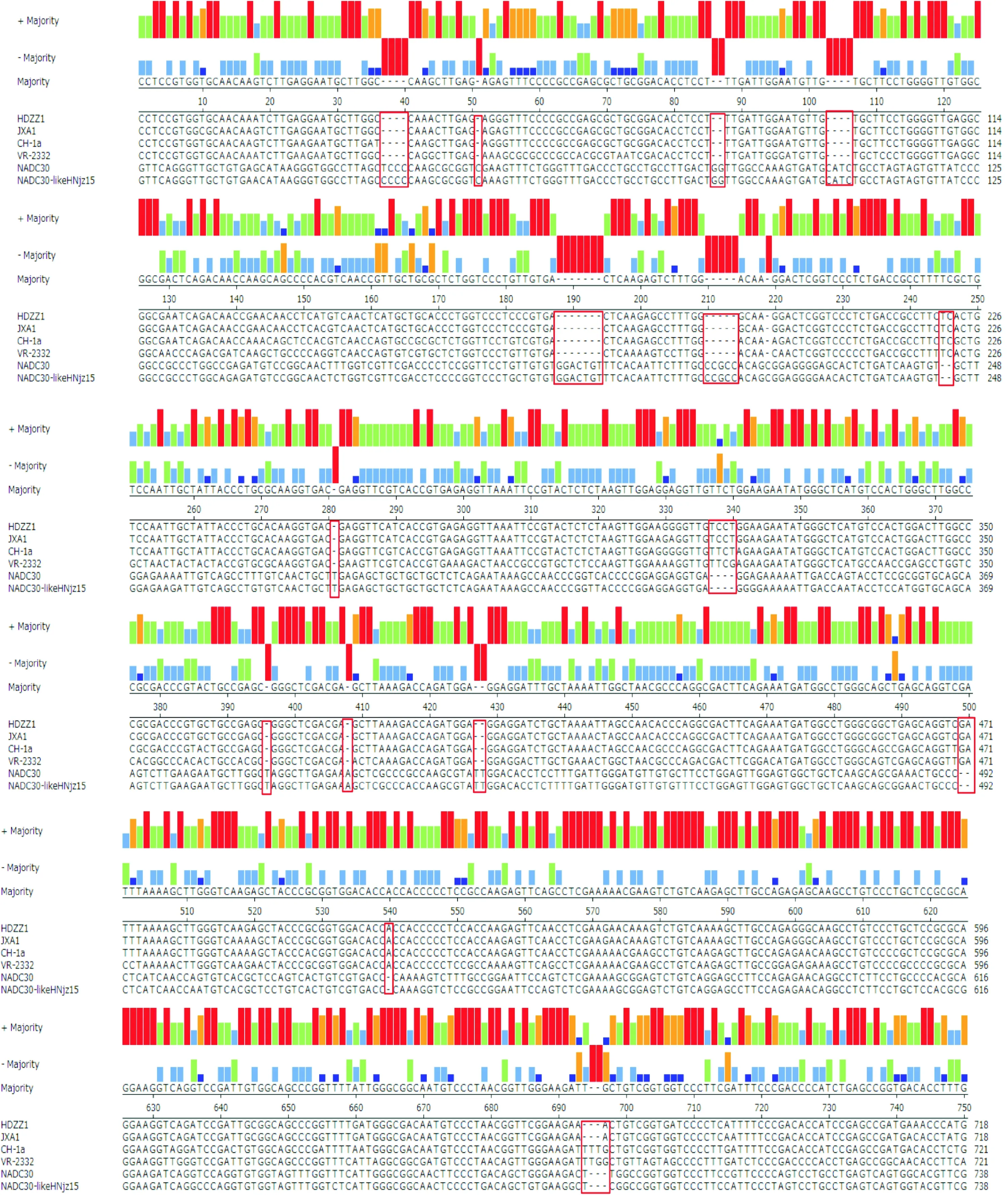

图3 分离株与PRRSV参考株Nsp2基因序列对比结果
2.2.3 推导氨基酸序列同源性分析 HDZZ1株与PRRSV欧洲型毒株Lv的Nsp2部分基因的同源性为14.5%,与PRRSV疫苗株MLV的Nsp2部分基因同源性为68.1%。与美洲原型毒株VR-2332的Nsp2部分基因的同源性为68.9%。与22株PRRSV美洲型毒株nsp2部分基因的氨基酸序列进行比较,发现HDZZ1株与4株经典毒株CH-1a、CC-1、BJ-4、S1的同源性分别为87.6、69.7%、67.8%、67.2%,与13株高致病07BJ、BJ0708、GD、GDYC、HEB1、Hennan-1、HN-HW、HPBEDV、HUB1、HUN4、JXA1、SX-1、SX2009的同源性分别为97.7%、97.2%、98.3%、98.0%、95.8%、94.9%、97.2%、96.9%、96.3%、97.2%、96.9%、94.4%、94.4%,见图4。
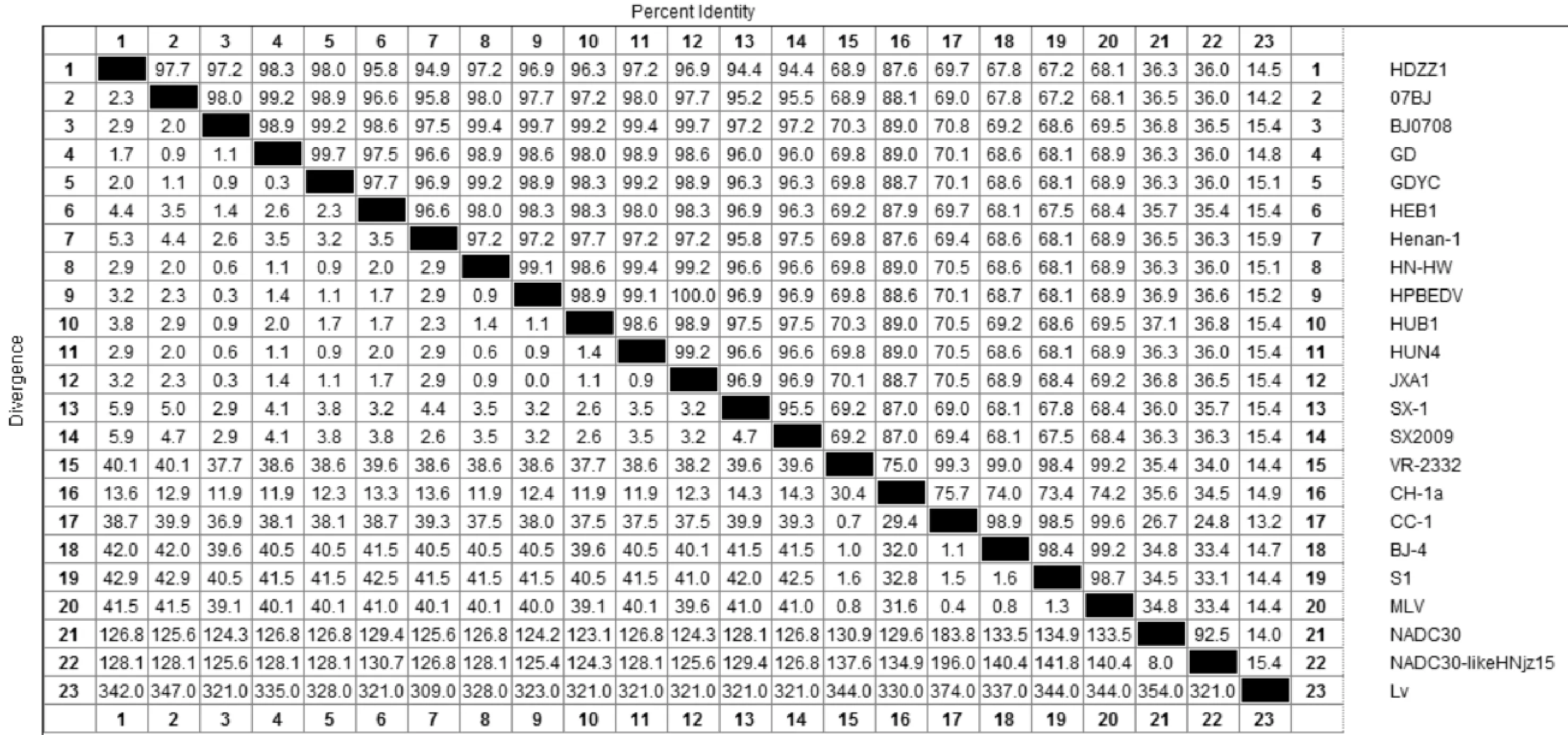
图4 分离毒株与参考毒株Nsp2氨基酸序列同源性对比
2.2.4 推导氨基酸序列差异分析 HDZZ1株与美洲原型毒株VR-2332和经典毒株BJ-4进行比较,发现在氨基酸缺失上:1处氨基酸单个缺失和1处氨基酸连续缺失,缺失氨基酸位于222位和274位~302位,共30个氨基酸,与江西省所分离的PRRSV JXA1株的Nsp2氨基酸缺失情况完全相同(图5红框所圈);在氨基酸突变上:与美洲原型毒株VR-2332相比有127处差异,与高致病性毒株PRRSV JXA1株对比,第6位S→N,第37位V→E,102位E→G,第225位G→D,第228位N→I,第237位T→K,第245位V→M,第256位T→K,第270位T→I,第293位E→G,第330位A→T,共11处差异(图5蓝框所圈)。
2.3 构建Nsp2序列的系统发生树
将该地区PRRSV分离株HDZZ1与其他地区PRRSV分离毒株的Nsp2氨基酸序列进行系统发生树构建与分析。结果表明,该分离毒株与其他高致病性PRRSV毒株(如JXA1、07BJ等)位于同一大分支中,亲缘关系比较近,其中与北京株(07BJ)关系最近,但与国内较早分离的经典病毒株的亲缘关系相对较远。与欧洲代表株(Lv)、美国NADC30株和国内NADC30-like型HNjz15株处于不同分支中,可见亲缘关系甚远(见图6)。

图5 分离株与PRRSV参考株Nsp2氨基酸序列对比结果

图6 分离株与PRRSV参考株Nsp2序列的系统发生树
2.4 推导蛋白质亲水性和疏水性的对比
分离株HDZZ1 Nsp2部分氨基酸与GenBank已登录的高致病性PRRSV进行亲水性和输水性对比,发现其与高致病性并无较大差异(图7和图8)。
3 讨论
猪繁殖与呼吸道综合征是造成我国养猪产业重大经济损失的疾病之一。我国自2006年报道由PRRSV变异毒株引起的高致病性猪繁殖与呼吸道综合征以来,多地区陆续发现了PRRSV的变异毒株,并且临床上呈现持续感染、适应力不断增强的趋势[7,8]。许多研究发现,与经典型PRRSV相比,高致病性PRRSV存在着30个氨基酸不连续的缺失,以此推测这就是高致病性PRRSV的重要特征,但经后续的实验证明,该氨基酸缺失与毒力无直接关系[9,10],但在病毒的复制和宿主免疫调节这两方面有着重要的作用。

图7 分离株与高致病性PRRSV推导氨基酸亲水性对比结果

图8 分离株与高致病性PRRSV推导氨基酸疏水性对比结果
目前,常用PCR法检测猪场PRRSV感染情况,检测靶基因一是选择结构基因ORF5和ORF7[11,12],另一个是以最易变异的nsp2基因为靶基因[13]。由于以nsp2基因为靶基因进行PCR检测可以区分经典PRRSV毒株、HP-PRRSV毒株及PRRSV类NADC30毒株等不同亚型和亚群[14]。因此,本实验设计了nsp2基因高变区的特异性引物,以分离湖北某猪场的毒株HDZZ1为模板,扩增获得了nsp2部分高变区基因,经序列测定及变异分析,结果显示,分离毒株HDZZ1与2006年以来发现的高致病性PRRSV相同,均存在90个核苷酸不连续缺失,分别为第481和533~561位,与Genbank登记的高致病性PRRSV核苷酸序列同源性高达96.4%,与美国NADC30株、国内NADC30型HNjz15株比较,同源性为55%,说明HDZZ1毒株为高致病性PRRSV,而不属于NADC30毒株,与曹素芳等[14]研究结果一致。推导的氨基酸序列表明,获得的Nsp2蛋白氨基酸序列具有高致病性毒株的经典缺失,即30个不连续氨基酸缺失,与白云等[15]研究结果不一致,可能是不同地区PRRSV流行毒株有差异。遗传进化树分析结果显示,HDZZ1毒株与高致病性PRRSV同属一分支,与北京株(07BJ)关系最近,再次证明该毒株为高致病性PRRSV,但该毒株的毒力是否比其他毒株更强,还需进一步深入研究。
