基于比较基因组学分析的方法定位及注释双峰驼MHC基因
2018-10-11支立康额尔敦木图安希文王超王瑞包花尔王秀珍
支立康,额尔敦木图,安希文,王超,王瑞,包花尔,王秀珍
基于比较基因组学分析的方法定位及注释双峰驼MHC基因
支立康,额尔敦木图,安希文,王超,王瑞,包花尔,王秀珍
(内蒙古农业大学兽医学院/农业部动物疾病临床诊疗技术重点实验室,呼和浩特 010018)
【目的】定位并注释双峰驼主要组织相容性复合体(major histocompatibility complex,MHC)基因序列,为进一步研究双峰驼MHC基因提供科学依据。【方法】 运用比较基因组学方法,提取人类MHC(HLA)基因编码序列和牛MHC(BoLA)基因编码序列并分别与双峰驼转录本进行blastn基因序列比对,识别出相似度较高的scaffolds,通过分析HLA、BoLA基因序列比对在这些scaffolds上的位置顺序,对多条scaffolds进行拼接,得到双峰驼MHC的Pseudo chromosome;再分别提取HLA、BoLA全基因组序列与双峰驼已拼接的scaffolds进行基因组共线性分析,利用lastz建立起的Pseudo chromosome与HLA、BoLA全基因组序列的线性关系判断筛选出的scaffolds是否准确;然后通过分析MHC基因在两物种间的线性关系,在双峰驼参考基因组中提取出MHC基因序列,并对这些序列进行基因注释;最后根据得到的双峰驼MHC基因绘制系统进化树,研究其基因间的进化关系。【结果】通过对HLA、BoLA基因编码序列与双峰驼转录本用blastn进行序列比对,识别出了相似度较高的3条scaffolds,即NW_011511766.1(全长4.1M)、NW_011515227.1(全长1.2M)和NW_011514613.1(全长15K),对其拼接得到双峰驼MHC的Pseudo chromosome;利用lastz共线性分析,识别出HLA基因序列和BoLA基因序列并比对出其在双峰驼MHC基因的共线性区域。该区域与拼接得到的Pseudo chromosome一致,证明筛选出的scaffolds是准确的。并且发现Class-Ⅰ类和Class-Ⅲ类基因集中分布在NW_011515227.1上,而Class-Ⅱ类基因集中分布在NW_011511766.1和NW_011514613.1上,进一步分析得知Class-Ⅱ类基因主要分布在NW_011511766.1 的3.5—4.1M的位置;将存在共线性区域的序列提取出来,与比对到双峰驼上的MHC基因的编码序列进行blat分析,结果在双峰驼基因组中共识别出24个与牛BoLA基因高度相似的基因,其中Ⅰ类基因1个,Ⅱ类10个, Ⅲ类基因13个。对双峰驼这24个MHC基因进行信息注释并绘制系统进化树,结果显示注释的Class-Ⅰ类和Class-Ⅱ类基因在同一分支。【结论】通过比较基因组学方法定位并注释了双峰驼的MHC基因,将双峰驼MHC基因序列定位到了3条scaffolds上,找到并注释了24个MHC基因,绘制了双峰驼MHC的Pseudo chromosome,为进一步研究双峰驼MHC基因奠定了理论基础。
双峰驼;MHC;CBLA;BoLA;HLA;比较基因组学
0 引言
【研究意义】MHC(major histocompatibility complex)是所有生物主要组织相容性复合体的统称,常见类型有3类,即Class-Ⅰ、Class-Ⅱ和Class-Ⅲ类基因。MHC不仅控制着同种移植排斥反应,更重要的是与生物机体免疫应答、免疫调节及某些病理状态的产生均密切相关。不同种类哺乳动物的MHC基因序列结构、名称存在差异[1-4]。双峰驼因其应对恶劣环境挑战的能力而闻名。关于MHC的研究,双峰驼是一种具有实际意义的生物模型[5-6]。定位并注释双峰驼中的MHC基因,对双峰驼MHC的进一步研究,以及对双峰驼机体免疫机制的研究有重要的科学意义。【前人研究进展】对于MHC的研究工作最早可以追溯到1936年,国外学者在研究小鼠肿瘤细胞排斥反应中首次发现了MHC基因;1951年,研究者发现了小鼠的MHC-H2系统;1958年,Dausset首次发现了人类的白细胞抗原;1961年SCHIERMAN和NORDSKOG确定了家禽中的MHC为一种红细胞抗原,并将其命名为B[2,7];到了20世纪70代,人们发现在遗传上与H2同源的系统同样存在于其它动物中[8-10];近年来已确定几乎所有的脊椎动物都存在MHC基因,且各国学者不断丰富着不同种属动物MHC基因的研究。2012年吉日木图教授等首次完成双峰驼全基因组序列图谱绘制和解析。2016年捷克科学家通过FISH技术将单峰驼MHC基因定位到了20号染色体短臂上(20q12)[11]。【本研究切入点】目前双峰驼参考基因组的组装及注释水平不完善,在双峰驼基因组中MHC基因并没有得到组装和注释[12]。传统的基因定位方法普遍采用的是杂交,侧交和自交,分别求出基因间的交换率和相对距离,然后在染色体上确定基因间的排列顺序,这些方法操作繁琐、复杂,成本也很昂贵。【拟解决的关键问题】本研究通过利用目前已知组装水平最好的人类MHC基因和与双峰驼亲缘关系较近的牛MHC基因为种子序列,采用比较基因组学方法来探究物种间基因的同源性关系,准确定位并注释到双峰驼中的MHC基因,为双峰驼MHC基因的进一步研究奠定理论基础。
1 材料与方法
试验于2017年6—9月在内蒙古农业大学的内蒙古自治区基础兽医学重点实验室(农业部动物疾病临床诊疗技术重点实验室)进行。
1.1 序列来源
双峰驼MHC基因序列从家养阿拉善双峰驼的参考基因组中获得,参考基因组Camelus bactrianus (assembly Ca_bactrianus_MBC_1.0)在NCBI上下载得到,人类HLA、牛BoLA基因序列都是从ensemble网站下载得到。相关信息如下(表1)。
1.2 方法
1.2.1利用blast v2.3.0软件将分别从表1所示网址提取的HLA、BoLA基因编码序列,分别与双峰驼转录本进行blastn序列比对,识别出相似度较高的scaffolds;再通过分析HLA、BoLA比对在scaffolds的位置顺序,从而对多条scaffold进行拼接。
1.2.2 分别提取HLA、BoLA基因组序列与双峰驼已拼接的scaffolds进行基因组共线性分析,利用lastz v.1.04.00软件建立起已拼接的scaffolds与HLA、BoLA基因组序列的线性关系,以判断筛选出的scaffolds是否准确。

表1 双峰驼、人类、牛基因序列及数据来源
1.2.3 通过分析MHC在两物种间的线性关系,利用BLAT v.35软件提取出MHC在双峰驼基因组的基因序列,并对这些基因序列进行基因注释。
1.2.4 利用MEGA 7.0.26软件对得到的双峰驼MHC基因绘制系统进化树。
2 结果
2.1 blastn序列对比及拼接
分别以人类HLA、牛BoLA基因编码序列与双峰驼转录本进行blastn序列对比,结果识别出了相似度较高的3条scaffold,即NW_011511766.1(全长4.1M)、NW_011515227.1(全长1.2M)和NW_011514613.1(全长15K),进一步通过分析HLA、BoLA比对在scaffolds的位置顺序,发现CBLAⅠ类和Ⅲ类基因分布在NW_011515227.1上,而CBLAⅡ类基因分布在NW_011511766.1和NW_011514613.1上对上述3条scaffolds进行拼接,得到双峰驼MHC的Pseudo chromosome,其结构示意图如图1。
2.2 共线性分析
2.2.1 人类HLA与双峰驼Pseudo chromosome共线性分析 提取人类MHC基因(HLA)的基因组DNA序列,与双峰驼基因组进行共线性分析。在基因组共线性分析中一个共线性基因对应一个点,而密集的共线性基因可以连接成片段,并以线条的形式呈现在图中。绘制双峰驼MHC基因与人类HLA基因共线性关系图(图2)。可以看出,有很多双峰驼MHC基因片段和人类HLA基因片段是共线性关系,即存在较长的线性片段,有力地支持了本研究所定位到的双峰驼MHC基因的准确性,从而说明双峰驼的MHC基因家族主要分布在笔者拼接的scaffolds上。
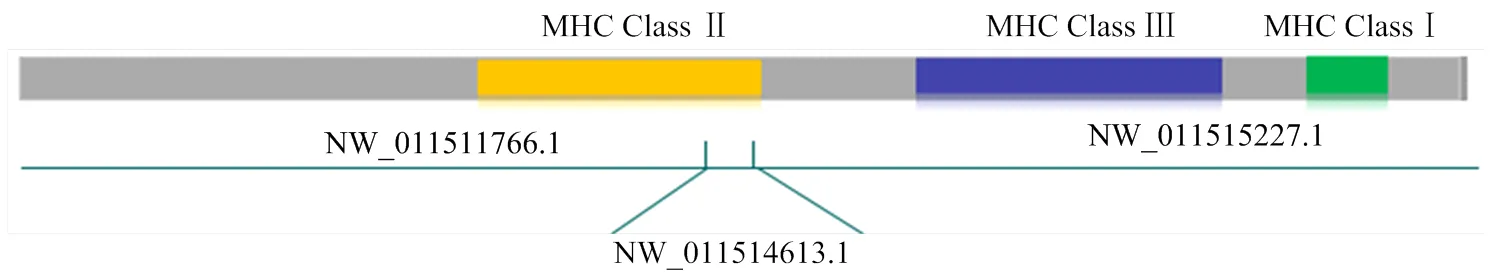
图1 双峰驼MHC-Pseudo chromosome结构示意图
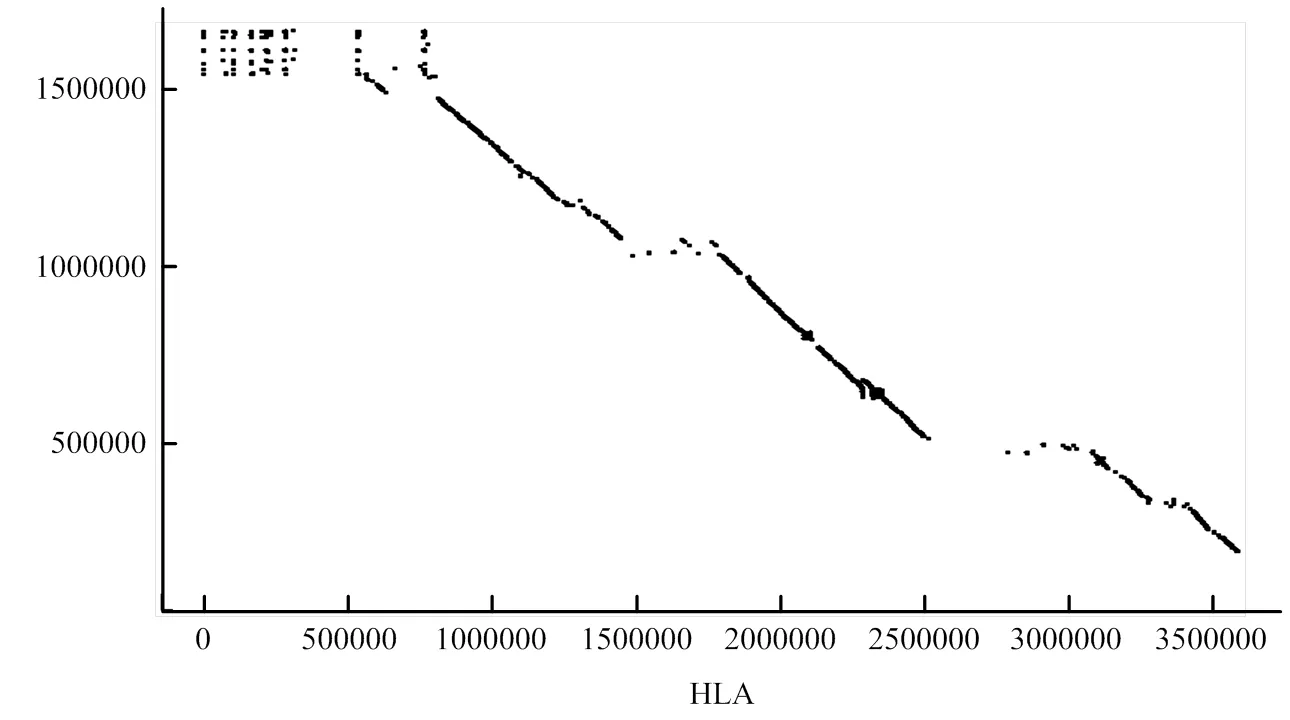
横轴为人HLA的基因序列,纵轴为双峰驼MHC的基因序列
2.2.2 牛BoLA与双峰驼pseudo chromosome共线性分析 同理,提取与双峰驼亲缘关系最近的牛的MHC基因(BoLA)[13]的基因组DNA序列,与双峰驼基因组进行共线性分析,结果显示牛BoLA基因与双峰驼的NW_011511766.1(全长4.1M)、NW_011515227.1(全长1.2M)和NW_011514613.1(全长15K)相似度最高,且Ⅰ类和Ⅲ类基因分布在NW_011515227.1上,而Ⅱ类基因分布在NW_011511766.1和NW_011514613.1上,进一步分析得知Ⅱ类基因主要分布在NW_011511766.1 的3.5—4.1M的位置。在基因组共线性分析中一个共线性基因对应一个点,密集的共线性基因可以连接成片段,并以线条的形式呈现在图中。绘制双峰驼MHC基因与牛BoLA基因共线性关系图(图3)。可以清楚的表明,有很多双峰驼MHC基因片段和牛BoLA基因片段是共线性关系,即存在较长的线性片段,有力地支持了本研究所定位到的双峰驼MHC基因的准确性,更加说明双峰驼的MHC基因家族主要分布在拼接的scaffolds上,该结果与结果1的一致。但在局部双峰驼的片段呈现更多的碎片,即当纵坐标低于4 000 000时牛BoLA与双峰驼pseudo chromosome 共线性区域出现倒置现象。
2.3 BLAT分析
将存在共线性区域的基因组序列提取出来,与比对到双峰驼上的CBLA基因的编码序列运用BLAT v.35进行分析,从而把双峰驼CBLA编码定位在基因组上。比对分析结果显示,在双峰驼基因组中共识别出24个与牛BoLA基因高度相似的基因,即在本次试验中发现双峰驼的CBLA包含24个基因,其中Ⅰ类基因1个,Ⅱ类10个, Ⅲ类基因13个(表2)。并由此可绘制人/牛/双峰驼 MHC基因个数统计表(表3)。
2.4 基因注释
对照从ensemble上获取的人类HLA、牛BoLA已知MHC基因注释信息对找到的这24个基因进行注释,可绘制双峰驼CBLA基因注释信息表(表4)。

横轴为牛BoLA的基因序列,纵轴为双峰驼MHC的基因序列
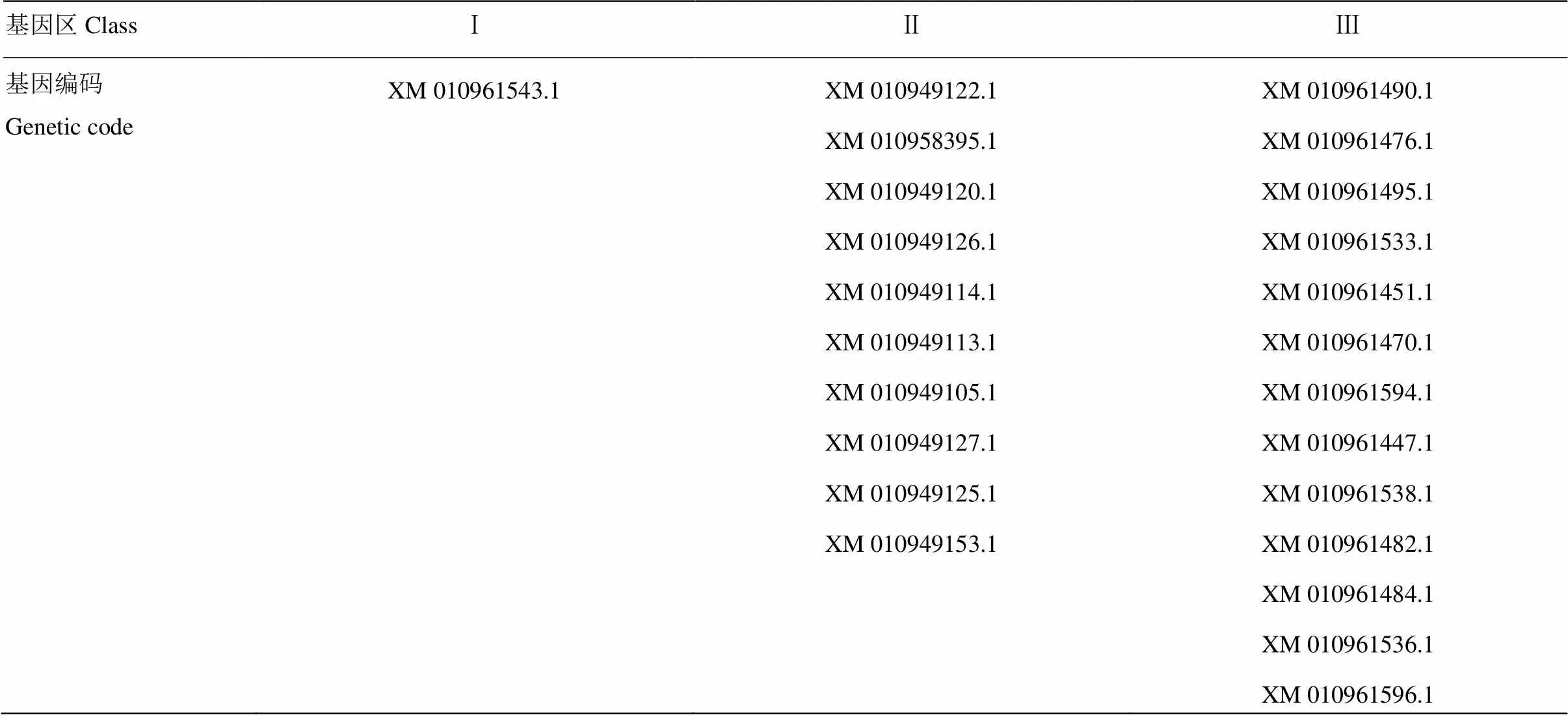
表2 blat分析找到的CBLA基因

表3 人/牛/双峰驼 MHC基因个数统计表
36/106表示人类HLA中共发现106个class-Ⅰ类基因,其中36个为真基因;同理:33/59表示人类HLA中共发现59个class-Ⅱ类基因,其中33个为真基因;59/59表示人类HLA中共发现59个class-Ⅲ类基因,其中59个为真基因
36/106 indicates that there are 106 class-I genes in human HLA, 36 of which are true genes; Similarly, 33/59 indicates that there are 59 class-II genes in human HLA, 33 of which are true genes. 59/59 indicates that 59 class-III genes were found in human HLA, 59 of which were true genes
2.5 双峰驼MHC基因绘制系统进化树
对得到的双峰驼MHC基因绘制系统进化树,结果显示注释的CBLA Class-Ⅰ类和Class-Ⅱ类基因在同一分支(图4),说明CBLA Class-Ⅰ类和Class-Ⅱ类基因进化关系最近。
3 讨论
近年来出现了很多MHC基因的定位方法,常用的方法有体细胞杂交法、克隆嵌板法、原位杂交和荧光原位杂交法(FISH)、连锁分析法。如Martin Plasil在2016年通过荧光原位杂交法将单峰驼的12个MHC基因定位到了20号染色体短臂(20q12)上的具体位置,并绘制了物理图谱。本研究通过利用目前已知组装水平最好的人类HLA基因和与双峰驼亲缘关系较近的牛BoLA基因[14]作为种子序列来定位并注释双峰驼的MHC基因。通过与两个参考物种的比较基因组学分析,最终在双峰驼基因组序列中找到了24个MHC基因(其中Ⅰ类基因1个,Ⅱ类10个, Ⅲ类基因13个),并绘制出了双峰驼MHC的Pseudo chromosome。相比于以前的MHC基因的定位方法,该方法操作简便、检测迅速、成本低廉,有望推广应用于其他物种MHC基因的定位和注释。
共线性描述了由同一祖先型分化而来的不同物种间基因的类型及相对顺序的保守性,是物种间的一种关系,体现了基因的同源性以及基因的排列顺序。本研究通过使用lastz软件分别对人类HLA、牛BoLA基因与拼接出来的双峰驼CBLA基因序列进行共线性分析发现,其绝大部分基因具有很高的共线性,说明这3种动物MHC基因组同源性很高[15]。对比图2、3,可以看到牛BoLA基因序列与拼接出来的双峰驼CBLA基因序列具有更好的共线性,即牛与双峰驼的的MHC基因同源性更高。说明了相比于人类,牛与双峰驼亲缘关系更近。这一结果与WU等[16]所得结论一致,从而证明本试验所用方法的结果是准确的。再对3个物种进行blat分析,发现其MHC基因组上的等位基因种类、结构和位置也极其相似,因此可以认定MHC基因为直向同源基因的一种[17-18]。对于像MHC基因这种功能基因的研究,通过选取其他近缘生物基因组为参照进行比较基因组学研究,利用计算机的强大处理信息能力鉴别存在于各物种的直向同源基因,并选择具有重要生物学功能或价值的候选标记基因进行试验研究,可以迅速实现动物基因组比较信息的物种间转移,发掘更多新的功能基因和定位信息,从而加快对其他物种基因组学研究[19]。
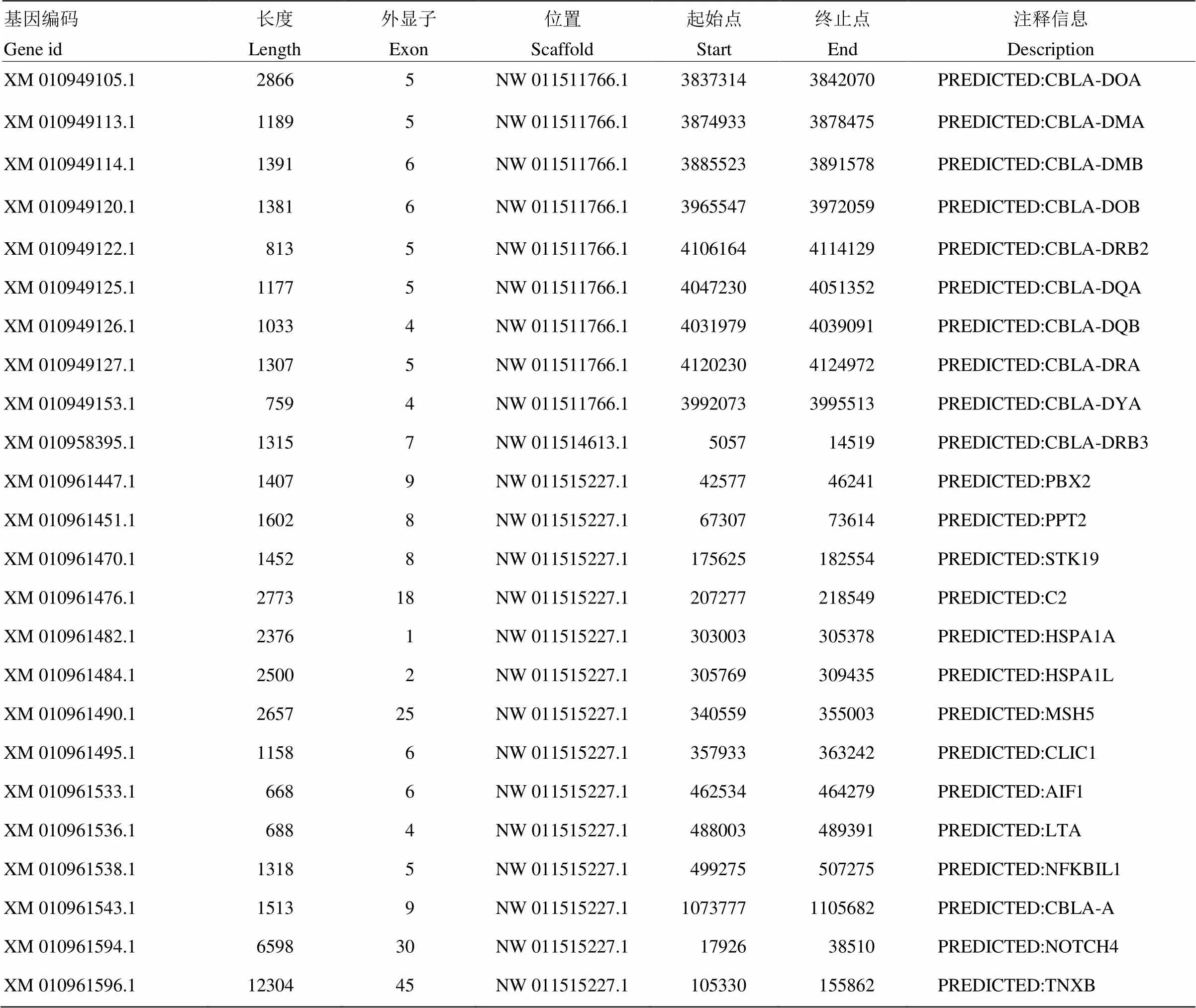
表4 双峰驼CBLA基因注释信息

蓝色线表示CBLA-Ⅰ类基因,红色线表示CBLA-Ⅱ基因,黑色线表示CBLA-Ⅲ类基因
目前,人类已知的HLA-Ⅰ类基因共有106个,其中36个编码基因,其余为假基因;HLA-Ⅱ类基因59个,其中编码基因33个,其余为假基因;HLA-Ⅲ类基因59个,均为编码基因[20-21]。与人类HLA基因数量相比,双峰驼中所注释到的MHC基因数量相差较大[22-23],推测原因可能有以下几点:1)双峰驼参考基因组的组装水平较差;2)双峰驼MHC基因具有低多样性特点[24-25];3)人类与双峰驼亲缘关系较远,基因在物种进化过程中发生改变[26-28]。而与亲缘关系较近的牛BoLA相比,双峰驼MHC基因数量基本一致(表3)。并且,从表3中能够明显观察到牛和双峰驼的MHCⅠ类基因的个数显著少于人类MHCⅠ类基因的个数[29-31]。因此,比较研究双峰驼与人类MHC基因在进化过程中发生的差异将是一个重要的研究课题,这对于进一步揭示双峰驼MHC的作用具有重要意义,也将是今后重点研究的新方向,同时,也将对牛和双峰驼的基因组研究起推动作用。
在对牛BoLA与双峰驼pseudo chromosome 进行共线性分析后发现当纵坐标低于4 000 000时牛BoLA与双峰驼pseudo chromosome 共线性区域出现倒置现象,说明双峰驼CBLA部分基因出现倒置。这与2016年PLASIL通过FISH技术绘制的物理图谱结果相似[11]。因此推测该部分基因出现倒置的原因可能是在物种进化过程中发生倒置改变,这种倒置改变增加了MHC基因的多态性。关于这一现象的发生机制以及意义仍需进一步的研究。
4 结论
本研究运用比较基因组学方法,利用目前已知组装水平最好的人类HLA基因和与双峰驼亲缘关系较近的牛BoLA基因作为种子序列,成功构建了一种新型的用于定位并注释双峰驼的MHC基因的方法。使用该方法找到了存在于双峰驼基因组中的24个MHC基因,其中Ⅰ类基因有1个,Ⅱ类基因有10个,Ⅲ类基因有13个。分别分布在NW_011511766.1(全长4.1M)、NW_011515227.1(全长1.2M)和NW_011514613.1(全长15K)3条scaffolds上,且Ⅰ类和Ⅲ类基因分布在NW_011515227.1上,而Ⅱ类基因分布在NW_011511766.1和NW_011514613.1上,进一步分析得知Ⅱ类基因主要分布在NW_011511766.1 的3.5—4.1M的位置,可绘制双峰驼MHC的Pseudo chromosome长约1.8M,并对双峰驼这24个MHC基因进行了信息注释,为双峰驼MHC的进一步研究奠定了理论基础。
[1] 亢孝珍, 额尔敦木图, 姜建强, 包花尔, 王瑞, 王秀珍, 李盈. 阿拉善驼与苏尼特驼MHC-DRB3exon2基因克隆及序列分析. 内蒙古农业大学学报(自然科学版), 2015(5): 5-11.
KANG X Z, ERDEMTU, JIANG J Q, BAO H, WANG R, WANG X Z, LI Y. Cloning and analysis of sequences of MHC-DRB3exon2 genes in alxa bactrian camel and sunit bactrian camel., 2015(5): 5-11. (in Chinese)
[2] 李文娟, 李岩, 李美玉, 牟凯, 刘思思, 潘庆杰. MHC基因结构及其功能的研究进展. 青岛农业大学学报(自然科学版), 2014(3): 167-171.
LI W J, LI Y, LI M Y, MOU K, LIU S S, PAN Q J. Research Progress of MHC Gene and Its Function., 2014(3): 167-171. (in Chinese)
[3] KUMANOVICS A, TAKADA T, LINDAHL K F. Genomic organization of the mammalian MHC.2003(21): 629-657.
[4] CHAVES L D, KRUETH S B, REED K M. Defining the turkey MHC: sequence and genes of the B locus.2009, 183(10): 6530-6537.
[5] GARBUZ D G, ASTAKHOVA L N, ZATSEPINA O G, ARKHIPOVA I R, NUDLER E, EVGEN′EV M B. Functional organization of HSP70 cluster in camel () and other mammals.2011, 6(11): e27205.
[6] SEQUENCING T B C G, CONSORTIUM A. Genome sequences of wild and domestic bactrian camels., 2012, 3: 1202.
[7] LONGJAM L A, DAS D. Major histocompatibility complex and its importance towards controlling infection.2017, 8(2): 1-13.
[8] 亢孝珍, 额尔敦木图, 姜建强, 陈泽明, 刘图雅, 伊特格勒图, 沙日扣, 图雅. 主要组织相容性复合体(MHC)基因研究进展. 中国畜牧兽医, 2014, 41(05): 28-33.
KANG X Z, ERDEMTU, JIANG J Q, CHEN Z M, LIU T Y, Yitegeltu, Sharhu, TU Y. Research progress on major histocompatibility complex (MHC) gene.2014, 41(05): 28-33. (in Chinese)
[9] KLEIN J. George Snell's first foray into the unexplored territory of the major histocompatibility complex.2001, 159(2): 435-439.
[10] EDWARDS S V, HEDRICK P W. Evolution and ecology of MHC molecules: from genomics to sexual selection., 1998, 13(8): 305-311.
[11] PLASIL M, MOHANDESAN E, FITAK R R, MUSILOVA P, KUBICKOVA S, BURGERE P A, HORIN P. The major histocompatibility complex in Old World camelids and low poly-morphism of its class Ⅱ genes.2016, 17(1): 167.
[12] AVILA F, DAS P J, KUTZLER M, OWENS E, PERELMAN P, RUBES J, HORNAK M, JOHNSON W E, RAUDSEPP T. Development and application of camelid molecular cytogenetic tools., 2014, 105(6): 858-869.
[13] DAVIES C J, ANDERSSON L, ELLIS S A, HENSEN E J, LEWIN H A, MIKKO S, MUGGLI-COCKETT N E, POEL J J, RUSSELL G C. Nomenclature for factors of the BoLA system, 1996: report of the IS AG BoLA Nomenclature Committee., 2015, 28(3): 159-168.
[14] BALMUS G, TRIFONOV V A, BILTUEVA L S, O'BRIEN P C M, ALKALAEVA E S, FU B Y, SKIDMORE J A, ALLEN T, GRAPHODATSKY A S, YANG F T, FERGUSON-SMITH M A. Cross-species chromosome painting among camel, cattle, pig and human: further insights into the putative Cetartiodactyla ancestral karyotype.2007, 15(4): 499-514.
[15] 贾震虎, 夏春. 硬骨鱼类MHCⅠ基因结构及表达研究. 中国兽医杂志, 2008, 44(11): 54-55.
JIA Z H, XIA C. Study on the structure and expression of MHCⅠgene in teleosts.2008, 44(11): 54-55. (in Chinese)
[16] WU H G, GUANG X M, Al-FAGEEH M B, CAO J W, PAN S K, ZHOU H M, Zhang L, ABUTARBOUSH M H, XING Y P, XIE Z Y, ALSHANQEETI A S, ZHANG Y R, YAO Q L, AL-SHOMRANI B M, ZHANG D, LI J, MANEE M M, YANG Z L, YANG L F, LIU Y Y, ZHANG J L, ALTAMMAMI M A, WANG S Y, YU L L, ZHANG W B, LIU S Y, BA L, LIU C X, YANG X K, MENG F H, WANG S W, LI L, LI E L, LI X Q, WU K F, ZHANG S, WANG J Y, YIN Y, YANG H M, AL-SWAILEM A M, WANG J. Camelid genomes reveal evolution and adaptation to desert environments., 2014, 5(5): 5188.
[17] ANTCZAK D. Major histocompatibility complex genes of the dromedary camel. 2013(2013): BIOP 015.
[18] SIDDLE H V, DEAKIN J E, COGGILL P, WHILMING L, HARROW J, KAUFMAN J, BECK S, BELOV K. The tammar wallaby major histocompatibility complex shows evidence of past genomic instability.2011, 12(1): 421.
[19] 潘增祥, 许丹, 张金璧, 林飞, 吴宝江, 刘红林. 基于直向同源序列的比较基因组学研究. 遗传, 2009, 31(05): 457-463.
PAN Z X, XU D, ZHANG J B, LIN F, WU B J, LIU H L. Reviews in comparative genomic research based on orthologs.2009, 31(05): 457-463. (in Chinese)
[20] CONSORTIUM T M S. Complete sequence and gene map of a human major histocompatibility complex.1999, 401(6756): 921-923.
[21] SAMBROOK J G, FIGUEROA F, BECK S. A genome-wide survey of Major Histocompatibility Complex (MHC) genes and their paralogues in zebrafish.2005, 6(1): 152.
[22] KELLEY J, WALTER L, TROWSDALE J. Comparative genomics of major histocompatibility complexes.2005, 56(10): 683-695.
[23] DIDINGER C, EIMES J A, LILLIE M, WALDM B. Multiple major histocompatibility complex class I genes in Asian anurans: Ontogeny and phylogeny.2017, 70: 69-79.
[24] CHAVES L D. The Major Histocompatibility Complex of the Turkey. [D]. Twin Cities: University of Minnesota. 2010.
[25] 郭秀丽, 代红星, 李祥龙, 周荣艳, 王立泽. 不同物种MHC-DQA1基因部分序列的生物信息分析. 中国畜牧兽医, 2007, 34(1): 65-67.
GUO X L, DAI H X, LI X L, ZHOU R Y, WANG L Z. Bioinformatics analysis of part of mhc-dqa1 gene in different species., 2007, 34(1): 65-67. (in Chinese)
[26] BEHL J D, VERMA N K, TYAGI N, MISHRA P, BEHL R, JOSHI B K. The major histocompatibility complex in bovines: A review.2012, (872710): 1-12.
[27] LIAN X D, ZHANG X H, DAI Z X, ZHENG Y T. Characterization of classical major histocompatibility complex (MHC) classⅡgenes in northern pig-tailed macaques ()., 2017, 56: 26-35.
[28] JIANLIN H, MBURU D, OCHIENG J, KAUFMANN B, REGE JE O, HANOTTE O. Application of New World Camelidae microsatellite primers for amplification of polymorphic loci in Old World camelids.2010, 31(6): 404-406.
[29] VILA C, SEDDON J, ELLEGREN H. Genes of domestic mammals augmented by backcrossing with wild ancestors., 2005, 21(4): 214-218.
[30] SATO A, FIGUEROA F, O'HUIGIN C, REZNICK D N, KLEIN J. Identification of major histocompatibility complex genes in the guppy,.1995, 43(1-2): 38-49.
[31] FITAK R R, MOHANDESAN E, CORANDER J, BURGER P A. The de novo genome assembly and annotation of a female domestic dromedary of North African origin.2016, 16(1): 314-324.
(责任编辑 林鉴非)
Mapping and Annotating of Bactrian Camel MHC Gene by Using the Comparative Genomic Approach
ZHI LiKang, Erdemtu, AN XiWen, WANG Chao, WANG Rui, BAO Huar, WANG XiuZhen
(College of Veterinary Medicine, Inner Mongolia Agricultural University/Key Laboratory of Clinical Diagnosis and Treatment Technology in Animal Disease, Ministry of Agriculture P. R. China, Hohhot 010018)
【Objective】The objective of this study was to locate and annotate the major histocompatibility complex (MHC) gene sequence of Bactrian camel in order to provide scientific basis for further study on Bactrian camel MHC gene. 【Method】This study used comparative genomics method. The human MHC (HLA) gene coding sequence and bovine MHC (BoLA) gene coding sequence were extracted, compared with Bactrian camel transcripts on the gene sequences through blastn, to identify the scaffolds with higher similarity. By analyzing the sequence of HLA and BoLA gene sequences on their positions on these scaffolds, multiple pieces of scaffolds were spliced to obtain the Pseudo chromosome of Bactrian camel MHC. Then, the human MHC (HLA) gene coding sequence and bovine MHC (BoLA) gene coding sequence were extracted and analyzed with the spliced scaffolds of Bactrian camels through the genomic collinearity analysis. The selected scaffolds could be judged whether or not it was accurate, based on the linear relationship between Pseudo chromosome established by lastz and HLA and BoLA genome sequences; then by analyzing the linear relationship between MHC genes in the two species, MHC gene sequences were extracted from Bactrian camel genomes, and these sequences were genetically annotated; finally, according to the obtained Bactrian camel MHC gene, the phylogenetic tree was drawn to study the evolutionary relationship between their genes. 【Result】By comparing the HLA and BoLA gene coding sequences with the Bactrian camel transcripts through blastn, three scaffolds with high similarity were identified, namely NW_011511766.1 (full-length 4.1M), NW_011515227.1 (full-length 1.2 M) and NW_011514613.1 (15K in total length), and spliced to obtain Bactrian camel MHC Pseudo chromosome; By using the lastz colinear analysis, the HLA gene sequence and the BoLA gene sequence were identified and compared with MHC gene of the Bactrian camel to obtain the colinear region. It was consistent with the spliced Pseudo chromosome, which proved that the selected scaffolds was accurate. It was found that Class-I and Class-III genes were distributed on NW_011515227.1, while Class-II genes were distributed on NW_011511766.1 and NW_011514613.1. Further analysis revealed that Class-II genes were mainly distributed in NW_011511766.1 3.5 to 4.1M position; the sequences that existed in the collinear region were extracted and subjected to blat analysis, namely aligned with the coding sequence of the MHC gene on the Bactrian camel. Results reveal that a total of 24 genes highly similar to bovine BoLA gene were identified in Bactrian camel genome, including 1 of class I gene, 10 of class II gene and 13 of class III gene. The 24 MHC genes of Bactrian camels were annotated and phylogenetic trees were mapped. The results showed that the annotated Class-I and Class-II genes were on the same branch. 【Conclusion】The method of locating and annotating the MHC gene sequence in Bactrian camel was established by comparative genomics. The MHC gene sequence of Bactrian camel was mapped to three scaffolds, 24 MHC genes were found and annotated, and the Pseudo chromosome of the MHC gene of the Bactrian camel was drawn, which laid the foundation for further study of Bactrian camel MHC gene.
Bactrian camel; MHC; CBLA; BoLA; HLA; comparative genomic
10.3864/j.issn.0578-1752.2018.18.015
2017-12-13;
2018-07-06
国家自然科学基金(31360591)
支立康,E-mail:15849121059@163.com。通信作者额尔敦木图,Tel:0471-4309179;E-mail:eedmt@imau.edu.cn
