基于染色体片段置换系的野生稻粒长QTL——qGL12的精细定位
2018-10-11丁膺宾张莉珍许睿王艳艳郑晓明张丽芳程云连吴凡杨庆文乔卫华兰进好
丁膺宾,张莉珍,许睿,王艳艳,郑晓明,张丽芳,程云连,吴凡,杨庆文,乔卫华,兰进好
基于染色体片段置换系的野生稻粒长QTL——的精细定位
丁膺宾1,2,张莉珍1,2,许睿2,王艳艳2,郑晓明2,张丽芳2,程云连2,吴凡2,杨庆文2,乔卫华2,兰进好1
(1青岛农业大学农学院,山东青岛 266109;2中国农业科学院作物科学研究所,北京 100081)
【目的】利用野生稻染色体片段置换系以及次级群体,精细定位与水稻粒长相关的QTL,发掘野生稻中影响粒长的新基因,为水稻育种提供遗传材料和基因资源。【方法】利用中国农业科学院作物科学研究所野生稻实验室已构建的野生稻染色体片段置换系群体,定位到一个与粒长相关的QTL——。在此基础上,选择粒长与受体亲本9311差异显著,且携带片段的置换系CSSL141与9311回交构建次级分离群体,设计区间内分子标记引物,筛选交换单株,结合粒型表型分析与基因型鉴定,对进行精细定位。通过扫描电子显微镜检测颖壳细胞,观察细胞的长宽变化,对定位区间内的基因进行基因注释以及测序分析,预测候选基因。【结果】根据整套置换系多个环境下的表型鉴定,初定位于第12染色体标记RM28621附近;选取携带的置换系CSSL141作为精细定位的亲本,CSSL141携带4个野生稻导入片段,在多年多点的田间试验中,CSSL141粒长、粒宽、粒重均明显高于9311;利用构建的CSSL141/9311 F2群体将定位于第12染色体标记RM5479与RM28621之间,影响粒长、粒宽以及粒重,对粒长的贡献率最高,为44.61%。在定位区间内设计了7个多态性分子标记引物,选择目标区间基因型杂合的植株种植F3,通过筛选交换单株,结合交换单株的基因型与表型,将定位于RM5479与RM28586之间50 kb区间内,为进一步缩短定位区间,在此区间内设计了4个多态性分子标记引物,选择交换单株种植下一代,筛选后得到20株交换单株,结合交换单株基因型与籽粒表型,最终将定位到第12染色体15.69 kb区间,该区间内有3个候选基因,其中编码一种微管蛋白,编码一种质膜钙转运ATP酶,尚未有明确功能,通过测序分析发现,、在编码区内存在变异;对亲本以及后代交换单株的颖壳细胞进行电镜扫描,发现9311颖壳细胞的长度与宽度均比CSSL141小CSSL141/9311 F4代群体中,目标区间为野生稻基因型的交换单株颖壳细胞的长度与宽度均比目标区间为9311基因型的交换单株大,表明通过调控颖壳细胞的大小影响水稻粒长。【结论】利用野生稻染色体片段置换系,将野生稻粒长QTL——定位于第12染色体15.69 kb区间内,通过调控水稻颖壳细胞的大小影响粒长。该区间内有3个基因。来自野生稻的与与栽培稻等位基因相比,存在自然变异,确定为的候选基因。
野生稻;染色体片段置换系;粒型;QTL定位
0 引言
【研究意义】普通野生稻中含有水稻驯化过程中丢失或者削弱的大量优异基因;粒长是水稻首选的育种目标之一。利用以普通野生稻为供体亲本构建的染色体片段置换系进行粒长QTL定位,可以挖掘出野生稻中的优异基因,为水稻育种以及粒长形成的分子机理研究提供新的基因资源。【前人研究进展】水稻是世界上重要的粮食作物之一[1]。中国是稻米消耗大国,65%以上的人口以大米为主食[2]。提高水稻产量对确保中国粮食安全和社会稳定发展有重要意义。水稻产量主要由3个因素构成:单株穗数、每穗粒数和粒重。粒重指标由粒长、粒宽、长宽比和粒厚来综合评价,这四个性状与千粒重成正相关[3-4]。因此,籽粒形状是水稻首选的育种目标之一。与大多数农艺性状一样,水稻粒型是受多基因控制的数量性状[5-6],它不但影响水稻的产量,还影响加工品质与外观品质。对籽粒形状相关数量性状位点(quantitative trait locus,QTL)的鉴定以及新基因的发掘是水稻育种与遗传研究的重要目标。随着分子生物学技术以及现代遗传学的发展,许多研究者通过全面解析粒型相关的遗传机制和挖掘功能基因/QTL位点,定位了越来越多的粒型QTL,并克隆出一些控制粒型性状的重要基因。林鸿宣等[7]利用RFLP图谱对控制籼稻粒型性状QTL进行定位,在特三矮2号/CB1128群体中定位到5个控制粒长QTL;在外引2号/CB1128群体中定位到5个控制粒长QTL,5个控制粒宽QTL。近年来,很多研究通过构建染色体片段置换系进行水稻优异基因的挖掘定位。周丽慧等[8]通过9311/日本晴CSSL群体定位到10个QTL,与单株产量、单株实粒数、千粒质量、每穗颖花数、结实率、单株总颖花数6个性状相关。朱文银等[9]利用染色体片段置换系定位到3个控制水稻粒型的主效QTL,其中有2个粒长相关的QTL,分别为和。被定位在水稻第3染色体RM5551与RM6832之间,遗传距离为14.8 cM;被定位在RM6832与RM3513之间,其遗传距离为5.3 cM。随着第一个控制水稻粒型的基因3[10]被克隆,许多控制粒型的基因如2[11]、2[12]、2[13]、3.1[14]、5[15]、5[16]、7[17]和[18]等相继被克隆,这些研究为理解调控粒型的机理提供了理论基础,也对作物的遗传改良具有重要意义。【本研究切入点】目前,很多研究都是以栽培稻为研究对象,被挖掘出的QTL大多来源于栽培稻,对于野生稻中优异基因的发掘鲜见报道。野生稻异质性强,构建合适的遗传群体是对其利用的关键。构建一套可以永久性利用的染色体片段群体,不仅建立了一个野生稻基因组学研究的平台,还可以为水稻育种提供新的种质资源。【拟解决的关键问题】本研究以海南普通野生稻为供体亲本,9311为受体亲本,利用中国农业科学院作物科学研究所野生稻实验室已构建的染色体片段置换系群体,通过多年多点表型鉴定试验,选择粒型差异明显的置换系构建次级群体,设计分子标记结合基因型与表型鉴定进行精细定位,发掘与水稻粒长相关的QTL,寻找野生稻中影响粒长的新基因。
1 材料与方法
1.1 试验材料
以中国农业科学院作物科学研究所野生稻实验室构建的一套野生稻染色体片段置换系为研究材料。以来自海南的普通野生稻材料为供体亲本,栽培稻品种9311为受体亲本,使用119个SSR标记和62个Indel标记,通过多代回交与分子标记辅助选择,构建出覆盖水稻染色体的200个染色体片段置换系[19]。齐兰等[19]利用构建好的置换系群体定位到了37个粒型相关QTL,其中粒长相关QTL——定位于RM28621标记附近,且能在4个环境中检测到。选择粒长与受体亲本9311差异显著,且携带片段的置换系CSSL141与9311回交,构建次级分离群体,分别于2016年南京、2016年三亚、2017年北京和2017年南京进行种植进行粒长QTL——的精细定位。
1.2 田间种植
2016年5月在南京基地种植200株CSSL141/9311 F2群体作为初定位群体,10月单株收获籽粒检测籽粒表型。从初定位结果中挑选出在定位区间内存在基因型差异,而其他区间的基因型为父本或母本的纯合基因型的单株于2016年12月在海南试验基地种植得到400株F3群体,2017年4月收获F3单株,筛选交换单株缩短定位区间,2017年5月从F3中选择目标区间杂合基因型的种子在北京、南京试验基地种植,分别得到2 400株、1 200株F4群体,2017年10月单株收获F4群体籽粒。田间种植规格为,行距16 cm×26 cm,每行种植10株。
1.3 表型鉴定
水稻成熟后单株收获种子,自然晒干,籽粒的表型鉴定均用万深SC-G自动考种及千粒重仪(杭州万深检测科技有限公司)进行,测定籽粒的粒长、粒宽以及千粒重。每份材料2次重复,每次测量150—200粒种子。
1.4 群体基因型鉴定
所用的SSR引物来源于美国Cornell大学公布的研究成果,定位区间内引物的设计是根据已经测序完成的日本晴和9311的序列信息,使用primer5软件进行引物设计,在Gramene网站进行在线比对,验证其所在染色体位置,确保标记的准确性。利用PCR扩增检验在两亲本间的多态性,选择多态性良好的引物作为精细定位的标记引物。试验自行设计的定位引物序列见表1。
1.5 粒型性状QTL定位
根据SSR分析的结果,与亲本CSSL141一致的带型记为2,与9311带型一致的带型记为0,同时具有两亲本的杂合带型记为1,由于某种原因造成带型缺失或模糊的记为-1。用QTL Ici-Mapping V4.1软件中的完备区间-加性模型(ICIM-ADD)定位方法,扫描步长为1 cM,以LOD值2.5作为阀值,在全基因组范围内进行扫描,判断可能存在的控制粒型性状的QTL,QTL的命名遵循McCouch等的原则。
1.6 颖壳细胞扫描电镜观察
选取9311、CSSL141籽粒,F4群体中定位区间基因型分别为野生稻、9311植株的籽粒,将颖壳分离,剪取中间部分的外颖,固定后将颖壳外颖进行喷金镀膜,使用扫描电镜观察颖壳外表面表皮细胞,检测颖壳细胞的长度与宽度。
1.7 候选基因预测
根据QTL精细定位结果,利用Rice Genome Browser网站(http://rice.plantbiology.msu.edu/cgi-bin/ gbrowse/rice/#search)查找区间内的所有基因,通过Rice Genome Annotation Project对基因注释进行分析,筛选出与目标性状相关的候选基因。在NCBI数据库(https://www.ncbi.nlm.nih.gov/)中下载候选基因序列,用Primer5设计测序引物对候选基因进行扩增、测序。比较候选基因在9311与CSSL141间的序列差异。

表1 用于精细定位的引物序列
2 结果
2.1 亲本CSSL141的选择以及基因型与表型分析
利用染色体片段置换系群体,在多个环境下进行了粒型QTL的定位[19]。在第12染色体RM28621标记附近检测到一个能够在多个环境下稳定表达,与粒长相关的QTL,命名为(表2)。选择携带片段且粒型与9311差异显著的置换系CSSL141作为亲本,进行下一步的精细定位。CSSL141染色体基因型如图1所示,携带4个来自野生稻的置换片段,1个片段位于第2染色体,3个片段位于第12染色体,其中第12染色体上的一个置换片段位于RM28621标记附近(图1)。
根据整套置换系材料在2014—2016年3年不同环境中的调查结果,发现CSSL141和亲本9311在粒型上有明显差异(图2-a和图2-b)。其中,9311在北京、南京、三亚粒长均值分别为9.61、9.77和9.35 mm,CSSL141粒长均值分别为10.10、10.19和9.90 mm;9311千粒重均值分别为31.22、32.60和32.22 g,CSSL141千粒重均值分别为33.83、35.04和35.08 g;粒长、千粒重差异均达到极显著水平(<0.001)。 9311粒宽均值在北京、南京、三亚分别为2.79、2.79和2.83 mm,CSSL141粒宽均值分别为2.82、2.86和2.93 mm(图2-c)。此外,CSSL141与9311在株高、穗长、有效穗数等农艺性状方面差异不显著。CSSL141中置换片段较少,籽粒表型与9311差异较大而其他农艺性状与9311没有显著差异,而且其置换片段中含有实验室已经定位到的QTL——,因此选为亲本进行的精细定位。
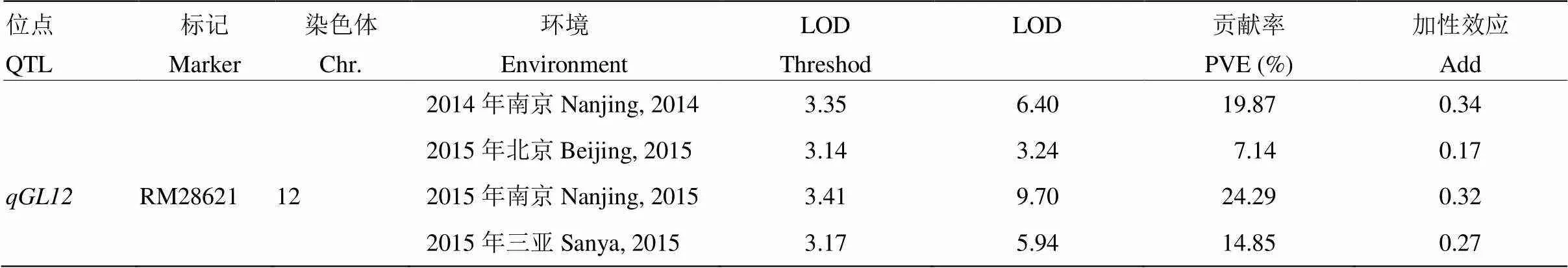
表2 不同环境下qGL12的检测
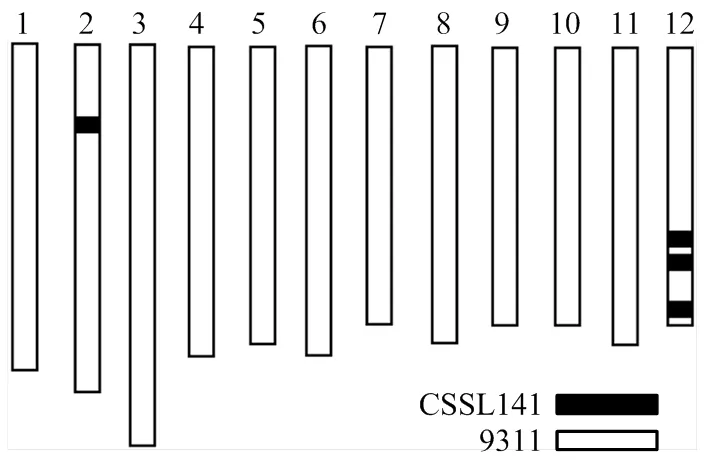
图1 CSSL141置换片段分布
2.2 F2群体定位结果
CSSL141/9311 F2群体种植于南京试验基地。粒长、粒宽以及千粒重3个粒型性状在F2群体的分布均呈连续正态分布(图3)。F2群体粒长介于9.58—10.80 mm,主要集中在9.81—10.27 mm;粒宽介于2.57—2.84 mm,主要集中在2.68—2.72 mm;千粒重介于30.50—36.63 g,主要集中在32.76—35.12 g。说明这三个性状是由多基因调控,属于数量遗传性状,适合QTL分析。
根据CSSL141染色体基因组中野生稻置换片段的分布,使用置换片段区间或周围的分子标记设引物RM5338、RM5479、RM28621、RM28642、Indel12-12作为初定位标记引物。利用QTL IciMapping分析软件将定位于第12染色体标记RM5479—RM28621,影响粒长、粒宽以及粒重。对粒长的贡献率最高,为44.61%(表3)。
2.3 qGL12精细定位
利用F2群体将定位于第12染色体标记RM5479与RM28621之间。F2群体中选择两标记基因型杂合的植株进行种植得到F3。在定位区间RM5479—RM28621两边及中间增加7个多态性分子标记引物,分别为RM28583、RM28586、RM28590、RM28591、RM28597、RM28607和RM28642。对400株海南F3单株进行基因型分析,筛选出18株交换单株。结合交换单株粒长表型与基因型,将定位于RM5479和RM28586之间,区间长度约为50 kb(图4和图5)。
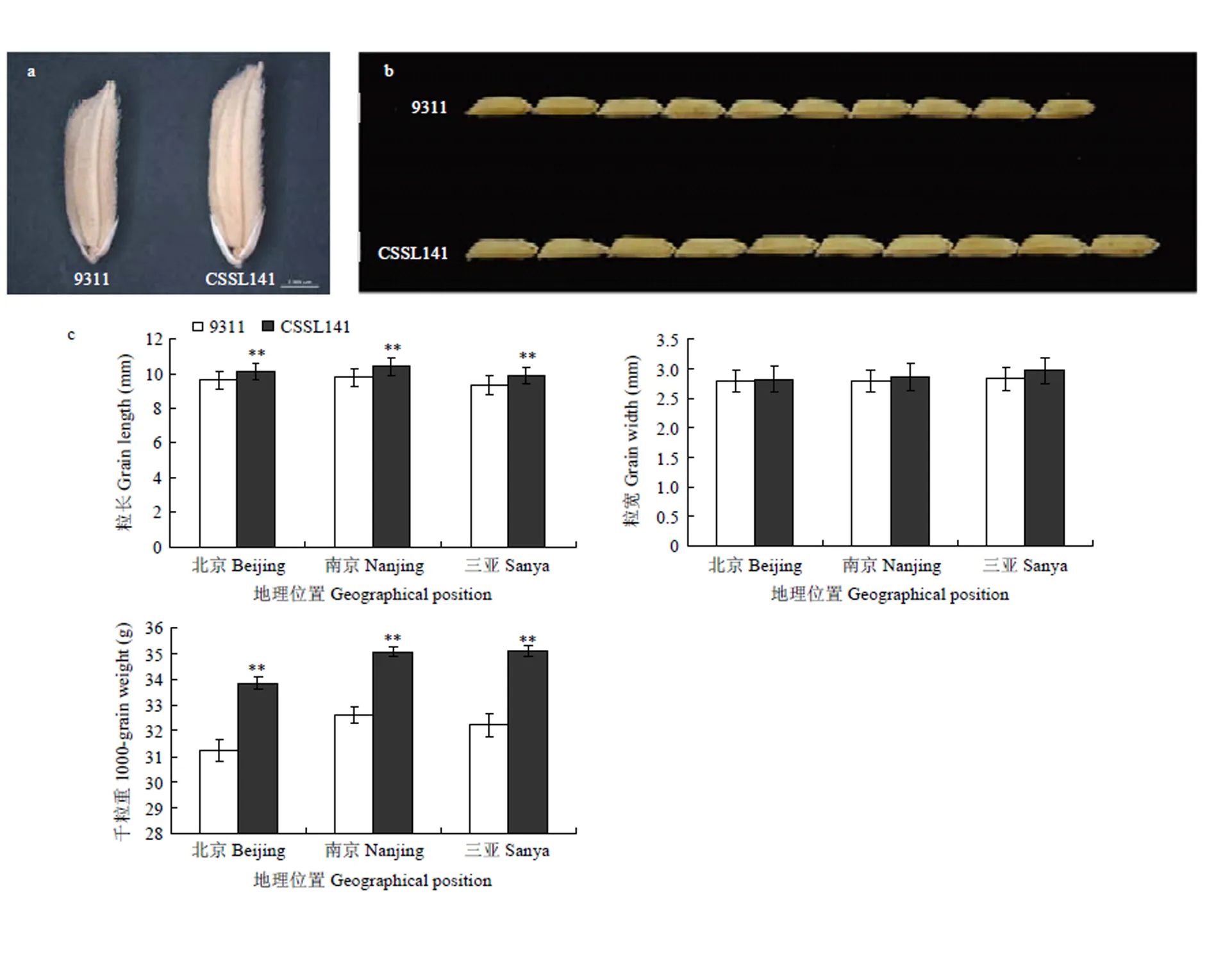
a:单粒种子;b:粒长比较;c:不同环境下的粒型性状比较。**差异极显著(<0.001)
a: The seeds of 9311 and CSSL141; b: 10 Comparison of grain length; c:Comparison of grain traits under multiple environments. **significantly different at<0.001
图2 9311与CSSL141籽粒性状比较
Fig. 2 Comparison of kernel traits between 9311and CSSL141
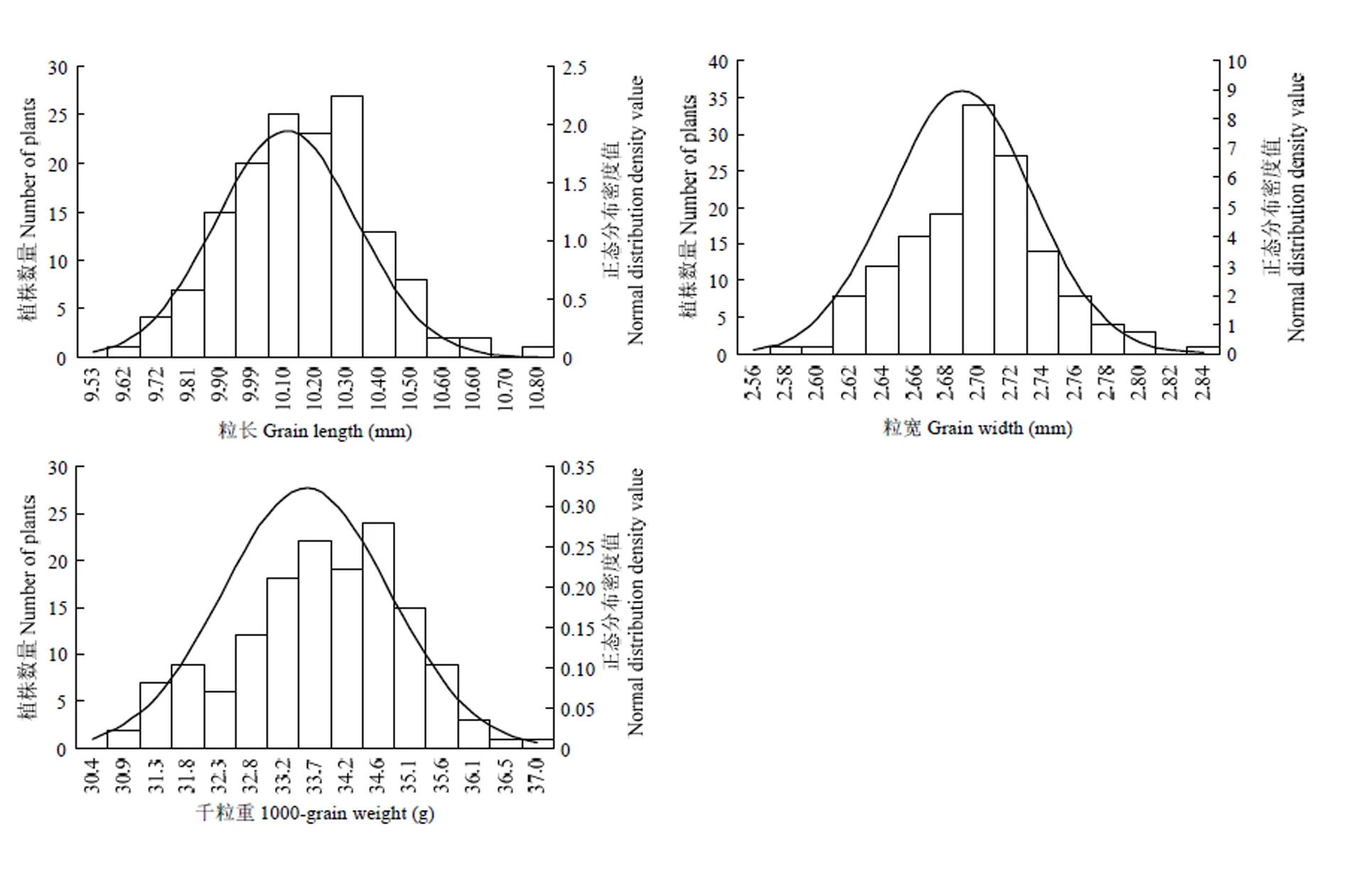
图3 F2群体粒长、粒宽、千粒重频次分布图
Fig. 3 Frequency distribution diagram of grain length, grain width and 1000 grain weight in CSSL141/9311 F2population
表3 CSSL141/9311 F2群体粒型QTL初定位结果
为了进一步精细定位,将F3群体中目标区间基因型杂合的单株种子分别在北京顺义、南京进行种植得到F4。在NCBI数据库(https://www.ncbi. nlm.nih.gov/)中找到RM5479与RM28586区间片段的序列,利用Primer5软件设计分子标记引物,设计好的引物在两亲本间进行多态性检测,最后得到4个多态性良好的分子标记引物DYB1.1、DYB6.1、DYB7.1和DYB9.1(表1)。对2 400株种植于北京顺义的F4单株以及1 200株种植于南京的F4单株进行定位分析,南京和北京的群体都在RM5479和RM28586之间发现粒长相关的QTL。通过筛选交换单株,得到20株交换单株,结合基因型与表型的结果,根据交换单株信息,将的左边界定于DYB9.1标记处,右边界定于RM28586,区间长度约15.69 kb(图4和图6)。
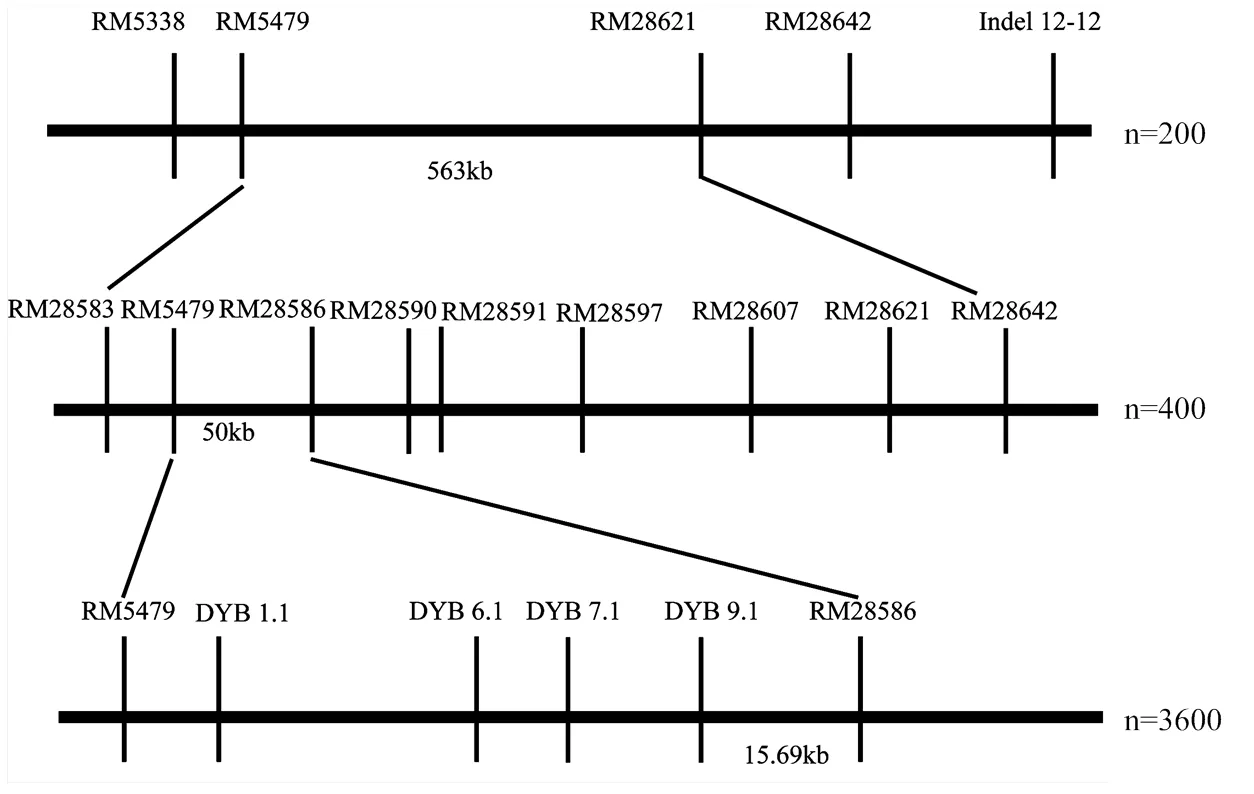
图4 qGL12的精细定位
2.4 颖壳细胞学观察
取同一时期、同一环境下9311与CSSL141的籽粒,以及F4群体中2个交换单株的籽粒,2个交换单株目标区间染色体片段分别为野生稻基因型以及9311基因型。扫描电镜观察颖壳的外表皮细胞。结果显示,在400×的视距下,亲本CSSL141的平均细胞长度为40.82 μm(图7-a);9311平均细胞长度为34.48 μm(图7-b);F4交换单株籽粒中标记区间为野生稻基因型的籽粒平均细胞长度为46.51 μm(图7-c);标记区间为9311基因型的交换单株籽粒平均细胞长度为35.71 μm(图7-d)。因此推断通过调控水稻颖壳细胞大小控制籽粒形状。
2.5 候选基因预测
最终定位于第12染色体15.69 kb的区间,两侧标记为DYB9.1和RM28586。根据Rice Genome Browser网站(http://rice.plantbiology.msu.edu/cgi-bin/ gbrowse/rice/#search)序列分析,该区间存在3个候选基因,分别为、和。
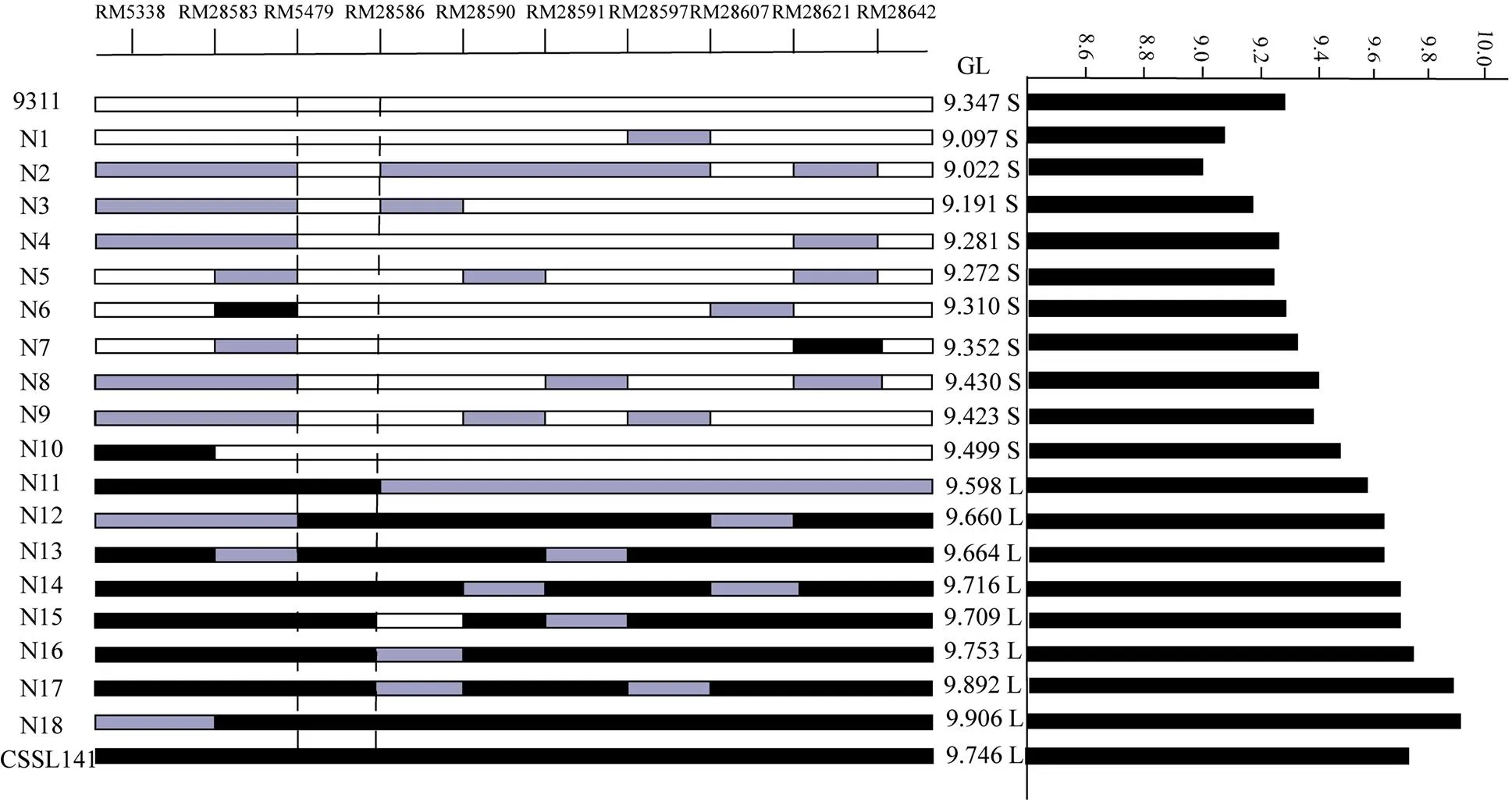
S:短粒;L:长粒;白色区域:9311基因型;黑色区域:野生稻基因型;灰色区域:杂合基因型。下同

图6 RM5479与RM28586区间内交换单株基因型与表型
Rice Genome Annotation Project网站基因注释为:编码一种微管蛋白,编码一种质膜钙转运ATP酶,尚未有明确功能(表4)。将9311与CSSL141中3个候选基因的测序结果进行对比,在两亲本编码区中有8个SNP,其中5个导致氨基酸发生改变;第403个碱基中T/A碱基发生替换导致半胱氨酸替换成丝氨酸;第427个碱基中G/T碱基发生替换导致天冬氨酸替换成酪氨酸;第460个碱基中G/A碱基发生替换导致缬氨酸替换成异亮氨酸;第1 450个碱基中G/A碱基发生替换导致丙氨酸替换成苏氨酸;第1 672个碱基中T/C碱基发生替换导致半胱氨酸替换成精氨酸。在两亲本编码区中有17个SNP,其中有2个导致氨基酸发生改变;第433个碱基中T/C碱基发生替换导致半胱氨酸替换成精氨酸;第2 754个碱基中T/A碱基发生替换导致天冬氨酸替换成谷氨酸。

a:亲本CSSL141;b:亲本9311;c:标记区间为野生稻基因型的交换单株;d:标记区间为9311基因型的交换单株

表4 qGL12定位区间内候选基因
3 讨论
3.1 染色体片段置换系在水稻QTL定位中的应用
传统的QTL定位一般采用的是初级定位群体,例如RIL等,受遗传背景的影响,QTL定位的区间都很大,通常是10 cM以上,难以确定 QTL的作用效应是单个主效QTL的作用还是多个微效QTL的作用。对于精细定位,最关键的环节就是构建合适的定位群体,筛选与目标基因紧密连锁的分子标记。理想的定位群体是除了目标基因所在座位的局部区间外,其他部分的遗传背景是相同的[20]。染色体片段置换系仅在置换片段上发生分离,其余遗传背景与受体亲本完全一致,因此,消除了遗传背景的干扰,尤其是能够检测出遗传效应较小的QTL,是QTL定位的理想工具[21]。
本研究中,粒型QTL的定位是建立在染色体片段置换系多年多点田间试验的基础上,利用构建的置换系群体定位到了37个粒型相关QTL,其中粒长相关的QTL在2年3点多环境试验中在4个环境中均被检测到,说明是可以在不同环境下稳定遗传的QTL。从CSSL群体中挑选粒型差异与9311较大而且携带置换片段的CSSL141与9311进行回交,通过F2、F3以及F4群体进行精细定位。最终将定位于第12染色体15.69 kb的区间,影响粒长、粒宽以及粒重,对粒长的贡献率最高。利用野生稻染色体片段置换系进行精细定位,能够充分消除遗传背景的影响,发掘野生稻中的优异基因,将成为一种主流方法。
3.2 qGL12通过调控颖壳细胞大小影响粒长
稻米的形状和大小受到颖壳形状和大小的严格控制,这表明颖壳的形态影响水稻的外观品质和产量,而颖壳的形态则是由细胞的大小、数目以及排列方式决定的。基因影响水稻粒型的方式可能有两种,一种为影响颖壳细胞的数量。例如[22]通过改变细胞分裂模式来控制籽粒形状,使纵向细胞分裂增加横向细胞分裂减少形成细长的籽粒。一种为影响颖壳细胞的大小。如[23]通过调控颖壳细胞的长度与宽度影响水稻粒型;[24]和[25]主要通过影响颖壳细胞的长度影响籽粒形状。本研究利用扫描电镜对水稻9311、CSSL141以及2个F4交换单株进行水稻颖壳细胞观察,发现9311颖壳细胞的长度与宽度均比CSSL141小,2个交换单株中,目标区间为野生稻基因型的交换单株颖壳细胞的长度与宽度均比目标区间为9311基因型的交换单株大,这表明通过调控细胞的长度与宽度影响水稻粒型。
3.3 qGL12应用前景
区间内含有3个基因,3个基因均未见报导与水稻粒长有关,测序结果表明2个候选基因在编码区均有非同义突变。下一步的研究将利用转基因等方法对2个候选基因进行功能验证,确定野生稻中控制粒长的基因。置换系CSSL141,或者CSSL141/9311 F4群体中携带的株系,可以直接作为水稻品种改良的育种材料,DYB9.1与RM28586可以作为分子标记用于辅助选择。本研究将为水稻粒长形成的分子机理研究提供新的基因资源,同时,为水稻粒长的改良提供了新的育种材料。
4 结论
被定位于第12染色体15.69 kb的区间,两侧标记分别为DYB9.1和RM28586。区间内与2个基因在编码区均有非同义突变,被确定为的候选基因,并且通过影响水稻籽粒颖壳细胞的大小影响粒长。
[1] 景春艳, 张富民, 葛颂. 水稻的起源与驯化——来自基因组学的证据. 科技导报, 2015, 33(16): 27-32.
Jing C Y, Zhang F M, Ge S. Genomic evidence of the origin and domestication of Asian cultivated rice (L.)., 2015, 33(16): 27-32. (in Chinese)
[2] Khush G S. What it will take to feed 5.0 billion rice consumers in 2030., 2005, 59: 1-6.
[3] 涂坦, 付洪. 水稻主要粒型基因及其遗传调控的研究进展. 山地农业生物学报, 2016, 35(5): 58-65.
Tu T, Fu H. Research progress in major genes controlling grain shape of rice and their genetic regulation., 2016, 35(5): 58-65. (in Chinese)
[4] 梁云涛, 潘英华, 徐志健. 利用野栽分离群体定位水稻粒型相关QTL. 西南农业学报, 2017, 30(10): 2161-2167.
Liang Y T, Pan Y H, Xu Z J. QTLs mapping of grain shape of rice by using offspring derived from cross ofGriff. and cultivated rice., 2017, 30(10): 2161-2167. (in Chinese)
[5] 邢永忠, 谈移芳, 徐才国, 华金平, 孙新立. 利用水稻重组自交系群体定位谷粒外观性状的数量性状基因. 植物学报, 2001(8): 840-845.
Xing Y Z, Tan Y F, Xu C G, Hua J P, Sun X L. Mapping quantitative trait loci for grain appearance traits of rice using a recombinant inbred line population., 2001(8): 840-845. (in Chinese)
[6] 余守武, 樊叶杨, 杨长登, 李西明. 水稻第1染色体短臂粒长和粒宽QTL的精细定位. 中国水稻科学, 2008(5): 465-471.
Yu S W, Fan Y Y, Yang C D, Li X M. Fine mapping of quantitative trait loci for grain length and grain width on the short arm of rice chromosome1., 2008(5): 465-471. (in Chinese)
[7] 林鸿宣, 闵绍楷, 熊振民, 钱惠荣, 庄杰云, 陆军, 郑康乐, 黄宁. 应用RFLP图谱定位分析籼稻粒形数量性状基因座位. 中国农业科学, 1995(4): 1-7.
Lin H X, Min S K, Xiong Z M, Qian H R, Zhuang J Y, Lu J, Zheng K L, Huang N. RFLP mapping of QTLs for grain shape traits in indica rice., 1995(4): 1-7. (in Chinese)
[8] 周丽慧, 张亚东, 朱镇, 陈涛, 赵庆勇, 姚姝, 赵凌, 赵春芳, 于新, 王才林. 利用染色体片段置换系群体检测水稻产量相关性状QTL. 江苏农业学报, 2015, 31(1): 1-9.
Zhou L H, Zhang Y D, Zhu Z,CHEN T, ZHAO Q Y, YAO S, ZHAO L, ZHAO C F, YU X, WANG C L. Quantitative trait locus detection for rice yield-related traits using chromosome segment substitution lines., 2015, 31(1):1-9. (in Chinese)
[9] 朱文银, 杨德卫, 林静, 赵凌, 张亚东, 朱镇, 陈涛, 王才林. 利用染色体片段置换系定位水稻粒型QTL. 江苏农业学报, 2008, 3: 226-231.
Zhu W Y, Yang D W, Lin J, Zhao L, Zhang Y D, Zhu Z, Chen T, Wang C L. Substitution mapping of QTLs for grain shape using chromosome segment substitution lines in rice(L.)., 2008, 3: 226-231. (in Chinese)
[10] Fan C C, Xing, Y Z, Mao H L, Lu T T, Han B, Xu C G, Li X H, Zhang Q F., a major QTL for grain length and weight and minor QTL for grain width and thickness in rice, encodes a putative transmembrane protein., 2006, 112(6): 1164-1171.
[11] Hu J, Wang Y X, Fang Y X, Zeng L J, Xu J, Yu H P. A rare allele ofenhances grain size and grain yield in rice., 2015, 10(8): 1455-1465.
[12] Song X J, Huang W, Shi M, Zhu M Z, Lin H X. A QTL for rice grain width and weight encodes a novel RING-type E3 ubiquitin ligase., 2007, 39(5): 623- 630.
[13] Che R H, Tong H M, Shi B H, Liu Y Q, Fang S R, Liu D P. Control of grain size and rice yield by-mediated brassinosteroid responses., 2015, 2(1): 15195.
[14] Qi P, Lin Y S, Song X J, Shen J B, Huang W, Shan J X, Zhu M Z, Jiang L W, Gao J P, Lin H X. The novel quantitative trait locuscontrols rice grain size and yield by regulating Cyclin- T1; 3., 2012, 22(12): 1666-1680.
[15] Weng J F, Gu S H, Wan X Y, Gao H, Guo T, Su N, Lei C L, Zhang X, Cheng Z J, Guo X P, Wang J L, Jiang L, Zhai H Q, Wan J M. Isolation and initial characterization of, a major QTL associated with rice grain width and weight., 2008, 18(12): 1199-1209.
[16] Li Y B, Fan C C, Xing Y Z, Jiang Y H, Luo L J, Sun L, Shao D, Xu C J, Li X H, Xiao J H, He Y Q, Zhang Q F. Natural variation inplays an important role in regulating grain size and yield in rice., 2011, 43(12): 1266-1269.
[17] Wang Y X, Xiong G S, Hu J, Jiang L, Yu H, Xu J, Fang Y X, Zeng L J, Xu E B, Xu J, Ye W J, Meng X B, Liu R F, Chen H Q, Jing Y H, Wang Y H, Zhu X D, Li J Y, Qian Q. Copy number variation at thelocus contributes to grain size diversity in rice., 2015, 47(8): 944-948.
[18] Wang S K, Wu K, Yuan Q B,LIU X Y, LIU Z B, LIN X Y, ZENG R Z, ZHU H T, DONG G J, QIAN Q, ZHANG G Q, FU X D. Control of grain size, shape and quality by, 2012, 44(8): 950-954.
[19] Qi L, Sun Y, Li J, SU L, ZHENG X M, WANG X N, LI K M, YANG Q W, QIAO W H. Identify QTLs for grain size and weight in common wild rice using chromosome segment substitution lines across six environments., 2017, 472: 482.
[20] 朱文银, 王才林. 作物染色体片段置换系研究进展. 江苏农业学报, 2008, 24(6): 963-968.
Zhu W Y, Wang C L. A review on chromosomal segment substitution lines in crops., 2008, 24(6): 963-968. (in Chinese)
[21] Qiao W H, Qi L, Cheng Z J, SU L, LI J, SUN Y, REN J F, ZHENG X M, YANG Q W. Development and characterization of chromosome segment substitution lines derived fromin the genetic background ofspp.cultivar 9311., 2016, 17: 580.
[22] Wang S K, Li S, Liu Q, Wu K, Zhang J Q, WANG S S, WANG Y, CHEN X B, ZHANG Y, GAO C X, WANG F, HUANG H X, FU X D. Theregulatory module determines grain shape and simultaneously improves rice yield and grain quality., 2015, 23(3): 12.
[23] Abe Y, MieDa K, Ando T, KONO I, YANO M, KITANO H, IWASAKI Y. The SMALL AND ROUND SEED1 (SRS1/DEP2) gene is involved in the regulation of seed size in rice., 2009, 85(5): 327-339.
[24] Segami S, Kono I, Ando T, YANO M, KITANO H, MIURA K, IWASAKI Y.gene encodes alphatubulin regulating seed cell elongation in rice., 2012, 5(1): 1-10.
[25] Heang D, Sassa H. Antagonistic actions of HLH/bHLH proteins are involved in grain length and weight in rice., 2012, 7(2): e31325.
(责任编辑 李莉)
Fine Mapping of Grain Length Associated QTL,(L.) Using a Chromosome Segment Substitution Line
Ding Yingbin1,2, Zhang Lizhen1,2, Xu Rui2, Wang Yanyan2, Zheng Xiaoming2, Zhang Lifang2, Cheng Yunlian2, Wu Fan2, Yang Qingwen2, Qiao Weihua2, Lan Jinhao1
(1College of Agriculture of Qingdao Agricultural University, Qingdao 266109, Shandong;2Institute of Crop Sciences, Chinese Academy of Agricultural Sciences, Beijing 100081)
【Objective】Fine mapping of a grain length QTL from wild rice using a chromosome segment substitution line and secondary population, exploring new genes affecting grain length and providing genetic materials and gene resources for rice breeding.【Method】Our laboratory had preliminary mapped a grain length related QTL,using the chromosome segment substitution line (CSSL) population. On this basis, we choose one CSSL, CSSL141 which harborssubstitution segment and has significant difference of grain length compared with receptor parent 9311. CSSL141 was backcrossed with 9311. The secondary separation population was constructed for fine mapping of. Detection of length of glumes cells was performed by scanning electron microscope.【Result】CSSL141 has 4 introgressive segments from wild rice, its grain length, grain width and grain weight was significantly higher than 9311 under multi-conditions. Using CSSL141/9311 F2population,was localized to the interval between RM5479 and RM28621 on chromosome 12.affected grain length, grain width and grain weight, and the phenotypic variation explained of grain length was 44.61%. Seven polymorphic molecular marker primers in the location interval were designed, F3plants which had heterozygous genotype in the target interval were investigated.was mapped to a 50 kb region between RM5479 and RM28586. Four polymorphic molecular marker primers were designed in this interval, individuals which harbor heterozygous genotype in this interval were selected for next generation F4. Eventuallywas narrowed to a 15.69 kb region between DYB9.1 and RM28586 on chromosome 12. There are 3 genes in the interval. Two candidate genes,and, which encode a tubulin protein and a calcium-transporting ATPase respectively, with variations in their coding regions. The results of electron microscopes scanning of glumes cell showed that the length and width of 9311’s glumes cell was smaller than CSSL141, indicated thatregulate grain size of rice by controlling the size of glumes cells.【Conclusion】The wild rice grain length associated QTLwas fine mapped to a 15.69 kb region on chromosome12.control grain length via regulate the size of glumes cells. Two candidate genes,and, were found and would be used for further research.
rice; chromosome segment substitution line; grain size; QTL mapping
10.3864/j.issn.0578-1752.2018.18.001
2018-04-11;
2018-05-26
国家自然科学基金(31471471)、作物生物学国家重点实验室开放课题(2016KF08)、中国农业科学院创新工程、国家重点研发计划(2016YFD0100101)
丁膺宾,E-mail:913299099@qq.com。通信作者杨庆文,E-mail:yangqingwen@caas.cn。通信作者乔卫华,Tel:010-62186687;E-mail:qiaoweihua@caas.cn。通信作者兰进好,E-mail:jinhao2005@163.com
