串联重复序列在克莱门柚基因组中的特征研究
2018-09-11赵志新
赵志新,张 蒙
(1.商洛学院生物医药与食品工程学院,陕西 商洛 726000;2.福建农林大学生命科学学院,福建 福州 350000)
【研究意义】克莱门柚(Citrusclementina) 别名“文旦”,为芸香科柑橘属植物[1]。果皮甚厚而光滑,果肉酸甜可口,维生素C含量丰富,兼营养、食用、药用、加工等多种功效,是南方重要的经济热带水果,主产于福建省漳州、厦门,我国柚类种植面积和产量居世界首位[2]。【前人研究进展】克莱门柚作为芸香科植物,其单倍体基因组为301.37 Mbp,遗传背景复杂,一直以来很难建立精确的物理图谱[3]。DNA重复序列包括串联重复序列、散在重复序和片段重复序列。串联重复序列(Tandem repeats, TRs),通常指1~200 bp的重复DNA单元组成,重复单元之间首尾依次相连成串排列[4]。根据重复单元和重复次数分为卫星、小卫星和微卫星等[5]。【本研究切入点】对重复序列的深入研究能进一步了解重复序列在基因组进化中的作用,及其在基因组中的生物学功能等[6-7]。【拟解决的关键问题】本实验通过研究串联重复序列在克莱门柚全基因组的密度及模体特征,以便阐明重复序列在克莱门柚基因组中可能的生物学功能。
1 材料与方法
1.1 克莱门柚全基因组数据的获得
从植物基因组数据库Phytozome (http//www.Phytozome.net/)下载克莱门柚(C.clementina)的全基因数据及基因组注解(Gene annotation)数据,得到其全基因组(whole genome size)大小为301.37 Mbp。
真核生物的基因结构包括启动子,转录起始点,增强子,编码区,终止子及上下游区域。为了便于对克莱门柚基因组中串联重复序列的分析,本研究依据图1的真核基因结构对每个区域分别进行分析,主要包括基因内区域(intragenic regions)和基因间区域(intergenic regions),其中基因内区域包括5′UTR(非翻译区)、CDS(编码区)、intron(内含子)和 3′UTR(非翻译区)。串联重复序列的密度(density)被定义为,每兆碱基对含有的串联重复序列的碱基对数(bp/Mbp),表示串联重复序列长度在总检测序列长度中所占的比例。依据图1,分别计算、分析克莱门柚基因组UI1000、UI500、UI200、5′UTR、CDS、Intron、3′UTR、DI200、DI500、DI1000区域中的串联重复序列特征。
1.2 串联重复序列的检测和分析
为了对健全和不完善的串联重复序列的检测,利用串联重复序列的搜索工具(Phobos version 3.3.12)。考虑所需处理的基因组的计算资源和执行时间,采用1~50 bp作为重复单位的大小,所需检测重复的的最小长度被设定为12。对于循环的串联重复序列,按照字母表的顺序只有一个基序被选择为代表[6-7],例如AAG、AGA和GAA为(AAG)n的重复单元,但只有AAG被选择为代表的重复序列。此外,串联重复序列以及它的反向互补序列(例如,AAG和CTT)应该分别检测,这是因为基因注解在不同的链上(正链和负链),最近有大量报道许多基因的正义和反义转录[8],强调在基因组注解基因定位的重要性,类似的策略已经被他人采用[9]。
2 结果与分析
2.1 高粱全基因中1~50 bp串联重复序列的密度分析
图2显示,在克莱门柚基因组中,从上游序列UI1000到5′UTR,重复密度缓慢上升,5′UTR处出现最高值7958 bp/Mbp,至CDS处出现最低值1628 bp/Mbp,其次3′UTR中密度也较低,为3737 bp/Mbp;在基因下游区域,重复密度在6500 bp/Mbp左右。现在就重复序列的密度做以下分析。
图3表示,在整个克莱门柚基因组(301.37 Mb)中,1~50 bp 串联重复序列密度排在前7位从高到低分别是单碱基、二碱基、六碱基、三碱基、七碱基、四碱基、22碱基。其中单碱基、二碱基和六碱基为主要的重复单元(每种碱基的重复密度大于5 %)。单碱基、二碱基和六碱基的重复密度分别为33.13 %、9.50 %、6.66 %。单核苷酸重复单元以A及其互补模体T为主,占总重复模体的90.68 %。G及其互补模体C密度最小,占总重复模体的9.32 %。

图1 串联重复序列分析的基因Fig.1 The gene in TR analysis

图2 克莱门柚基因组不同区域串联重复序列密度Fig.2 The densities of TRs in different regions in C.clementina genome
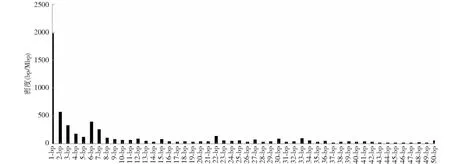
图3 克莱门柚基因组中1~50 bp串联重复序列密度Fig.3 The densities of 1-50 bp TRs in C.clementina genome
2.2 串联重复序列1-50 bp在基因内的密度分布
2.2.1 串联重复序列1~50 bp在5′UTR中的密度分布 图4显示,单碱基、二碱基、六碱基、三碱基串联重复序列密度较高,分别为2289、1238、1080和904 bp/Mbp。单碱基重复单元以A(1305 bp/Mbp)及其互补模体T(870 bp/Mbp)为主,占95.02 %。二碱基重复单元以CT(697 bp/Mbp)及其互补模体AG(291 bp/Mbp)为主,占79.94 %。六碱基中以CTTTTT(50 bp/Mbp)最高,以AAGATC(15 bp/Mbp)最低。三碱基重复单元以CTT(180 bp/Mbp)及其互补模体AAG(150 bp/Mbp)为主,占37.02 %,CGG最低(3 bp/Mbp)。5′UTRs的重复序列可能与启动子区的识别有关,转录起始点上游TATA区与CAAAT区(CAAT box)结合RNA聚合酶启动转录。
2.2.2 串联重复序列1~50 bp在CDS中的密度分布 CDS (coding sequence),即编码区,包含相间排列的Intron(内含子)和Exon(外显子),两者均可转录,转录后内含子经加工剪切,外显子连接后翻译出蛋白质[4]。因此内显子突变,对生物无意义,生物主要靠外显子起作用,因此不能轻易突变,否则对生物将会产生不可预测的影响。图5显示,三碱基和六碱基串联重复密度最大,分别是414和337 bp/Mbp,在总密度的比例分别为25.43 %和20.71 %;其次就是三碱基倍数的重复单元密度较高,如9-bp (79 bp/Mbp),12-bp (70 bp/Mbp),33-bp (64 bp/Mbp)等。三碱基重复单元以AAG密度最高(57 bp/Mbp),占13.80 %。六碱基中ACCGTG密度最高(14 bp/Mbp)。
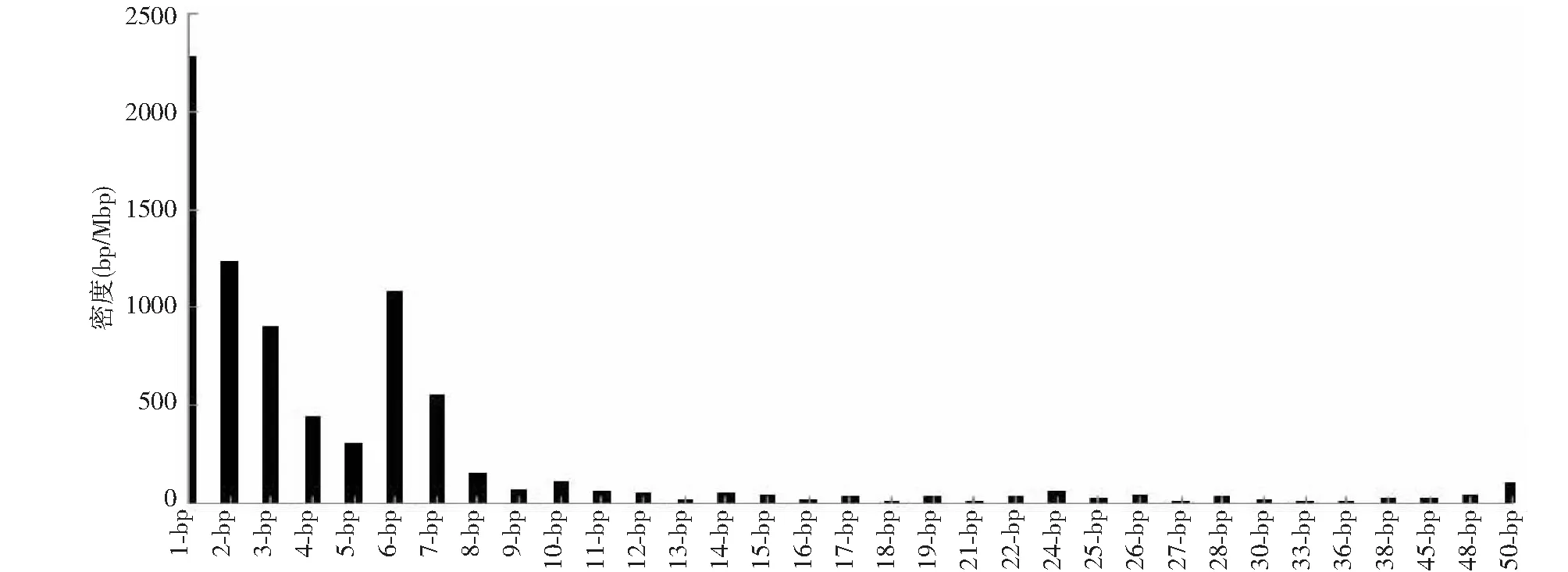
图4 5′UTR中1~50 bp串联重复序列密度Fig.4 The densities of 1-50 bp TRs in 5′UTR s

图5 CDS中1~50 bp串联重复序列密度Fig.5 The densities of 1-50 bp TRs in CDS
2.2.3 串联重复序列1~50 bp在内含子中的密度分布 Intron为内含子,即翻译生成蛋白时需要被剪切掉的部分。单碱基和二碱基串联重复密度最大,分别为2319和557 bp/Mbp,占总重复序列的42.06 %和10.10 % (图6)。单碱基重复以T(1401 bp/Mbp)及其互补模体A(524 bp/Mbp)为主。二碱基重复中以AT(154 bp/Mbp)和CT最高(146 bp/Mbp), CG最低(4 bp/Mbp)。

图6 内含子中1~50 bp串联重复序列密度Fig.6 The densities of 1-50 bp TRs in introns
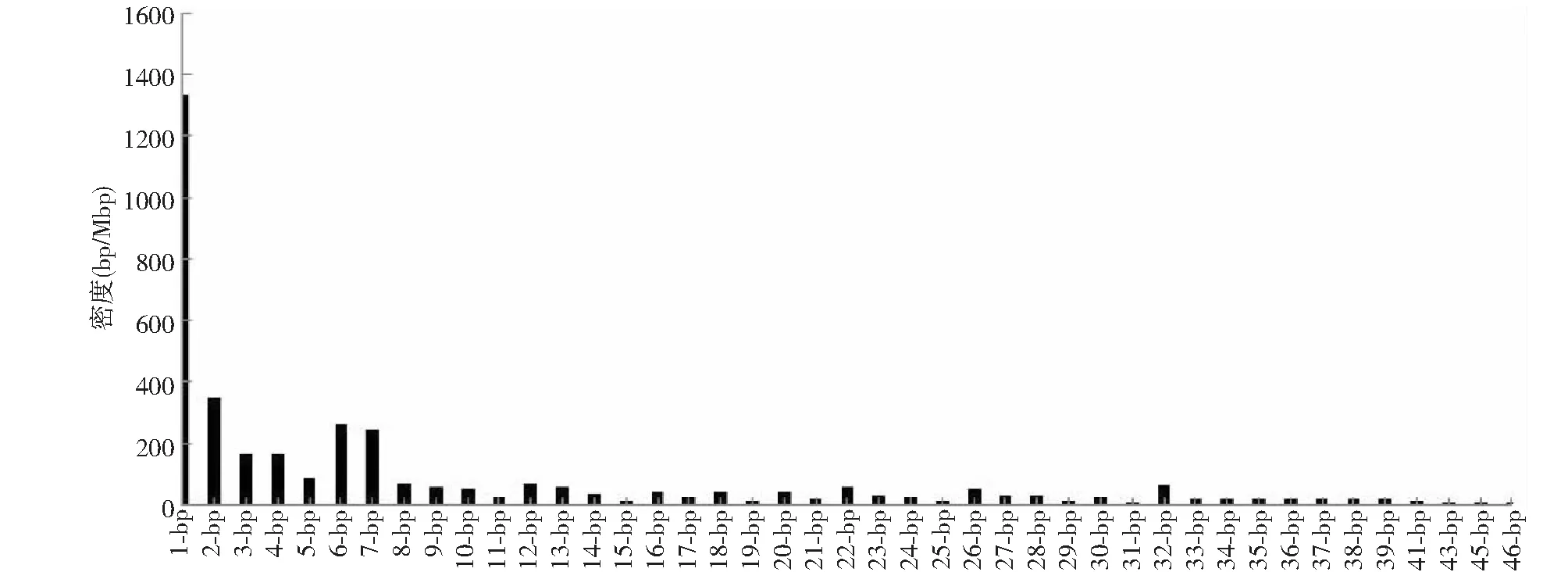
图7 3′UTR中1~50 bp串联重复序列密度Fig.7 The densities of 1-50 bp TRs in 3′UTRs
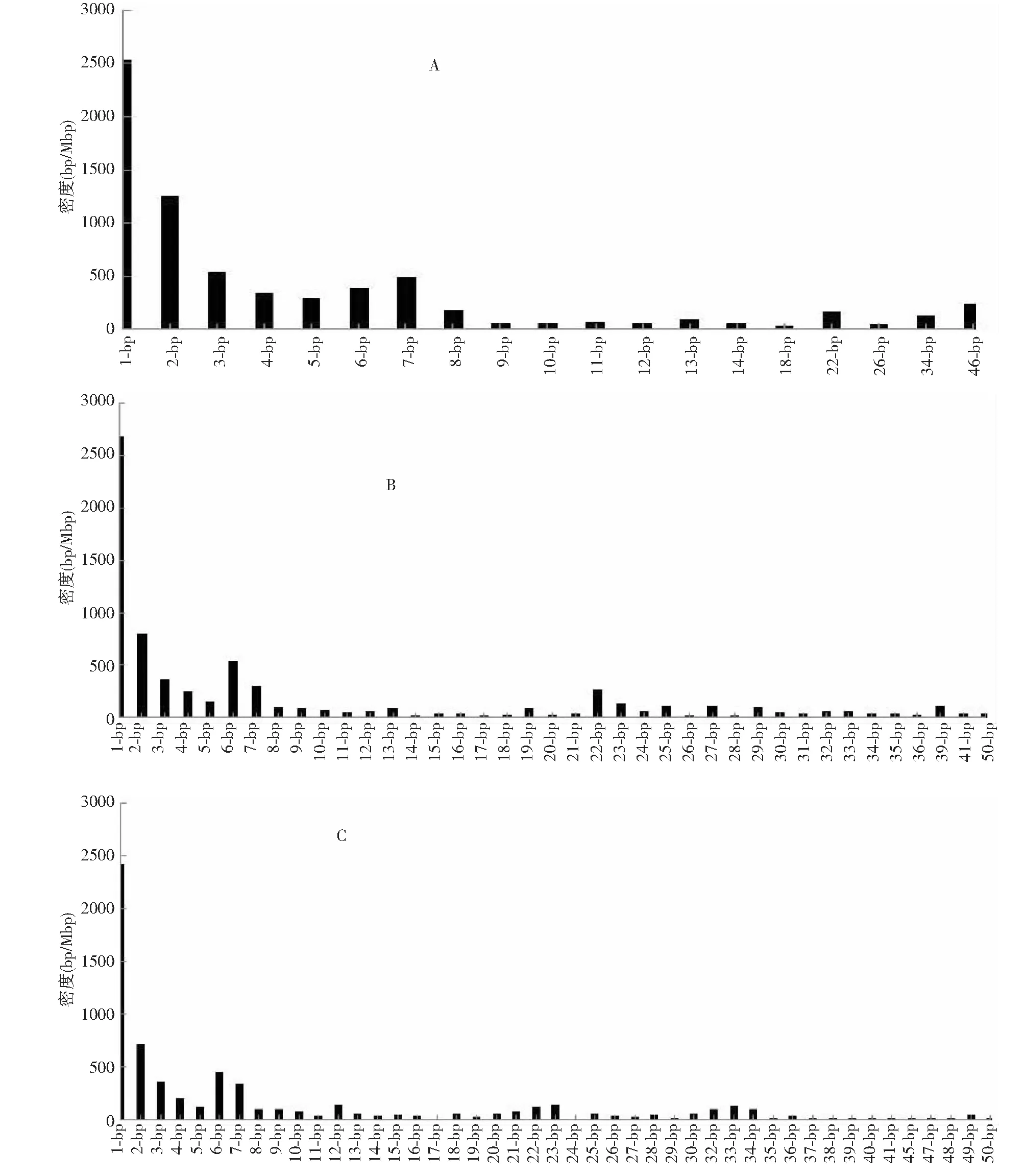
(A)UI200, (B)UI500, (C)UI1000图8 上游基因间隔区1~50 bp串联重复序列密度Fig.8 The densities of 1-50 bp TRs in upstream intergenic regions
2.2.4 串联重复序列1~50 bp在3′UTR中的密度分布 3′UTR为结构基因的3′-端非编码区,包括促使转录终止的终止子序列和真核生物的加尾序信号[4]。单碱基串联重复密度高达1337 bp/Mbp,其次为二碱基(350 bp/Mbp)、六碱基(261 bp/Mbp)和七碱基(248 bp/Mbp)重复(图7)。单碱基重复单元以T(846 bp/Mbp)及其互补模体A(391 bp/Mbp)为主,占总的92.45 %。这可能与3′UTR末端聚腺苷酸化形成poly(A)尾巴有关。
2.3 串联重复序列1~50 bp在基因间的密度分布
2.3.1 串联重复序列1~50 bp在基因上游区域的密度分布 在基因上游UI200、UI500和UI1000区域内,单碱基串联重复序列密度都是最高(>2400 bp/Mbp),其次为2~7 bp的重复序列,相比较而言五碱基重复序列密度在这些微卫星中则最低(<300 bp/Mbp)(图8)。
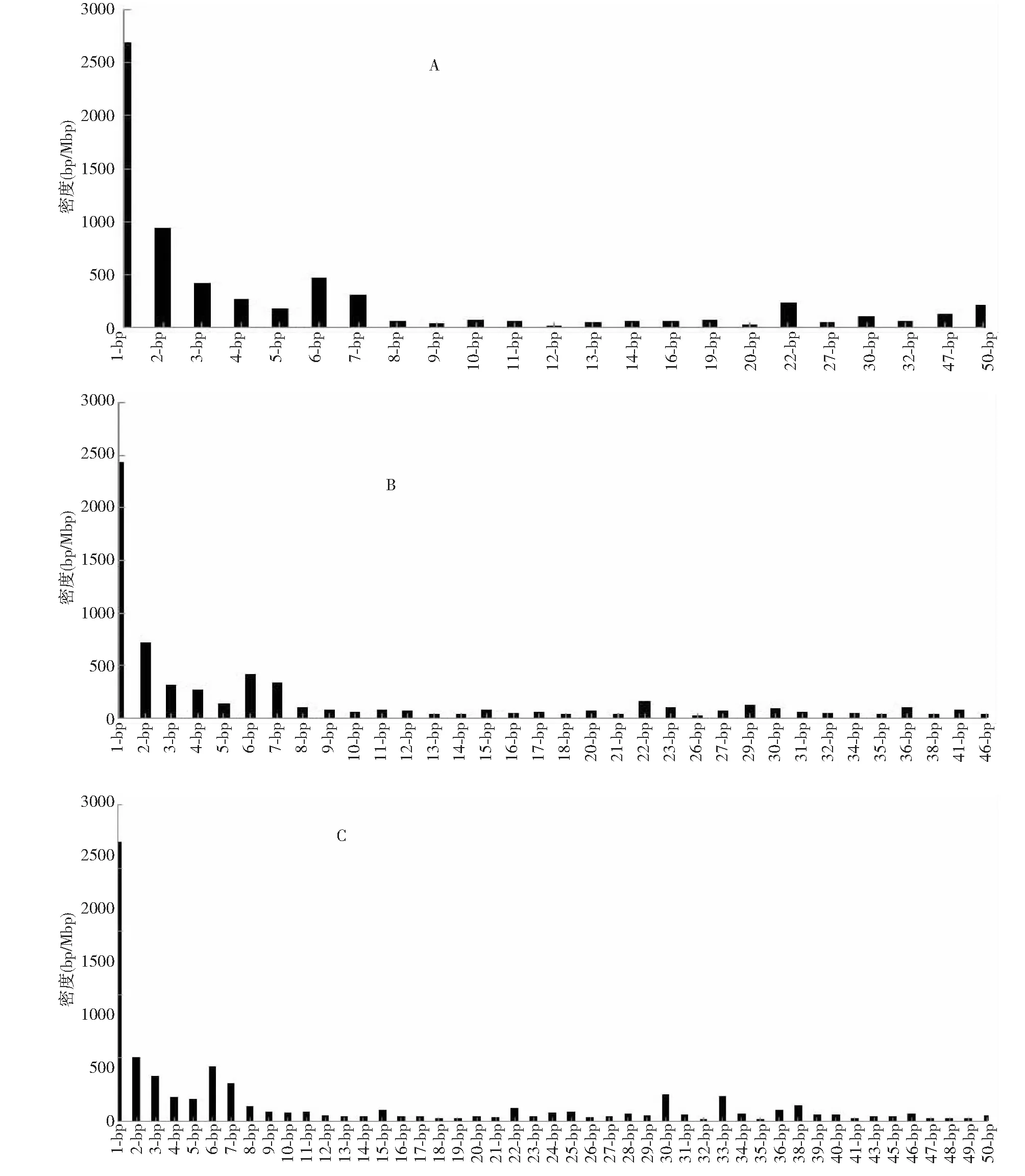
(A)DI200, (B)DI500, (C)DI1000图9 下游基因间隔区1~50 bp串联重复序列密度 Fig.9 The densities of 1-50 bp TRs in downstream intergenic regions
在UI200区域中(图8-A),单碱基和二碱基串联重复密度最大,分别是2532 bp/Mbp和1256 bp/Mbp。单碱基重复单元以A(1424 bp/Mbp)及其互补模体T(859 bp/Mbp)为主,占总的90.13 %。二碱基中以AT为最高(424 bp/Mbp),占总的33.76 %。
UI500区域中(图8-B),单碱基和二碱基密度最大,分别为2686和805 bp/Mbp。单碱基重复单元以A(1267 bp/Mbp)及其互补模体T(1206 bp/Mbp)为主,占总的92.07 %。二碱基中以AT为最高(287 bp/Mbp),占总重复模体的35.65 %。
UI1000区域中(图8-C),单碱基和二碱基密度最大,分别是2415 bp/Mbp和712 bp/Mbp。单碱基重复单元以A(1123 bp/Mbp)及其互补模体T(1090 bp/Mbp)为主,占总的91.64 %。二碱基中以AT为最高(316 bp/Mbp),占总的44.38 %。
2.3.2 串联重复序列1~50 bp在基因下游区域的密度分布 类似于基因上游区域,在基因下游(DI200、DI500和DI1000)区域,单碱基密度最高(>2200 bp/Mbp),其次为2~7 bp重复序列,而五碱基重复序列密度在这些微卫星中则最低(<200 bp/Mbp)(图9)。
在DI200区域中(图9-A),单碱基和二碱基串联重复密度最大,分别为2695和937 bp/Mbp。单碱基重复以T(1428 bp/Mbp)及其互补模体A(1078 bp/Mbp)为主,占总的92.99 %。二碱基中以AT和CT最高,分别是300和256 bp/Mbp,占总的59.34 %。
在DI500区域中(图9-B),单碱基和二碱基串联重复密度最大,分别是2439和722 bp/Mbp。单碱基重复单元以T(1130 bp/Mbp)及其互补模体A(1090 bp/Mbp)为主,占总的90.98 %。二碱基中以AT为最高(287 bp/Mbp),占总的39.56 %。
在DI1000区域中(图9-C),单碱基和二单碱基串联重复密度最大,分别是2211和505 bp/Mbp, 占总的34.15 %。单碱基重复单元以A(1075 bp/Mbp)及其互补模体T(969 bp/Mbp)为主,占总重复模体的92.49 %。
3 讨 论
在克莱门柚的基因组中,本文主要研究的特征区域包括UI1000、UI500、UI200、5′UTR、CDS、Intron、3′UTR、DI200、DI500和DI1000等。数据显示克莱门柚基因组串联重复序列高密度的主要为短序列重复单元(1~7 bp),主要重复类别是单碱基、二碱基、六碱基、三碱基、七碱基、四碱基、22碱基等,且主要以A和T重复为主。研究显示,克莱门柚基因组中最高和次高的串联重复序列密度在5′UTR和它的直接上游区域,即UI500和UI200区,而这个区域通常为转录起始调控区域,大量重复序列的存在有利于保证转录起始的稳定性[10]。5′UTR被认为是串联重复序列的热点区域,之前的研究表明,5′UTR中的串联重复序列可参与转录或翻译的调控[6-7,11];而在家蚕基因组中,5′UTR区域却拥有最少的SSR数量[12],这可能是物种差异造成的。CDS中串联重复序列的密度最低,低密度的重复序列会降低蛋白质的复杂性从而增强其保守度,已经证实CDS的突变会导致蛋白功能改变,功能丧失和蛋白截短[13];同时CDS中主要以3n模体 (如3、9、12 bp等)作为主要的重复单元,应该与翻译的三联体密码子有关,以避免框移。3′UTR和内含子中的串联重复序列密度也较低,可能暗示重复序列在这些区域保守度高,参与的生物学功能也可能较少[7];3′UTR重复序列变异将会导致转录提前终止或延后[4]。
4 结 论
本文研究串联重复序列在克莱门柚基因组不同区域的特征,结果显示重复序列在基因不同区域具有明显的数量(密度)及模体类型差异,说明重复序列很可能参与克莱门柚不同区域基因表达与调控。生物能够稳定遗传和进化与串联重复序列的存在有很重要的关系,而克莱门柚中串联重复序列具体的生物学功能还有待进一步研究。
