基于高通量测序分析年糕菌群结构和优势菌属
2018-09-03蔡怀依雷丽萍娄永江李勇勇
蔡怀依 雷丽萍 娄永江 李勇勇
(宁波大学海洋学院,宁波 315211)
年糕(rice pastry)是一种具有悠久历史的半方便性的米制品,有浓郁的地方传统和特色[1]。其营养丰富,水分含量高,水分活度大,且整个加工过程中年糕原辅料半成品及成品都直接与水或空气接触,这也直接为微生物提供了适宜的生长条件[2]。通过分析年糕储藏过程中的菌群结构,从而有目的的选用合适的抑菌剂,能为年糕品质劣化控制提供基础资料。
目前,分析微生物群体的多样性及群落结构的经典方法是分离、培养以及鉴定,需要进行一系列繁杂的形态特征和生理生化实验,且能够培养分离出的微生物仅占样品的1%~10%,无法解析微生物的组成及丰度情况[3]。随着宏基因组学概念的提出和高通量测序技术的发展,使样品中不可培养的微生物、低丰度的微生物均能被检测出,更完整地反映样品中微生物的群落特征,使菌群的分析更准确和快速[4-7]。本研究采用宏基因组结合高通量测序技术分析年糕菌群结构和优势菌属,为进一步探讨微生物与年糕品质的相关性提供参考。
1 材料与方法
1.1 材料与试剂
年糕:宁波江北五桥粮油食品有限责任公司提供;E.Z.N.A.Soil DNA Kit D5625-01:OMEGA;Qubit2.0 DNA检测试剂盒Q10212:Life;Taq DNA Polymerase Ep0406:Thermo;Agencourt AMPure XP A63882:Beckman;实验用水:经Milli-Q Integral 5超纯水系统纯化。
1.2 仪器与设备
Pico-21台式离心机:Thermo Fisher;GL-88B漩涡混合器:海门市其林贝尔仪器制造有限公司;TND03-H-H混匀型干式恒温器:深圳拓能达科技有限公司;DYY-6C型电泳仪、DYCZ-21电泳槽:北京市六一仪器厂;凝胶成像系统:美国UVP;Q32866 Qubit© 2.0荧光计:Invitrogen;T100TM Thermal Cyeler PCR仪:BIO-RAD;Research plus移液器: Eppendorf。
1.3 实验方法
1.3.1 样品预处理
新鲜年糕采取液氮研磨的方法将样品磨成粉末状,用于基因组DNA的提取。
1.3.2 宏基因组DNA的提取
采用E.Z.N.ATM Mag-Bind Soil DNA Kit的试剂盒(OMEGA公司)提取总DNA,提取的总DNA经1%琼脂糖凝胶电泳检测完整性,超微量分光光度计(Thermo NanoDrop 2000)检测浓度。检测合格后于-20 ℃保存,用于后续实验。
1.3.3 PCR扩增及高通量测序
利用Qubit2.0 DNA检测试剂盒对基因组DNA精确定量,以确定PCR反应加入的DNA量。PCR所用的引物已经融合了Miseq测序平台的V4-V5通用引物,其中515F引物:CCCTACACGACGCTCTTCCGAT CTN (barcode) GTGCCAGCMGCCGCGGTAA,909R引物AGTTCCTTGGCACCCGAGAATTCCACCCCGY CAATTCMTTTRAGT。
通过两轮PCR扩增并完成接头序列的连接。第一轮PCR反应体系为30 μL反应液,含2×Taq master Mix 15 μL,Bar-PCR primer F(10uM) 1 μL,Primer R (10uM) 1 μL,Genomic DNA 10 ng, H2O补足至30 μL 。PCR反应条件为:94 ℃预变性 3 min;94 ℃30 s,45 ℃20 s,65 ℃ 30 s进行5个循环;之后94 ℃20 s,55 ℃ 20 s,72 ℃30 s进行20个循环,最后72 ℃延伸5 min。第二轮扩增引入Illumina桥式PCR兼容引物, PCR 反应条件为:95 ℃预变性30 s,然后95 ℃15 s,55 ℃15 s,72 ℃30 s进行5个循环,最后72 ℃延伸5 min。PCR 结束后,利用琼脂糖电泳进行鉴定,之后进行DNA纯化回收。在25 μL PCR产物中加入体积0.8倍的磁珠(Agencourt AMPure XP),震荡充分悬浮后放在磁力架上吸附5 min,小心的用移液枪吸出上清。加入30 μL0.8倍的磁珠洗涤液,震荡充分悬浮后放在磁力架上吸附5 min,小心吸出上清。加入90 μL WashBuffer,反向放置在磁力架上,使磁珠吸附到PCR管的另外一面,充分吸附后吸出上清。将PCR管或8联管放在55 ℃烘箱5 min,使里面的酒精完全挥发。加入30 μL Elution Buffer洗脱。将PCR管放在吸附架上5 min,充分吸附,移出上清到干净的1.5 mL离心管中,定量备用。利用Qubit 2.0 DNA检测试剂盒对回收的DNA精确定量,以方便按照1∶1的等量混合后测序。等量混合时,每个样品DNA量取10 ng,最终上机测序浓度为20 pmol。
1.3.4 数据分析
Illumina MiseqTM得到的原始图像数据文件经CASAVA碱基识别 (Base Calling) 分析转化为原始测序序列(Sequenced Reads) ,其中包含测序序列 (reads) 的序列信息以及其对应的测序质量信息。Miseq测序序列中含有barcode序列,以及测序时加入的引物和接头序列。首先需要去除引物接头序列,再根据PE reads之间的overlap关系,将成对的reads 拼接 (merge) 成一条序列,然后按照barcode标签序列识别并区分样品得到各样本数据,最后对各样本数据的质量进行质控过滤,得到各样本有效数据。去除3′端测序引物接头,Read1 3′端测序接头为TGGAATTCTCGGGTGCCAAGGAACTC,根据PE reads之间的overlap关系将成对reads拼接(merge) 成一条序列,拼接序列的overlap区域允许的最大错配比率是0.1,根据各样本barcode序列从融合后数据中分割出各样本数据,去除各样本中reads尾部质量值在20以下的碱基。设置10 bp的端口,如果窗口内的平均质量值低于20,从窗口开始去除后端的碱基。切除reads中含N部分序列,并去除数据中的短序列,长度阈值200 bp,随后再对低复杂度的序列进行过滤。将多条序列按其序列间的距离进行聚类,对相似性在 0.97 以上的序列进行归并,生成操作分类单元(OTU)[8-10]。根据聚类分析结果,计算 ACE、Chao1、Shannon、Simpson 指数进行 alpha 多样性分析,其中 ACE、Chao1 指数是对菌群丰度进行评估,Shannon、Simpson 指数是对菌群多样性进行评估。采用 RDP classifier 软件对序列进行物种分类,根据分类学分析比对结果,在门、属等水平上对样品的菌群结构进行种类和丰度分析[11]。
2 结果与分析
2.1 序列长度分布
在年糕样品总DNA的16S rDNA V4-V5区中测得的原始reads数目34 778条,原始序列平均长度为414.32(图1)。质量控制之后剩余reads数目33 090条,序列平均长度为374.47(图2)。从序列长度的分布来看, 与16S rDNA- V4-V5 区序列长度大致吻合。去除嵌合体与靶区域之前序列总数33 090条,比对到细胞器组织序列数目6 763条,非靶区域序列数目0条,嵌合体数目159条,处理后剩余序列26 168条。
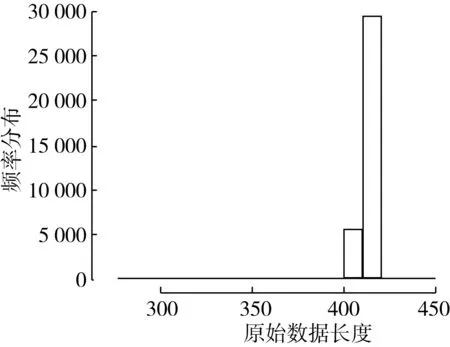
图1 原始数据长度分布图
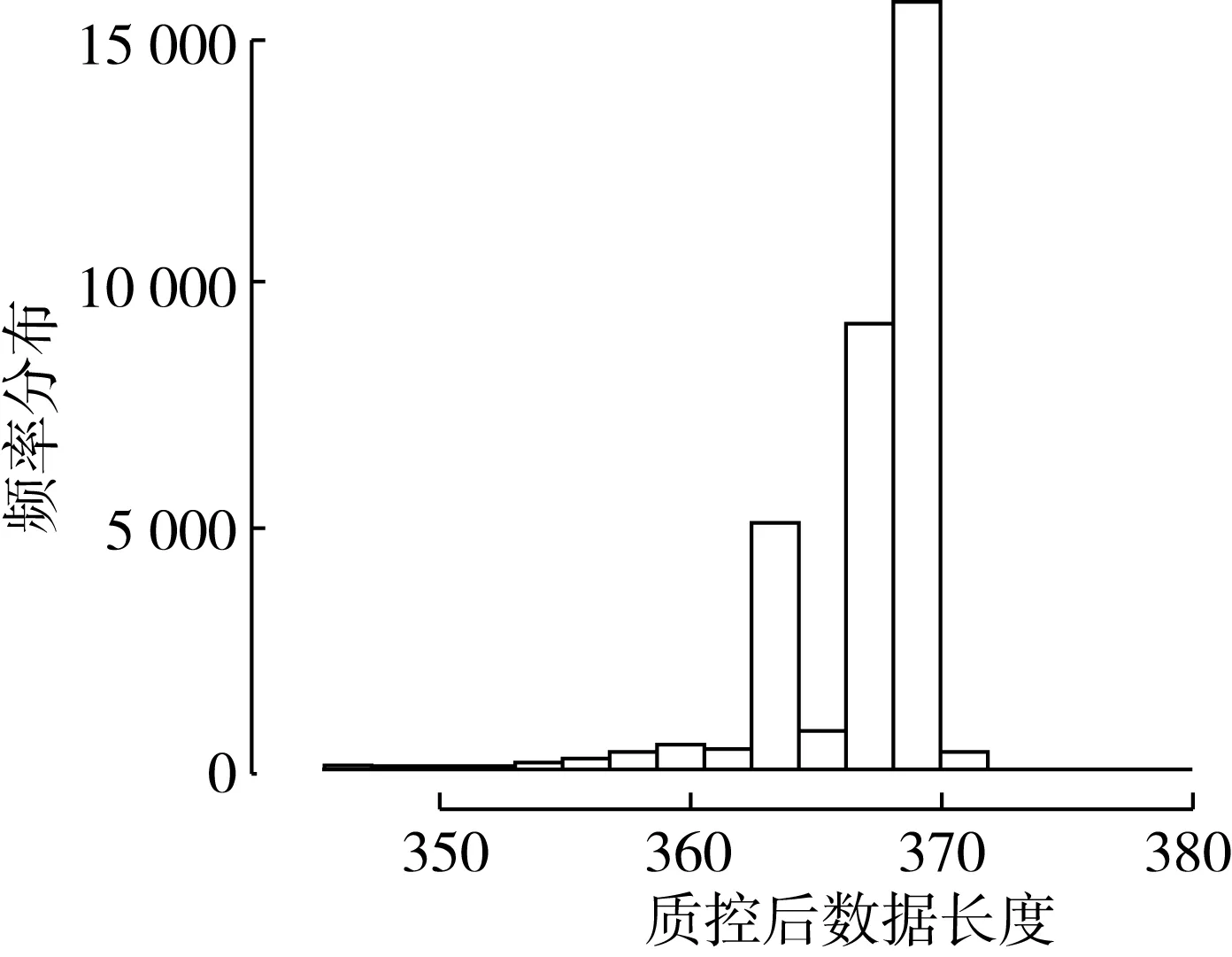
图2 质控后序列长度分布图
2.2 OTU多样性分析
通过绘制OTU数目变化与聚类similarity值之间的关系图,从中选择最佳的similarity值进行OTU分析和分类学分析,分析使用的similarity值为97%的序列相似性(图3)[12]。样品的基因组 DNA 经 PCR 扩增 16S rDNA V4-V5 区后进行高通量测序,经统计分析获得序列的 alpha 多样性,结果见表 1。Shannon值越大,说明群落多样性越高,而Simpson值越大说明群落多样性越低[13],由表1 可知样本群落多样性较高。此外,样本的测序覆盖率在1.00以上,表明样品中序列未被测到的概率较低。
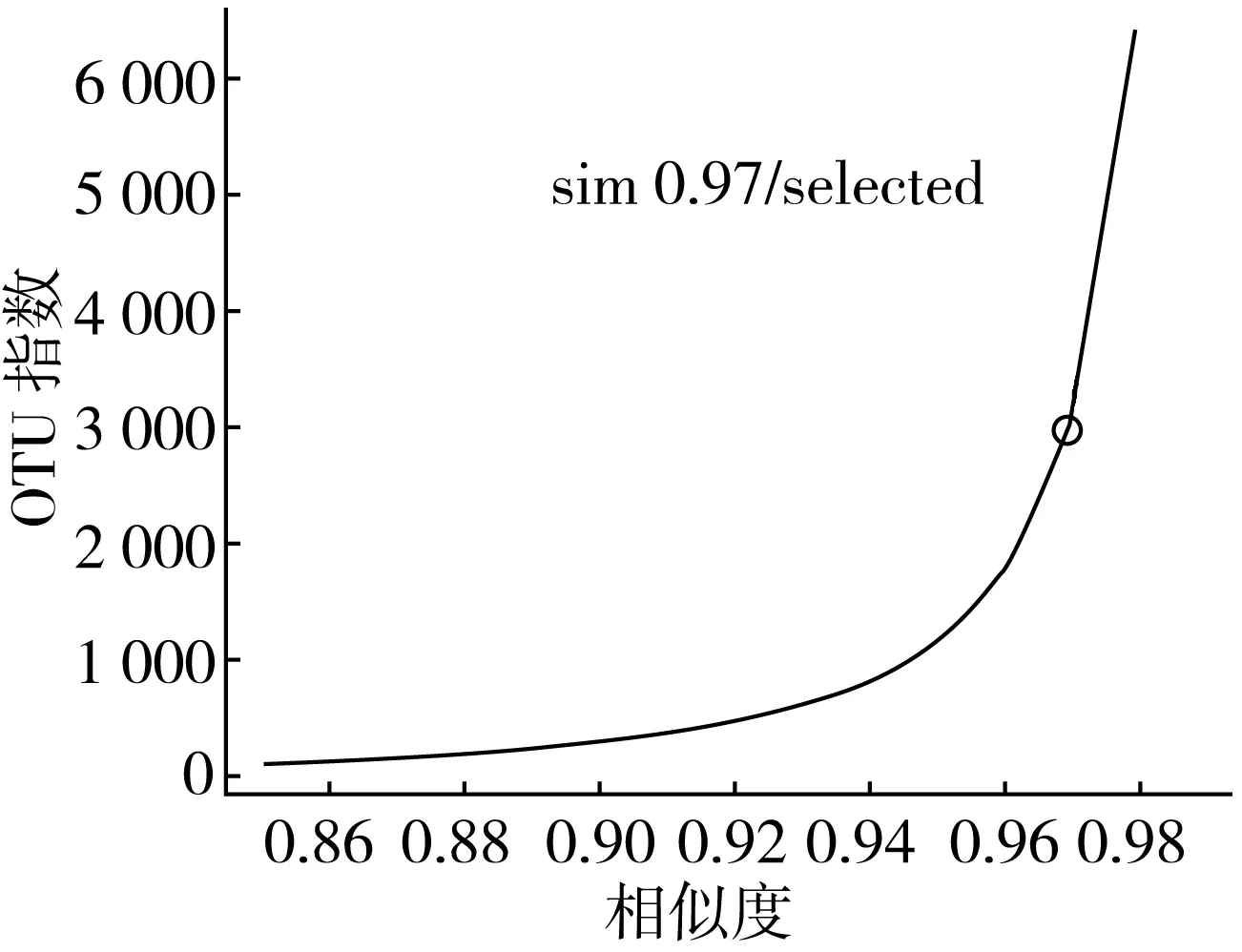
图3 OTU数目与聚类相似度值关系图
采用对测序序列进行随机抽样的方法,以抽到的序列数与它们所能代表OTU的数目构建曲线,即稀释曲线(Rarefaction Curve)。它可以用来比较测序数据量不同的样本中物种的丰富度,也可以用来说明样本的测序数据量是否合理[14]。当曲线趋向平坦时,说明测序数据量合理,更多的数据量只会产生少量新的OTU,反之则表明继续测序还可能产生较多新的OTU。样本的Shannon-Wiener曲线(图4), 发现曲线已到达平台期, 说明更大的测序量不会引起物种多样性的显著增长,基于现有数据量的分析结果准确可靠。

表1 年糕腐败样品菌群的alpha多样性比较
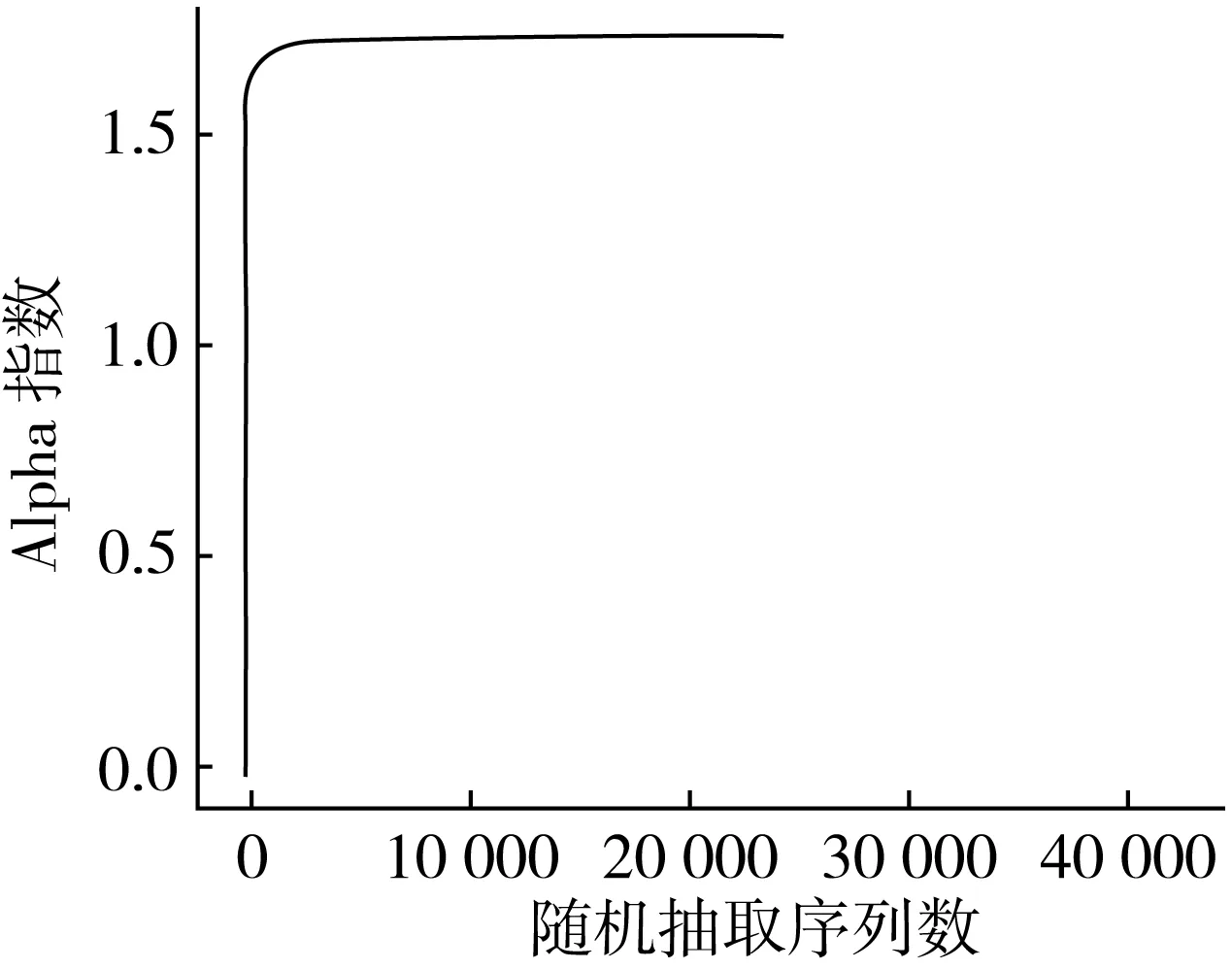
注:横坐标为样本中随机抽取序列数,纵坐标为所得相应的Alpha指数图4 Alpha指数稀疏曲线图
2.3 细菌种类多样性和丰度分析
利用blastn将OTU序列与对应数据库进行比对,筛选出OTU序列的最佳比对结果,并对比对结果进行过滤,默认满足相似度>90%且coverage>90%的序列被用来后续分类,不满足条件的序列则被归为unclassified。根据分类学分析结果,统计样本在各个分类层级水平上的群落组成如表2所示。以OTU的物种分类结果为依据, 分别在门、纲和属3个分类级别上对样本中的细菌种类和相对丰度进行统计分析。样本的细菌种类覆盖5个门,其中以变形杆菌门(Proteobacteria)为主, 占总数的69.23%,厚壁菌门(Firmicutes)28.26%次之,其中变形杆菌门genus水平上树状图如图5所示,厚壁菌门genus水平上树状图如图6所示。在纲的分类水平上, 包含8个纲,以变形菌纲(Betaproteobacteria,37.21%)和芽孢杆菌纲(Bacilli,28.25%)为主。在目的分类水平上,包含13个目,以伯克氏菌目(Burkholderiales,37.21%)和芽孢杆菌目(Bacillales,24.38%)为主,在科的分类水平上,包含了25个科,以丛毛单胞菌科(Comamonadaceae,36.80%)和芽孢杆菌科(Bacillaceae,24.38%)为主。在属的分类水平上, 包含29个属,以玫瑰色半光合菌属(Roseateles,36.75%)和芽胞杆菌属(Bacillus,24.38%)为主。丰度大于1%的还有寡养单胞菌属(Stenotrophomonas,9.36%)、藤黄色杆菌属(Luteibacter,1.45%)、乳球属(Lactococcus,2.83%)。而刘青梅等[15]通过传统培养方法获得散装年糕中疑似腐败菌,并采用分子生物学方法对细菌的进行扩增结合数据库比对等方法鉴定出优势腐败菌为芽孢杆菌和短芽孢杆菌两大属。胡庆松等[16]采用VITEK-32自动化微生物分析仪鉴定系统、 API微生物鉴定系统和手工法鉴定引起散装年糕腐败的典型菌株为波茨坦短芽胞杆菌、巨大芽孢杆菌、乙酰短杆菌、希氏短杆菌。俞科伟等[2]通过对发生腐败变质的真空包装年糕中微生物进行分离、纯化和反证,经过形态观察、 生理生化、微生物鉴定系统等方法,初步鉴定 2株细菌分属于枯草芽孢杆菌、不动杆菌属。黄丽金等[17]采用分子生物学方法对真空年糕的腐败菌进行分离和鉴定,确定其优势菌为枯草芽孢杆菌。陈挺[18]对真空年糕制品表面和内部的微生物进行分离纯化,并通过形态特征的观察和一些生理生化实验对其进行鉴定得出主要优势菌株为醋酸钙不动杆菌、乙酰短杆菌、枯草芽孢杆菌、蜡状芽孢杆菌、巨大芽孢杆菌和短芽孢杆菌。由此可以看出利用传统纯培养方法等和高通量测序得到的年糕腐败菌属具有基本一致的结果,但又不完全相同。此外,在属的水平上,尽管不同方法的分析数据有差异,造成分析数据差异的原因可能在于年糕的不同包装方式、不同培养时间和不同的腐败时期菌群结构鉴定也会造成优势菌属及丰度的不同,但不同方法所得出丰度较高的腐败菌属中均含有芽胞杆菌属。这可能是由于芽孢杆菌属的微生物能够形成芽孢的特性使得它们能够抵抗各种极端环境如高温、极酸、极盐,而且对各种杀菌剂具有抵抗力,因此,它们在各种自然环境中都能被分离到[19],并且引起年糕胀袋的微生物也很有可能是芽胞杆菌属(Bacillus)[20]。所以,如何抑制芽胞杆菌属对于真空年糕的贮运保鲜具有重要意义后续研究应重点关注。除此之外,新鲜年糕中另一种优势菌属玫瑰色半光合菌属(Roseateles)是伯克氏菌目(Burkholderiales)分支的一个属,而伯克氏菌目是一类能够利用自身固氮的微生物,在水稻与环境的互作过程中可能发挥着重要的作用,是水稻的内生有益菌[21]。故由稻米制作出的新鲜年糕可能携带该菌,造成该菌在年糕微生物丰度中所占比例较大。
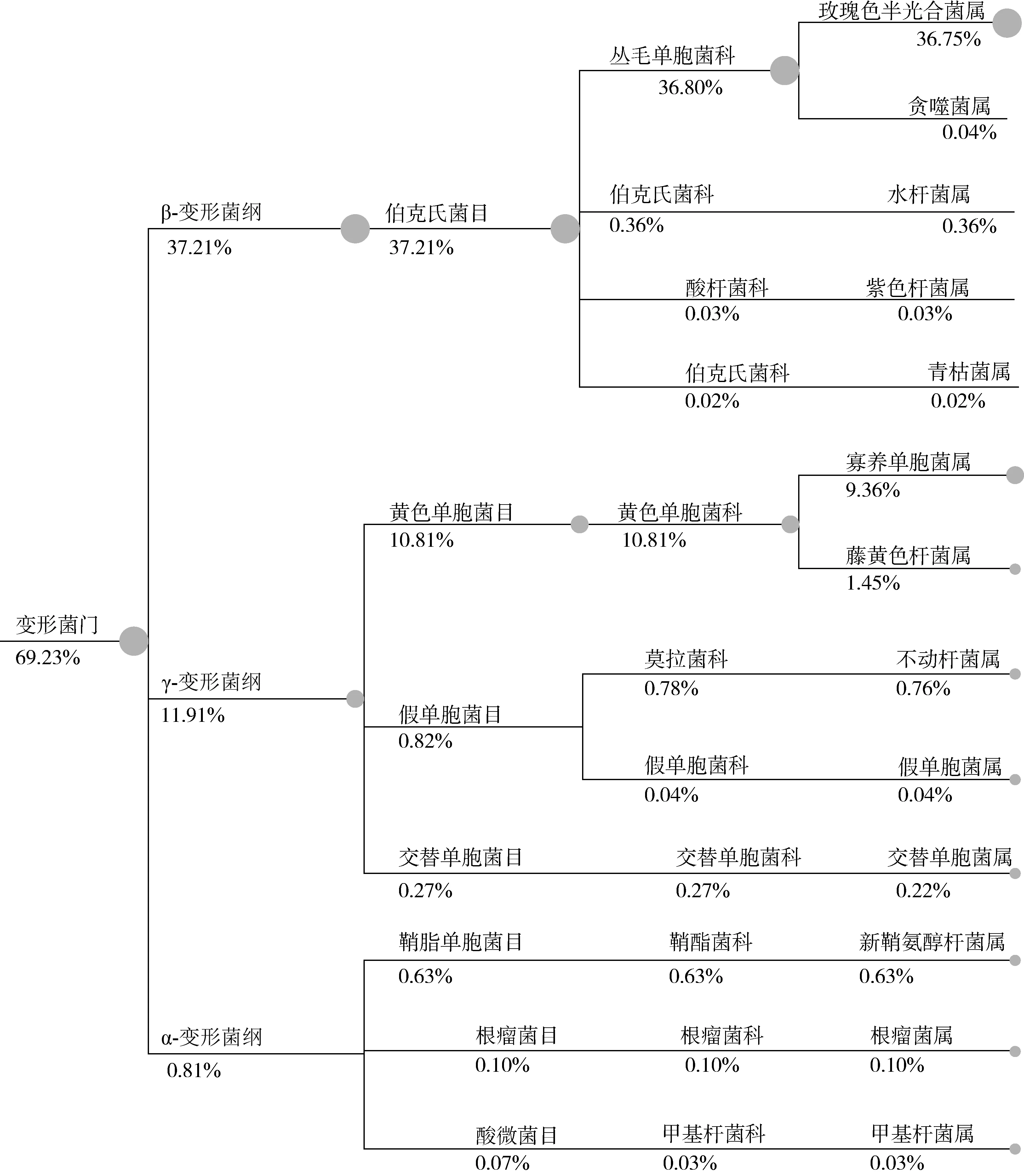
图5 样本genus水平上变形杆菌门树状图组成
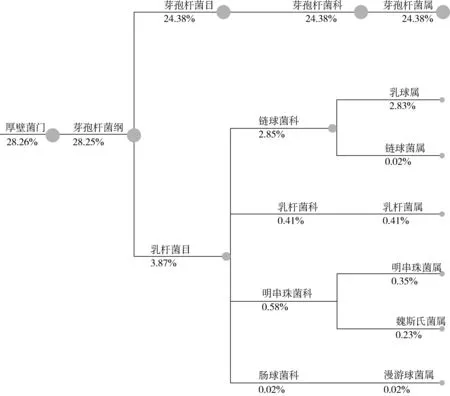
图6 样本genus水平上厚壁菌门树状图组成
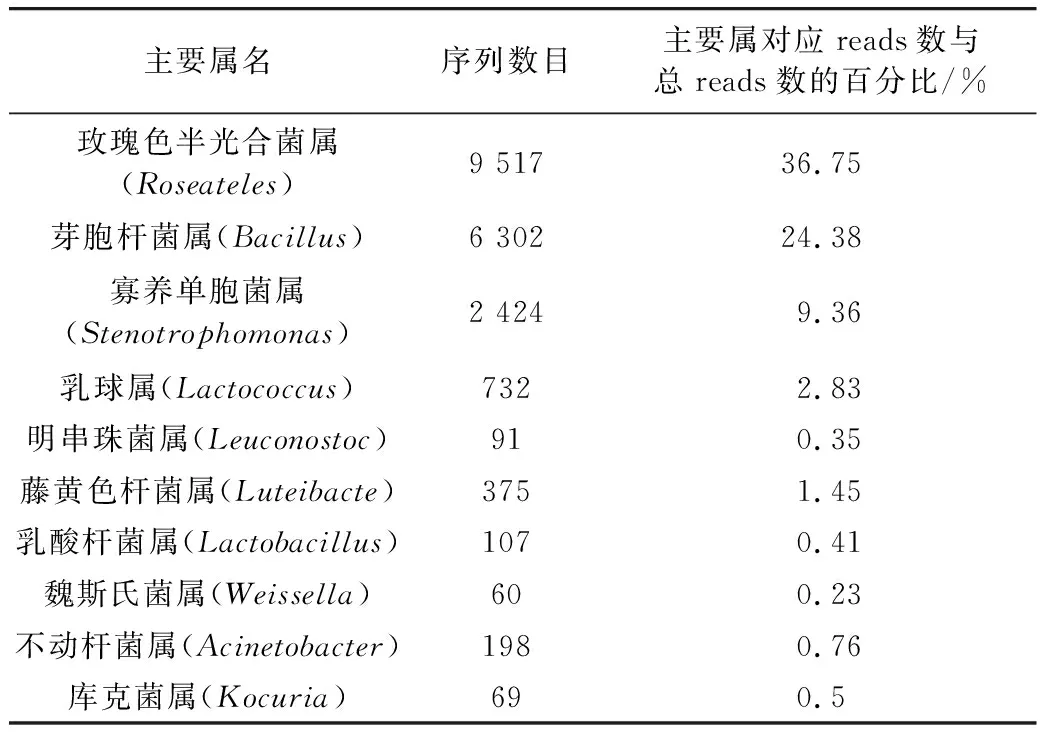
表2 genus水平上样本主要rank reads数目
3 结论
本研究通过高通量测序技术分析了新鲜年糕经真空包装后置于37 ℃下腐败后的总菌群结构和优势菌属。结果表明,年糕内生细菌主要分布于玫瑰色半光合菌属(Roseateles)和芽胞杆菌属(Bacillus)、寡养单胞菌属(Stenotrophomonas),这3个属是年糕的优势菌属。
