壳斗科植物叶绿体基因组结构及变异分析
2018-09-01张妍彤林丽梅冯若宣邢朝斌
张妍彤 黄 剑 宋 菊 林丽梅 冯若宣 邢朝斌*
(1.华北理工大学生命科学学院,唐山 063210; 2.百色学院农业与食品工程学院,百色 533000)
叶绿体是植物细胞内进行光合作用的重要细胞器,其拥有自身完整的一套基因组,可进行自主遗传[1]。在被子植物中,叶绿体基因组大多为双链环状DNA分子结构,包含大单拷贝区(large single copy,LSC)、小单拷贝区(small single copy,SSC)、反向重复区A(inverted repeats A,IRA)、反向重复区B(IRB)4个部分,其中两个IR区序列相同,方向相反[2]。基因组大小一般为120~180 kB,共编码100~130种基因,其中包括70~80种蛋白编码基因,30~32种tRNA,4种rRNA[3]。
通常情况下,叶绿体基因组的基因数量、基因顺序及结构组成相对稳定[4],但由于成长历程和遗传背景等方面的差异,不同类群间基因组有时会发生插入/缺失、重复、倒位、重排等不同形式的结构变异和基因丢失现象[5]。同时,相比于核基因,叶绿体基因组具有相对稳定、长度较短、易获取、包含信息量大、变异速率适中[6]等特点。这些基因组的结构变异和基因丢失现象对研究植物系统进化具有重要参考意义,基因组自身特点使其成为植物系统发育分析的优势选择。
壳斗科(Fagaceae)为双子叶植物,亦名山毛榉科,共8个属,900多个物种,其中多为重要的药用植物,如黄栎(Cyclobalanopsisdelavayi)、柯树(Lithocarpusglaber)等用作药材,具有解毒、止咳、安神、健胃等功效[7];栗子不仅可食用,且具有健脾补肾、活血止血的疗效;栗花可用于治疗泻痢、便血,栗树皮可用于治疗丹毒、口疮等。同时,关于壳斗科植物化学成分的研究报道显示,壳斗科植物内含有单宁类化合物、三萜、黄酮、蛋白、色素、有机酸、生物碱等成分,这些成分具有抗菌、抗炎、抗氧化等生物活性[8]。
本研究利用壳斗科已测序发表的叶绿体基因组进行比较分析,初步揭示壳斗科叶绿体基因组的结构特征和变异,阐明IR区边界的扩张与收缩问题,展示壳斗科物种间进化关系,为进一步研究壳斗科基因组进化奠下基础。
1 数据和方法
1.1 数据收集
在GenBank中检索壳斗科已发表叶绿体基因组序列的物种信息,下载其基因组序列,获取注释信息。检索所得用于分析的物种名称、基因组序列GenBank登录号列于表1,共计14个物种。
表1所选取的壳斗科物种及其叶绿体基因组GenBank登录号
Table1TheselectedFagaceaespeciesandtheirchloroplastGenBankaccessionnumber

科Family属Genus种SpeciesGenBank登录号GenBank No.壳斗科Fagaceae栎属Quercus L.栗属Castanea Mill.锥属Castanopsis(D. Don) Spach青冈属Cyclobalanopsis Oerst.柯属Lithocarpus Bl.三棱栎属Trigonobalanus Forman橿子栎Quercus baroniiKT963087.1栓皮栎Quercus variabilisKU240009.1匙叶栎Quercus dolicholepisKU240010.1北美红栎Quercus rubraJX970937.1槲栎Quercus alienaKU240007.1川滇高山栎Quercus aquifolioidesNC_026913.1刺叶高山栎Quercus spinosaNC_026907.1锥栗Castanea henryiNC_033881.1Castanea pumilaKM360048.1华南锥Castanopsis concinnaNC_033409.1短刺锥Castanopsis echidnocarpaNC_023801.1华南青冈Cyclobalanopsis edithiaeKU382355.1猴面柯Lithocarpus balansaeKP299291.1三棱栎Trigonobalanus doichangensisNC_023959.1
1.2 基因组统计和分析
通过注释信息获得14个物种的基因组大小、GC量、各部分(LSC、SSC、IR)长度、基因类型及长度等基本信息,利用Excel进行统计,分析各物种基因组间差异。对于未知LSC/SSC/IRs长度的叶绿体基因组(如猴面柯、短刺锥、华南青冈等)首先使用Blast进行自身序列比对,获取IR区长度信息,然后确定IR区边界,进而得出LSC和SSC的长度信息。
1.3 系统发育树构建和分析
使用14个物种的叶绿体全基因组序列进行系统发育分析。采用ClustalX 2.0做多重序列比对,选择默认参数,Jukes-Canter进行序列间的差异度校正。应用MEGA 7.0的NJ(neighbor-joining)法中的p-distance模型构制系统进化树,进行1 000次重复,选取近缘种桦木科榛(Corylusheterophylla,GenBank登录号KX822769.2)作为外类群。
2 结果与分析
2.1 基因组结构基本特征
壳斗科叶绿体基因组相对保守,均为包含1个LSC、1个SSC、2个IR的四分体结构,未检测到区域碱基大片段的缺失。全基因组大小在159 938~161 304 bp,其中北美红栎最大,三棱栎最小,仅相差1 366 bp。LSC长度在89~90 kB,差异1 222 bp,SSC为18~19 kB,相差557 bp,IR大小均为25 kB,不同物种间仅有数百个碱基差异。大多数物种的2个IR区长度一致,但川滇高山栎、刺叶高山栎、三棱栎的IRa与IRb区域相差1~3 bp,华南锥差异19 bp。叶绿体基因组GC含量全部为36%,不同物种间最大差异仅0.28%,栗属的2个物种则完全一致(表2)。

表2 壳斗科叶绿体基因组的结构比较
2.2 基因数量分析
基因数量在壳斗科各物种间有所变化,总基因数在115~137个(表2),川滇高山栎数量最少,仅有115个,北美红栎最多,有137个。在所有基因中,rRNA的数量最为保守,均为8个。tRNA数目在29~46个,其中大多数物种约为40个,但川滇高山栎、刺叶高山栎、华南青冈等3个物种的tRNA数目约为30个,引起差异的主要原因是缺少trnK、trnM、trnL、trnV、trnT、trnI、trnA等基因;华南锥为46个,因其基因组中trnG、trnF、trnP、trnC、trnN等基因的数量较多。蛋白编码基因为78~89个,检测到infA、petG、rpl22、ψycf1(ycf1假基因)、ycf15等多个基因发生缺失,infA基因在川滇高山栎、锥栗、C.pumila、三棱栎等4个物种的叶绿体基因组中发生丢失,petG基因在华南锥、短刺锥中丢失,rpl22基因在锥栗、C.pumila、华南青冈、三棱栎中丢失,ycf1假基因在川滇高山栎、刺叶高山栎、锥栗的叶绿体基因组中丢失,ycf15基因在除北美红栎、刺叶高山栎、华南青冈以外的11个物种中丢失。此外,川滇高山栎还丢失psbI、psaI、petL、psaJ、rps16、rpl32等6个基因,华南锥丢失rps12基因和petD基因,三棱栎丢失了ycf1基因。
北美红栎、华南锥、短刺锥、猴面柯、三棱栎等5个物种在进化过程中发生了基因替换事件,由lhbA基因替换了原有的psbZ基因。华南青冈的rps7基因和ycf15基因位置发生了颠倒。猴面柯物种的叶绿体基因组插入了1个orf188蛋白编码基因。
2.3 基因长度变异
对壳斗科14个物种的叶绿体基因组蛋白编码基因长度进行分析,发现主要有32个蛋白编码基因长度发生变异,其中包括12个含内含子基因和20个无内含子基因。华南锥物种的变异程度较高,有55个蛋白编码基因长度发生了变化。
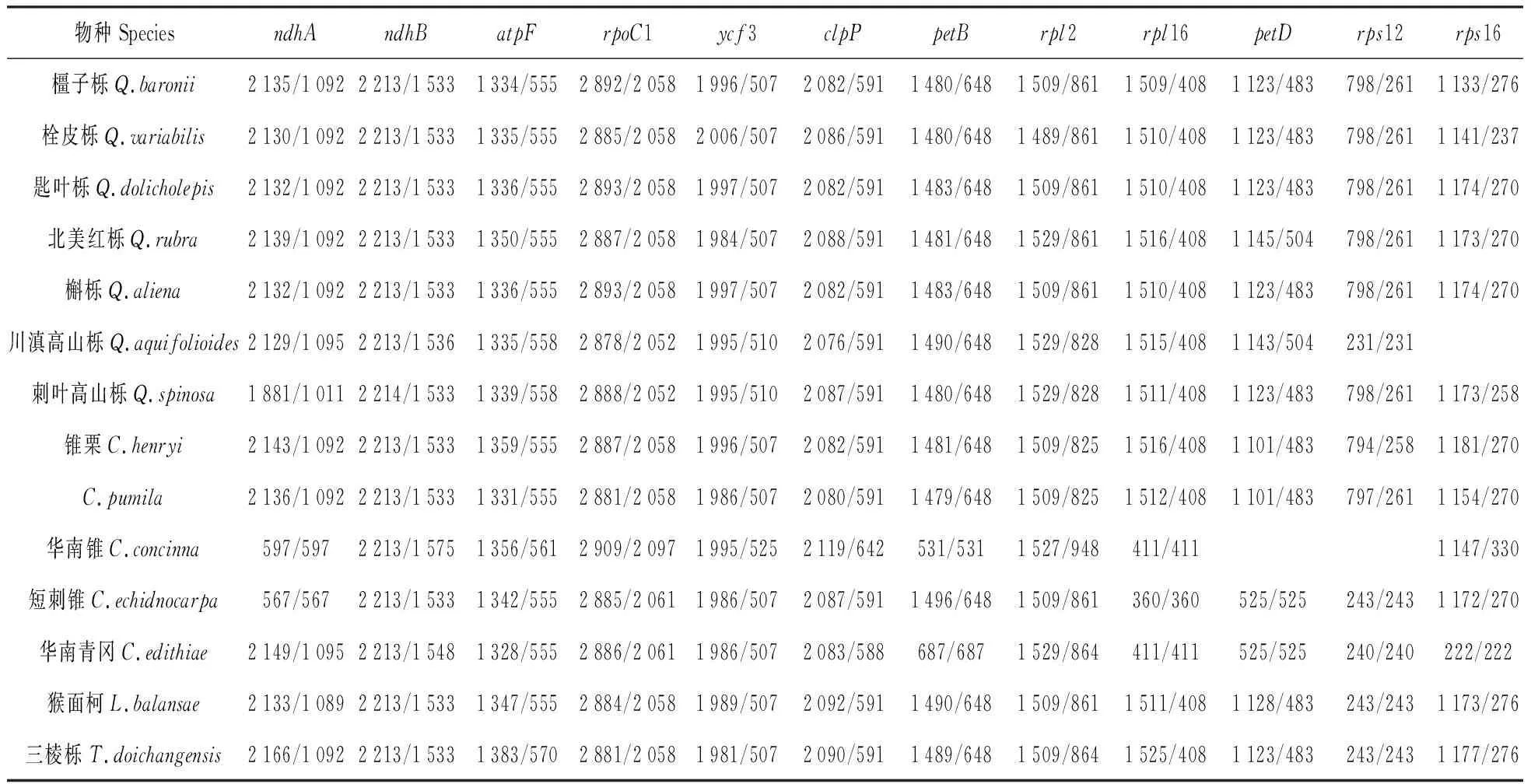
在12个含内含子的蛋白编码基因中,基因长度变异的主要类型是内含子的丢失、编码区或内含子长度的改变(表3)。华南锥的叶绿体基因组缺失了petB、rpl16、ndhA基因的内含子,短刺锥物种缺失了rps12、petD、rpl16、ndhA基因的内含子,华南青冈的叶绿体基因组缺失了rps16、rps12、petD、petB和rpl16基因的内含子,猴面柯、三棱栎、川滇高山栎等3个物种丢失了rps12基因内含子。ndhA基因在大多数物种中基因长度和编码区长度为2 140 bp和1 100 bp左右,而在刺叶高山栎中基因长度和编码区长度均发生了变化,在华南锥和短刺锥2个物种中编码区长度为597和567 bp,缺失了约500 bp。rpl16、ycf3、petB等基因的编码区在大部分物种中具有一致长度,但其内含子区长度的改变导致了基因总长的变化。
表3壳斗科叶绿体基因组中含内含子蛋白编码基因的全长和编码区长度
Table3ThechloroplastgenomeofFagaceaecontainsthefull-lengthcodingregionofintronproteinandthelengthofcoding
物种SpeciesndhAndhBatpFrpoC1ycf3clpPpetBrpl2rpl16petDrps12rps16橿子栎Q.baronii2135/10922213/15331334/5552892/20581996/5072082/5911480/6481509/8611509/4081123/483798/2611133/276栓皮栎Q.variabilis2130/10922213/15331335/5552885/20582006/5072086/5911480/6481489/8611510/4081123/483798/2611141/237匙叶栎Q.dolicholepis2132/10922213/15331336/5552893/20581997/5072082/5911483/6481509/8611510/4081123/483798/2611174/270北美红栎Q.rubra2139/10922213/15331350/5552887/20581984/5072088/5911481/6481529/8611516/4081145/504798/2611173/270槲栎Q.aliena2132/10922213/15331336/5552893/20581997/5072082/5911483/6481509/8611510/4081123/483798/2611174/270川滇高山栎Q.aquifolioides2129/10952213/15361335/5582878/20521995/5102076/5911490/6481529/8281515/4081143/504231/231刺叶高山栎Q.spinosa1881/10112214/15331339/5582888/20521995/5102087/5911480/6481529/8281511/4081123/483798/2611173/258锥栗C.henryi2143/10922213/15331359/5552887/20581996/5072082/5911481/6481509/8251516/4081101/483794/2581181/270C.pumila2136/10922213/15331331/5552881/20581986/5072080/5911479/6481509/8251512/4081101/483797/2611154/270华南锥C.concinna597/5972213/15751356/5612909/20971995/5252119/642531/5311527/948411/4111147/330短刺锥C.echidnocarpa567/5672213/15331342/5552885/20611986/5072087/5911496/6481509/861360/360525/525243/2431172/270华南青冈C.edithiae2149/10952213/15481328/5552886/20611986/5072083/588687/6871529/864411/411525/525240/240222/222猴面柯L.balansae2133/10892213/15331347/5552884/20581989/5072092/5911490/6481509/8611511/4081128/483243/2431173/276三棱栎T.doichangensis2166/10922213/15331383/5702881/20581981/5072090/5911489/6481509/8641525/4081123/483243/2431177/276

表4 壳斗科叶绿体基因组中部分不含内含子的蛋白编码基因长度

图1 壳斗科物种叶绿体基因组的4部分IR区边界比较 灰色方框代表基因,紫色方框代表基因重叠区域,蓝色方框代表LSC区,绿色方框代表SSC区,橙色方框代表IR区;图形大小与实际长度不成比例,且基因与LSC、SSC和IR区长度不成比例。Fig.1 Comparisons of 4 regions IR region boundary of chloroplast genomes in Fagaceae Gray boxes represent genes,purple boxes represent overlapping regions of genes,blue boxes represent LSC regions,green boxes represent SSC regions,orange boxes represent IR regions; size of the profile is not proportional to actual length,and gene length is not proportional to the lengths of LSC,SSC and IR regions.
20个无内含子的蛋白编码基因长度在各个物种间均发生不同程度的变异(表4)。同属之间,变异程度相近,锥栗和C.pumila2个同属间物种的matK、atpI、psbC、ndhK、rbcL、ycf1等基因在变异后长度一致。ndhF基因长度在三棱栎中为1 506 bp,而在其他13个物种中均为2 250 bp左右,缺失了约750 bp的片段。ndhH基因在刺叶高山栎中为972 bp,在华南锥中1 197 bp,而在其他12个物种中均为1 182 bp,则刺叶高山栎的ndhH基因长度缺失210 bp。ycf1假基因长度约1 100 bp,而在三棱栎中为795 bp,推测其是在复制过程中发生了丢失。rpl22基因在部分物种中完全缺失,在未缺失物种中具有不同的长度,说明基因序列是在进化过程中逐渐退化的。
2.4 IR边界变化
叶绿体基因组的环状结构特征使2个反向复制的IR区与LSC及SSC间存在4个边界,既IRb-LSC、IRb-SSC、IRa-SSC和IRa-LSC。在基因组进化过程中,4个边界会发生扩张或收缩,使得有些基因进入到IR区域,或者使有些基因进入到单拷贝区,每个物种边界处序列复制的程度不同。通过比较发现,壳斗科叶绿体基因组的绝大多数边界较为保守,但极少数边界存在扩张或收缩现象(图1)。IRb-LSC边界在大部分物种中与rps19基因存在8~32 bp的间隔,而在锥栗、C.pumila和华南锥3个物种中则位于rps19基因内,且三棱栎物种的该边界与rps19基因距离423 bp。大多数物种的IRb-SSC边界处于ycf1假基因上,但C.pumila的该边界位于ycf1假基因和ndhF基因的重叠区内,锥栗的位于ndhF基因内;IRb-SSC边界和ndhF基因间的间隔一般为1~25 bp,而在三棱栎物种中间隔为1 023 bp。IRa-SSC边界在除三棱栎外的其他所有物种中均位于ycf1基因内,ycf1基因存在于IRa区中的序列长度在828~1 107 bp,差异较小。IRa-LSC边界与LSC上的trnH基因间具有较短间隔,长度分别为1、8、12、15和16 bp,其中16 bp为主要类型。
2.5 系统发育分析
以壳斗科14个物种的叶绿体全基因组序列为研究基底,以榛为外类群,构建了系统发育树。如图2所示,在所构建的系统发育树中,绝大部分的节点都具有较高的支持率,壳斗科的14个物种间展示出了明确的系统关系。三棱栎物种位于系统发育树的基部,是壳斗科植物中最早分离出来的物种,其余的13个壳斗科植物共同聚到1个大分支中,该分支又可分为2个亚分支,亚分支Ⅰ和亚分支Ⅱ。亚分支Ⅱ仅包括猴面柯,作为1个基部分支,说明猴面柯是壳斗科物种中较早分化出来的类群。亚分支Ⅰ则由栎属、栗属、锥属、青冈属等4个属的12个物种聚成,其中栗属和锥属的2个分支呈姐妹关系,不同属的刺叶高山栎与华南青冈物种聚为一支,且该支较早分离出来并获得100%的高支持率。

图2 基于壳斗科叶绿体全基因组序列构建的系统发育树Fig.2 Phylogenetic tree based on chloroplast genome sequence of Fagaceae
3 讨论
被子植物叶绿体基因组多以典型的环式双链结构形式存在,2个反向复制区将基因组分隔成大单拷贝区和小单拷贝区,基因组大小在120~180 kB,其结构和序列相对保守,极少发生大程度变异[9]。14个壳斗科物种的叶绿体基因组结构均包含1个LSC区、1个SSC区、2个IR区,大小为159~161 kB,与上述被子植物叶绿体基因组基本结构特征相吻合,与双子叶原始类群烟草相比较,基因组有所增大。同时发现,壳斗科14个物种间基因组大小差异1 366 bp,LSC长度差异1 222 bp,则LSC区域变异占全基因组变异的绝大部分,因此认为整个基因组长度的差异主要是由LSC长度的差异引起的[10~11]。
叶绿体基因组具有较高的基因转换能力,以确保2个IR反向复制区序列的一致与稳定,从而增强其自身的稳定性和保守性[12],但在物种不断进化过程中,仍有一些类群的IR区发生较大程度变异。如被子植物叶绿体基因组IR区大小多为20~30 kB,而天竺葵属的IR区长达75 kB[13],老鹳草属的短至11 kB,牻牛儿苗属和一些豆科植物的则完全丢失[14]。壳斗科叶绿体基因组的IR区相对稳定和保守,大小均为25 kB,没有出现区域过长、过短或者丢失的现象,且大部分物种中2个IR区长度一致,但川滇高山栎、刺叶高山栎、三棱栎和华南锥4个物种由于IRa区或者IRb区内若干碱基的插入/缺失而导致长度不一致。
壳斗科叶绿体基因组基因总数在115~137个,各物种间数量有所差异,其中rRNA基因均为8个,因此差异主要来自tRNA基因和蛋白编码基因,trnK、trnM、trnF、trnP等多个tRNA基因在叶绿体基因组中缺失或增多,infA、petG、rpl22、ycf1、ycf15等多个蛋白编码基因在部分物种中丢失。在叶绿体基因组中,基因丢失现象较为常见,如rpl22、rpl23、accD、ycf1、rps16、infA等基因在豆科植物的叶绿体基因组中发生部分或全部丢失[15],ycf1、ycf2、accD基因在整个禾本科植物中全部丢失[16]。ycf1基因是一个较大的开放阅读框,在大部分被子植物中具有2个拷贝,一个拷贝为完整基因,另一个拷贝则由于被IRb-SSC边界所截断,为一个假基因[10],而在壳斗科的川滇高山栎、刺叶高山栎、锥栗和三棱栎4个物种的叶绿体基因组中则完全丢失了ycf1的一个拷贝,结合壳斗科植物特征推断,其可能是与IRs的扩张或收缩有关。rpl22基因在壳斗科部分类群的叶绿体基因组中丢失,但在部分类群的核基因组中出现,研究推测是从叶绿体基因组中转移而来,在细胞核中进行转录,然后再将编码的蛋白转运回叶绿体参与生命活动[17]。
壳斗科植物的80多个蛋白质编码基因中,主要有32个基因的长度发生变异,其变异的主要原因是内含子的丢失、编码区或内含子长度的改变。如rps16、ndhA、rps12、petD等基因在不同物种的叶绿体基因组中发生内含子的丢失,rpl16、ycf3、petB等基因的编码区在大部分物种中具有一致的长度,但因其内含子区长度的改变而导致了基因长度的变化,ndhA基因在壳斗科植物中内含子的丢失、编码区和内含子长度改变等均有发生。同时,壳斗科植物的一些基因长度变化表现出了一定的系统进化关系,如matK、atpI、psbC、ndhK、rbcL等基因的长度在大部分物种中较为稳定,只在少数物种中发生变化,而这些物种多具有很近的亲缘关系。
在叶绿体基因组进化过程中,IR区为延展或者收缩的区域,即4个IR边界在同属或不同属物种间产生滑动的现象时有发生,使得叶绿体基因组大小出现较大差异。在十字花科[10]和五加科[11]中,IRb-LSC边界均位于rps19基因内部,而在壳斗科中,IRb-LSC边界在大多数植物中与LSC上的rps19基因存在8~32 bp的较小间隔,但在锥栗、C.pumila、华南锥中则由于边界的扩张而位于rps19基因内部,在三棱栎中由于边界的收缩而与rps19基因具有较大间隔。与五加科[11]相同,大多数壳斗科植物的IRb-SSC边界位于ycf1假基因内部,而C.pumila、锥栗2个物种的边界由于扩张而分别位于ycf1假基因与ndhF基因重叠区内和ndhF基因内。IRa-SSC边界均位于ycf1基因上,但边界的收缩或扩张使得ycf1基因存在于IRa中的长度具有一定差异。壳斗科的IRa-LSC边界与LSC上的trnH基因间具有稳定间隔,且trnH基因仅有1个拷贝,而剪股颖、大麦、高粱等单子叶植物的trnH基因虽同样在LSC上,却具有2个拷贝,这是由于边界扩张使得trnH基因首先进入到IR区,从1个拷贝变成2个拷贝,之后边界又发生收缩,使2个拷贝的trnH基因回归到LSC区内[18],这表明双子叶植物与单子叶植物边界变化存在较大差异。
前人通过现有化石资料、地理信息系统技术等对壳斗科及其各属的起源时间、地理分布进行了大量研究,结果显示,三棱栎属的物种化石最早出现,青冈属较栎属更接近祖先类群[19];柯属、锥属、青冈属、栎属、栗属间的空间分布区较相似,三棱栎属与其他属的空间分布区相似性较低[20]。本研究利用壳斗科植物6个属14个物种的叶绿体全基因组序列构建了系统发育树,该发育树具有良好的分辨率,各个进化支获得较高的支持率,各个属间表现较明确的发育关系,各属归类与传统分类学基本一致。三棱栎位于发育树的基部进化支,说明三棱栎是14个壳斗科物种中较早分化的一个类群,与前人研究结果相同。不同属的刺叶高山栎与华南青冈物种聚为一支,并位于所在小分化支的基部,说明刺叶高山栎较其他栎属物种更早分化出来,且与华南青冈物种有很近的亲缘关系,与传统分类学中两者原为一属[21]的说法相一致。
本研究以壳斗科14个已测序物种的叶绿体基因组为基础来进行比较研究,揭示了壳斗科叶绿体基因的一般结构及变异特征,并基于叶绿体全基因组序列构建系统发育树,阐明了物种间的发育关系。但目前已测序的物种仅仅占整个壳斗科物种的极小部分,未来还将需要获得更多壳斗科物种的叶绿体基因序列,从而进一步了解壳斗科叶绿体基因组基本特征和进化规律。
