基于生物信息学分析肺炎链球菌LytR蛋白特征*
2018-08-29吴凯峰张競之童华波王正蓉
吴凯峰, 张競之,查 何,刘 雁,童华波,潘 卫,王正蓉
(1.贵州省遵义市第一人民医院/遵义医学院第三附属医院检验科 563000; 2.贵州医科大学检验医学院,贵阳 550004)
肺炎链球菌是社区获得性肺炎最常见的细菌性病原菌,其所致的肺炎也是重症社区获得性肺炎入住重症监护室最常见的原因[1]。2013年,全球约有40万6岁以下儿童死于肺炎链球菌感染[2]。此外,全球也正面临较为严峻的肺炎链球菌抗生素耐药,以及7价或13价肺炎链球菌蛋白结合疫苗在地区之间保护效果不一和保护率有限等问题[3-4]。为切实提高对肺炎链球菌感染的防治能力,需要深入了解肺炎链球菌的分子致病基础。
磷壁酸是革兰阳性细菌细胞壁重要的多糖分子。在肺炎链球菌中,磷壁酸也是一种极为重要的毒力因子[5-6],该分子在感染过程中有着与其他细菌磷壁酸相似的生物学功能,且可能具有某些新特征。当细菌磷壁酸显著减少时,相应的肺炎链球菌的生理学功能将严重受损,细菌致病力也将明显降低[5]。由此可见,研究磷壁酸的合成过程对指导设计新型抗菌药物或疫苗具有重要的理论价值。本课题组前期研究发现RafX蛋白参与了磷壁酸的生物合成,体内外研究显示,RafX蛋白与肺炎链球菌毒力有密切关系,RafX基因缺陷的肺炎链球菌毒力明显下降[7]。然而,肺炎链球菌磷壁酸并未因RafX蛋白的缺失而完全减少[7],因此,本课题组猜测还存在替代途径参与磷壁酸的合成。
肺炎链球菌存在一个名为LytR_cps_psr (LCP)蛋白家族,该家族蛋白被认为与细菌胞外多糖共价连接至细胞壁肽聚糖有关[8-9],该家族成员包括spr1759 (LytR)、Cps2A和Psr等。其中,LytR蛋白功能尚未完全清楚。闵迅等[10]前期已利用原核表达方式对肺炎链球菌LytR蛋白(肺炎链球菌R6菌株即spr1759)进行过表达,晶体生长及衍射分析,然而由于技术原因,尚未对结构进行解析和功能研究。因此,本研究试图利用生物信息学方法,从美国国立生物技术信息中心(NCBI)数据库中提取肺炎链球R6标准菌株的spr1759基因及其编码蛋白序列,分析其基本理化性质、结构功能特征等,为肺炎链球菌的LytR蛋白功能研究及细菌致病机制和防治策略研究奠定基础。
1 材料与方法
1.1肺炎链球菌spr1759基因及其编码氨基酸序列和生物信息学策略 从NCBI数据库检索并下载肺炎链球菌R6标准菌株spr1759及其编码全长氨基酸序列(protein ID 为NP_359351.1)。本研究主要利用BioEdit Sequence Alignment Editor、TMHMM、ExPASy、Megalign等多种软件对LytR进行分析。所用在线工具的网址链接参见前期文献报道[11]。
1.2方法
1.2.1肺炎链球菌LytR蛋白的理化性质预测 利用Bioedit Sequence Alignment Editor软件对spr1759蛋白氨基酸组成进行分析。从NCBI数据库Protein数据字库搜索NP_359351.1获得FASTA格式的全长蛋白序列,使用Bioedit软件打开spr1759蛋白序列文件,点击Sequence,找到Protein,再选择Amino Acid Composition,获得蛋白构成信息。利用ProtParam预测相关spr1759蛋白原子组成、等电点、不稳定系数进行预测。进入ExPASy ProtParam网站,输入spr1759蛋白序列,点击Compute parameters,即可获取相关参数,其中,GRAVY值为正,提示疏水性强;GRAVY为负,提示亲水性强。此外,再利用ProtScale预测spr1759蛋白的疏水性/亲水性,进入ExPASy ProtScale网站,输入spr1759蛋白序列,选择Kyte & Doolittle,点击Submit,获得Kyte和Doolittle疏水分析信息,预测值为正,表明疏水性强;反之,负值表示亲水性强。
1.2.2肺炎链球菌spr1759蛋白的结构功能预测 分别利用Tmpred和TMHMM对spr1759蛋白进行跨膜螺旋结构预测。进入ExPASy TMpred网站,输入spr1759蛋白序列,点击Run TMpred;进入TMHMM Server V.2.0,输入spr1759蛋白序列,点击Submit,获得跨膜螺旋结构信息。利用SignalP预测其信号肽;进入SignalIP 4.1 Server,输入spr1759蛋白序列,点击Submit,获得信号肽信息。利用InterProScan和SMART对spr1759蛋白的功能域进行分析。进入EMBL-EBI InterPro网站,输入spr1759蛋白序列,点击Submit,获得功能域信息;进入SMART主页,输入spr1759蛋白序列,点击Sequence SMART,获得功能域信息。利用SOPMA和Predictprotein预测蛋白质长spr1759蛋白的2级结构。进入SOPMA的2级结构预测网站,输入spr1759蛋白序列,点击Submit,获得α-螺旋、β-折叠、β-转角和无规则卷曲比例。进入Predictprotein网站,输入spr1759蛋白序列,点击PredictProtein,登录获得二级结构信息。将来源不同细菌的序列输入Megalign软件进行序列间比对。
2 结 果
2.1spr1759蛋白的理化特征 肺炎链球菌spr1759蛋白理论等电点为5.11,相对分子质量为37.56×103,由338个氨基酸残基构成,包含31个Leu(9.2%),30个Glu(8.9%),30个Gly(8.9%),29个Ile(8.6%);该蛋白碱性氨基酸和酸性氨基酸总数分别为39个(11.5%)和45个(13.3%)(图1A)。全长spr1759蛋白氨基酸脂肪指数97.16,GRAVY值为-0.218,稳定指数为44.95,属于不稳定蛋白。从图1B可见,全长spr1759蛋白肽链,亲水性氨基酸多于疏水性氨基酸,第214位氨基酸残基分值最低(-3.000),第10位氨基酸残基分值最高(2.700)。
2.2一级结构中所含结构和功能域特征序列分析 InterProScan分析结果显示,spr1759蛋白仅含有1个结构功能域,属于细胞壁相关转录衰减因子(图2A)。另外,用NCBI网站分析软件对全长spr1759蛋白进行了保守结构域的分析,发现S.pneumoniae spr1759蛋白属于LytR_cpsA_psr超家族蛋白(图2B),其E值为1.24e-93,提示一种阴离子细胞壁多聚物合成相关酶的功能。SMART分析进一步支持了上述分析结果,spr1759蛋白该结构域属于LytR_cps_psr结构域。
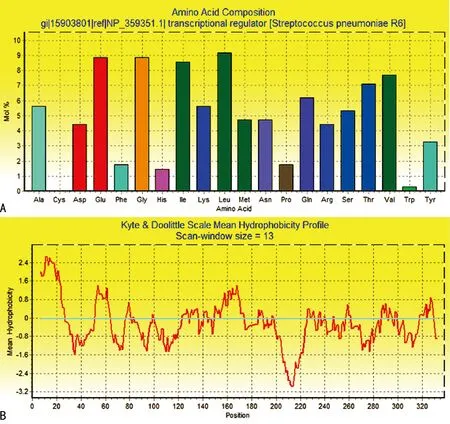
A:spr1759氨基酸组成;B:Kyte和Doolittle疏水性分析
图1肺炎链球菌spr1759蛋白氨基酸组成分布和疏水性
2.32级结构特征 spr1759蛋白SOPMA预测结果显示,该蛋白α-螺旋占48.52%,β-折叠占17.16%,β-转角占7.40%,无规则卷曲占26.92%。Predictprotein预测结果与SOPMA基本一致,从图3A可见,α-螺旋和β-折叠散在分布蛋白中,作为整个蛋白的刚性支撑结构,α-螺旋占31.95%,β-折叠13.91%;无规则卷曲结构为蛋白的柔性区域,占54.14%。Tmpred预测跨膜结构提示,该蛋白总分值大于0,最高值达2500,在氨基端具有跨膜结构(图3B)。LytR蛋白进行信号肽分析发现,该蛋白存在信号肽切割位点,位于蛋白第24和第25位氨基酸之间,cut-off值为0.420(图3C),InterProScan分析结果进一步证实了SignalP和Tmpred的预测结果。利用TMHMM软件对S.pneumoniae spr1759蛋白(图3D)与LytR家族蛋白成员B.anthracis BAS5115(图3E)和B.subtilis DJ97_1211(图3F)进行了2级结构跨膜区域的比对,发现3者均含有1个跨膜结构(TM)和胞外结构。

A:InterProScan预测肺炎链球菌R6 spr1759蛋白一级结构中的保守功能域;B:NCBI蛋白保守结构域分析
图2生物信息学分析肺炎链球菌spr1759的保守结构域

A:以Predictprotein预测蛋白结构;B:使用Tmpred预测蛋白跨膜结构;C:SignalP预测信号肽,利用TMHMM分析;D:Streptococcus pneumoniae spr1759跨膜结构;E:Bacillus anthracis BAS5115跨膜结构;F:Bacillus subtilis DJ97_1211跨膜结构
图3肺炎链球菌R6 LytR的拓扑结构、二级结构特征
2.4系列比对 为了进一步明确肺炎链球菌spr1759蛋白的功能,本研究将其与LytR-Cps-Psr家族其他成员的一级结构进行比对,结果显示,Streptococcus pneumoniae LytRvs.Bacillus anthracis BAS5115,Streptococcus pneumoniae LytRvs.Bacillus subtilis DJ97_1211蛋白序列一级结构比对spr1759与BAS5115和DJ97_1211序列相似性分别为35.7%和32.5%(图4),相似性超过30.0%。

图4 LytR家族蛋白成员一级结构对比
3 讨 论
任何一种抗菌药的使用总是会伴随着耐药菌的逐渐增加而失去功效,例如,我国肺炎链球菌临床分离株对红霉素几乎全部耐药[12]。因此,为能长期有效控制细菌感染,人类需要不断研发与原有抗菌药物无交叉耐药性的新抗菌药,这就需要不断挖掘新的作用靶点。
LCP蛋白家族在很多阳性细菌中具有重要作用,主要参与细胞壁合成[8,13]。通过系列分析本课题组发现肺炎链球菌也存在类似蛋白,LytR蛋白就是其中之一。为了探究其可能的生物学功能,本研究利用生物信息学软件对蛋白进行分析,结果发现该蛋白预测相对分子质量为37.56×103,具有跨膜结构,脂溶性差,亲水性好,这一预测结果与闵迅等[10]前期研究是吻合的。闵迅等利用原核表达系统对蛋白进行过表达,结果发现带有跨膜结构(信号肽部分片段)无法实现蛋白游离表达,而将跨膜结构(氨基端30氨基酸)去除后,成功实现了蛋白的可溶表达,一方面证实了该蛋白具有跨膜结构,位于氨基端;另一方面也提示使用TMHMM和Tmpred等生物信息学软件分析蛋白跨膜结构域是一种快速、简便和正确的方式。
使用InterProScan和SMART对LytR蛋白结构域进行分析发现,该蛋白具有Lcp蛋白家族功能结构域,属于细胞壁代谢相关转录衰减因子,然而从细胞定位分析和跨膜结果预测并不支持其作为转录因子的功能,此外,EBERHARDT等[14]已经利用GFP蛋白示踪的方法显示肺炎链球菌LytR蛋白(spr1759)位于细菌细胞膜,因此,该蛋白是一种转录因子的可能性较小。序列比对分析结果显示,肺炎链球菌LytR蛋白与炭疽杆菌的BAS5115二级结构相似,都含有1个跨膜结构域和1个功能结构域;二者一级结构相似性超过30%,说明二者具有较高的同源性。查阅文献发现,炭疽杆菌BAS5115具有酶学功能[15],因此,本课题组认为spr1759可能也具有酶学的功能。那么,肺炎链球菌spr1759蛋白功能究竟如何,仍需进一步研究。
综上所述,本课题组利用生物信息学对肺炎链球菌LytR蛋白的理化性质,跨膜结构和功能进行了分析,结果提示LytR蛋白可能参与肺炎链球菌细胞壁多糖的组装连接过程。目前,通过抑制LytR蛋白发挥抗菌作用的药物尚未见报道,本研究提示LytR可能具有这一重要酶学功能,将为后续LytR蛋白的定位和功能实验室研究奠定基础,也将为未来针对LytR蛋白靶点药物的开发铺垫。
