黑斑侧褶蛙Temporin基因内含子在PCR扩增、克隆和Sanger测序过程中的复制跳跃
2018-08-21徐艳春杨淑慧
崔 杰 林 煜 徐艳春 杨淑慧
(东北林业大学野生动物资源学院,哈尔滨,150040)
聚合酶链式反应(Polymerase Chain Reaction,PCR)是指在DNA聚合酶的催化下,以母链DNA为模板,以特定引物为延伸起点,通过变性、退火、延伸等步骤,体外复制出与母链模板DNA互补的子链DNA的过程[1],是一项能够快速、特异地在体外扩增目的DNA的技术,是生物学研究中应用最为广泛的实验室技术之一[2-4]。
PCR产物经常需要经过克隆后进行测序或其他分析。克隆时将PCR产物与特定载体DNA分子连接,形成重组DNA分子,转入感受态细胞中通过细胞和质粒的不断复制,生成大量目标分子[5-7]。随着新型载体的不断出现,如T-载体等,在很大程度上简化了PCR产物的克隆过程,同时也提高了克隆效率[8-10]。
但是,PCR并不总是完全忠实的,除了引物与模板的非特异结合产生非特异性扩增产物以外,因为模板的高级结构、聚合酶的某些属性等,还经常会介导DNA的重组(recombination)、复制滑动(slippage)和跳跃(jumping)等[11]。
重组是DNA聚合酶在按照模板合成新链的时候,因为另一条与模板序列高度相似的片段与模板在空间上接近,聚合酶从原来的模板链上移动到该片段上继续合成新链,最终使PCR产物成为两个不同模板链的重组产物[12]。复制滑动在微卫星等短串联重复序列的扩增中十分常见,DNA聚合酶在合成新链时越过一个或几个重复单位,造成PCR产物比模板链缩短重复单位整数倍[13]。
跳跃扩增是当模板DNA自身因为同原序列形成较为稳定的环形局部二级结构,新链合成时DNA聚合酶移动到该区域后,就可能跨过这个环形区继续向下合成新链,从而使PCR产物较模板丢失了一个片段[14]。这种现象最早报道的是Cariello et al(1991)在扩增人18S rDNA基因时发现了中间缺失54 bp的异常产物[15]。与微卫星等短串联重复序列的滑动相比,较大片段的跳跃更难发生,在实验中也十分罕见。正因如此,PCR的跳跃问题常常被忽视,造成错误结果。
PCR产物的克隆是利用细菌内的DNA聚合酶等复制体系,将目标DNA片段进行在体复制。因为细菌复制体系的忠实性远高于一般的TaqDNA聚合酶(无3′-5′外切酶活性),所以利用质粒复制进行分子克隆通常认为是十分精准的。然而,在特殊情况下,如目标分子中含有某些特殊的回文结构时,细菌质粒在复制过程中也会出现跳跃或滑动[16]。
我们在研究黑斑侧褶蛙(Pelophylaxnigromaculatus)抗菌肽Temporin基因结构时,曾遇到在PCR扩增和扩增产物T-A克隆过程中同时出现跳跃而引起长达560 bp的片段缺失,并发现与一段6 bp的重复单元有关。我们报道这一实验结果,为更多的研究提供借鉴。
1 材料与方法
1.1 样品
2015年,在吉林、长白山、佳木斯、富锦、银川、西安、太原等20个地区采集成年黑斑侧褶蛙,每个地区采集5只,野外双毁髓处死后立即采集后腿肌肉样品于液氮中保存,然后转移至-80℃冰箱中保存。
1.2 总DNA的提取
取20 mg肌肉组织剪碎后分别加入25 μL Proteinase K(10 mg/mL)、35 μL SDS(10%)、295 μL TNE buffer(pH=8),充分混匀后瞬时离心,56℃水浴消化1 h。消化液采用AxyPrep基因组DNA提取试剂盒(AxyPrep,杭州)提取总DNA,操作按照说明书进行。
1.3 PCR反应
采用DNAstar软件包的Primer Select软件,以黑斑侧褶蛙抗菌肽 Temporin基因序列为模板设计该基因内含子的扩增引物[17-18],上游引物C2:GCAGCAGCTGAAACACTGAAT,下游引物D2:AGTTATACCAATTGTGTCAGGTC。反应体系为:PimeSTAR MAX(Takara,大连)25μL,上下游引物(10 μmol/L)各1.0 μL,DNA模板(10 ng/μL)5.0 μL,ddH2O 18 μL。反应程序为:94℃/5 min,(94℃/30 s,55℃/1 min,72℃/2 min)×35cycle,72℃/10 min。
1.4 PCR产物的克隆
PCP产物经1.5%的琼脂糖凝胶电泳后,用AxyPrep(AxyPrep,杭州)DNA纯化回收试剂盒回收,用pEASY-T5 Zero试剂盒(TransGen Biotech,北京)进行克隆,反应体系为5 μL,组分包括PCR产物1~4 μL、pEASY-T5 Zero cloning vector 1 μL。在25℃恒温条件下反应20~30 min。转化到DH5α大肠杆菌感受态细胞后,在LB液体培养基中37℃下培养过夜。阳性质粒经过1.3中相同的PCR方法鉴定后,采用Sanger法,使用M13+/M13-测序引物进行双向测序。
1.5 质粒提取与限制性双酶切
取700 μL过夜培养的菌液于2 mL离心管中10000 xg离心5 min,弃上清液。用350 μL PBS悬浮菌体细胞,具体操作按照AxyPrep质粒小量DNA提取试剂盒(AxyPrep,杭州)使用说明书进行。
根据pEASY-T5 Zero cloning vector自带酶切位点,采用PstI(5′...CTGCA↓G...3′)和NotI(5′...GC↓GGCCGC...3′)对质粒进行双酶,反应体系为:PstI 1 μL、NotI 1 μL、10 xFlyCut Buffer 5 μL、质粒DNA 20μL和ddH2O 25 μL,37℃孵育10 min。加入10 xDNA Loading Buffer至终浓度为1 x终止反应,用1.5%琼脂糖凝胶电泳检测酶切产物。
1.6 测序结果分析
测得的原始序列用DNAstar软件包中EditSeq去掉隆载体序列和引物序列。用MegAlign软件将获得的DNA序列与已有的参考序列进行对比,确定序列的正确性。至少3个克隆序列完全一致时才被确定为一个最终的DNA序列。采用RepeatAround 2.1软件查找和统计DNA序列中重复单元的性质、种类和重复次数[19]。
2 结果
如图1,PCR产物经琼脂糖电泳发现,大约有73%的个体在1200 bp和550 bp处同时出现清晰的两条带,18%个体较短的条带信号稍弱,9%的个体只有1200 bp的条带。将两处条带分别回收后并测序,结果显示较长的条带为1126 bp,较短的条带为562 bp,后者是在前者198 bp处开始缺失564 bp而成。
在后续实验中,对22个个体进行PCR扩增,虽然退火温度经过稍许调整,使用SuperMix(TransGen Biotech,北京)、High Fidelity DNA Polymerase(TransGen Biotech,北京)、LA Taq(Takara,大连)等不同的DNA聚合酶进行扩增,发现依然会有3种情况出现,基本没有变化,由此将二者推断为两个不同的等位基因。
但在对长片段进行Sanger测序时发现一种异常现象,模板实际长度为1126 bp,但测序结果中有时会只得到562 bp的短片段序列。该短序列是长片段在的198 bp处开始缺失564 bp而成。这表明短片段极有可能是PCR造成的一种假象,即在扩增反应中发生了片段缺失。
为了进一步验证这个缺失来自PCR过程,我们用回收的长片段作为模板进行PCR扩增,琼脂糖凝胶电泳检测PCR产物时发现3种情况,一是仅得到1126 bp的长片段,一是仅得到562 bp的短片段,再者两个片段同时出现(图2),对PCR产物测序也证明了这一点。

图2 以1126 bp的长片段为模板的PCR扩增产物电泳图Fig.2 Electrophorogram of the products of PCR templated with a 1126 bp fragment M为Marker DL2000,N为阴性对照(H2O);显示绝大多数扩增产物含有1126 bp的目的条带以外,还出现一个短片段(测序显示为560 bp),否定了图1中关于两个等位基因的推断 Showing that the PCR products from most samples contained two fragments,again corresponding to 1126 bp and 562 bp.This conflicts with the hypothesis that these are two different alleles because there is also a short fragment(sequencing showed 560 bp)that invalidates the deduction of two alleles as suggested by Fig 1.M is molecular marker DL2000,N is negative control(H2O)
为了完全排除模板污染的可能性,我们对长片段纯化回收之后进行了T-A克隆。提取阳性克隆的质粒后,用限制性内切酶Pst I和Not I把PCR产物从载体上切下来,再经琼脂糖电泳检测发现,阳性克隆中含有两种克隆片段,一种是含有与长片段完全相同的克隆片段,一种是与短片段完全相同的克隆片段(图3)。也就是说,1126 bp长的目的片段在不同菌落中,变成大小两种片段。测序表明,短片段仍然是长片段在198 bp处缺失564 bp而成。这表明,这个1126 bp长的片段不仅在PCR扩增过程中可以发生缺失,而且在大肠杆菌内的质粒复制过程中也能够发生缺失,这种现象十分罕见。
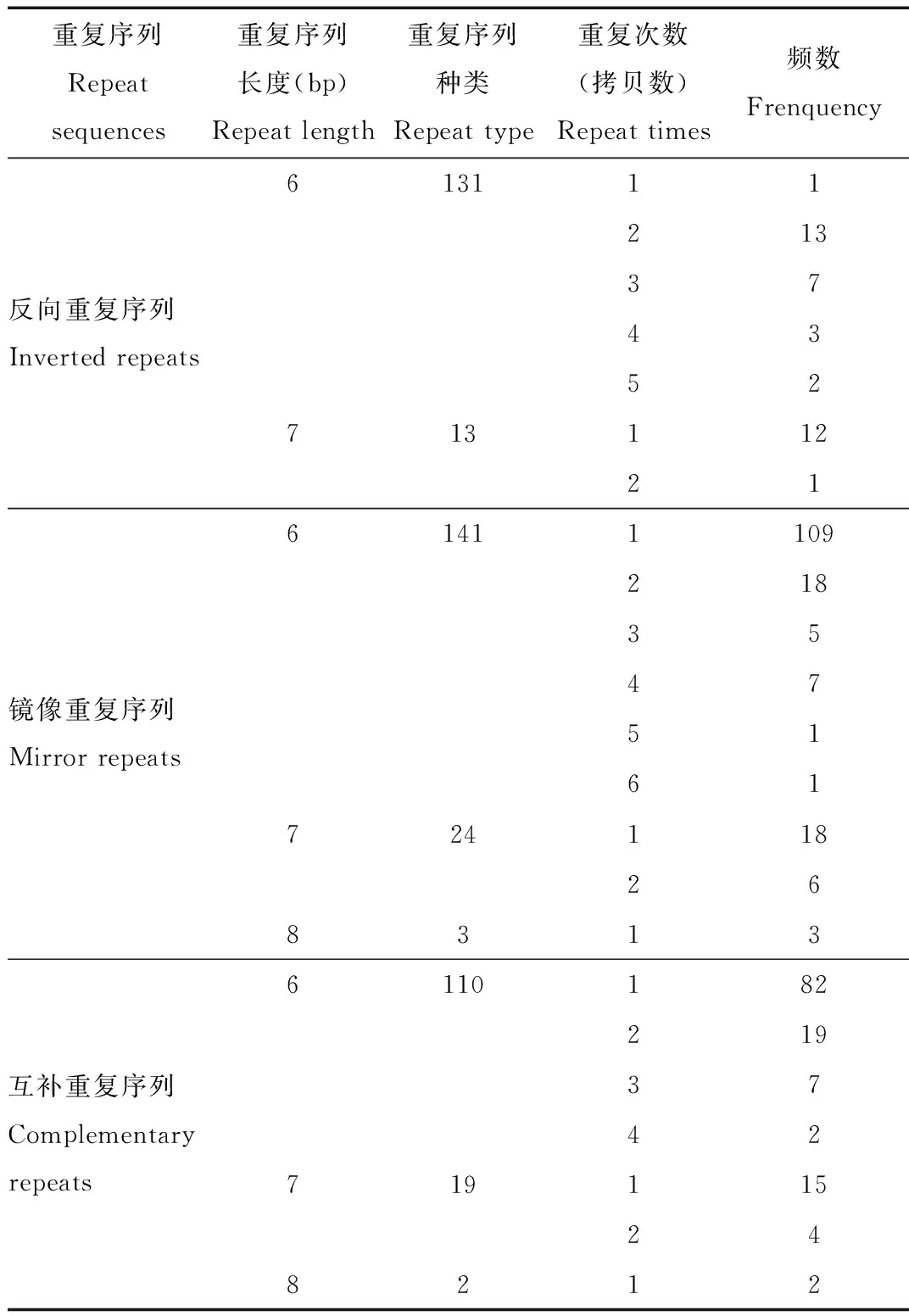
利用软件RepeatAround 2.1对1126 bp的片段进行重复序列分析,结果显示,该序列具有同向重复序列(direct repeats)70种、反向重复序列(inverse repeats)13种、镜像重复序列(mirror repeats)21种和互补重复序列(complementary repeats)21种(表1)。将1126 bp和526 bp的两个片段的序列进行比对发现,两个序列在缺失片段的上游和末端,分别有一个6 bp的同向重复序列5′-AGACCT-3′(图4),是序列缺失发生的位置,至此我们推测DNA复制到第二个重复序列的时候发生了跳跃(PCR Jumping)。
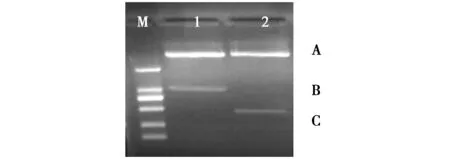
图3 阳性质粒的酶切电泳图Fig.3 Electrophorogram electrophoresis of positive plasmids 以1126 bp片段作为插入片段在质粒中进行克隆后,用限制性内切酶Pst I和Not I切割质粒,发现插入片段在一些克隆里保持1126 bp(1中的条带B),而在另一些克隆里变成526 bp(2中的条带C),条带A为酶切后的载体;M为Marker DL2000 While a 1126 bp fragment was cloned using pEASY-T5 Zero cloning vector,plasmids extracted from different positive clones contained two fragments of different sizes,i.e.,a 1126 bp true fragment(B)and a 526 bp pseudo-fragment generated during replication jumping(C)after double digested with the restriction enzymes Pst I and Not I.Band A is the vector,M is molecular marker DL2000
此外,序列比对还发现,在对应于长片段的894 bp位置开始,有一个4 bp(5′-TGCT-3′)的短片段的插入,而且无论采用总DNA作为模板扩增,还是采用单克隆长片段作为模板扩增,还是将单克隆长片段进行再克隆,只要出现片段缺失,就会出现这一插入序列,提示这一插入与片段缺失密切相关。
表1 黑斑侧褶蛙Temporin基因内含子1126 bp的长片段中可能引起跳跃的重复序列的情况统计

Tab.1 Repeat sequences predicted capable of inducing replicationjumping in the 1126 bp fragment on the intron of the Temporingene of black-spotted frog(Pelophylax nigromaculatus)
续表1
3 讨论
DNA序列的重复方式可以分为同向重复,反向重复,镜像重复和互补重复,如果在适当条件下形成必要的二级或更高级结构,其中一些序列可能导致复制跳跃或者滑动[20]。本研究中的1126 bp片段中检测到665种不同的重复序列(表1),但是多次反复扩增中,只在第194~199 bp位置上和第757~762 bp的位置上出现的重复序列5′-AGACCT-3′处发生了跳跃,使得扩增产物缺失了564 bp。 令人惊讶的是,由这对重复引起的跳跃在体外和体内复制都会产生。
Canceill等人提出了两种复制跳跃模型[21]。一种是与亲本双链形成发夹结构相关。子链合成在形成发夹结构的碱基处被封闭,然后在双链瞬时打开后经由DNA聚合酶的链置换活性继续,最后滑过发夹结构。然而,亲本双螺旋模型似乎不适用于我们的复制跳跃事件,因为在我们的研究中,子链中缺失了一个重复序列。这表明重复序列之一被并入发夹结构中,但是,在我们的研究中,第一次重复之后或第二次重复之前的序列都不能与重复序列形成稳定的发夹结构(图5A和5B)。
另一种模型就是异双螺旋模型,在第一次重复的时候阻断了子链合成,同时DNA聚合酶和子链从模板上脱落,下一个循环中,新链作为互补链,与第二个重复单位相配对、退火,并成为引物,DNA聚合酶再次结合,从第二个重复单位的下游开始完成新链的延伸[22]。这种情况很符合我们研究中的缺失现象,因为这种情况下子链中缺失一个重复序列,与我们的情况一致。我们的结果和模型之间唯一的差异是发夹底部的弱双链体。据报道,DNA聚合酶的脱落和链延伸的阻断可能不是真正由于发夹导致的,而是由DNA模板的许多二级和高级结构导致[22-23]。由于在DNA复制的时候出现了这样的结构,才导致了跳跃的发生(图5C)。我们无法对这一预测做进一步测试,但我们推测高级结构诱导的子链合成阻断和聚合酶脱落都与重复序列相关,而且这种现象在体内和体外复制时都会发生。
DNA聚合酶产生跳跃的能力与其链置换活性有关,链置换活性越高,跳跃能力越低,反之亦然[21]。在细菌体内进行质粒复制时,DNA聚合酶III(复制早期有时是DNA聚合酶I)[24]进行新链延伸时,因其链置换活性不是很高[25],在遇到模板上合适的高级结构时更容易脱落下来,脱落后也可以在新链与附近的另一个同源序列(本研究中的第二个重复单位)重新退火后再次结合上去,并完成链的延伸而产生跳跃。本研究中单克隆DNA片段再次克隆时,所发生的跳跃很可能属于这一情形(图5C)事实上,常用于PCR,Sanger测序或分子克隆的DNA聚合酶的类型比如TaqDNA聚合酶,DNA聚合酶II,T4聚合酶等不具有高的链置换活性[21,26]。 因此,当模板序列具备促进形成易于跳跃的高结构,在这些酶参与复制的情况下也容易发生复制跳跃。
本研究所遇到的复制跳跃不仅发生在Temporin基因内含子双向扩增中,也发生在Sanger测序时的单链扩增中,甚至也发生在细菌体内质粒复制的过程中。基于我们做了确认实验,使用纯化的1126 bp片段作为模板用于再扩增,并且双酶消化阳性质粒,得出了这个结论,否则,跳跃可能被误解为独立的等位基因。所以,我们建议在利用片段大小进行二倍体或多倍体基因分型时,应该首先选取片段较大的等位基因进行克隆制备成单一模板,再用单一模板和已经开发出的具有强置换活性的热稳定DNA聚合酶[24,26]进行PCR扩增或再克隆后做质粒酶切,来确认是否发生复制跳跃,避免分型错误。

图4 Temporin基因内含子发生复制跳跃前后的序列比较Fig.4 Allignment of the true and jumped sequences of the intron of the Temporin gene of black-spotted frog(Pelophylax nigromaculatus) TempLong为原初序列;TempShort为复制跳跃导致TempLong丢失了564 bp;方框中是丢失片段之前和末尾的6 bp同向重复序列,圆圈中是4 bp的插入序列 TempLong is the true sequence;TempShort is the jumped sequence from TempLong.Two 6 bp direct repeat sequences are enclosed in square boxes and the 4 bp insert is enclosed in an oval

图5 产生扩增跳跃和克隆跳跃的机制示意图Fig.5 Schematic diagram of the mechanism of replication jumps on the intron fragment of the Temporin gene of black-spotted frog(Pelophylax nigromaculatus) A.第一个重复单位下游的序列与第二个重复单位配对后形成发夹环形结构,DNA聚合酶直接越过发夹延伸新链,形成缺失的新链;B.第二个重复单元的下游序列与第一个重复单元配对形成一个发夹环,而DNA聚合酶将新链直接穿过发夹,形成一个缺失的新链;C.模板上两个重复单位下相互靠近,形成特定的高级结构,使DNA聚合酶脱落下来,新链延伸被阻断。在进行PCR扩增时,下一个循环中新链的第一个重复单位与模板的第二个重复单位互补配对并退火,成为引物,DNA聚合酶结合后继续延伸,形成缺失的PCR产物;在质粒复制时,新链的重复单位移动到模板的第二个重复单位并退火,DNA聚合酶再次结合并完成延伸,形成缺失的新链 A.The sequence downstream of the first repeat unit is paired with the second repeat unit to form a hairpin loop,and DNA polymerase extends the new strand directly across the hairpin to form a missing new strand.B.The sequence downstream of the second repeat unit is paired with the first repeat unit to form a hairpin loop,and DNA polymerase extends the new strand directly across the hairpin to form a missing new strand.C.The two repeat units of the template approach each other to form a specific high-level structure.The DNA polymerase sheds and the new chain extension is blocked.The first repeat unit of the new strand in the next cycle anneals to the second repeat unit acting as a primer for strand extension when DNA polymerase is reloaded.As a result,the segment between the two repeats is missing from the PCR product.In the case of plasmid replication,the repeat unit of the new strand moves to the second repeat unit of the template and annealed allowing reloaded DNA polymerase complete the extension that leads to the segment missing from the new strand
致谢:首先,感谢国家自然科学基金项目(31370393)对本研究的大力支持。同时,感谢国家林业局野生动物检测中心为本研究提供实验设备,也感谢检测中心马跃老师的悉心帮助。
