北京港口水产品中非O1/O139群霍乱弧菌MLST分型研究
2018-08-01付溥博张西萌魏咏新魏海燕
付溥博, 张 琳, 曾 静, 张西萌, 魏咏新, 魏海燕, 耿 荣
(1.北京出入境检验检疫局, 北京 朝阳 100026 ; 2.河北出入境检验检疫局, 河北 石家庄 050051)
霍乱弧菌为革兰阴性菌,是人类霍乱病的病原体。根据霍乱弧菌O抗原的不同,可分为210多种血清型,过去O1和O139型被认为是引起霍乱流行的主要血清型[1],所以在检验霍乱弧菌时,常常检验此2种血清型,但随着研究的深入,人们发现非O1/O139群霍乱弧菌也会导致疾病[2]。非O1/O139群霍乱弧菌是指除O1群霍乱弧菌和O139群霍乱弧菌以外的霍乱弧菌[3],是一种全世界都有分布的水生性细菌,广泛存在于自然界的水体中,常常在水体或水产品中检出。近年来,越来越多的研究表明,散发或局部暴发的腹泻是由非O1/O139群霍乱弧菌引起的,此类菌群引起了细菌学家们越来越多的关注[4]。
多位点序列分型(Multilocus sequence typing,MLST)起源于多位点酶凝胶电泳(Multilocus enzyme electrophoresis,MLEE)[5],是一种针对微生物群体中缓慢基因位点变异进行分型的方法。此方法较简单快速,有着较高的分辨率,最主要是其可以通过互联网实现实验室间数据的共享与比较,并利于实验结果的保留,实验结果可与多年前的数据相比较[6]。此方法正逐渐被认作沙门、耶尔森等细菌分子分型的“金标准”[7],有部分学者认为此方法是进行流行病学研究最为可靠的工具[8-9],其结果比脉冲场凝胶电泳(PFGE)方法有着更好的分辨率[10]。本研究采用现有的MLST标准程序,对本实验室保存的67株分离自进出口水产品中的霍乱弧菌进行分析,并汇总MLST数据库中菌株信息,进行了基因分型和系统进化的分析。
1 材料与方法
1.1 材料 67株霍乱弧菌为本实验室保存的分离自2007-2011年北京港口进出口水产品中的菌株。所有菌株活化后,经VITEK 2 Compact鉴定符合霍乱弧菌的生化特征,经血清试验验证均为非O1非O139血清型。菌株来源见表1。

表1 菌株来源与ST型
1.2 仪器与试剂 PCR仪(美国罗克韦尔自动化公司);水平式核酸电泳仪和凝胶成像分析仪(美国伯乐生命医学产品有限公司)。
碱性蛋白胨水(Alkaline Peptone Water ,APW)(北京陆桥生物技术有限责任公司,161207);荧光PCR用引物,由上海生工生物工程技术服务有限公司合成(20161018);7对管家基因引物,由宝生物工程(大连)有限公司合成(20170108);TaqDNA聚合酶PCR试剂盒(普洛麦格生物技术有限公司,20170517)。
1.3 方法
1.3.1 基因组DNA的提取 将保存菌株在APW中培养过夜,取菌悬液1 mL于Eppendorf管中,10 000 r/min离心2 min,弃去上清,加50 μL无菌水,煮沸裂解10 min,冰浴5 min,10 000 r/min离心2 min,上清液即为DNA模板。立即用于PCR扩增或短期保存于-20 ℃。
1.3.2 致病基因的检测 应用实时荧光PCR检测菌株的CTX、toxR和hlyA基因,其20 μL体系为:10 μL 2 ×TaqMan Universal Master Mix,目标菌上游引物、下游引物(10 μmol/L)各1 μL:,探针(10 μmol/L)1 μL,模板50 ng ~100 ng,用灭过菌的双蒸水补足20 μL。引物探针具体序列见表2。PCR反应条件:50 ℃,2 min; 95 ℃,10 min;95 ℃,15 s; 60 ℃,60 s;40个循环。反应体系为20 μL。具

表2 致病基因检测用引物探针
1.3.3 MLST目标基因的选择和引物的设计 选择霍乱弧菌的adk、gyrB、metE、mdh、pntA、pyrC、purM7对管家基因作为MLST分析基因。PCR 扩增和测序引物引用http://pubmlst.org/vcholerae/info/primers.pdf 网站上发表的PCR引物序列。
1.3.4 PCR 扩增体系与条件 PCR反应50 μL体系包括:双蒸水38 μL,TaqDNA Polymerase(5 U/μL)1μL,PremixExTaqTM(10×, 含Mg2+) 5 μL,dNTP 1 μL,上游引物、下游引物(25 μmol/L)各1 μL,模板DNA 3 μL。PCR反应条件:96 ℃预变性10 min;96 ℃变性1 min,56 ℃退火1 min,70 ℃延伸1 min,30次循环后70 ℃延伸10 min。扩增产物在加入溴化乙锭(EB)的1%琼脂糖凝胶上电泳,后在成像仪的紫外光下观察扩增效果。
1.3.5 管家基因的测序及序列分析 将条带单一且位置正确的PCR产物委托上海生工生物工程技术服务有限公司进行双向测序。将测得序列与NCBI上查询到的7种管家基因的进行比较,应用软件DNAStar-MegAlign7.0将测得的序列裁剪到合适的大小。将裁剪好的序列按adk-gyrB-mdh-metE-pntA-purM-pyrC的顺序进行拼接,应用软件DNAStar-SeqMan 7.0 连接为3 215 bp的片段。将所得片段提交至MLST数据库比对,获取等位基因谱(Allele profile)和序列型(Sequence types,STs)。若发现新的等位基因及ST,则将新的信息上报至管理员,以获得等位基因号码和ST号码。应用eBURSTv3.0软件对我实验室分离菌株的STs和pubmlst上的数据,进行克隆群(Clonal Complex,CC)、组(Groups)和单体(singletons)分析;并应用goeBURST软件进行分析。
2 结果
2.1 致病基因CTX、toxR、hlyA检测结果 67株菌株针对CTX、toxR、hlyA3种毒力基因扩增结果均为阴性。
2.2 管家基因扩增结果 所有菌株均扩增出7个管家基因位点的目的片段结果,扩增产物的核酸浓度及纯度均符合测序要求。
2.3 霍乱弧菌MLST分型结果 将等位基因谱提交至MLST网站,结果显示,67株霍乱弧菌可分为62个STs。其中原有STs 2个,新发现STs 60个。其中相同的ST型有ST377 2株,均来源于中国;ST380 3株,均来源于中国;ST383 2株,均来源于泰国;ST388 2株,均来源于中国。原有ST型为ST271、ST338;新发现STs经MLST网站管理员确认编号为ST369~ST428。
2.4 本试验结果与pubmlst数据库相比较的eBURST分析结果 由于本试验用菌株均为非产毒株,基因变异性较大,分布分散(图1)。用Seqman软件分别对这些菌株的7对管家基因进行分析,其基因相似度分别为90.4%~100%(adk)、91.8%~100%(gyrB)、88.6%~100%(mdh)、94.0%~100%(metE)、96.6%~100%(pntA)、41.9%~100%(purM)、86.5%~100%(pyrC),用Mega 7.0 分别对这7对管家基因进行系统发生分析,用Neighbor-Joining(NJ)法绘制进化树,其中adk、gyrB、mdh、metE、pntA、pyrC基因相似度较接近,结果以adk可以为例见图2,purM基因差异最大,结果见图3。以上结果说明这些菌株的管家基因,尤其是purM基因发生了较大的遗传变异。继续将pubmlst数据库中全部霍乱弧菌的STs进行分析,截止到2017年2月,数据库中霍乱弧菌共有STs 505个,做eBURST分析,本实验数据与数据库中数据可归为7个组别(图4)。其中明显成克隆群的CC71-75、CC176-173、CC430,根据Pubmlst数据库中信息可知,均为产毒株。根据上面数据分组情况,将Pubmlst数据库中(包括本实验数据)与本实验相关的数据做goeBURST分析,共将71个STs归为7个组别(图5),其中方形框为本实验室结果具体结果为ST-88~ST-8~ST-384,其中菌株来源于中国和澳洲,本试验结果为ST-384,来源于中国;ST-423~ST-204~ST-294,其中菌株来源于泰国和法国,本试验结果为ST-423来源于泰国;ST-403~ST-267,其中菌株均来源于中国,本试验结果为ST-403,来源于中国;ST-411~ST-15,其中菌株来源于中国和澳洲,本试验结果为ST-411,来源于中国;ST-416~ST-205,其中菌株来源于中国和泰国,本试验结果为ST-416,来源于中国;ST-338~ST-348,其中菌株来源于中国和非洲,本试验结果为ST-338,来源于中国;ST-385~ST-326,其中菌株来源于中国和法国,本试验结果为ST-385,来源于中国。

图1本试验STs的eBURST分析结果
注:每个圆点代表一个ST型,上标数字即为其具体的ST型

图2adk基因的NJ进化树
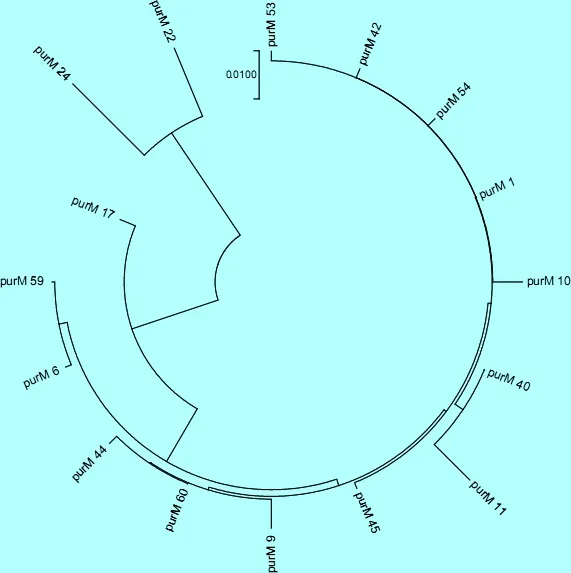
图3purM基因的NJ进化树

图4Pubmlst数据库中STs的eBURST分析结果

图5Pubmlst数据库中的相关STsgoeBURST分析结果
3 讨论
早期研究认为,霍乱弧菌的致病基因主要为CTX基因,但也有毒力基因阴性却引发腹泻暴发的病例[11]。近年来,越来越多的研究表明,散发或局部暴发的腹泻是由非O1/O139群霍乱弧菌引起的,此类菌群引起了科研人员越来越多的关注[4],研究人员发现,非O1/O139群霍乱弧菌相比O1、O139群霍乱弧菌的菌体多糖分泌的变异,使得前者能形成生物膜,从而能更好的抵御不良环境,更适于在其在水产品中生存与繁殖[12],而在外界某些条件的作用下非O1/O139群霍乱弧菌会通过抗原漂移来获得毒力基因[13]。故此只有对霍乱弧菌的分子分型进行深入的研究,才能了解该细菌的分型特征及进化、以及溯源情况,并避免产品间或实验室内的相互污染。
本试验共从非O1/O139群霍乱弧菌中新发现了60个STs,通过对结果应用eBURST、Paup4.0和goeBURST软件进行分析可以看出,本试验的菌株没有成组,均为单体,将本实验的ST结果与Pubmlst数据库中相关的ST一起进行MLST分析,本次试验的结果也并没有与数据库中数据成群的ST型,形成7组,这与本实验室样品来源较广,菌株分离自世界各地的水产品有关。而且,本试验菌株均为非产毒株,也说明环境非产毒株基因的变化要大于产毒株,此结论与此前有学者报道的“非产毒性霍乱弧菌基因组间差异较大”相符[1]。
应用MLST方法对霍乱弧菌进行研究是较新兴的项目,自2013年4月29日开始建立霍乱弧菌的数据库,迄今此数据库中数据尚不够完善,对分组造成了较大影响。在Pubmlst霍乱弧菌数据库建立之前,有学者对霍乱弧菌进行MLST分析时,选取了与现数据库不同的管家基因,据报道共涉及了管家基因40个[14],从而导致了无法与很多早些时候的学者们研究成果相比较,也说明了充分丰富这种全球数据库的重要性。
从本次试验结果来看,具有相同ST型的菌株均来源于相同国家,这说明MLST方法擅长于显示不同地理区域的菌株的进化关系,此方法可以建立一个可靠的食品污染溯源体系,也说明进化有一定的地域性;而成组的ST型则来源于中国、泰国、澳洲、非洲和法国,说明不同地理来源的菌株也可能有着类似的克隆株,说明随着国际贸易的日益发展,不同大洲之间的细菌正发生着基因的水平转移。
但在对Pubmlst数据库中的霍乱弧菌进行研究时发现,霍乱弧菌没有像其他致病菌那样,产不同毒素的菌株或不同血清型会明显成不同群。有学者就曾报道,由于霍乱弧菌产毒菌株的高度克隆化,使得MLST方法对其致病血清的分析效果并不理想[15],这也是今后研究中值得注意并要解决的问题。
总之,本试验利用MLST这种操作简单、易于保存数据,便于比较不同实验室间的结果,擅长于显示不同地理区域的菌株的进化关系分子方法,对保存的67株霍乱弧菌进行了分子特征的分析,发现了新的ST型,并向Pubmlst数据库上传了数据,扩充了数据库。通过分析,发现本次试验数据的克隆组,主要来源于不同国家,有遗传多样性;进境水产品中分离菌株多为单体,但相同ST型来源地一致。
