盐藻microRNAs高通量测序和生物信息学分析
2018-07-14娄素琳朱秀兰曾志勇胡章立
娄素琳,朱秀兰,曾志勇,李 辉,4,胡章立,4
1) 深圳大学生命与海洋科学学院,深圳市海洋生物资源与生态环境重点实验室,广东深圳 518060;2) 深圳大学光电工程学院,光电子器件与系统教育部/广东省重点实验室,广东深圳 518060;3) 广东省植物表观遗传学重点实验室,广东深圳 518060;4) 深圳大学龙华生物产业创新研究院,深圳市海洋藻类生物工程技术研究中心,广东深圳 518060
杜氏盐藻(Dunaliellasalina)也称盐藻,是一种生长于海水或高盐环境的单细胞光合绿藻,长约14~22 μm,宽约3~14 μm,无细胞壁,有两条鞭毛可游动.盐藻属于极端生物,可适应高盐、高光、低温和低pH值等极端环境,是研究植物耐高盐分子机制的重要模式生物.盐藻细胞营养丰富,含有大量类胡萝卜素和甘油,β-胡萝卜素含量最高,质量分数可达干质量的14%,商业价值极高[1].盐藻基因组草图拼接数据(Dunaliellasalinagenome sequencing project)已于2017年公布(http://phyto zome.jgi.doe.gov/)[2],为本研究探索盐藻非编码小核糖核酸(micro ribonucleic acid, microRNA 或miRNA)提供了参考.miRNA是一类内源性长约21~22个核苷酸碱基(nucleotide, nt)的非编码小RNA,通过碱基互补配对的方式介导靶mRNA的降解或阻遏靶mRNA的翻译,从而对基因表达进行转录后调控.miRNA于1993年首次在线虫体内被发现[3],2007年首次在低等单细胞莱茵衣藻中被发现[4-5].这让人们意识到miRNA很可能是一种古老的调控机制,且比多细胞的出现更早.2011年,胡章立等[6]分离鉴定到一些硫胁迫相关miRNAs.近年来,在低等生物团藻、三角褐指藻和微拟球藻中相继发现有miRNA存在[7].本研究利用Illumina HiSeqTM2500高通量测序方法对盐藻进行miRNA测序,探究单细胞微藻是否普遍存在miRNA.
1 材料与方法
1.1 藻株和培养条件
盐藻藻株购自英国(https://www.ccap.ac.uk/).盐藻培养基为RAMARAJ,培养条件为25 ℃,光照强度为50 μmol·m-2·s-1.盐藻置于恒温光照培养箱培养至对数生长期,收集藻细胞用于总 RNA提取.
1.2 小RNA的分离和cDNA文库的构建
用TRIzol提取盐藻总 RNA,琼脂糖凝胶电泳切胶选择18~30 nt的片段作为小RNA测序.分别连接3′和5′接头,进行反转录和聚合酶链式反应(polymerase chain reaction,PCR)扩增.回收并纯化约140 bp的条带,完成文库构建.使用2100 TOF LC-MS质谱仪(Agilent,美国)及定量PCR(real-time PCR, RT-PCR)进行质控,并上机测序.
1.3 盐藻miRNA的鉴定和靶基因预测分析
下机数据经初步过滤,得到用于后续分析的小RNA clean tags序列.比对GenBank、Rfam数据库及盐藻参考基因组数据,筛选后的clean tags同miRBase中已知的植物或动物miRNA进行比对,鉴定得到已知miRNA.结合盐藻基因组序列,进行发卡结构预测,鉴定得到新的miRNA.
使用RNAhybrid(v2.1.2)+svm_light(v 6.01)、Miranda(v3.3a)和TargetScan(version: 7.0)3种方法进行靶基因预测,然后取3种方法得到的靶基因预测的结果的交集作为miRNA靶基因预测的结果.
2 结果与分析
2.1 盐藻miRNA的测序信息分析
对盐藻小RNA原始下机数据,通过过滤中低质量、不带3′接头、含有5′接头、不含插入片段和插入片段长度小于18 nt,以及含有polyA的reads,得到盐藻小RNA clean tags序列11 542 169条,数据质量良好,用于后续分析. 比对GnenBank数据库、Rfam数据库及杜氏盐藻的参考基因组(包括内含子、外显子和重复序列),3个生物学重复的盐藻clean tags注释统计分析如表1(取3者平均值),没有比对上任何注释信息的tags为unann.
比对miRBase数据库和筛选后的clean tags数据,3个生物学重复获得盐藻已知miRNA 分别为42、41和44个.比对盐藻参考基因组及其miRNA前体发卡结构预测,3个重复分别获得盐藻新的miRNA 920、880和957个.
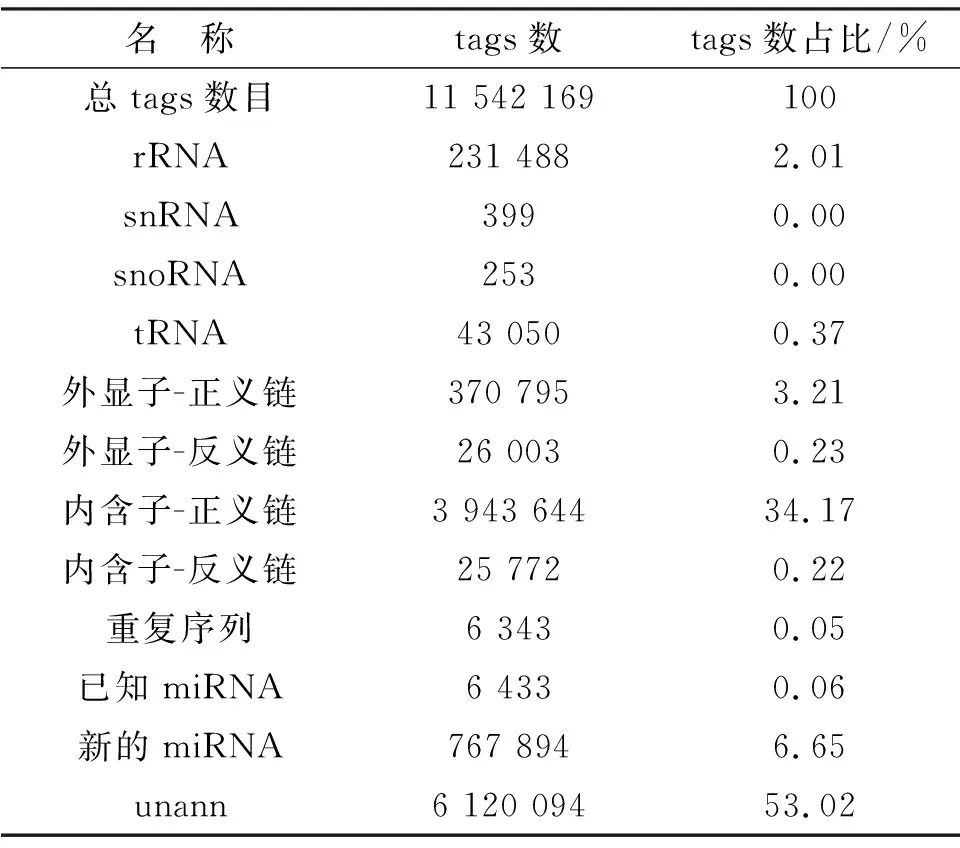
表1 盐藻小RNA测序的tags丰度统计
2.2 盐藻miRNA靶基因富集分析
对盐藻所有miRNA进行靶基因预测分析,并对靶基因进行GO功能分析和KEGG Pathway分析.对靶基因进行功能注释和归类,功能富集分析发现,靶基因主要集中在细胞代谢进程(约450个靶基因)、分子功能的结合(约500个靶基因)和催化活性(约500个靶基因)等生物途径,说明盐藻miRNA活跃参与调节这几个途径的基因表达.
3 讨论
本研究在杜氏盐藻中发现miRNA的存在,不仅丰富了人们对微藻miRNA的认识,也为揭示miRNA这一古老调控机制广泛存在于低等单细胞微藻提供了依据,为后续研究miRNA是如何调控盐藻积累β-胡萝卜素的分子机制研究奠定了基础.关于盐藻miRNA靶基因的预测,本研究结合了动植物两套模式来预测,因为最新的研究表明微藻和动物类似,miRNA的种子序列与靶基因配对就足以导致靶基因表达抑制[8].后续的研究将对靶基因进行RT-PCR和RACE(rapid-amplification of cDNA ends)实验验证.
