丙型肝炎患者血清病毒宏基因组学研究*
2018-06-29周成林
李 旺,倪 萍,周成林
(江苏省泰州市人民医院检验科 225300)
病毒宏基因组学方法在病毒学研究方面具有独特优势,它既能够检测标本中的已知病毒,又能够检测标本中的未知病毒。目前,病毒宏基因组学方法已得到广泛应用。KRABERGER等[1]应用病毒宏基因组学方法从污水处理池发现了许多新的单链环形DNA病毒。杨凡力等[2]同样应用病毒宏基因组学方法在蝙蝠体内发现腺病毒、圆环病毒、细小病毒、博卡病毒等新病毒。ZHANG等[3]应用病毒宏基因组学方法在牛肉中检测与感染灵长类多瘤病毒亲缘关系最近的牛多瘤病毒(BPyV2-SF)。用病毒宏基因组学方法对腹泻儿童及健康儿童的粪便标本进行检测,发现腹泻标本中主要存在指环病毒、杯状病毒和Salivirus,而正常儿童粪便标本中最多的病毒依次为指环病毒、Tymovirus和人双埃可病毒。此外,两组标本中病毒序列数量相差甚远,并且首次在中国儿童腹泻粪便标本中发现了第4基因群型(诺如病毒)。应用病毒宏基因组学方法对儿童呼吸道标本和脑脊液样本进行研究显示,不同标本及不同健康状态下病毒群体的组成存在一定差异。丙型肝炎是由丙型肝炎病毒(HCV)引起的一种传染性疾病,全球有1.3~2.1亿人感染HCV并转变为慢性感染者,对人类的健康造成严重威胁[4]。因此,进行HCV致病机制及相关性研究对于预防和治愈丙型肝炎有重要意义。本研究应用病毒宏基因组学方法对HCV-RNA阳性血清标本进行研究,以了解HCV-RNA阳性血清样本中病毒群落的组成,并与健康人群血清病毒群落的组成进行比较,发现二者之间病毒组成的差异性,为探索和研究与丙型肝炎相关的其他病毒提供前期研究基础,现报道如下。
1 资料与方法
1.1标本采集 选取2015年4月至2016年7月泰州市人民医院收集的30份HCV-RNA阳性患者血清标本,男、女性各15份;另选取10份健康人群血清标本,男、女性各5份。
1.2标本分组设计 考虑到构建病毒文库成本较高,本研究根据性别及病毒载量将每10份标本作为一组,各组内每份标本取150 μL混合制成混合液,构成4个标本文库。详细信息见表1。

表1 标本组合信息
1.3核酸酶消化 在4支1.5 mL的无RNA酶离心管内分别加入10×Buffer Cocktail 20 μL、Cocktail of DNases 7 μL、 Micrococcal Nuclease 5 μL、RNase A 2 μL及混合血清166 μL,混匀后37 ℃ 反应60 min,反应至30 min时轻轻混匀几次使反应充分。
1.4病毒核酸提取 采用病毒核酸提取对核酸酶消化处理后的标本进行病毒核酸提取,最后病毒核酸溶于50 μL buffer AVE中再加入0.5 μL RNA酶抑制剂,混匀冰上放置待用。
1.5反转录及合成双链DNA 应用SuperScript Ⅲ反转录试剂盒进行反转录反应,取4支无RNA酶的聚合酶链反应(PCR)管分别加入dNTP(10 nmol/L)1 μL、含接头的6碱基随机引物(100 μmol/L) 1 μL、RNA 1 μL,涡旋混匀后瞬时离心后置于PCR仪上,65 ℃ 反应5 min,迅速置于冰上>2 min。随后向各管中分别加入Buffer Cocktail 4 μL、ScriptⅢ®RT Enzyme Mix Ⅰ 1 μL、DTT 1 μL,涡旋混匀后瞬时离心后,于25 ℃ 10 min、50 ℃ 60 min、85 ℃ 5 min、95 ℃ 2 min后迅速置于冰上放置≥2 min。再向各反应管中加入Klenow酶1 μL,涡旋混匀后瞬时离心,置于PCR仪上进行37 ℃ 60 min,75 ℃ 20 min反应。最后将得到的双链DNA产物保存待用。
1.6构建病毒文库 取4支无RNA酶的PCR管分别加入10 μL TD buffer、5 μL ATM及5 μL双链DNA,混匀后2 100 r/min离心1 min后置于PCR仪上进行55 ℃、5 min反应,反应结束后当温度降至10 ℃时迅速向每管中加入5 μL NT buffer,混匀,2 100 r/min离心1 min。随后分别转移至4个预先分别加有Illumina index primer(5 μL N primer和5 μL S primer)和15 μL NPM buffer的混合液中混匀,2 100 r/min离心1 min,置于PCR仪上进行72 ℃ 3 min、95 ℃ 30 s、94 ℃ 10 s、55 ℃ 30 s、72 ℃ 30 s,共15个循环;72 ℃ 5 min反应,即得到病毒文库。
1.7筛选DNA片段 使用Qiaquick公司生产的PCR核酸纯化试验盒纯化病毒文库以去除引物二聚体。随后利用Ampure磁珠(Beads)对病毒文库DNA长度进行筛选:取4支1.5 mL离心管分别向各管加入30 μL文库DNA和27 Beads,加样枪上下轻轻混匀,室温静置5 min,随后再置于磁力架上5 min后吸弃上清液。再向管内加入80%乙醇溶液500 mL的洗涤Beads,洗涤2次后吸弃乙醇溶液,打开管口室温静置5 min,使乙醇完全挥发。随后从磁力架上取下离心管加入30 μL无菌水,加样枪上下轻轻混匀,静置2 min。再将离心管置于磁力架上静置3 min,吸取20 μL上清液于0.2 mL的PCR管中用于Miseq测序。
1.8Miseq深度测序及生物信息学分析应用 Illumina测序平台对文库进行Miseq深度测序,随后将测序原始数据传至美国加州大学旧金山分校,通过其病毒宏基因组学分析平台对原始数据进行生物信息学分析。应用Bowtie 2软件滤除数据中真核及原核基因组序列。再应用Phred、VecScreen CAP3、meta-Velvet、ABySS和SOAPdenovo 2等一系列软件实施序列尾部剪切、引物序列和接头去除、序列拼接等操作。随后将序列放入本地病毒蛋白组数据库中进行BLASTx搜索。再将所有可能为病毒基因的核酸序列放在平台内的NVNR蛋白组数据库中进行BLASTx搜索,最后筛选出病毒核酸序列。
2 结 果
2.1各文库中病毒群落组成 对4个病毒文库中病毒群落序列进行整理,文库HCV01、HCV02、HCV03、HCV04中病毒群落构成见图1~4。数据显示,HCV01文库中总病毒序列数为1 514条,主要由黄病毒科(Flaviviridae)48%、指环病毒科(Anelloviridae)29%、未分类科病毒(None)8%、反转录病毒科(Retroviridae)7%、微小噬菌体科(Microviridae)2%构成;HCV02文库中总病毒序列数为2 820条,主要由Anelloviridae 61%、Flaviviridae 18%、Retroviridae 8%、None 3%、Microviridae 1%构成;HCV03文库中总病毒序列数为3 539条,主要由Anelloviridae 78%、None 5%、Flaviviridae 4%、Retroviridae 4%、人类内源性反转录病毒科(HERV)2%、Microviridae 1%构成;HCV04文库中总病毒序列数为3 549条,主要由Anelloviridae 90%、None 4%、Retroviridae 2%、藻类脱氧核糖核酸病毒科(Phycodnaviridae)1%、其他病毒科1%构成。

图1 HCV01病毒群落构成
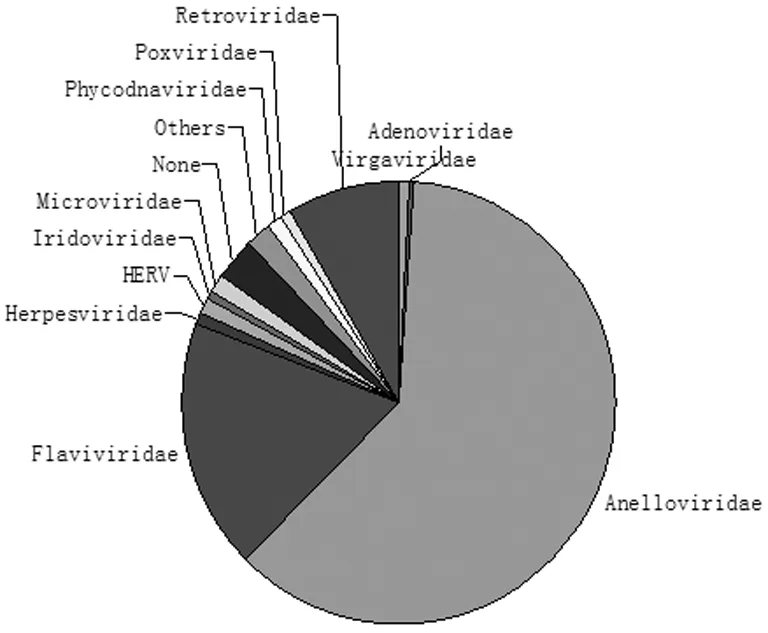
图2 HCV02病毒群落构成
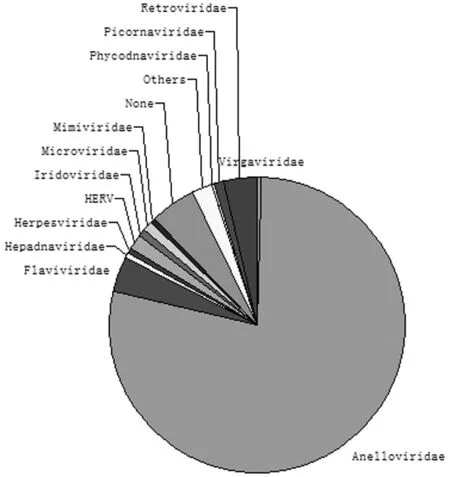
图3 HCV03病毒群落构成

图4 HCV04病毒群落构成
2.2HCV序列检测 HCV01和HCV03文库中分别检测到的719条、138条Flaviviridae序列均为HCV序列;HCV02文库中检测到的515条Flaviviridae序列中有504条为HCV序列,11条为瘟疫病毒(Pegivirus);HCV04文库中检测到的3条Flaviviridae序列均为Pegivirus。
2.3各文库中病毒群落构成比较 见表2。
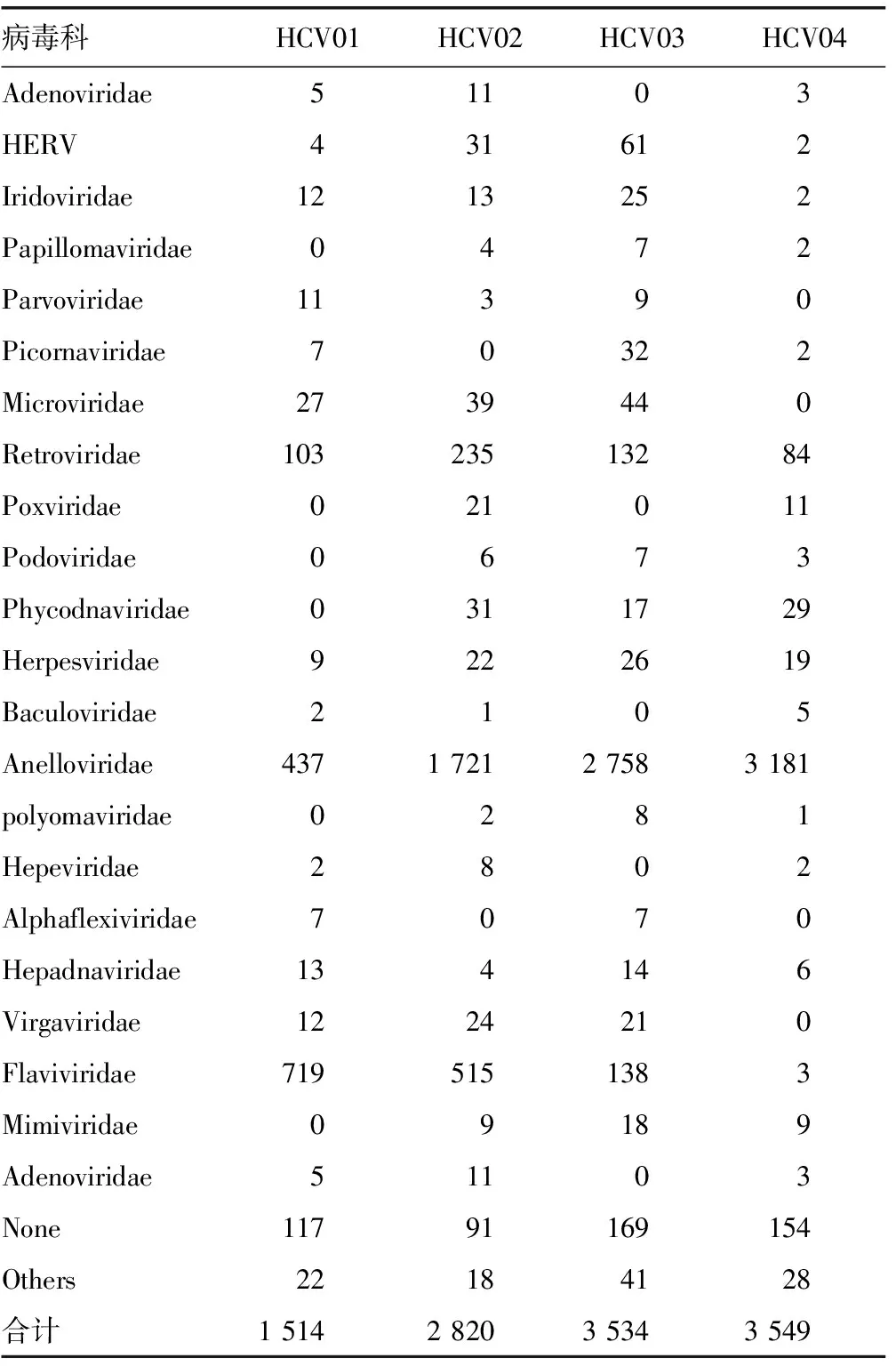
表2 各文库中病毒群落构成比较(n)
3 讨 论
传统研究病毒的方法具有一定的局限性,如PCR只能扩增已知病毒或低变异病毒,而高变异病毒及未知序列病毒此法则难以实施;血清学方法则难以获得新病毒的特异抗体;细胞培养法,很多病毒缺乏特异的细胞系及大多数病毒不能进行细胞培养。病毒宏基因组学专注于病毒群体,能够检测标本中已知和未知病毒再发现新病毒,在检测病毒流行情况方面展现出强大的优势。
本研究应用病毒宏基因组学方法对江苏省泰州地区健康者及HCV-RNA阳性患者血清病毒群落组成进行研究。结果显示,4个病毒文库中Flaviviridae序列水平由多至少分别是HCV01、HCV02、HCV03、HCV04,该结果与HCV-RNA检测结果相符。文库HCV04为健康对照组,该文库中仅检测到3条Flaviviridae序列为Pegivirus;4个文库中总病毒序列数及Anelloviridae病毒序列数由多至少分别是HCV04、HCV03、HCV02、HCV01,这可能是由于血清中高HCV水平抑制了Anelloviridae病毒的复制,从而导致文库中Anelloviridae病毒序列水平与HCV水平呈相反现象。4个文库中Anelloviridae病毒序列均占有相当大的比例,除文库HCV01外,其他3个文库中Anelloviridae病毒序列数最多,说明正常情况下Anelloviridae病毒是构成血液中病毒群落的主体。
ABUODEH等[5]研究显示,人类感染Anelloviridae病毒可高达90%,说明人类普遍感染Anelloviridae病毒,可能该病毒科病毒是人体内的正常病毒群落。近年来,一定数量的新型Anelloviridae病毒被发现,且这些新型病毒可能与人类的某些疾病有一定关系。如GALMS等[6]研究显示,Anelloviridae病毒的一种新型TTMV病毒可能与严重的儿童肺炎有关;ZHANG等[7]通过对患有慢性牙龈炎孕妇的研究发现了一种新的Anelloviridae,并发现该病毒可能与牙龈炎的发生有密切关系。ALAVI等[8]研究显示,Anelloviridae与丙型肝炎有一定关系,但其对肝脏的影响甚微。本研究中Anelloviridae序列构成了病毒文库的主体,该病毒科中是否存在一些新型的并与疾病相关的病毒还有待进一步研究。同时,丙型肝炎患者血清中是否存在与疾病相关的其他未知的病毒还需要今后的深入研究。
目前病毒宏基因组学已广泛应用于很多领域,但还存在很多技术难题和缺陷。(1)标本过滤处理时导致病毒颗粒的丢失[9]。(2)核酸酶不能完全消化游离核酸,从而增加了测序成本及对测序数据分析的难度[10]。(3)目前病毒宏基因组学方法成本高、历时长,作为紧急检测方法应用于临床检测还有很大难度。(4)一次测序获得的数据有限,如标本量较多,就会导致部分数据被掩盖;反之,如标本量较少,则增加了测序成本。相信随着科技的不断进步,以上技术难题将会逐步得以解决,病毒宏基因组学技术将来一定能够成为临床上的常规检测项目,更好地为人类的健康生活服务。
[1]KRABERGER S,ARGÜELLO-ASTORGA G R,GREENFIELD L G,et al.Characterisation of a diverse range of circular replication-associated protein encoding DNA viruses recovered from a sewage treatment oxidation pond[J].Infect Genet Evol,2015,31(1):73-86.
[2]杨凡力,王意银,郑文成,等.中国部分地区蝙蝠携带病毒的宏基因组学分析[J].生物工程学报,2013,29(5):586-600.
[3]ZHANG W,LI L,DENG X,et al.What is for dinner? Viral metagenomics of US store bought beef,pork,and chicken[J].Virology,2014,9(16):303-310.
[4]GAYET-AGERON A,COMBESCURE C,LAUTENSCHL-AGER S,et al.Comparison of diagnostic accuracy of PCR targeting the 47-Kilodalton protein membrane gene of treponema pallidum and PCR targeting the DNA polymerase Ⅰ gene:systematic review and meta-analysis[J].J Clin Microbiol,2015,53(11):3522-3529.
[5]ABUODEH R,AL-MAWLAWI N,AL-QAHTANI A A,et al.Detection and genotyping of torque teno virus (TTV)in healthy blood donors and patients infected with HBV or HCV in Qatar[J].J Med Virol,2015,87(7):1184-1191.
[7]ZHANG Y,LI F,SHAN T L,et al.A novel species of torque teno mini virus(TTMV) in gingival tissue from chronic periodontitiis patients[J].Sci Rep,2016,25(6):26739.
[8]ALAVI S,SHARIFI Z,VALESHABAD A K,et al.Clinical outcomes of Torque teno virus-infected thalassemic patients with and without hepatitis Cvirus infection[J].Korean J Hematol,2011,46(2):123-127.
[9]THURBER R V,HAYNES M,BREITBART M,et al.Laboratory procedures to generate viral metagenomes[J].Nat Protoc,2009,4(4):470-483.
[10]DJIKENG A,HALPIN R,KUZMICKAS R,et al.Viral genome sequencing by random priming methods[J].BMC Genomics,2008,9(1):5-9.
