芝麻发芽期耐盐性鉴定方法研究及耐盐候选基因的挖掘
2018-06-29张玉娟游均刘爱丽黎冬华于景印王燕燕周瑢宫慧慧张秀荣
张玉娟,游均,刘爱丽,黎冬华,于景印,王燕燕,周瑢,宫慧慧,张秀荣
芝麻发芽期耐盐性鉴定方法研究及耐盐候选基因的挖掘
张玉娟1,2,游均1,刘爱丽1,黎冬华1,于景印1,王燕燕1,周瑢1,宫慧慧2,张秀荣1
(1中国农业科学院油料作物研究所/农业部油料作物生物学与遗传育种重点实验室,武汉 430062;2山东棉花研究中心,济南 250100)
【目的】确定芝麻发芽期耐盐性鉴定适宜的NaCl胁迫浓度和评价指标,发掘耐盐种质和耐盐相关重要基因,为芝麻耐盐性大规模鉴定、遗传改良和耐盐机理研究提供方法借鉴和优异基因资源。【方法】以8份耐盐性差异较大的芝麻种质为材料,在不同浓度的NaCl(0、50、100、150、200和250 mmol·L-1)胁迫下发芽,测定其发芽势、成苗率、根长、芽长和鲜重等指标,通过对指标值的方差分析、主成分分析、隶属函数和相关性分析等,筛选芝麻发芽期耐盐性鉴定适宜的NaCl处理浓度和评价指标。以100 mmol·L-1NaCl溶液为处理浓度,相对成苗率为鉴定指标对71份芝麻核心种质进行发芽期耐盐性鉴定和全基因组关联分析,并对获得的候选基因进行功能注释;通过NaCl胁迫下芝麻幼苗叶片转录组测序和荧光定量PCR分析候选基因的表达模式,筛选耐盐相关候选基因。【结果】对不同浓度NaCl胁迫下8份芝麻材料发芽各指标进行统计和方差分析表明,100 mmol·L-1的NaCl处理下,除发芽势外,发芽指数、活力指数、成苗率、根长、芽长和苗鲜重的标准偏差值均较大,所有指标在0.05水平上均存在显著差异,100 mmol·L-1的 NaCl溶液可以作为芝麻发芽期耐盐性鉴定的适宜胁迫浓度;相对成苗率、相对发芽势、相对发芽指数、相对活力指数、相对芽长、相对根长和相对鲜重7个指标与芝麻发芽期耐盐性关系密切,贡献率较高,可以作为芝麻发芽期耐盐性鉴定的评价指标。71份芝麻核心种质材料的相对成苗率变异比较丰富,符合正态分布;通过对71份芝麻核心种质相对成苗率与SNP标记进行全基因组关联分析,检测到7个与芝麻发芽期耐盐性显著关联的SNP标记(LG5:688003、LG7:9582027、LG10:5274091、LG10:10788493、LG11:11924186、LG14:2128695和LG16:3930301),SNP标记上、下游各100 kb区间内共有基因67个,其中有功能注释的基因34个;通过耐盐材料S04在盐胁迫下的转录组测序和荧光定量PCR对预测的候选基因进行表达模式分析,共有21个候选基因受到盐胁迫诱导显著差异表达。【结论】芝麻发芽期耐盐性鉴定的适宜NaCl胁迫浓度为100 mmol·L-1,相对成苗率等7个指标可以作为适宜的评价指标;检测到与芝麻发芽期耐盐性显著关联的SNP标记7个,并鉴定出耐盐候选基因21个。
芝麻;耐盐性;全基因组关联分析;耐盐基因
0 引言
【研究意义】盐碱化和次生盐碱化土壤广泛分布于世界各地,特别是沿海、内陆干旱和半干旱地区较为严重,土壤盐碱化造成了作物生境条件的变化,影响其生长过程和干物质积累,重度盐碱土壤甚至无法种植利用,严重制约农业生产的可持续发展[1]。当前,中国盐碱地总面积约1×108hm2,主要分布在华北、西北和东北等内陆干旱地区及沿海地区,且面积逐年增加[2]。芝麻是中国五大油料作物之一,年消费量(约1.5×106t)和进口量(2016年达到9.3×105t)均居世界之首,但国内芝麻生产总供给不足50%。因此,加强西部和内陆干旱、半干旱地区次生盐碱地和滨海土壤的开发利用,对扩大芝麻种植面积、提高自给率具有重要意义。【前人研究进展】国内外诸多学者对其他植物耐盐性研究比较深入,建立了针对不同作物的耐盐性鉴定方法,开展了大量的耐盐作物种质资源评价工作,对多种作物如小麦、水稻、玉米、棉花等种质资源进行了耐盐性鉴定筛选工作[3-6]。植物对盐胁迫的响应、抵御机理是一个非常复杂的过程,包括盐信号的转导、特异转录因子的激活和下游应答基因的表达等[7]。迄今研究较清楚的盐胁迫应答机制是SOS(salt overly sensitive)信号转导途径,包含6个关键基因,在盐胁迫下通过不同的作用方式调控植物对盐胁迫的抵御[8]。通过QTL定位和全基因组关联分析(genome-wide association study,GWAS)挖掘耐盐基因应用较多。Xu等[9]以小麦耐盐重组自交系群体为材料,定位到11个与耐盐性显著关联的QTL位点;Yu等[10]对198份苜蓿地方品种进行了GWAS分析,发现了23个与耐盐性显著关联的SNP位点,获得2个耐盐基因;Thudi等[11]对盐胁迫下拟南芥群体进行耐盐相关性状的连锁分析和GWAS分析,获得4个耐盐关键基因;、、、和等耐盐基因先后被验证了其提高耐盐性的功能[12-16]。【本研究切入点】目前,芝麻耐盐性研究严重滞后于小麦、玉米、棉花等其他作物[17-19],缺乏完善的耐盐性鉴定方法,分子基础研究极其薄弱,基本处于空白,可用的耐盐基因资源未见报道,制约着芝麻耐盐育种的发展。【拟解决的关键问题】种子发芽期不仅是植物生长发育过程中最关键的一个生长阶段,有研究表明在盐胁迫逆境环境下,种子发芽期的耐盐性最敏感,随着植物的生长,其耐盐性逐渐提高[20-21]。本研究以芝麻核心种质为研究材料,筛选芝麻发芽期耐盐性鉴定适宜的NaCl处理浓度和评价指标,发掘耐盐优异种质和相关重要基因,为芝麻耐盐性大规模鉴定发掘和耐盐育种提供方法和基因资源支持。
1 材料与方法
1.1 材料
试验于2017年在中国农业科学院油料作物研究所芝麻种质资源培养间进行。供试芝麻核心种质材料共71份,包括53份国内种质(来自吉林、新疆和福建等18个省份)和18份国外种质(来自美国、日本和印度等10个国家),由中国农业科学院油料作物研究所芝麻种质中期库提供。
1.2 种子发芽期的适宜胁迫浓度和评价指标
1.2.1 NaCl胁迫适宜浓度的确定 挑选耐盐性差异较大的种质材料8份,设置0(对照,去离子水)、50、100、150、200和250 mmol·L-1的NaCl溶液,共6个处理,每处理重复3次,每重复用种50粒;将2%次氯酸钠消毒后的种子均匀放在铺有2层滤纸、直径为10 cm、高7 cm的塑料培养皿中,加入NaCl溶液15 mL,置于28℃、湿度60%的培养箱内黑暗发芽。第2天开始记录每皿的发芽种子数(发芽的标准为胚根长度≥种子长度),第6天调查统计幼苗根长、芽长、鲜重和正常苗数;计算各材料发芽势、成苗率、发芽指数、活力指数、根长、芽长和鲜重的均值。
正常苗判定标准:各器官生长良好、匀称、健康的幼苗;带有轻微缺陷的幼苗(某一器官生长稍迟缓,但其他与健康幼苗相近);轻微感染的幼苗(由真菌或细菌感染导致幼苗发病腐烂,但被感染前各器官生长基本正常[22]。
各指标按以下公式计算:
发芽势(%)=(第3天发芽的种子数/供试种子数)×100;
成苗率(%)=(第6天正常苗数/供试种子数)×100;
发芽指数=∑(Gt/Dt),Gt表示时间t的发芽数,Dt表示对应的发芽日数;
活力指数=发芽指数×鲜重;
指标相对值=处理平均测定值/对照平均测定值。
1.2.2 测定数据统计分析和适宜评价指标的确定 利用Excel2010对数据进行整理统计计算,利用SAS9.1软件对测定指标数据进行方差分析、主成分分析和相关性分析。耐盐性综合评价采用隶属函数法[23]。
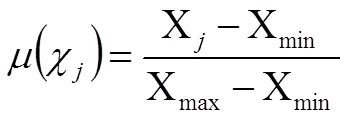
公式(1)中计算每一个材料各综合指标的隶属函数值,X表示第个综合指标的测定值;Xmin、Xmax分表表示第个综合指标的最小值和最大值。公式(2)计算综合指标的权重,代表各材料第个综合指标的贡献率。公式(3)计算各材料的耐盐性综合评价值。
1.3 全基因组关联分析及耐盐候选基因的预测
调查71份核心种质群体在适宜浓度NaCl处理后的相对成苗率表型数据,基于本课题组前期芝麻核心种质全基因组重测序获得的SNP[24],利用TASSEL 5.0软件[25],采用混合线性模型进行关联分析,并利用qqMan绘制曼哈顿图;显著关联SNP宽松阈值设为-lg()=3,严谨阈值设为-lg()= 4.5;在显著关联的SNP位点上下游各100 kb的序列作为候选区间,对区间内候选基因进行功能注释。
1.4 耐盐候选基因表达分析
取一份耐盐材料(S04),用1/2 Hoagland营养液培养至第一对真叶展开时,用150 mmol·L-1NaCl营养液处理幼苗根部,分别于处理0(CK)、6、12和24 h各取10株幼苗叶片混合后用于转录组测序分析和荧光定量PCR(qRT-PCR)分析。转录组测序分析由北京诺禾致源生物信息科技有限公司完成,显著差异基因筛选的阈值为|log2FoldChange(处理/对照)|≥1。采用天根RNA提取试剂盒提取样品总RNA,反转录利用HiScriptⅡ 1st Strand cDNA Synthesis kit(诺唯赞,南京)试剂盒,利用ChamQTM SYBR®qPCR Master Mix试剂(诺唯赞,南京)在罗氏LightCycler480系统上进行qRT-PCR。内参基因为Histone H3.3 (LOC105176435)[26],引物设计利用Primer 5软件(表1)。
2 结果
2.1 芝麻发芽期耐盐性鉴定适宜NaCl浓度的确定
在不同浓度的NaCl胁迫下,8份芝麻种质材料的发芽均受到不同程度的影响(图1,图2),在50 mmol·L-1的NaCl胁迫下,发芽势、发芽指数和活力指数与对照相比均未出现明显变化,成苗率和芽长略有下降,而5份材料的根长和6份材料的鲜重却略有升高,说明低浓度的NaCl胁迫对某些材料的幼苗生长有一定促进作用。在100 mmol·L-1的NaCl胁迫下,7份材料的发芽势与对照相比未出现明显变化,仅S38显著下降,所有材料的成苗率、发芽指数、活力指数、根长、芽长和鲜重与对照相比均出现显著下降。当NaCl浓度升高到150 mmol·L-1,所有材料的发芽均受到明显抑制,各发芽指标值均显著下降;多数材料的胚根虽然能够萌动,但发芽后期多为畸形、坏死、子叶不能正常伸展等等。在200和250 mmol·L-1NaCl胁迫下,多数材料不能萌动。

表1 候选基因的引物序列

对不同浓度NaCl胁迫下8份芝麻材料发芽各指标进行统计和方差分析表明(表2):50 mmol·L-1的NaCl处理与对照相比,发芽势、根长和鲜重均无显著差异,说明盐胁迫程度较轻;100 mmol·L-1的NaCl处理下,除发芽势外,发芽指数、活力指数、成苗率、根长、芽长和苗鲜重的标准偏差值均较大,所有指标在0.05水平上均存在显著差异,说明这些指标在8份芝麻材料间分散程度均较大:150、200和250 mmol·L-1的NaCl处理下,发芽势、发芽指数、活力指数、成苗率、根长、芽长和苗鲜重与对照相比均差异显著,但大多数种质材料的绝大多数种子不能成长为正常苗(图1,图2),说明盐胁迫程度过重。因此,100 mmol·L-1的NaCl可以作为芝麻发芽期耐盐性鉴定的适宜处理浓度。
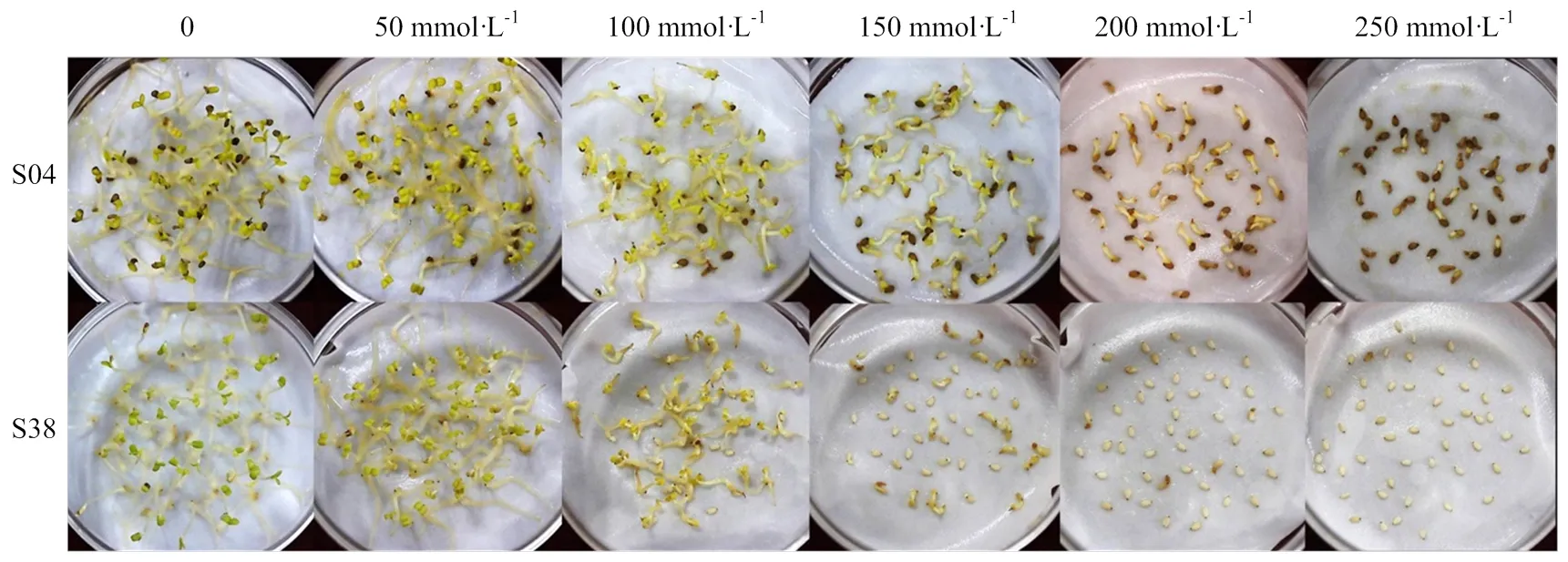
图2 不同浓度NaCl胁迫下耐盐(S04)和敏感(S38)芝麻种质材料发芽第6天的表现
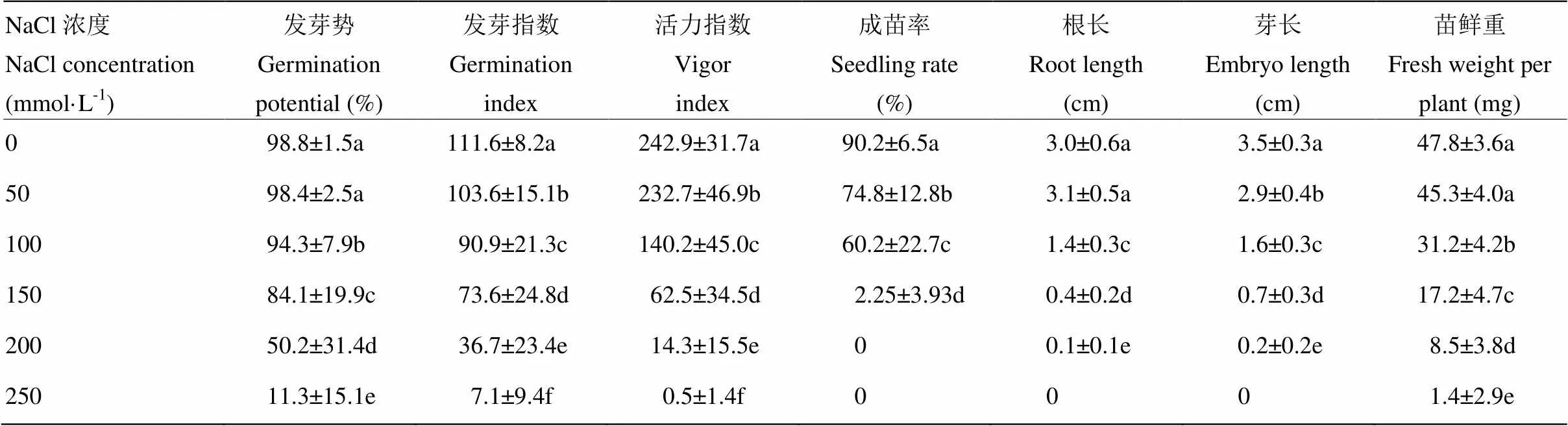
表2 不同浓度NaCl胁迫下8份芝麻材料发芽各指标的统计分析
同一列不同字母表示在=0.05水平差异显著
Different letters in the same column for each variable indicates a significant difference at 0.05 level
2.2 芝麻发芽期耐盐性鉴定适宜评价指标的筛选
对100 mmol·L-1的NaCl胁迫下芝麻发芽各指标的相对值进行统计分析、相关性分析、主成分分析和隶属函数分析。8份芝麻材料的各发芽指标差异明显,其中相对成苗率变化幅度最大,为28.97%—88.63%,标准偏差值也最大,表明该指标在各材料间分散程度最大(表3)。相关性分析表明(表4),各指标间均存在一定的相关性,其中相对鲜重、相对活力指数、相对芽长和相对成苗率之间的相关性在0.05水平上极显著,相关系数分别为0.815*、0.787*和0.759*。主成分分析结果显示(表5),第一主成分贡献率为76.920%,第二主成分贡献率为9.984%,前2个主成分的累计贡献率达到86.904%,包含了原始指标的绝大部分信息。第一主成分主要包括相对活力指数、相对芽长、相对发芽指数和相对发芽势,第二主成分主要包括相对根长、相对鲜重和相对成苗率,表明这7个指标均能反映芝麻的耐盐性。
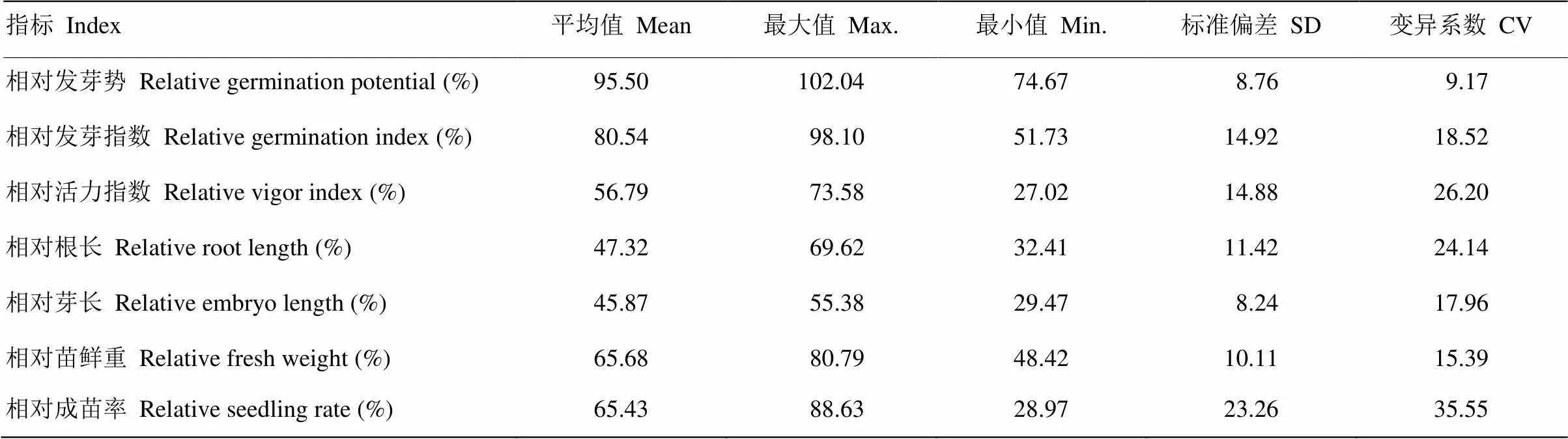
表3 100 mmol·L-1的NaCl处理条件下芝麻发芽期各指标的统计分析
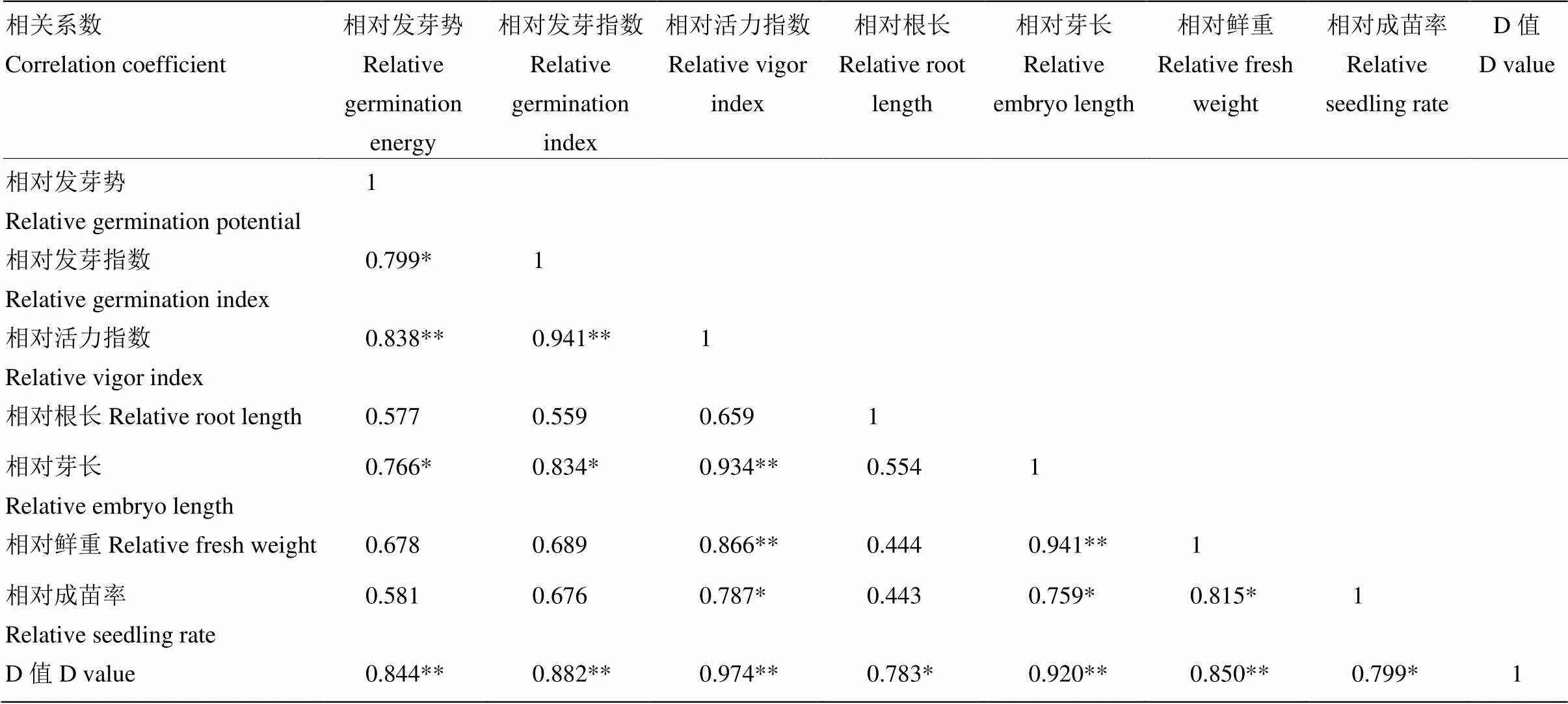
表4 100 mmol·L-1的NaCl处理条件下芝麻发芽期各指标的相关性分析
** 在0.01水平差异极显著;*在0.05水平差异显著
** indicates a significant relationship at 0.01 level; * indicates a significant relationship at 0.05 level
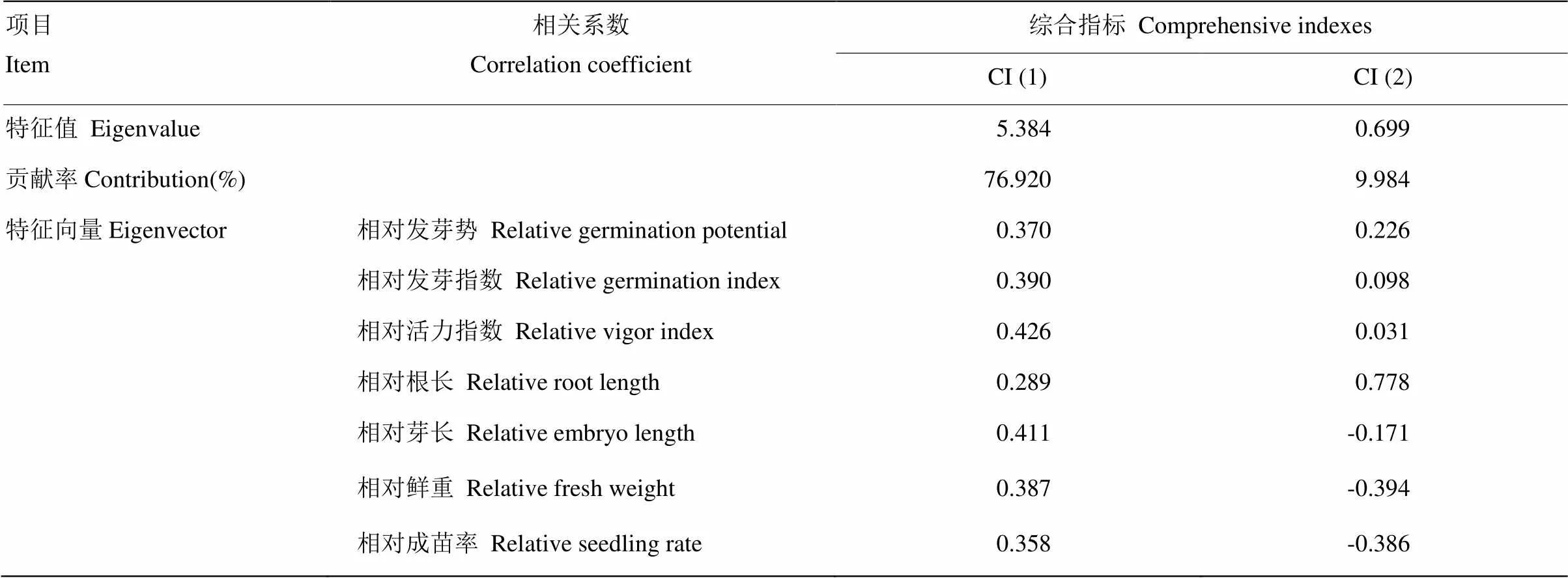
表5 2个综合指标的特征值、贡献率及特征向量
基于前2个主成分的综合指标值,采用隶属函数法综合评价8份芝麻种质材料的耐盐性。先根据公式(1)获得各材料所有综合指标的隶属函数值,通过公式(2)计算出各综合指标的权重,再根据公式(3)计算出各材料在100 mmol·L-1NaCl胁迫下耐盐性综合评价D值(表6)。D值越大,材料的综合耐盐性越强,从中评选出2份高耐盐种质S11和S10,其D值分别为0.991和0.907,S04种质耐盐性较强,D值为0.721。相关性分析显示D值与7个耐盐鉴定指标间均呈显著正相关(表4),表明这7个指标能较好的区分芝麻不同种质的耐盐性差异。
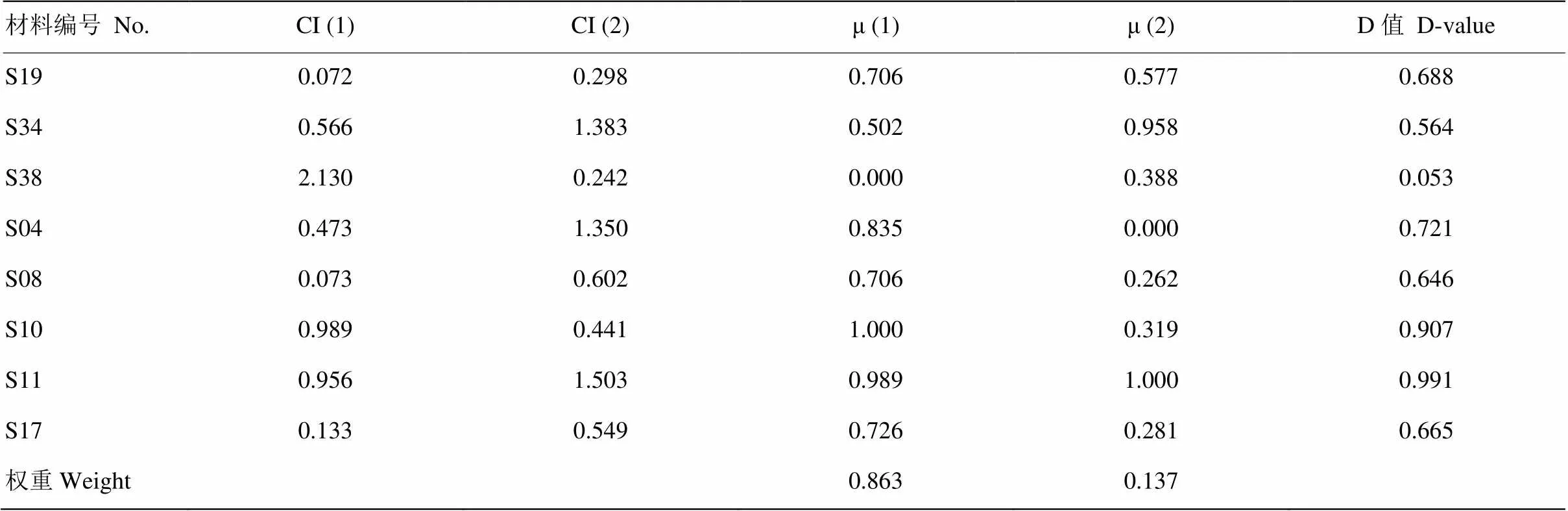
表6 各材料综合指标值、权重、μ(x)值和D值
2.3 芝麻核心种质群体全基因组关联分析及耐盐候选基因的预测
对71份(含以上8份)芝麻核心种质在100 mmol·L-1NaCl胁迫下发芽期的相对成苗率调查记载,数值范围为16.67%—88.26%,平均值为51.04%,变异系数为30.08。当样本数量n>50时,参考国家标准GB4882-85进行正态检验,D检验结果为0.0785(大于0.05),符合正态分布,且在各个等级均有分布,耐盐性变异比较丰富(图3),该群体适合做耐盐性全基因组关联分析。
71份核心种质相对成苗率与SNP标记进行GWAS分析,共检测到7个与相对成苗率显著关联的SNP标记(LG5:688003、LG7:9582027、LG10:5274091、LG10:10788493、LG11:11924186、LG14:2128695和LG16:3930301),分别位于LG5、LG7、LG10、LG11、LG14和LG16连锁群上(图4),SNP标记上、下游各100 kb区间内共有基因67个,其中有功能注释的基因34个,包含了7个参与植物非生物逆境胁迫应答的基因,如甲基转移酶(LOC105171975和LOC105171977)、尿苷二磷酸葡萄糖基转移酶(LOC105172099)、红素氧还蛋白(LOC105176797)、丝氨酸/苏氨酸蛋白磷酸酶(LOC105176801)和富亮氨酸重复类受体蛋白激酶(LOC105176803)等基因,涉及多种生物学功能。候选基因中还包含了5个去甲乌药碱合成酶类似蛋白基因和4个去甲乌药碱合成酶2类似蛋白基因,均位于第7连锁群,其中LOC105166829、LOC105166830、LOC105166831和LOC105166832、LOC105166836和LOC105166837为两组串联重复基因。
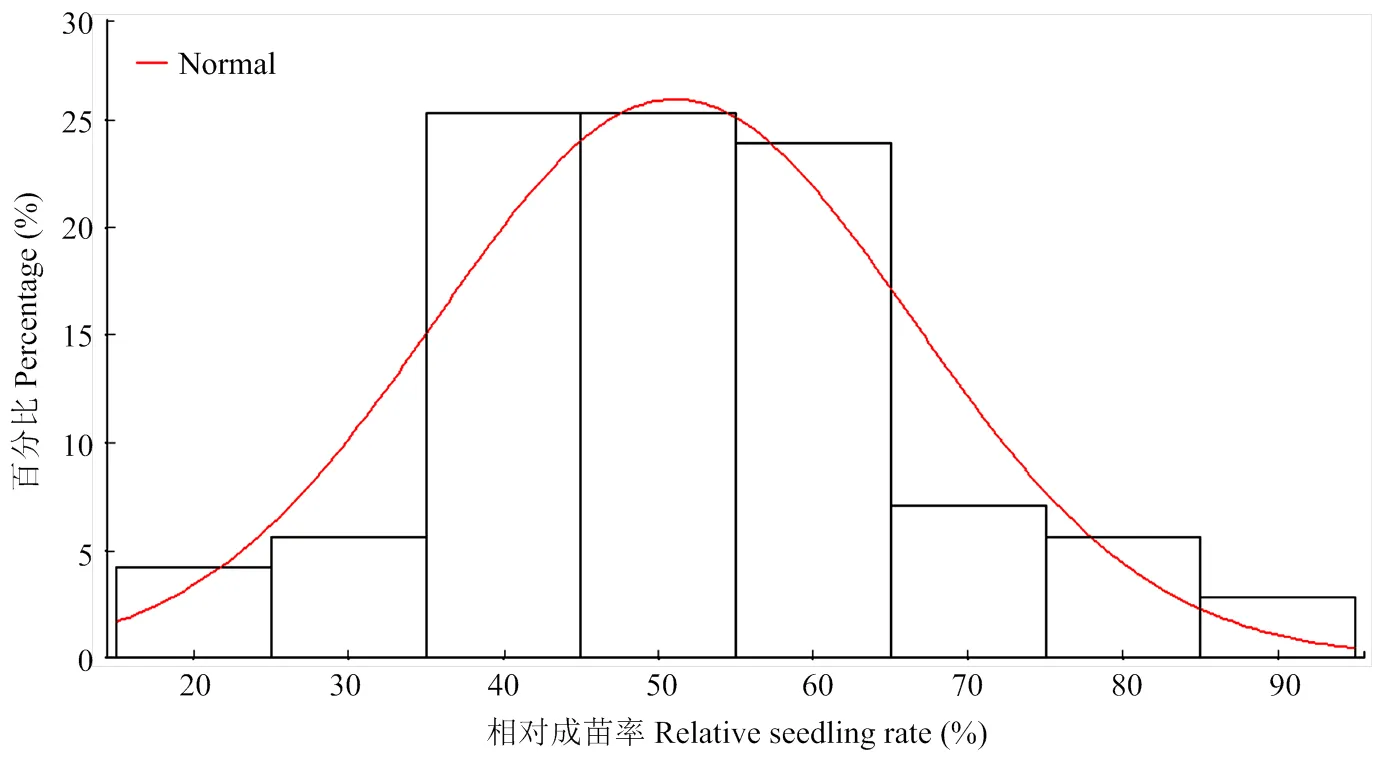
图3 核心种质群体相对成苗率正态分布图
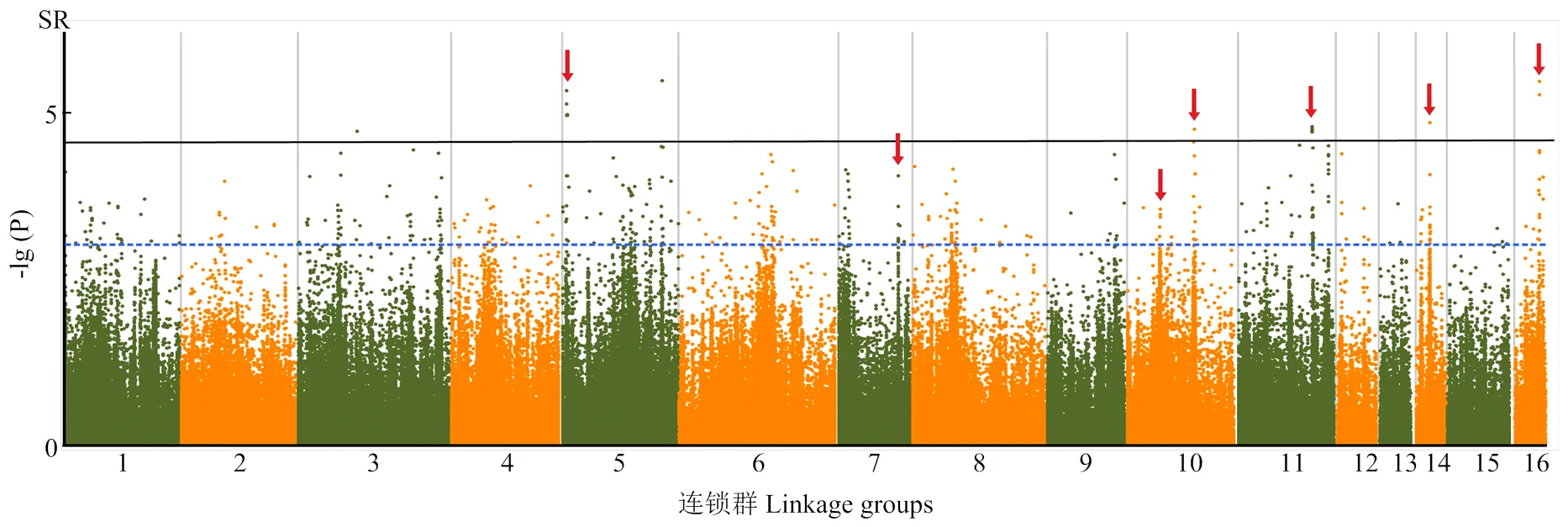
图4 盐胁迫下芝麻发芽期相对成苗率全基因组关联分析的曼哈顿图
2.4 耐盐候选基因的表达分析
利用耐盐材料S04在盐胁迫下的转录组结果(未发表数据)对以上预测的34个候选基因进行表达模式分析,其中21个候选基因在芝麻叶片中受到盐胁迫诱导显著差异表达(图5,表7),有12个基因在盐胁迫24与0 h相比表达量显著下降,9个基因显著上调表达,基因LOC105172100和LOC105166820分别编码了多聚半乳糖醛酸酶和ATP酶,在NaCl胁迫6、12和24 h均显著上调表达,基因LOC105166829、LOC105172096、LOC105166932、LOC105166836、LOC105166833、LOC105166827和LOC105176796在NaCl胁迫6或12 h显著上调表达(图5)。
从21个候选基因中随机选取8个基因进行qRT- PCR分析,结果显示,LOC105166820、LOC105166829、LOC105166932和LOC105172100在NaCl胁迫后6—24 h上调表达,LOC105172099、LOC105176797、LOC105176803和LOC105166823在NaCl胁迫后下调表达(图6),表达量变化趋势与转录组分析结果一致。
3 讨论
发芽期耐盐性鉴定快速、简便、高效,适用于资源和育种群体的大规模评价筛选,已被广泛应用于其他作物。目前,国内小麦种质资源芽期耐盐性鉴定参考农业部行业标准《小麦耐盐性鉴定评价技术规范》NY/PZT001-2002进行,该标准以350 mmol·L-1NaCl为筛选浓度,以相对盐害率为鉴定指标;《棉花耐盐性鉴定评价技术规范》DB13T1339-2010规定了棉花发芽期耐盐性鉴定方法,该方法以0.5%的NaCl或0.4%的地下咸水为盐胁迫浓度,以相对盐害率作为评价指标;水稻和玉米种质芽期耐盐性鉴定目前在国内尚未有统一标准,田蕾等[4]利用125 mmol·L-1NaCl溶液对64份粳稻种质资源耐盐性进行鉴定评价,相对盐害率、发芽指数和相对根长可以作为粳稻芽期耐盐性快速鉴定的重要指标;张海艳等[5]以220和270 mmol·L-1的NaCl两种胁迫浓度对47份玉米品种进行发芽期耐盐性综合评价,认为发芽率在所有玉米发芽期鉴定指标中与耐盐性关系最密切,在品种间的表现也最稳定。本研究筛选的芝麻发芽期耐盐性鉴定以100 mmol·L-1NaCl为适宜胁迫浓度,与以上介绍的几种作物均不同,可能与不同作物对盐胁迫的耐受能力不同有关;本研究发现相对活力指数、相对芽长、相对发芽指数、相对发芽势、相对根长、相对鲜重和相对成苗率与耐盐性关系均较为密切,与报道的其他作物类似,当然,各指标的贡献率存在一定差异,表明芝麻耐盐性是一个较复杂的性状,为研究制订综合评价方法或标准奠定基础。
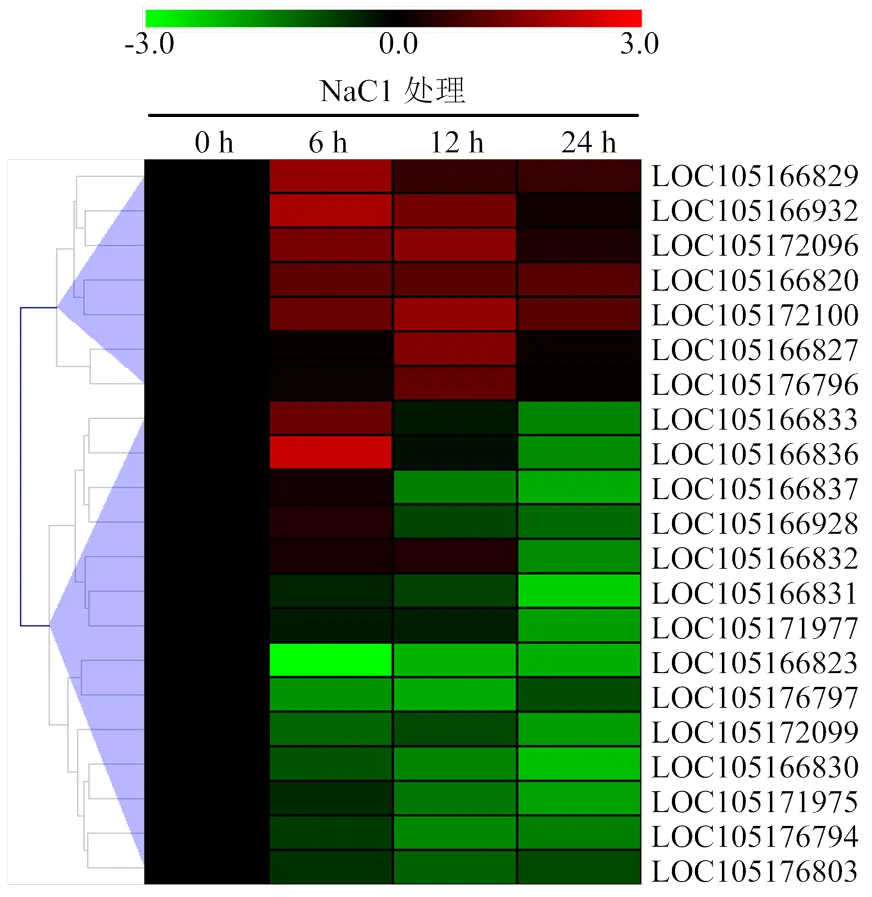
图5 NaCl胁迫下21个候选基因的差异表达模式
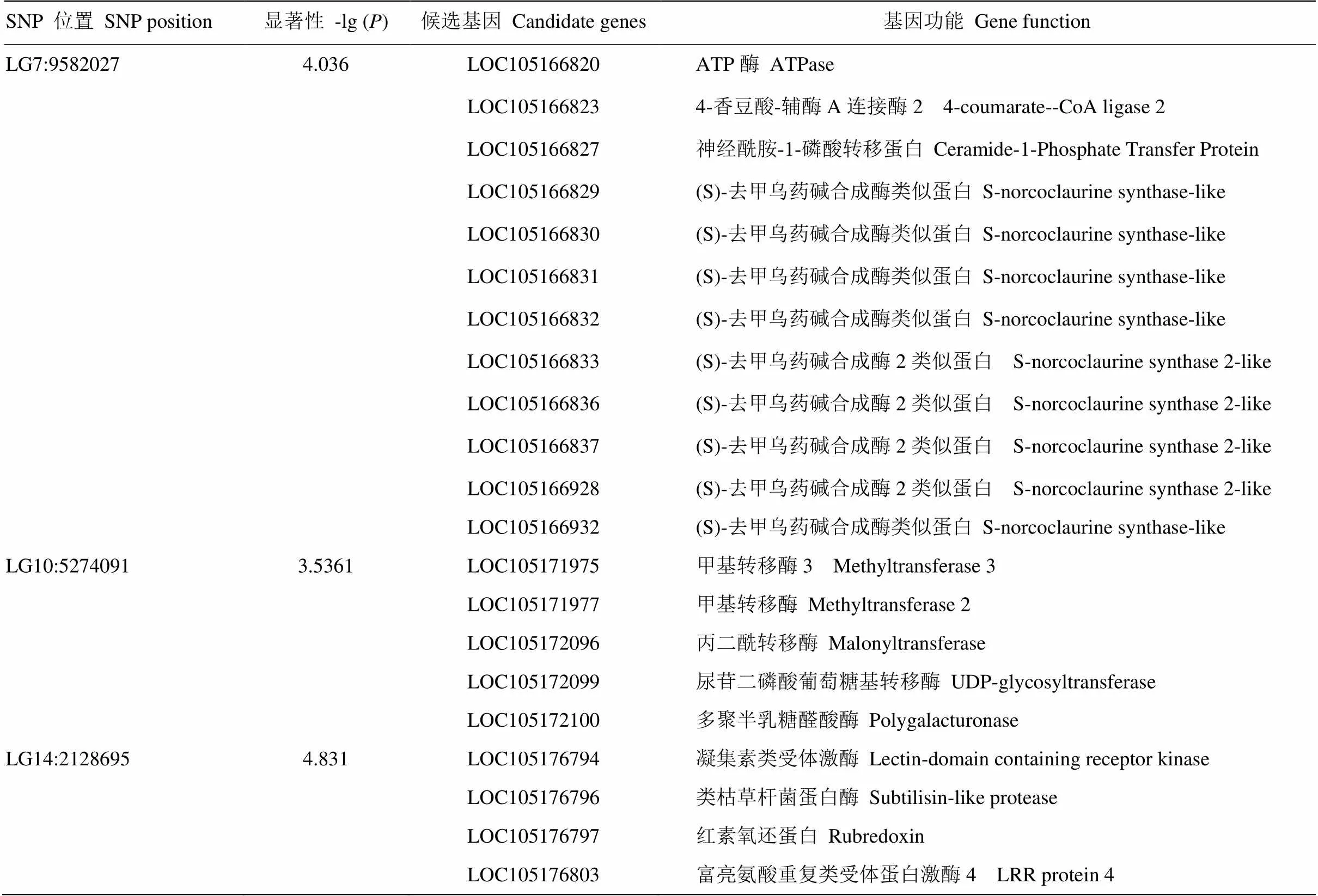
表7 芝麻耐盐性显著相关的SNP位点和耐盐候选基因
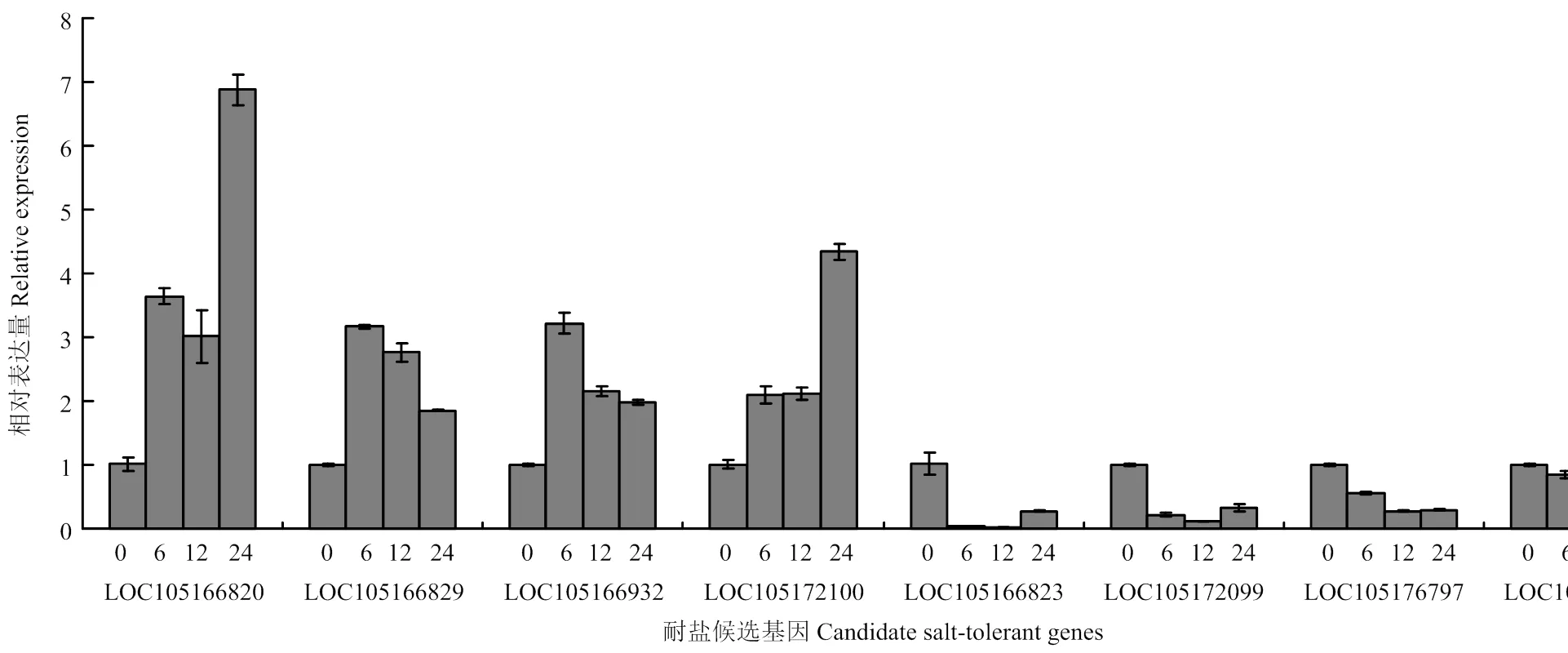
图6 NaCl胁迫后8个耐盐候选基因的表达模式
本研究在前期芝麻核心种质全基因组重测序[24]的基础上,利用GWAS方法,获得了7个与耐盐性显著关联的SNP标记,可优先选择贡献率较大的标记,进一步验证后开发耐盐分子标记,用于芝麻耐盐品种的遗传改良。本研究挖掘到21个与芝麻耐盐性有关的重要候选基因,其中,SNP 5274091候选区间的候选基因LOC105172100位于10号连锁群,编码一个多聚半乳糖醛酸酶(PG),该酶是植物最大的水解酶家族之一,已被证实其参与植物非生物逆境胁迫的响应过程之中[27-29]。Orozcocárdenas等[28]研究发现基因在逆境胁迫下大量上调表达,PG催化的水解反应中间产物寡聚半乳糖醛酸可以快速触发H2O2反应,H2O2作为第二信使参与到植物的防御反应。寇晓虹[29]利用NaCl胁迫处理番茄幼苗和绿熟期的果实组织圆片,发现在NaCl处理1 h后显著上调表达,认为很有可能作为一个早期的信号基因参与NaCl引起的胁迫反应,并利用产物激发子传递信号,激发植物防卫反应信号途径中一系列基因的表达。本研究中LOC105172100在芝麻幼苗叶片中受盐胁迫快速显著上调表达,提示其可能以类似的方式参与到芝麻盐胁迫初期防御系统。
在本研究中,候选基因LOC105172099编码了一种尿苷二磷酸葡萄糖基转移酶(UDP-glycosyltransferase,UGT),UGT被发现可能通过调节次级代谢产物和体内激素平衡而参与植物的非生物逆境胁迫抵御过程[30-31],芝麻幼苗叶片中LOC105172099基因在盐胁迫下显著下调表达,说明它很可能以负调控方式参与到芝麻盐胁迫调控过程。研究表明红素氧还蛋白基因在植物抵御非生物胁迫方面发挥着重要的作用[32-33],Li等[34]研究发现红素氧还类似蛋白基因可以通过调节植物体内离子平衡和清除体内活性氧来提高拟南芥对NaCl胁迫的抵御,本研究筛选到一个红素氧还蛋白基因LOC105176797,位于第14连锁群,该基因很可能在芝麻抵御NaCl胁迫过程中也起到类似的作用。这些候选基因的发现,对芝麻耐盐定向育种具有重要的意义。除此之外,SNP 9582027候选区间LOC105166829、LOC105166830、LOC105166831和LOC105166832为一组串联重复基因,均编码一种去甲乌药碱合成酶(NCS)类似蛋白,NCS是苄基异喹啉类生物碱合成代谢途径初始反应步骤的关键酶[35],目前,尚未有研究发现其参与植物的逆境胁迫响应过程,但基因表达模式分析显示这些基因很可能与芝麻盐胁迫响应机制有关,有待进一步验证。总之,本研究鉴定得到的与芝麻耐盐性相关的SNP位点和候选基因为后续开展芝麻大规模耐盐等位基因发掘、耐盐基因验证和功能标记开发等相关研究奠定重要的工作基础。
4 结论
确定了100 mmol·L-1NaCl为芝麻发芽期耐盐性鉴定的适宜浓度,相对成苗率等7个指标与芝麻耐盐性关系密切。71份芝麻核心种质材料的耐盐性数值变异比较丰富,符合正态分布,共检测到与芝麻发芽期耐盐性显著关联的SNP位点7个,连锁不平衡区域包含候选基因67个,基因功能注释和盐胁迫表达分析获得耐盐候选基因21个。
[1] 杨真, 王宝山. 中国盐渍土资源现状及改良利用对策. 山东农业科学, 2015,47(4): 125-130.
YANG Z, WANG B S. Present status of saline soil resources and countermeasures for improvement and utilization in China., 2015, 47(4):125-130. (in Chinese)
[2] 冯琛, 党高兵, 解建仓, 孙博, 汪妮. 大米草玉米的耐盐效果及对盐渍化土壤的改良试验. 水土保持通报, 2011, 31(6): 47-50.
FENG C, DANG G B, XIE J C, SUN B, WANG, N. Salt-tolerant effect of rice grass-maize and improvement on saline soil., 2011, 31(6): 47-50. (in Chinese)
[3] 张鸿, 林静, 王立成, 王阳, 何威, 李卫华. 春小麦种质资源耐盐性鉴定及筛选. 种子, 2014, 33(6): 53-57.
ZHANG H, LIN J, WANG L C, WANG Y, HE W, LI W H. The identification and screening of salt tolerance of spring wheat germplasm resources., 2014, 33(6): 53-57. (in Chinese)
[4] 田蕾, 陈亚萍, 刘俊, 马晓刚, 王娜, 杨兵, 李莹, 郭海东, 李娟, 胡慧, 张银霞, 李培富. 粳稻种质资源芽期耐盐性综合评价与筛选. 中国水稻科学, 2017, 31(6): 631-642.
TIAN L, CHEN Y P, LIU J, MA X G, WANG N, YANG B, LI Y, GUO H D, LI J, HU H, ZHANG Y X, LI P F. Comprehensive evaluation and selection of rice () germplasm for saline tolerance at germination stage., 2017, 31(6): 631-642. (in Chinese)
[5] 张海艳, 赵海军. 不同品种玉米萌发期和苗期耐盐性综合评价. 玉米科学, 2016, 24(5): 61-67.
ZHANG H Y, ZHAO H J. Comprehensive evaluation of salt tolerance of different corn varieties at the germination and seedling stages., 2016, 24(5): 61-67. (in Chinese)
[6] 王俊娟, 樊伟莉, 叶武威, 王德龙. 陆地棉萌发出苗期耐盐鉴定方法的研究及应用. 江苏农业科学, 2010(2): 87-88.
WANG J J, FAN W L, YE W W, WANG D L. The research and application of the method of salt tolerance on upland cotton at the germination and seedling stages., 2010(2): 87-88. (in Chinese)
[7] 沈义国, 陈受宜. 植物盐胁迫应答的分子机制. 遗传, 2001, 23(4): 365-369.
SHEN Y G, CHEN S Y. Molecular mechanism of plant responses to salt stress., 2001, 23(4): 365-369. (in Chinese)
[8] ZHU J K. Cell signaling under salt, water and cold stresses., 2001, 4(5): 401-406.
[9] XU Y, LI S, LI L, ZHANG X, XU H, AN D. Mapping QTLs for salt tolerance with additive, epistatic and QTL×treatment interaction effects at seedling stage in wheat., 2013, 132(3): 276-283.
[10] YU L X, LIU X, BOGE W, LIU X P. Genome-wide association study identifies loci for salt tolerance during germination in(L.) using genotyping-by-sequencing., 2016, 7: 956.
[11] KOBAYASHI Y, SADUKHAN A, TAZIB T, NAKANOY, KUSUNOKI K, KAMARA M, CHAFFAI R, IUCHI S, SAHOO L, KOBAYASHI M, HOEKENGA OA, KOYAMA H. Joint genetic and network analyses identify loci associated with root growth under NaCl stress in., 2016, 39(4): 918-934.
[12] DHINGRA A, DANIELL H. Plastid-expressed betaine aldehyde dehydrogenase gene in carrot cultured cells, roots, and leaves confers enhanced salt tolerance., 2004, 136(1): 2843-2854.
[13] HMIDASAYARI A, GARGOURIBOUZID R, BIDANI A, JAOUA L, SAVOURE A, JAOUA S. Overexpression of delta¹-pyrroline-5- carboxylate synthetase increases proline production and confers salt tolerance in transgenic potato plants., 2005, 169(4): 746-752.
[14] QIAO W H, ZHAO X Y, LI W, LUO Y, ZHANG X S. Overexpression of AeNHX1, a root-specific vacuolar Na+/H+antiporter from, confers salt tolerance toandplants., 2007, 26(9): 1663-1672.
[15] 陈凤莲, 刘志斌, 王健美, 李旭锋, 杨毅. 油菜BnRCH基因提高转基因拟南芥的耐盐性研究. 四川大学学报(自然科学版), 2013, 50(3): 643-648.
CHEN F L, LIU Z B, WANG J M, LI X F, YANG Y. The studies of gene BnRCH from Brassica napus enhances the tolerance to salt stress in transgenic., 2013, 50(3): 643-648. (in Chinese)
[16] JING P, ZOU J, KONG L, HU S, WANG B, YANG J, XIE G. OsCCD1, a novel small calcium-binding protein with one EF-hand motif, positively regulates osmotic and salt tolerance in rice., 2016, 247: 104-14.
[17] 孙建, 乐美旺, 饶月亮, 颜廷献, 颜小文, 周红英. 芽期NaCl胁迫下不同基因型芝麻的反应差异研究. 干旱地区农业研究, 2014, 32(6): 75-81.
SUN J, LE M W, RAO Y L, YAN T X, YAN X W, ZHOU H Y. Genotypic variation in sesame salt tolerance during germination under nacl stress., 2014, 32(6): 75-81. (in Chinese)
[18] 徐芬芬, 杜佳朋. 干旱胁迫和盐胁迫对芝麻种子萌发的影响. 种子, 2013, 32(11): 85-86.
XU F F, DU J P. Effect of drought and salt stress on seed germination of sesame., 2013, 32(11): 85-86. (in Chinese)
[19] DIAS A S, LIMA G S D, GHEYI H R, NOBRE R G, SANTOS J B D. Emergence, growth and production of sesame under salt stress and proportions of nitrate and ammonium., 2017, 30(2): 458-467.
[20] FARISSI M, BOUIZGAREN A, FAGHIRE M, BARGAZ A, GHOULAM C. Agro-physiological and biochemical properties associated with adaptation ofpopulations to water deficit., 2013, 37(6): 1166-1175.
[21] 吴兰荣, 陈静, 许婷婷, 苗华荣, 胡文广, 禹山林. 花生全生育期耐盐鉴定研究. 花生学报, 2005(1): 20-24.
WU L R, CHEN J, XU T T, MIAO H R, HU W G, YU S L. Identification of salt tolerance in peanut growth duration., 2005(1): 20-24. (in Chinese)
[22] 支巨振, 毕辛华, 杜克敏, 常秀兰, 杨淑惠, 任淑萍, 吴志行, 李仁凤, 赵菊英. GB/T 3543.4-1995, 农作物种子检验规程发芽试验. 北京:中国标准出版社, 1995.
ZHI J Z, BI X H, DU K M, CHANG X L,YANG S H, REN S P, WU Z H, LI R F, ZHAO J Y. GB/T 3543.4-1995, Rules for agricultural seed testing-Germination test. Beijing: China Standard Press, 1995. (in Chinese)
[23] 刘文萍, 吕伟, 黎冬华, 任果香, 张艳欣, 文飞, 韩俊梅, 张秀荣. 芝麻种质资源成株期抗旱性关联分析. 中国农业科学, 2017, 50(4): 625-639.
LIU W P , LÜ W, LI D H, REN G X, ZHANG Y X, WEN F, HAN J M, ZHANG X R. Drought resistance of sesame germplasm resources and association analysis at adult stage., 2017, 50(4): 625-639. (in Chinese)
[24] WEI X, LIU K, ZHANG Y, FENG Q, WANG L, ZHAO Y, LI D, ZHAO Q, ZHU X, ZHU X, LI W, FAN D, GAO Y, LU Y, ZHANG X, TANG X, ZHOU C, ZHU C, LIU L, ZHONG R, TIAN Q, WEN Z, WENG Q, HAN B, HUANG X, ZHANG X. Genetic discovery for oil production and quality in sesame., 2015, 6: 8609.
[25] BRADBURY P J, ZHANG Z, KROON D E, CASSTEVENS T M, RAMDOSS Y, BUCKLER E S. TASSEL: Software for association mapping of complex traits in diverse samples., 2007, 23(19): 2633-2635.
[26] WEI L, MIAO H, ZHAO R, HAN X, ZHANG T, ZHANG H. Identification and testing of reference genes for Sesame gene expression analysis by quantitative real-time PCR., 2013, 237(3): 873-889.
[27] SÉNÉCHAL F, WATTIER C, RUSTÉRUCCI C, PELLOUX J. Homogalacturonan-modifying enzymes: structure, expression, and roles in plants., 2014, 65(18): 5125.
[28] Orozcocárdenas M, Narváezvásquez J, Ryan C. Hydrogen peroxide acts as a second messenger for the induction of defense genes in tomato plants in response to wounding, systemin, and methyl jasmonate., 2001, 13(1): 179-191.
[29] 寇晓虹. 番茄多聚半乳糖醛酸酶(PG)反义基因表达及相关功能研究[D]. 北京: 中国农业大学, 2003.
KOU X H. Antisense gene expression and gene function of polygalacturonase (PG) in tomato[D]. Beijing: China Agricultural University, 2003. (in Chinese)
[30] O’DONNELL P J, TRUESDALE M R, CALVERT C M, DORANS A, ROBERTS M R, BOWLES D J. A novel tomato gene that rapidly responds to wound- and pathogen-related signals., 1998, 14(1): 137-142.
[31] Roberts M R, Warner S A J, Darby R, Lim E K, Draper J, Bowles D J. Differential regulation of a glucosyl transferase gene homologue during defence responses in tobacco., 1999, 50(332): 407-410.
[32] 李莹, 柳参奎. 红素氧还蛋白研究进展. 草业科学, 2016, 33(6): 1118-1125.
LI Y, LIU S K. research progress on rubredoxin., 2016, 33(6): 1118-1125. (in Chinese)
[33] 林凡云, 陆琼娴, 徐剑宏, 史建荣. 两个与盐和赤霉病菌胁迫相关的小麦糖基转移酶基因的克隆与表达. 遗传, 2008, 30(12): 1608-1614.
LIN F Y, LU Q X, XU J H, SHI J R. Cloning and expression analysis of two salt and Fusarium graminearum stress associated UDP- glucosyltransferases genes in wheat., 2008, 30(12): 1608-1614.(in Chinese)
[34] ZHU J, FU X, KOO Y D, ZHU J K, FRANCIS E J J, MICHAEL W W A, ZHU Y, SHI H, YUN D J, HASEGAWA P M, BRESSAN R A. An enhancer mutant ofmediates both ion homeostasis and the oxidative stress response., 2007, 27(14): 5214.
[35] SAMANANI N, LISCOMBE D K, FACCHINI P J. Molecular cloning and characterization of norcoclaurine synthase, an enzyme catalyzing the first committed step in benzylisoquinoline alkaloid biosynthesis., 2004, 40(2): 302-313.
(责任编辑 李莉)
Screening Method for Salt Tolerance in Sesame (L.) and Identification of Candidate Salt-tolerant Genes
ZHANG YuJuan1,2, YOU Jun1, LIU AiLi1, LI DongHua1, YU JingYin1, WANG YanYan1, ZHOU Rong1, GONG HuiHui2, ZHANG XiuRong1
(1Oil Crops Research Institute of the Chinese Academy of Agricultural Sciences/Key Laboratory of Biology and Genetic Improvement of Oil Crops, Ministry of Agriculture, Wuhan 430062;2Cotton Research Center, Shandong Academy of Agricultural Sciences, Jinan 250100 )
【Objective】In order to evaluate salt tolerance of sesame germplasms and reveal the genetic basis of salt tolerance in sesame at the germination stage, optimum NaCl concentration and best salt tolerance indexes were determined, salt-tolerant sesame genotypes as well as candidate genes were identified in this study. 【Method】Eight sesame genotypes with different salt tolerance were germinated under different NaCl concentrations (0, 50, 100, 150, 200 and 250 mmol·L-1). Seven germination indexes including germination potential, germination index, vigor index, seedling rate, radicle length, embryo length and fresh weight of seedling were measured. The optimum NaCl concentration and salt tolerance indexes of sesame were determined using variance analysis, principal component analysis, membership function and correlation analysis based on the relative values of multiple indexes. In addition, genome-wide association analysis (GWAS) of salt tolerance related trait in 71 sesame germplasms was performed to find the SNP loci and candidate genes related to salt tolerance in sesame. Gene function annotation, transcriptome analyses and real-time quantitative reverse transcriptase PCR (qRT-PCR) were used to identify important candidate genes involved in salt tolerance. 【Result】The larger standard value deviations of germination indexes were observed at 100 mmol·L-1NaCl concentration, implying that 100 mmol·L-1is the optimum NaCl concentration for salt-tolerance screening in this study. The seven indexes were highly correlated with salt tolerance at germination stage, suggesting that, these indexes could be targeted for effective screening of salt-tolerance in large sesame germplasm. The genome-wide association analysis identified 7 SNP loci peaks (LG5:688003, LG7:9582027, LG10:5274091, LG10:10788493, LG11:11924186, LG14:2128695 and LG16:3930301) significantly associated with salt tolerance and 34 functional candidate genes. The transcriptome analysis and qRT-PCR showed that 21 candidate genes respond strongly to salt stress.【Conclusion】In this study, 100 mmol·L-1is the optimum NaCl concentration for salt-tolerance screening and seven indexes including relative seedling rate could be targeted for effective screening of salt-tolerance in large sesame germplasm. In addition, 7 SNP loci peaks significantly associated with salt tolerance were identified and 21 candidate salt-tolerant genes were discovered.
sesame; salt tolerance; genome-wide association study; salt-tolerant genes
2018-02-06;
2018-04-09
农业部油料作物生物学与遗传育种重点实验室开放课题(2017007)、国家特色油料产业技术体系(CARS-14)、中国农业科学院创新工程(CAAS-ASTIP-2013-OCRI)、农业部黄淮海棉花遗传改良与栽培生理重点实验室开放课题(2016KL03)、山东省农业科学院创新工程“棉花与特色经济作物学科团队建设”
张玉娟,E-mail:723949246@qq.com。
张秀荣,E-mail:zhangxr@oilcrops.cn
10.3864/j.issn.0578-1752.2018.12.002