人冠状病毒HKU1 N和S蛋白基因序列及进化分析
2018-06-21,,,,,,
,,,, ,,
呼吸道和下呼吸道感染是人类主要疾病之一,是儿童发病、住院和死亡的主要原因。引起人类呼吸道感染的病原十分复杂,已知大约有5%~30%是由人冠状病毒HCoV-229E和HCoV-OC43感染引起[1],主要导致普通感冒。2002-2003年“非典”暴发,SARS[2]冠状病毒的出现推进了人们对冠状病毒的研究。继SARS-CoV之后,科学家们又发现了两种新的引起呼吸系统疾病的HCoV(HCoV-NL63[3]和HCoV-HKU1[1])。HCoV-HKU1是2005年1月由香港科学家在一位老年肺炎患者体内发现的,同年9月,另一位澳大利亚学者[4]在儿童急性呼吸道感染中也发现了该病毒的存在。该病毒可引起急性呼吸道感染,症状有流涕、发热、咳嗽和喘鸣,严重者表现为支气管炎和肺炎[4-5],也可引起胃肠疾病[6],甚至和热性癫痫发作有关[6]。
冠状病毒(Human coronaviruses, HCoV)属冠状病毒科冠状病毒属,在自然界中广泛存在,人和多种动物易感。形态上呈圆形或椭圆形,因其在电镜下呈日冕状或皇冠状而得名。为单股正链RNA病毒,是目前已知RNA病毒中基因组最大的病毒,基因组全长约27~32 kb,使其在加工和修饰过程中增加了不稳定性;由于RNA聚合酶忠实性较低,RNA病毒在复制过程中基因序列较易发生点突变;同时由于冠状病毒具有独特的复制机制,在基因组复制过程中存在一个复制中间体导致其在进化过程中发生重组的概率增大,因而不断出现新的变异株而呈现遗传多样性。
HCoV-HKU1为无节段单股正链RNA病毒,GC含量为32%,是所有已知冠状病毒中GC含量最低的。根据病毒基因组的序列,HCoV-HKU1和HCoV-OC43同属β类冠状病毒[7]。病毒基因组(图1)5′ 端具甲基化的帽状结构,3′ 端具有 poly(A)尾,两端分别包含一个转录非转译区(Untranslated region, UTR),即 5′-UTR和 3′-UTR,其长度分别在200~600nt和200~500nt之间。其中,5′-UTR包含核糖体结合位点和转录起始信号,3′-UTR含有转录终止信号。距5′ 端约2/3的基因组包含两个大的重叠的开放读码框(Open reading frame, ORF)ORF 1a和ORF 1b,主要负责编码与病毒复制转录有关的酶类等非结构蛋白,比如RNA聚合酶(RNA-dependent RNA polymerase,pol),复制酶等和结构蛋白。结构蛋白包括血凝素酯酶糖蛋白(hemagglutinin-esterase, HE)、棘刺蛋白(spike, S)、膜蛋白(membrane, M)、包膜蛋白(envelop, E)和核衣壳蛋白(nucleocapsid, N)。在结构蛋白之间通常含有一些小的开放读码框ORFs。体外实验表明,有些ORF并非病毒复制所必须,但与病毒的致病性有关,同时决定着病毒的群特异性[8]。研究HCoV-HKU1病毒各个蛋白基因序列并进行比对和进化树分析是非常有必要的。HCoV-HKU1分型较为复杂,主要根据pol基因、S基因和N基因,香港科学家将HCoV-HKU1可分为A、B、C三型[9-10],并认为C型可能是A型和B型重组形成的。

图1 HCoV-HKU1病毒基因组结构Fig.1 Genome organization of HCoV-HKU1
本研究从2007年11月至2015年1月采集了在福建省福州市妇幼保健院因呼吸道感染住进儿科重症监护病房(PICU)的266例小儿鼻咽抽取物标本,对用RT-PCR法检测出的两例HCoV-HKU1进行了N基因和S基因的全基因序列测定、拼接和系统进化分析。
1 材料与方法
1.1 材料
1.1.1标本采集 采集266例因急性下呼吸道感染住院患儿(临床诊断为支气管炎或肺炎、毛细支气管炎)的鼻咽抽取物(Nasopharyngeal aspirates, NPA)标本,并征得患儿父母的知情同意和医院伦理委员会的批准。
1.1.2试剂仪器 病毒RNA提取采用德国QIAGEN公司的QIAamp Viral RNA Mini Kit,按手册所提供的方法提取病毒RNA。引物由TaKaRa(大连)有限公司合成。M-MLV反转录酶为Invitrogen公司产品(SuperScriptTM Ш Reverse Transcriptase), Taq酶为TaKaRa公司产品。RT-PCR和PCR扩增主要仪器为ABI公司的VeritiTM Dx 96 Well Thermal Cycler基因扩增仪。荧光定量RT-PCR检测试剂盒和荧光定量PCR检测试剂盒均购自上海之江生物技术有限公司。
1.2 方法
1.2.1标本处理 将采集的NPA标本加入3 mL 左右DMEM采样液中,在4 ℃条件下送达实验室后,于生物安全柜中反复吹打标本,并加入双抗(含200 U/mL青霉素,200 U/mL链霉素),分装数管,置-70 ℃保存。
1.2.2病毒核酸提取 将标本冻融两次后,10 000 g离心15 min,吸取上清。采用德国Qiagen 公司的QIAamp Viral RNA Mini Kit,按其手册所提供的方法提取病毒RNA。
1.2.3HCoV-HKU1 RT-PCR产物的扩增 采用一对人冠状病毒的通用引物[1,11]进行扩增,扩增产物为pol基因的一段,长440 bp。反应条件为:50 ℃逆转录30 min, 95 ℃ 15 min, 然后94 ℃ 30 s, 50 ℃ 1 min, 72 ℃ 1 min,共45个循环,最后72 ℃ 10 min;同时采用一对新型人冠状病毒HCoV-HKU1的特异性引物[11-12]进行扩增确认, 扩增产物长950 bp。反应条件为:50 ℃逆转录30 min,95 ℃ 15 min,然后94 ℃ 40 s,50 ℃ 40 s,72 ℃ 1 min,共45个循环,最后72 ℃ 10 min。
1.2.4RNA的体外转录 提取的RNA, 用随机引物和反转录酶逆转录成cDNA,以便进行后续的各种PCR反应。逆转录反应条件为: 65 ℃变性5 min,50 ℃逆转录60 min,70 ℃ 10 min。
1.2.5HCoV-HKU1N基因和S基因的扩增与序列测定 根据文献[13]合成N基因和S基因序列引物。引物以GenBank中NC-006577为参考序列,N基因全长1 326 bp,扩增片段为参考株的28 119~29 682区域,扩增片段长度为1 564 bp;S基因全长4 071 bp,扩增部分为参考株的22 642~27 075区域,总共分3个片段进行扩增。S1片段扩增长度为1 738 bp,S2片段扩增长度为1 625 bp,S3片段扩增长度为1 443 bp。N基因和S基因的S2、S3片段PCR反应条件为:94 ℃,5 min预变性;再以94 ℃,45 s,54 ℃,45 s, 72 ℃,1 min 30 s进行35个循环;最后72 ℃延伸10 min。S1片段PCR退火温度则为52 ℃。各扩增片段用1.2%的琼脂糖凝胶电泳进行检测鉴定后送铂尚生物技术(上海)有限公司进行纯化和序列测定。
1.2.6其它几种呼吸道病毒的检测 对检测出HCoV-HKU1的2例阳性标本也进行了4种常见呼吸道病毒—呼吸道合胞病毒(RSV)、流感病毒(包括甲1、甲3和乙型)、人副流感病毒(HPIV)1-4型、呼吸道腺病毒(R-ADV)和4种呼吸道新病毒(包括人偏肺病毒hMPV、人博卡病毒HBoV、WU多瘤病毒WUPyV和KI多瘤病毒KIPyV)的检测[11]。
1.2.7基因比对分析 将所测得的序列在GenBank中进行BLAST初步比对分析。系统进化分析采用MEGA6.06软件,以邻接法(Neighbor-Joining method)构建进化树,Bootsrap值设定为1000。
2 结 果
2.1病毒检测 从266例患儿中检测到8例HCoV感染阳性病例[11],经基因序列测定和BLAST比对分析得到证实,检出率为3.0%。其中2例检出HCoV-HKU1(FZ90, FZ96),检出率0.75%;还有1例检出HCoV-NL63,4例检出HCoV-OC43,1例检出HCoV-229E。检测出2例HCoV-HKU1与人副流感病毒3型(HPIV-3)混合感染。
2.2HCoV-HKU1阳性病例的临床症状 HCoV-HKU1感染患儿为一男一女,男婴4个半月大,诊断为支气管炎,连续咳嗽加剧;女婴5个月大,诊断为急性重症肺炎伴心衰。他们体温正常,都在3月底发病。
2.3序列比对和系统进化分析 根据pol基因RT-PCR产物序列测定与BLAST比对分析显示,在福州地区检测出的8株人冠状病毒中,2株为人冠状病毒HCoV-HKU1,序列比对表明福州的2株HCoV-HKU1pol基因片段碱基序列有4处不同。1株FZ90与北京BJ01-p9株(NO. KT779556)和香港N18基因型A株(NO. DQ415914)等的同源性为99%;另1株FZ96与北京BJ01-p9株和香港N18基因型A株等的同源性也为99%,但与泰国CU-H2809/2010株(NO. JX513214)的1abpol基因的同源性为100%[11]。用于确认HCoV-HKU1的950 bp的核酸片段含有部分M基因和N基因,福州2株HCoV-HKU1的950 bp的核酸片段序列完全相同。
2.4N基因进化分析 N蛋白基因序列全长为1 326个核苷酸, 编码441个氨基酸。序列比对表明福州的2株HCoV-HKU1 N基因片段碱基序列完全相同。BLAST比对分析显示,2株福州株与北京BJ01-p9株和香港N18基因型A株等的同源性都为100%。
将在福州地区检测出的2株HKU1 N基因核苷酸序列与基因库中HKU1的3种亚型的一些代表性毒株序列进行比对分析(图1),这些毒株包括香港(HK)、北京(BJ)、美国(USA)、法国(FR),系统进化分析采用MEGA6.06软件。图1显示2株FZ90和FZ96与HKU1的A基因型聚成一簇。
2.5S基因进化分析 S蛋白基因序列全长为4 071个核苷酸, 是编码1 356个氨基酸的糖蛋白。将检测出的S基因的3个片段进行拼接和序列分析。序列比对表明福州的2株HCoV-HKU1S基因片段碱基序列有2处不同,但所编码的氨基酸序列却完全相同。BLAST比对分析显示,这2株与北京BJ01-p9株和香港N18基因型A株等的同源性为99%。
将在福州地区检测出的2株HKU1S基因核苷酸序列与基因库中HKU1的3种亚型的一些代表性毒株序列进行比对分析。比对所采用的毒株比N基因多了3株巴西(BRA)的。图2显示2株FZ90和FZ96与HKU1的A基因型聚成一簇。
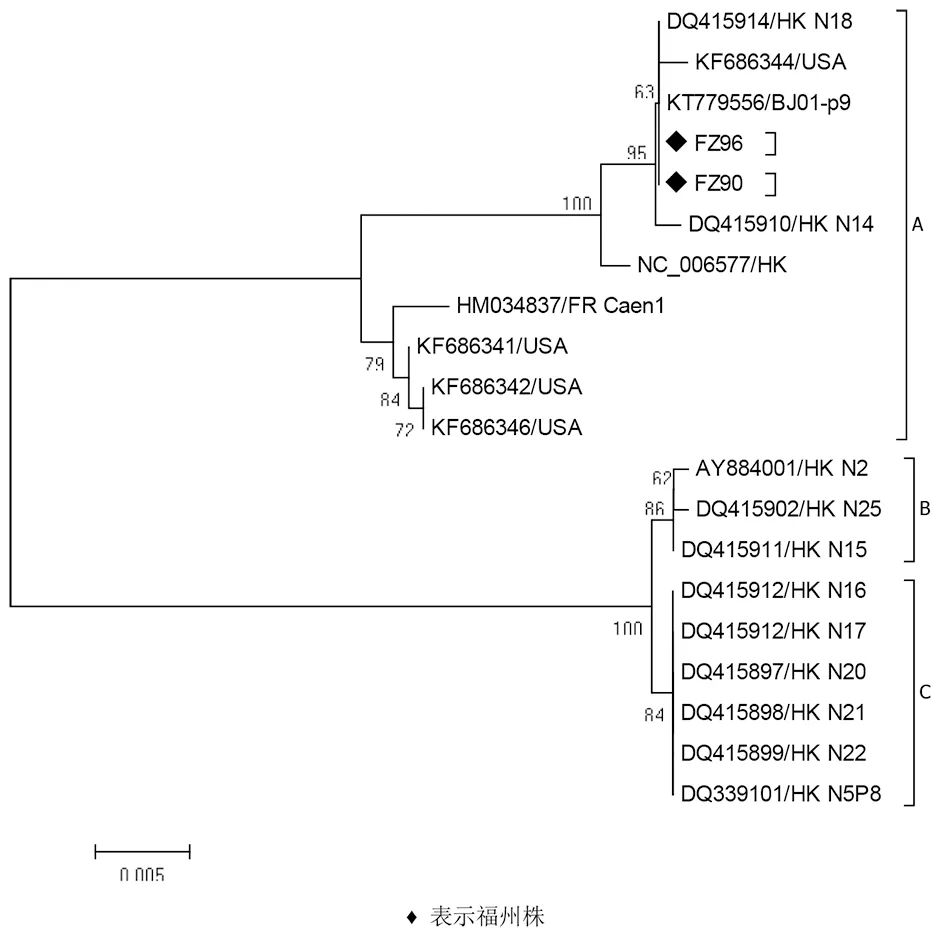
图2 福州2株HKU1 N基因系统进化分析Fig.2 Phylogenetic analysis of N genes in Fuzhou HKU1 strains

图3 福州2株HKU1 S基因系统进化分析Fig.3 Phylogenetic analysis of S genes in Fuzhou HKU1 strains
3 讨 论
HCoV-HKU1和同属β群的HCoV-OC43一样, 均为普通感冒病毒, 通常对人没有生命危险, 但在婴幼儿、老年人和免疫缺陷者可引起较严重的临床症状。目前只报道2例因HCoV-HKU1感染导致肺炎死亡病例[9],这2例都伴有基础疾病,尤其是呼吸系统和心血管系统疾病。香港同一研究表明, 7名感染HKU1 A基因型的病人都有基础疾病,而2名感染HKU1 B基因型的病人没有基础疾病。在法国, 6名HKU1感染者中有3名伴有基础疾病[6],HKU1的感染可能会加重患者基础疾病病情,导致其需要入院治疗。本研究发现2例HKU1感染患儿都伴有HPIV-3的混合感染,这可能加重了呼吸道感染的病情。美国学者的研究[14]表明59%HKU1阳性的科罗拉多呼吸道感染病人伴有其它病毒的混合感染,其中24%混合感染鼻病毒,20%混合感染呼吸道合胞病毒,11%混合感染腺病毒,7%混合感染HCoV-NL63。58%为2岁以下婴幼儿。
2012年,又出现一种新型HCoV—中东呼吸综合症(Middle East Respiratory Syndrome, MERS)病毒[15],使已知感染人的冠状病毒增加到了6种,MERS-CoV和SARS-CoV也归类为β类冠状病毒。它们的出现意味着在野生动物宿主中新的、对人类有毒性的HCoVs不断出现的可能。在2003年SARS出现之前,总共已知19种冠状病毒,包括2种人类,13种哺乳动物和4种禽类冠状病毒。SARS流行之后短短3年内,科学家们又发现了20种新的冠状病毒,包括3种人类,11种哺乳动物和6种禽类冠状病毒[10]。香港科学家曾在蝙蝠体内发现了8种不同种类的冠状病毒。由于冠状病毒存在跨种属传播机制,且是一类较为独特的、具有高频重组特点的RNA病毒[16],高重组和高突变率的倾向使其容易去适应新的宿主和生态小环境,并跨越宿主屏障,导致动物源性传染病的暴发并带来灾难性的后果。
从HCoV-HKU1的N基因和S基因的进化树分析可以看出福州的2株都属于HKU1的A基因型,与部分pol基因的进化树分析结果[11]一致。ORF(open reading frams)7编码N蛋白,冠状病毒的N蛋白是一种磷酸化蛋白,与病毒基因组的RNA相结合,可能对RNA的转录、病毒的形态和在基因组的包装中起重要的作用[17];同时,在大多数的冠状病毒中, 磷酸化的N蛋白可能对细胞周期起抑制作用[18]。ORF3编码预测的S糖蛋白[1],大多数S糖蛋白(残基15-1300)暴露在病毒的外部,C端跨膜区为残基1301-1356。S基因是基因组中最容易发生改变的地区之一,决定了病毒的进化枝(clade)。但在各亚型中,S基因序列是相对稳定的。病毒在选择压力的作用下, 通常在其表面蛋白上的某些位点发生糖基化,从而改变其空间结构。糖基化分析显示, 在S蛋白上有28个潜在的糖基化位点[13]。有研究认为在美国科罗拉多,HKU1亚型随着年份交替出现[16],福州这2株都出现在同一年份。临近的泰国[19],HKU1的检出率相近,为0.32%,3种基因型都有,多为基因型B;位于热带地区的马来西亚[20],HKU1的检出率为1.1%,其中27.3%属于基因型A,72.7%属于基因型B。北京地区A[21]、B[22]两种基因型都有检出;在韩国检出的11株中,3株为基因型A,8株为基因型B[23]。
显然,目前的研究证明HCoV-HKU1在世界各国人群中持续流行并引发感染,儿童感染多于成人,并且多与其它呼吸道病毒一起混合感染人类。我们下一步将对成人严重急性呼吸道感染(SARI)患者中HCoV-HKU1感染情况进行检测和分析,以便全面了解该病毒在本地区人群中的感染状况、流行特征和系统进化情况。
[1] Woo PCY, Lau SK, Chu CM, et al. Characterization and complete genome sequence of a novel coronavirus HKU1 from patients with pneumonia[J]. J Virol, 2005, 79: 884-895. DOI: 10.1128/JVI.79.2.884-895.2005
[2] Drosten C, Gunther S, Preiser W, et al. Identification of a novel coronavirus in patients with severe acute respiratory syndrome[J]. N Engl J Med, 2003, 348:1967-1976. DOI: 10.1056/NEJMoa030747
[3] Van der Hoek L, Pyrc K, Jebbink MF, et al. Identification of a new human coronavirus[J]. Nat Med, 2004, 10: 368-373. DOI: 10.1038/nm1024
[4] Sloots TP, McErlean P, Speicher DJ, et al. Evidence of human coronavirus HKU1 and human bocavirus in Australian children[J]. J Clin Virol, 2006, 35(1): 99-102. DOI: 10.1016/j.jev.2005.09.008
[5] Lau SK, Woo PC, Yip CC, et al. Coronavirus HKU1 and other coronavirus infections in Hong Kong[J]. J Clin Microbiol, 2006, 44:2063-2071. DOI: 10.1128/JCM.02614-05
[6] Vabret A, Dina J, Gouarin S, et al. Detection of the new human coronavirus HKU1: a report of 6 cases[J]. Clin Infect Dis, 2006, 42:634-639. DOI: 10.1086/500136
[7] Virus taxonomy, classification and nomenclature of viruses. Ninth report of the International Committee on Taxonomy of Viruses[Z]. San Diego, CA: Academic Press, 2012.
[8] Lai MC, Cavanagh D. The molecular biology of coronaviruses[J]. Adv virus Res, 1997, 48: 1-100.
[9] Woo PCY, Lau SKP, Tsoi HW, et al. Clinical and molecular epidemiological features of coronavirus HKU1-associated community-acquired pneumonia[J]. J Infect Dis, 2005, 192 (11): 1898-1907. DOI: 10.1086/497151
[10] Woo PCY, Lau SK, Yip CC, et al. Comparative analysis of 22 coronavirus HKU1 genomes reveals a novel genotype and evidence of natural recombination in coronavirus HKU1[J]. J Virol, 2006, 80 (14): 7136-7145. DOI: 10.1128/JVI.00509-06
[11] 修文琼, 郑奎城, 吴冰珊, 等. 福州地区重症呼吸道感染患儿中4种人冠状病毒的检测与分析[J]. 中华实验和临床病毒学杂志, 2017, 31(5): 429-433. DOI:10.3760/cma.j.issn.1003-9279.2017.05.011
[12] 陆柔剑, 张陵林, 谭文杰, 等. 人冠状病毒HCoV-NL63和HCoV-HKU1常规RT-PCR与实时荧光定量RT-PCR检测方法的建立及应用比较[J]. 病毒学报, 2008, 24(4):305-311. DOI:CNKI:SUN:BDXB.0.2008-04-013
[13] 段招军, 黄灿平, 瞿小旺, 等. 中国内陆发现HCoV-HKU1感染及N和S蛋白基因序列及进化分析[J]. 病毒学报, 2006, 22(4):241-247. DOI:10. 13242/j.enki. bingduxuebao. 001727
[14] Dominguez SR, Shrivastava S, Berglund A, et al. Isolation, propagation, genome analysis and epidemiology of HKU1 betacoronaviruses[J]. J Gen Virol, 2014, 95: 836-848. DOI: 10.1099/vir.0.059832-0
[15] Corman VM, Muller MA, Costabel U, et al. Assays for laboratory confirmation of novel human coronavirus (hCoV-EMC) infections[J]. Euro Surveill, 2012, 17(49): 1-9.
[16] Lai MM, Cavanagh D. The molecular biology of coronaviruses[J]. Adv Virus Res, 1997, 48: 1-100.
[17] 侯云德.分子病毒学[M].北京:学苑出版社, 1990: 381-394.
[18] Surjit M, Kumar R, Mishra RN .The severe acute respiratory syndrome coronavirus nucleocapsid protein is phosphorylated and localizes in the cytoplasm by 14-3-3-mediated translocation[J]. J Virol, 2005, 79(17): 11476-11486. DOI:10.1128/JVI.79.17.11476-11486.2005
[19] Soonnarong R, Thongpan I, Payungporn S, et al. Molecular epidemiology and characterization of human coronavirus in Thailand, 2012-2013[J]. Springer Plus, 2016, 5(1420): 1-8. DOI: 10.1186/s40064-016-3101-9
[20] AI-Khannaq MN, Ng KT, Oong XY, et al. Molecular epidemiology and evolutionary histories of human coronavirus OC43 and HKU1 among patients with upper tract infections in Kuala Lumpur, Malaysia[J]. Virol J, 2010, 13(33): 1-12. DOI: 10. 1186/ s12985-016-0488-4
[21] Cui LJ, Zhang C, Zhang T, et al. Human coronavirus HCoV-NL63 and HCoV-HKU1 in hospitalized children with acute respiratory infections in Beijing, China[J]. Adv Virol, 2011, 2011: 129134. DOI: 10.1155/2011/129134
[22] 段希洁, 陆柔剑, 俞晓燕, 等. 北京地区成人呼吸道感染患者中HCoV-HKU1感染的初步分析与基因型确定[J]. 生物技术通讯, 2013, 24(2):205-208. DOI:10.3969/j.issn.1009-0002.2013.02.014
[23] Lee WJ, Chung YS, Yoon HS, et al. Prevalence and molecular epidemiology of human coronavirus HKU1 in patients with acute respiratory illness[J]. J Med Virol, 2013, 85: 309-314.
