朗姆酒发酵过程中微生物群落结构及其动态演替
2018-06-08张苇莉杨慧敏胡景辉樊晓璐
张苇莉,杨慧敏,胡景辉,樊晓璐,邹 毅,李 楠*
(1.广西大学 生命科学与技术学院,广西 南宁 530004;2.广西大学 糖业工程研究中心,广西 南宁 530004)
朗姆酒是以甘蔗汁或甘蔗糖蜜为原料,经酒精发酵、蒸馏、陈酿、勾兑、过滤等一系列工序后得到的一种蒸馏型酒精饮品。其口感丰富,香醇芳郁,且有保健效果,深受人们喜爱[1-2]。朗姆酒的发酵是以酵母作为主发酵菌种,通过添加丹多液进行共发酵,丹多液中的多种微生物及甘蔗自带微生物发酵产生的代谢产物对酒体的影响和风味物质的形成具有重要意义,如乳酸杆菌在朗姆酒发酵过程中为酒体添香和风味物质形成提供前体物质[3-4],因此,了解发酵过程中微生物群落多样性的变化将有助于科学地认识朗姆酒酿酒微生物菌群代谢,对朗姆酒品质的提高具有重要意义[5]。传统微生物群落多样性主要通过分离培养技术、生物标记法和现代分子生物学免培养等技术获得[6-7]。进入21世纪,聚合酶链式反应(polymerase chain reaction,PCR)技术开始应用于白酒微生物群落结构的研究,但是该技术具有分辨率低(仅能检出少数优势种群)、条带回收困难、难以定量确定条带比例等缺点[8-9]。高通量测序技术是在近年来快速发展并广泛应用于多学科多领域的微生物群落多样性检测技术,如土壤[10]、湖泊[11]、淤泥[12]等生态环境,香肠[13]、米酒[14]、醋[15]等发酵食品,利用高通量测序技术不需要进行单菌落分离提取,便可直接在微生物基因水平上获取群落资源,该方法具有通量高,效率高,测序快,覆盖率高等优点[16],在保证测序深度和测序质量的条件下,可获得较为全面的微生物群落多样性评估信息[17]。
本研究主要通过高通量测序技术研究朗姆酒发酵中的微生物群落结构及其动态变化规律,确定发酵进程中各阶段的优势菌种,以期为朗姆酒生产与质量控制提供理论依据。
1 材料与方法
1.1 材料与试剂
朗姆酒发酵液:广西海酩威酿酒股份有限公司;土壤基因组提取试剂盒:美国MP Biomedicals公司;DNA聚合酶AP221-02、Trans DNA 15KMarker:北京全式金生物技术(TransGen Biotech)公司;产物回收试剂盒:天根生化科技(北京)有限公司。
1.2 仪器与设备
JulaboTW12恒温水浴锅:优莱博技术有限公司;Nano Drop 2000紫外可见分光光度计、ST16R高速冷冻离心机、PICO17小型台式离心机:赛默飞世尔科技(中国)有限公司;GeneAmpR9700型PCR仪:美国ABI公司;QuantiFluorTM-ST DNA定量仪:美国Promega公司;Illumina Miseq高通量测序仪:美国Illumina公司。
1.3 方法
1.3.1 样品采集
分别取发酵第1天、2天、3天、4天、5天的朗姆酒发酵液(记为样品A1、A2、A3、A4、A5),取样方法为5点取样法,从发酵罐(取样深度80 cm左右)四角及中心处取样品,在无菌条件下混合均匀后,后冷冻保存于-80℃。
1.3.2 样品总DNA提取、PCR扩增及检测
将朗姆酒发酵液样品送至上海美吉生物医药科技有限公司,由美吉公司采用土壤基因组提取试剂盒提取总DNA,具体操作方法按照试剂盒内说明书操作。先进行细菌和真菌特异区域的PCR扩增。16S rDNA V3-V4区的测序引物为338F(5'-ACTCCTACGGGAGGCAGCAG-3')和806R(5'-GGACTACHVGGGTWTCTAAT-3')[18-19];ITS1区的引物为ITS1F(5'-CTTGGTCATTTAGAGGAAGTAA-3')和ITS2(5'-GCTGCGTTCTTCATCGATGC-3')[20]。将全部实验样本进行重复试验。用凝胶回收试剂盒回收PCR产物,用Tris-HCl洗脱,并将PCR产物用DNA荧光定量系统进行检测定量。1.3.3测序文库构建、测序与拼接
构建Miseq文库时,需通过PCR将Illumina官方接头序列添加至目标区域外端;后使用凝胶回收试剂盒切胶将PCR产物进行回收;然后用Tris-HCl缓冲液来洗脱产物,并用2%的琼脂糖凝胶电泳进行检测;最后利用氢氧化钠对PCR产物变性的原理,产生单链DNA片段。利用Illumina Miseq高通量测序仪进行双端测序(paired-end sequencing)。将Miseq测序得到的PE reads根据overlap关系进行拼接,同时对序列质量进行质控和过滤,得到样品的有效序列,校正序列方向,使用FLASH和Trimmomatic软件得到优化序列[21]。1.3.4数据分析
将测序结果在美吉公司的I-sangerz在线生物信息分析平台上进行分析。样品标记区分后,采用Usearch软件进行操作分类单元(operational taxonomic unit,OTU)聚类分析。对于相似性水平≥97%的OTU进行生物信息统计,其中每个OTU代表一个物种[22]。基于OTU聚类分析和各个分类水平下物种的比对结果,利用Qiime软件对OTU进行多样性指数分析,以及对测序深度进行检测[23];基于分类学信息,在各个分类水平上进行群落结构的统计分析。即在各个分类水平上统计各样品微生物群落组成。其中,细菌16SrDNA V3-V4区采用Sliva细菌数据库比对;真菌ITS1区采用Unite真菌数据库比对。同时采用Qiime软件和RDP Classifier算法,选择置信度阈值≥0.9,进行物种注释和丰度分析[24]。为评估微生物分类与环境变量之间的相关性,基于分类学信息的基础上利用Qiime软件对微生物群落和环境因子的相关性进行Mantel text分析[25-26]。
Rank-abundance曲线是分析多样性的一种方式。构建方法是统计单一样品中,每一个OTU所含的序列数,将OTUs按丰度(所含有的序列条数)由大到小等级排序,再以OTU等级为横坐标,以每个OTU中所含的序列数(也可用OTU中序列数的相对百分含量)为纵坐标,使用97%相似度的OTU,利用R语言工具制作曲线图。Rank-abundance曲线可用来解释多样性的两个方面,即物种丰度和物种均匀度。在水平方向,物种的丰度由曲线的宽度来反映,物种的丰度越高,曲线在横轴上的范围越大;曲线平滑程度反映了样品中物种的均匀度,曲线越平缓,物种分布越均匀[27]。
2 结果与分析
2.1 测序数据统计及Alpha多样性统计
2.1.1细菌多样性分析
对5个样品的细菌群落进行测序分析,共得到187063条有效序列,样品的细菌群落多样性指数见表1。
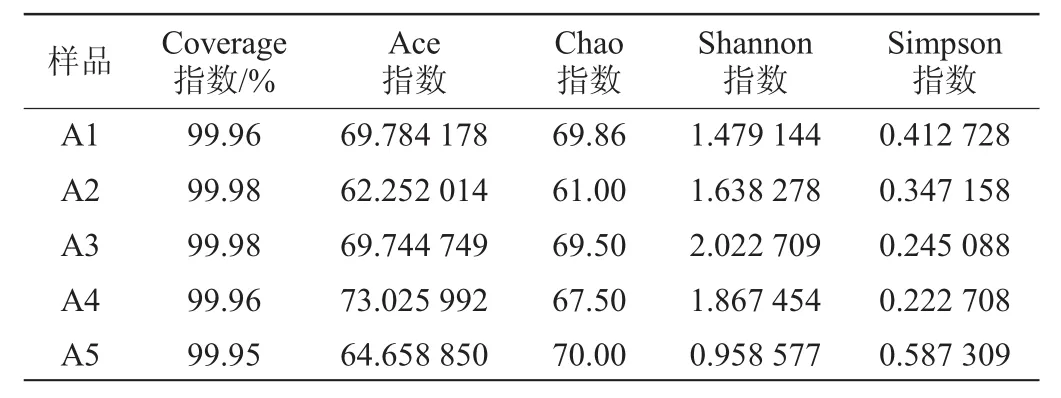
表1 细菌多样性指数分析Table 1 Index analysis of bacterial diversity
Coverage指数指各样本文库的覆盖率,反映测序结果是否代表了样本中微生物的真实情况,其数值越高,表示样本中序列被测出的概率越高;Chao指数和Ace指数用来估计样本中物种丰富度,Chao指数越大,表示总物种种类越多,Ace指数越大,表明样本的真实物种种类越多;Shannon指数和Simpson指数是物种均匀度和丰富度的综合指标,用来估算样本中群落多样性,Shannon指数越大,Simpson指数越小,表明群落多样性越高。由表1可知,样品的Coverage
指数均>99.9%,表明测序深度已覆盖到测试样品中的大部分物种,可以真实展示样品中的绝大数细菌;Chao指数和Ace指数总体上呈现先减小后增大的趋势,第2天最小,表明此时发酵液中细菌物种种类最少,第5天最大,表明此时发酵液中细菌物种种类最丰富;Shannon指数先增大后减小,Simpson指数先减小后增大,发酵第3天时,Shannon指数最大,发酵第3天和第4天时,Simpson指数小于第1、2、5天,表明此时发酵液中细菌群落多样性最大,第5天时,Shannon指数最小,Simpson指数最大,表明此时发酵液中细菌群落多样性最小。
2.1.2 样品细菌群落的Rank-abundance曲线
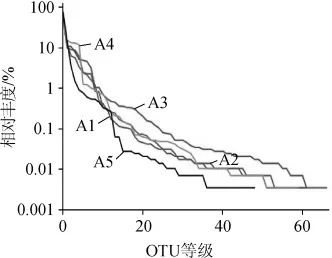
图1 细菌群落的Rank-abundance曲线Fig.1 Rank-abundance curves of bacterial community
图1 为朗姆酒发酵过程中5个阶段细菌的Rank-abundance曲线。由图1可知,细菌物种相对丰度:第3天>第4天>第2天>第5天>第1天;物种均匀度:第3天>第4天>第2天>第1天>第5天,第1天的细菌相对丰度最小,表明此时细菌物种种类最少,第5天均匀度最小,表明此时发酵液中各物种所占比例差异较大,第3天的朗姆酒发酵液中物种丰富度和均匀度均最大,表明此时发酵液中细菌种类最多,且各物种所占比例相似,群落多样性最大。
结合表1和图1可知,第1天开始,细菌群落多样性增加,这是因为起始阶段细菌大多来源于甘蔗表皮带入的天然微生物,这些细菌在适应新生境过程中,出现了种类和构成的急剧变化,第3天,丹多液的添加带来了大量的细菌,使得此时发酵液中细菌物种最丰富,群落多样性最大。2.1.3真菌的多样性指数分析
对5个样品的真菌群落进行测序分析,共得到168185条有效序列,样品的真菌群落多样性指数见表2。
由表2可知,样品的Coverage指数均>99.9%,可以真实展示样品中的绝大多数真菌;第1天时,Ace指数和Chao指数最大,表明此时真菌的物种种类最为丰富;第2天稍有减少,第3、4、5天,Ace指数和Chao指数出现了较大幅度的降低,表明此阶段真菌种类大幅减少,这可能是由于发酵液中微生物产生的代谢产物改变了发酵液的理化特性,不适宜真菌的生长,从而降低了真菌的多样性;第5天Ace指数和Chao指数最小,此时发酵液中真菌种类最少;第2天,Shannon指数最大,Simpson指数最小,说明此时发酵液中真菌群落多样性最大,第3、4、5天,Shannon指数大幅度降低,Simpson指数大幅度增加,表明此阶段真菌群落多样性大幅减少。
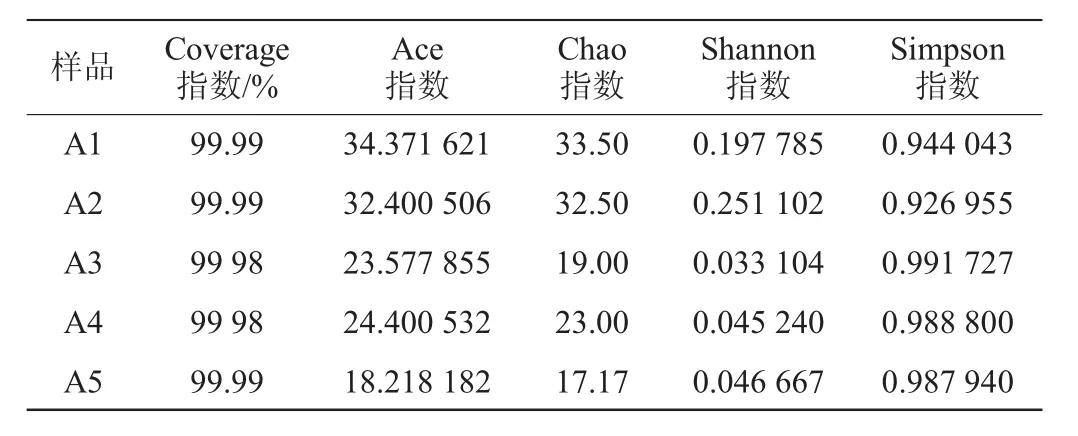
表2 在97%相似水平下真菌多样性指数分析Table 2 Index analysis of fungal diversity at 97%similar level
2.1.4 真菌的Rank-abundance曲线
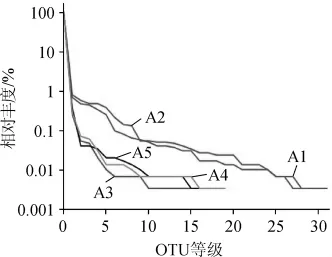
图2 真菌群落的Rank-abundance曲线Fig.2 Rank-abundance curves of fungal community
图2 为朗姆酒发酵过程中5个阶段真菌的Rank-abundance曲线,由图2可知,真菌物种相对丰度:第1天>第2天>第4天>第3天>第5天,物种均匀度:第2天>第1天>第4天>第5天>第3天,结合表2和图2可知,第1天真菌种类最多,但此时的群落多样性不是最大,这是由于各真菌物种所占比例差异较大,物种均匀度小,第2天真菌种类相较第1天有所减少,但此时的群落多样性最大,这是因为此时物种均匀度大,各真菌物种所占比例相似,第3天以后真菌物种丰富度和均匀度大幅降低。
结合细菌多样性和真菌多样性结果可知,细菌的Chao指数和Shannon指数均大于真菌,表明在朗姆酒发酵过程中细菌种类和群落多样性始终大于真菌。
2.2 朗姆酒发酵期间菌群群落结构变化分析
2.2.1 朗姆酒发酵期间细菌群落结构变化分析
采用RDP classifier对各样品中的OTU进行细菌门和属水平分析,获得7个门,包括放线菌门(Actinobacteria)、梭杆菌门(Fusobacteria)、拟杆菌门(Bacteroidetes)、变形菌门(Proteobacteria)、厚壁菌门(Firmicutes)、蓝细菌门(Cyanobacteria)和其他(others)相对丰度较小的门细菌;17个属,包括乳酸杆菌属(Lactobacillus)、酵单胞菌属(Zymomonas)、明串珠菌属(Leuconostoc)、泛菌属(Pantoea)、假单胞菌属(Pseudomonas)、普雷沃菌属(Prevotella)等。
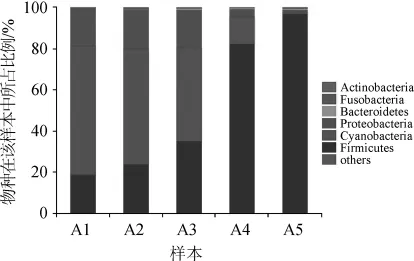
图3 门水平上细菌样本群落组成分析Fig.3 Composition analysis of bacterial community at the phylum level
门水平上的细菌群落结构如图3所示。由图3可知,发酵第1、2、3天朗姆酒发酵液中优势菌门为蓝细菌门(Cyanobacteria),次优势菌门为厚壁菌门(Firmicutes)、变形菌门(Proteobacteria),随着发酵的进行蓝细菌门(Cyanobacteria)的细菌比例逐渐减少,厚壁菌门(Firmicutes)的细菌比例逐渐增多,第4天开始,厚壁菌门(Firmicutes)继续增加迅速成为优势细菌,到发酵第5天比例达到最大值,在发酵液中比例为96.93%。
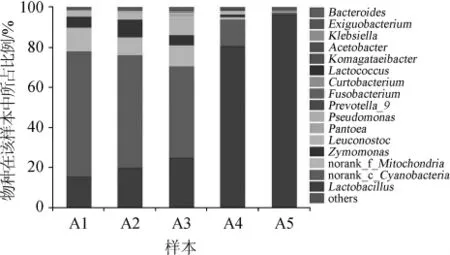
图4 属水平上细菌样本的群落组成分析Fig.4 Composition analysis of bacterial community at the genus level
属水平上的细菌群落结构如图4所示。由图4可知,发酵第1、2、3天,朗姆酒发酵液中的优势菌属为未分类的蓝细菌属(unclassified-Cyanobacteria),其次为乳酸杆菌属(Lactobacillus)、酵单胞菌属(Zymomonas)、明串珠菌属(Leuconostoc)、泛菌属(Pantoea)等。第1天,蓝细菌属在发酵液中所占比例最大,这可能是因为蓝细菌能较快的适应新环境,随后其比例开始逐步减小,但仍处于优势地位,而乳酸杆菌属所占比例从第1天开始逐步增加,第4天乳酸杆菌属取代蓝细菌属成为发酵液中优势菌属,且在发酵第5天所占比例达到最大值96.51%,这一现象现在泸型酒发酵过程中也同样存在,栗连会[28]的研究中,对泸型酒酒醅中乳酸菌群落进行功能预测并模拟发酵验证,发现乳酸菌在碳水化合物代谢中参与最多的是糖酵解途径,还具有合成丁酸、丙酸代谢的潜力。张艳[29]的研究也发现,通过乳酸菌与酵母的相互作用,乳酸菌可通过影响酵母菌的生长和代谢来调节酿造微生物区系的平衡和白酒呈味物质的产生,对酱香基酒的品质产生一定影响。结合朗姆酒生产工艺,朗姆酒在发酵过程中,乳酸杆菌属(Lactobacillus)通过发酵产生乳酸,并与酒精反应生成乳酸乙酯,乳酸乙酯与酵母菌发酵得到乙酸乙酯混合,这对朗姆酒特殊风味的形成具有重要意义。
2.2.2 朗姆酒发酵期间真菌群落结构变化分析
采用RDPclassifier对各样品中的OTU依次进行真菌门和属水平的信息分析,共获得4个门,包括子囊菌门(Ascomycota)、担子菌门(Basidiomycota)和未分类真菌(unclassi fied Fungi)和其他门真菌;11个属,主要有未分类的酵母菌属(unclassifiedSaccharomyces)、枝氯霉属(Ramichloridium)、隐球菌属(Cryptococcus)、镰刀霉属(Fusarium)、间座壳属(Diaporthe)等。
样品在门水平上的真菌群落结构如图5所示,由图5可知,子囊菌门(Ascomycota)在整个发酵过程中始终处于优势地位,第1天,其在发酵液中所占比例最小为99.47%,随后一直增加,第3天比例达到100%,第1、2天发酵液中有少量的担子菌门(Basidiomycota)一些未分类真菌以及其他门真菌,第3天以后,整个发酵液中全部为子囊菌门真菌。
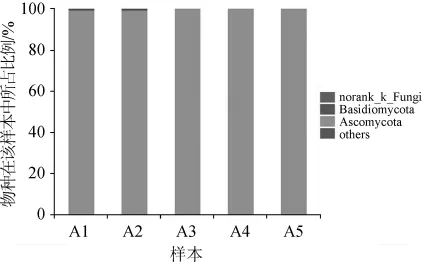
图5 门水平上真菌样本的群落组成分析Fig.5 Composition analysis of fungal community at the phylum level
样品在属水平的真菌群落组成如图6所示,由图6可知,第1天,发酵液中的优势真菌为酵母属,其所占比例>90%,其次为枝氯霉属(Ramichloridium)、隐球菌属(Cryptococcus)、镰刀霉属(Fusarium)、间座壳属(Diaporthe),随后酵母属在发酵液中的比例逐步增加,并一直处于优势地位,第5天比例最高,为99.77%,而其他真菌属比例逐步减小,第3天以后发酵液中只剩少量的镰刀霉属(Fusarium)、隐球菌属(Cryptococcus)和大量的酵母属(Saccharomyces)。
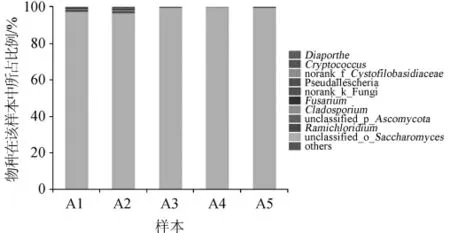
图6 属水平上真菌样本的群落组成分析Fig.6 Composition analysis of fungal community at the genus level
2.3 样品的Beta多样性分析
2.3.1 细菌属水平的PCA主成分分析
对发酵过程中5个样本的细菌进行属水平上的主成分分析(principal component analysis,PCA)分析,结果如图7所示。由图7可知,发酵第1天和第2天样品的距离较近,第4天和第5天样品的距离较近,表明这两个阶段细菌属组成相似度高,第3天和第5天距离最远,表明这一阶段细菌属多样性大,这是因为添加丹多液带来了大量的细菌,使得此时发酵液中细菌物种和种类极为丰富,组成最为复杂。

图7 属水平上细菌样本的PCA分析Fig.7 Principal component analysis of bacteria samples at the genus level
2.3.2 真菌属水平的PCA主成分分析
对发酵过程中5个样本的真菌进行属水平上的PCA分析,如图8所示。由图8可知,第1、2、3天样品间距离远且分散,表明此阶段发酵液中真菌组成相似度低,群落多样性大,第3、4、5天样品距离近且集中,表明此阶段发酵液中真菌组成相似度高,群落多样性小。这是因为第3天后发酵液中其他属的真菌逐渐消失,发酵液中主要真菌为酵母属。
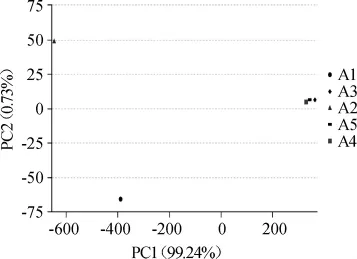
图8 属水平上真菌样本的PCA分析Fig.8 Principal component analysis of fungal samples at the genus level
2.4 样品的环境因子分析
2.4.1 OUT水平上细菌的Mantel test分析
为得到样品细菌群落与环境因子相关性,利用Qiime软件对细菌群落和各环境因子进行OUT水平上的Mantel text分析,结果如表3所示。
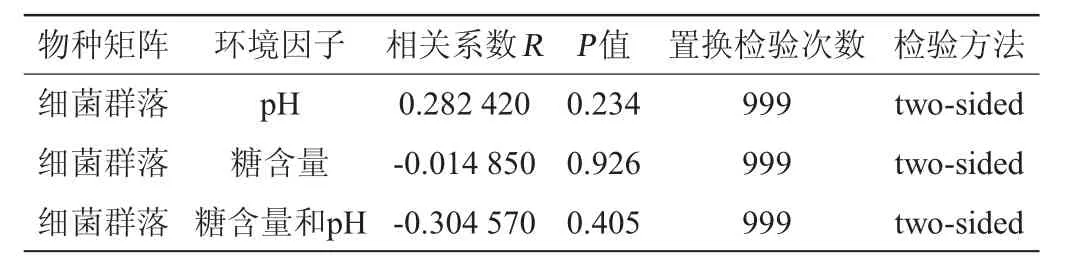
表3 OUT水平上细菌的Mantel test分析Table 3 Mantel test analysis of bacteria at the OUT level
Mantel text可用来分析微生物群落与环境因子的相关性,R值越接近1,表明微生物群落与环境因子越正相关,越接近-1表明两者越负相关,0表示两者不相关。
由表3可知,以OUT为分类水平,在置换检验次数为999次的情况下,pH值与细菌群落多样性呈正相关,糖含量、糖含量和pH与细菌群落多样性呈负相关,且pH值(P>0.05)、糖含量、糖含量和pH值对细菌群落多样性影响均不显著,但三种环境因子中,pH值对细菌群落多样性的显著性水平最高,因此是影响细菌群落多样性的主要因素。
2.4.2 OUT水平上真菌的Mantel test分析
为得到样品真菌群落与环境因子相关性,利用Qiime软件对真菌群落和各环境因子进行OUT水平上的Manteltext,结果如表4所示。
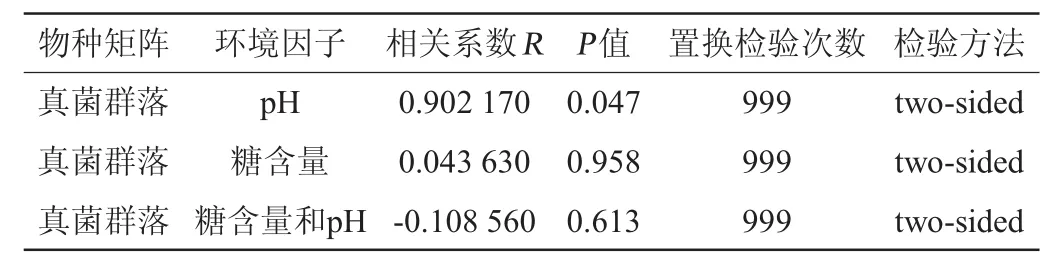
表4 OTU水平上真菌的Mantel test分析Table 4 Mantel test analysis of fungal at the OUT level
由表4可知,以OUT为分类水平,在置换检验次数为999次的情况下,pH值与真菌群落多样性呈正相关,且pH值对真菌群落多样性有显著的影响(P<0.05),糖含量、糖含量和pH值对真菌群落多样性影响均不显著(P>0.05)。
3 结论
本研究采用高通量测序方法系统鉴定了朗姆酒发酵过程中细菌和真菌微生物菌群多样性,共鉴定得到到53个细菌菌属和25个真菌菌属。高通量测序结果表明,朗姆酒发酵液中细菌群落多样性始终大于真菌,且细菌群落多样性在第3天添加丹多液后达到最大,此阶段中主要优势细菌为未分类的蓝细菌属(unclassified-Cyanobacteria),第5天的群落多样性最小,此阶段主要优势细菌为厚壁菌门的乳酸杆菌属(Lactobacillus);真菌群落多样性在第2天最大,第3天后真菌群落多样性一直减小,且其物种组成相似度高,整个发酵过程中,酵母菌始终属于优势真菌。通过Manteltext分析微生物群落与环境因子间的相关性,结果显示,在OUT水平上,三种环境因子中,影响细菌群落多样性和真菌群落多样性的主要是pH值,且真菌群落多样性与pH值之间具有显著正相关(P<0.05)。
本试验为研究微生物对朗姆酒风味物质的形成提供理论依据,对后续优势功能菌的分离筛选以及人工接种风味菌以提高朗姆酒质量奠定了坚实的基础。
[1]曾文生.朗姆酒工艺技术的探讨[J].轻工科技,2016,32(2):16-18.
[2]鲁 龙,张惟广.朗姆酒的香气与品质[J].酿酒科技,2013(11):104-108.
[3]邹 毅.低浓度糖浆生产重质朗姆酒的方法[P].CN 102952665A,2013-03-06.
[4]FASHRASMANE L,PARFART A,JOURET C,et al.Production of higher alcohols and short chain fatty acids by different yeast used in Rum fermentation[J].J Food Sci,1985(50):1427-1430,1436.
[5]侯小歌,土俊英,李学思,等.浓香型自酒糟酷及窖泥产香功能菌的研究进展[J].微生物学通报,2013,40(7):1257-1265.
[6]蔡雪梅,蒋英丽,吴联海,等.不同区域酱香型白酒人工窖底泥细菌多样性及其影响因子[J].食品科技,2017,38(10):87-96.
[7]陶 勇.浓香型曲酒窖池的微生物群落演替及其影响因子[C].大连:全国环境微生物学术研讨会,2012.
[8]ZHENG J,LIANGR,ZHANG L Q,et a1.Characterization of microbial communities in strong aromatic liquor fermentation pit muds of different ages assessed by combined DGGE and PLF analyses[J].Food Res Int,2013,54(1):660-666.
[9]ZHENG J,WU C,HUANG J,et a1.Spatial distribution of bacterial communities and related biochemical properties in Luzhou-flavor liquor fermented grains[J].J Food Sci,2014,79(12):M2491-M2498.
[10]ZHOU X,LI S,LI W,et al.Myxobacterial community is a predominant and highly diverse bacterial group in soil niches[J].Environ Microbiol Rep,2014,6(1):45-56.
[11]LI S,ZHOU X,LI P,et al.The existence and diversity of myxobacteria in lake mud-a previously unexplored myxobacteria habitat[J].Environ Microbiol Rep,2012,4(6):587-595.
[12]POLILA J,REBECCHI A,PISACANE V,et al.Bacterial diversity in typical Italian salami at different ripening stages as revealed by highthroughput sequencing of 16S rRNA amplicons[J].Food Microbiol,2015,46:342-356.
[13]WANG P,MAO J,MENG X,et al.Changes in flavour characteristics and bacterial diversity during the traditional fermentation of Chinese rice wines from Shaoxing region[J].Food Control,2014,44(44):58-63.
[14]吴红萍,陈喜蓉,甫秋燕,等.Biolog-ECO解析长裙竹荪栽培种覆土层土壤微生物群落功能多样性特征[J].基因组学与应用生物学,2017,36(10):4223-4228.
[15]METZGERJ,TONDA R,BELTRAN S,et al.Next generation sequencing gives an insight into the characteristics of highly selected breeds versus non-breed horses in the course of domestication[J].BMC Genom,2014,15(1):1-13.
[16]秦 楠,来东芳,杨瑞馥.高通量测序技术及其在微生物学研究中的应用[J].微生物学报,2011,54(4):445-457
[17]罗 雯,张倩颖,廖作敏,等.基于高通量测序技术的不同性状窖泥微生物组成研究[J].食品与发酵工业,2017,43(9):9-14.
[18]WALTER J,TANNOCK G W,TILSARA A,et al.Detection and identification of gastrointestinal lactobacillus species by using denaturing gradient gel electrophoresis and species-specific PCR primers[J].Appl Environ Microbiol,2000,66(1):297-303.
[19]YU Y,LEE C,KIM J,et al.Group-specific primer and probe sets to detect methanogenic communities using quantitative real-time polymerase chain reaction[J].Biotechnol Bioeng,2005,89(6):670-679.
[20]GARDES M,BRUNS T D.ITS primers with enhanced specificity for basidiomycetes-application to the identification of mycorrhizae and rusts[J].Mol Ecol,1993,2(2):113-118.
[21]何雨琦,李桂梅,周 鑫,等.用于Illumina测序平台不同试剂盒制备RNA-seq文库的方法比较[J].基因组学与应用生物学,2017,36(11):4607-4615.
[22]EDGAR R.Highly accurate OTU sequences from microbial amplicon reads[J].Nat Method,2013,10(10):996-998.
[23]PAUL F K,JOSEP Y A.Bacterial diversity in aquatic and other environments:what 16S rDNA libraries can tell us[J].FEMS Microbiol Ecol,2004,47(2):161-177.
[24]WANG Q,GATTITY G M,TIEDJE J M,et al.Naïve bayesian classifier for rapid assignment ofrRNAsequences into the new bacterial taxonomy[J].Appl Envirom Microbiol,2007,73(16):5261-5267.
[25]SHORT R A,CUSHMAN S A,MACE R,et al.Why replication is important in landscape genetic:American black bear in the Rocky Mountains[J].Mol Ecol,2011,20(6):1092-1107.
[26]GRAVES T A,BEIER P,ROYLE A J.Current approaches using genetic;distances produce poor estimates of landscape resistance to inter individual dispersal[J].Mol Ecol,2013,22(15):3888-3903.
[27]BATES S T,CLEMENTE J C,FLORES G E,et al.Global biogeographyof highlydiverse protistan communities in soil[J].ISME J,2013(7):652-659.
[28]栗连会.泸型酒酒醅中乳酸菌和乳酸降解菌的多样性和代谢特性[D].无锡:江南大学,2016.
[29]张 艳.酱香型白酒发酵中乳酸菌群结构及功能研究[D].无锡:江南大学,2015.
