甲基化状态风险分析对胶质母细胞瘤患者预后评价意义
2018-05-30康恩铭章薇章翔
康恩铭 章薇 章翔
(空军军医大学西京医院神经外科,陕西 西安 710032)
胶质母细胞瘤(glioblastoma multiforme, GBM)是最常见的原发性中枢神经系统恶性肿瘤,其患者的中位生存期仅有14个月左右[1]。DNA序列中胞嘧啶-磷酸-鸟嘌呤(cytosine-phosphate-guanine, CpG)双核苷酸的甲基化,可以通过影响转录因子的结合活性、改变基因稳定性、重塑基因结构从而对基因表达产生明显的影响。目前对DNA甲基化研究最多的是启动子内CpG岛(CpG island, CGI)甲基化。然而,只有大约70%的基因包含启动子CGI,并且只有6.8%的CpG双核苷酸位点在CGI中。GCI shores位于传统CGI两侧2 kb范围内区域,其CpG密度较GCI相对较低。有研究证实,组织和肿瘤特异性的DNA差异甲基化在CGI shores区域发生更频繁,而DNA甲基化也可以直接通过影响非CGI启动子区域来改变肿瘤细胞的甲基化模式[2]。目前,CGI shores甲基化状态与GBM患者预后的关系尚不明确。在本研究中,我们利用在线GBM数据库,建立并验证了与GBM患者临床预后密切相关的基于7个基因(AP3B1, RGS16, PPM1M, ENPP4, GOLGB1, CACNA2D2, TMEM16)的启动子所在GCI shores甲基化状态的风险评分方程,为进一步明确GBM甲基化改变模式以及探索GBM发病机制提供了有力依据。
对象与方法
一、GBM病例全基因组甲基化数据库
GBM病例全基因组甲基化芯片数据和对应的临床数据均通过以下在线数据库获得:癌症和肿瘤基因图谱计划数据库(The Cancer Genome Atlas, TCGA),数据库网址:https://cancergenome.nih.gov/。基因表达公共数据库(Gene Experssion Omnibus, GEO)中的GSE36278系列和GSE60274系列,数据库网址https://www.ncbi.nlm.nih.gov/geo/。
本研究选取成人GBM病例的Illumina Human甲基化450 k芯片测量数据,排除生存资料不全以及OS<7 d的非死亡病例,最后纳入TCGA病例组:136例,GSE36278病例组:37例,GSE60274病例组:62例。
二、甲基化芯片数据的处理
我们将各数据库提供的所有235例甲基化表达芯片(Illumina Human 450 k)样本的beta值数据转化为M值。随后根据以下标准对探针进行筛选:去除包含SNP位点的探针和去除没有UCSC位点信息的探针。在筛选后的探针中,我们选取了启动子相关的CGI shores探针进行研究(n=16 792)。
三、统计学分析
应用生物计量研究分支测量(Biometric Research Branch-Array, BRB)软件以及SPSS 19.0软件进行统计分析。甲基化探针的M值和病例组生存之间关系采用置换检验(permutation test)和单因Cox回归模型分析法。置换检验根据10 000次随机置换计算,并选取P<0.001做为显著性统计学意义的标准。筛选后的位点应用多元Cox回归模型分析,以此建立风险评分方程。我们使用实验组的中位风险评分值M将各病例组患者分成高风险阻和低风险组,随后采用Kaplan-Meier检验对高风险组、低风险组分别进行生存期分析。
结 果
一、GBM病例的一般资料
本研究共纳入235例来自TCGA数据库(136例),GEO数据库(99例,其中GSE60274系列 62例及GSE36278系列37例)的成人GBM病例,其中男144例,女91例;235例患者的平均年龄为(55.2±13.1)岁。各数据库病例的人口学特征及临床资料详见表1。
表1 所选GBM数据库的患者临床资料特征
Tab 1 Patients' characteristics of included GBM datasets
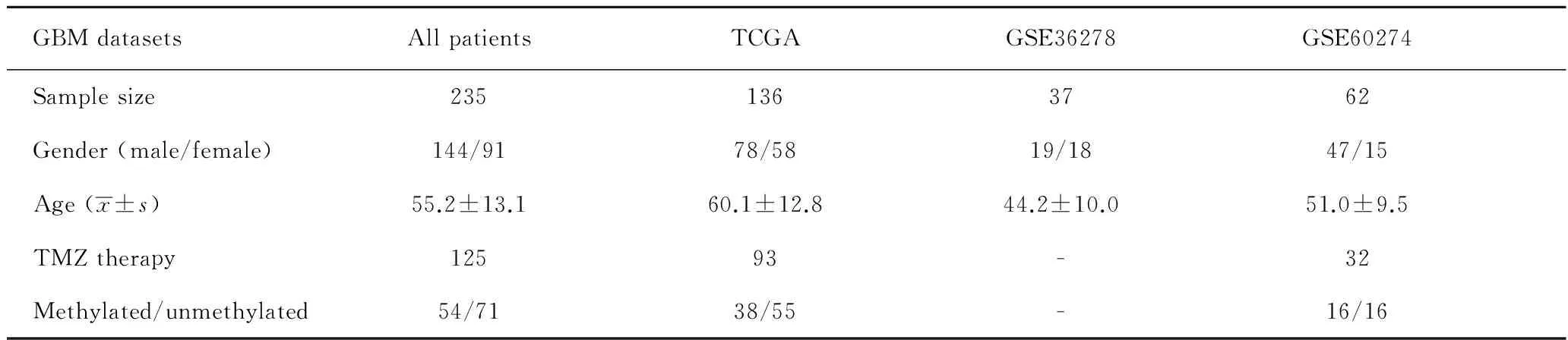
GBMdatasetsAllpatientsTCGAGSE36278GSE60274 Samplesize2351363762 Gender(male/female)144/9178/5819/1847/15 Age(x±s)55.2±13.160.1±12.844.2±10.051.0±9.5 TMZtherapy12593-32 Methylated/unmethylated54/7138/55-16/16
GBM: glioblastoma; TMZ: temozolomide.
二、生存相关CGI shores探针筛选
由于GSE36278病例组样本量较小,我们将TCGA病例组(n=136)和GSE36278病例组(n=37)合并为实验组(n=173)。通过使用BRB软件,对实验组的GCI shores位点甲基化M值进行单因Cox回归模型分析和置换检验。根据P<0.001标准,在16 792个CGI shores探针中得到112个与生存密切相关的探针。随后在实验组中对这112个探针的甲基化M值进行多元Cox回归模型分析,根据P<0.01标准,最后得到7个与生期相关的探针位点(表2)。
表2 与实验组病例生存相关的GCI shores探针
Tab 2 GCI shores probes were significantly associated with the overall survival in the training-set patients
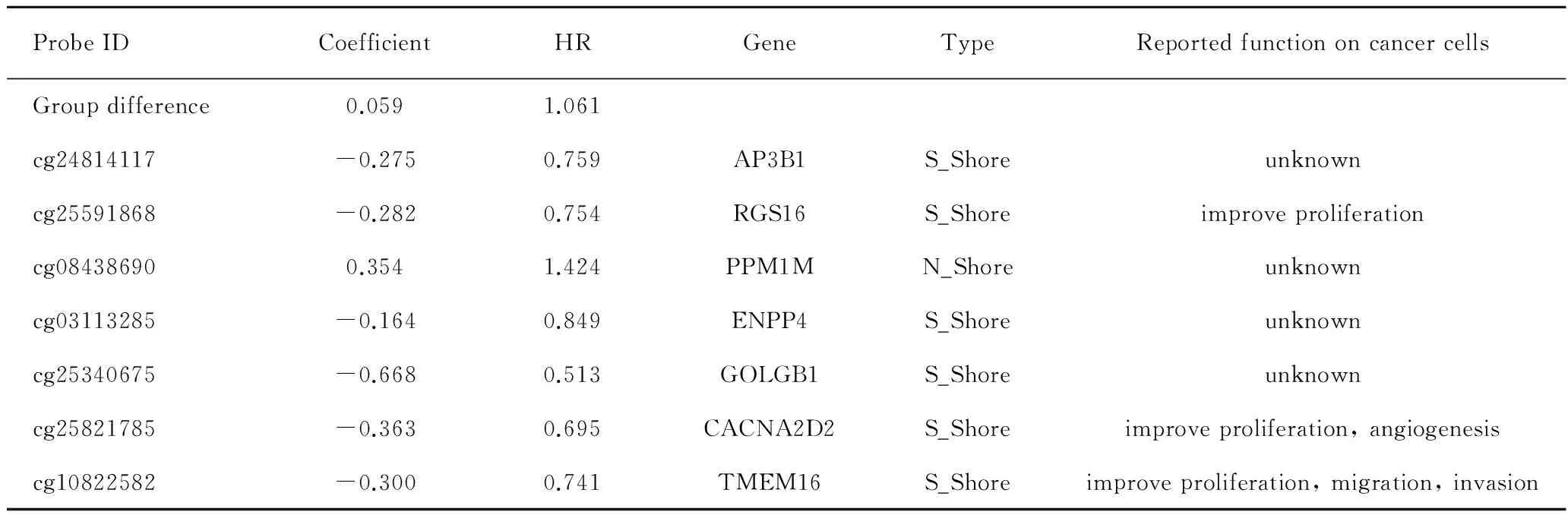
ProbeIDCoefficientHRGeneTypeReportedfunctiononcancercells Groupdifference0.0591.061 cg24814117-0.2750.759AP3B1S_Shoreunknown cg25591868-0.2820.754RGS16S_Shoreimproveproliferation cg084386900.3541.424PPM1MN_Shoreunknown cg03113285-0.1640.849ENPP4S_Shoreunknown cg25340675-0.6680.513GOLGB1S_Shoreunknown cg25821785-0.3630.695CACNA2D2S_Shoreimproveproliferation,angiogenesis cg10822582-0.3000.741TMEM16S_Shoreimproveproliferation,migration,invasion
HR: harzad ratio.
三、基于7-CGI shores甲基化探针M值风险评分方程的建立
根据cox结果的回归系数建立了基于这7个探针位点的风险评分方程:Risk score=(-0.275 M value of cg24814117)+(-0.282 M value of cg25591868)+(0.354 M value of cg08438690)+(-0.164 M value of cg03113285)+(-0.668 M value of cg25340675)+(-0.363 M value of cg25821785)+(-0.300 M value of cg10822582)。实验组病例根据风险评分的中位数的风险值(M=4.4)分为低风险组(n=86)和高风险组(n=87)。高风险组的生存时间(中位OS:8.5个月,95% CI:5.7~11.3月)明显短于低风险组(中位OS:16.2个月;95% CI:12.6~19.7个月),χ2=41.365,P<0.001。
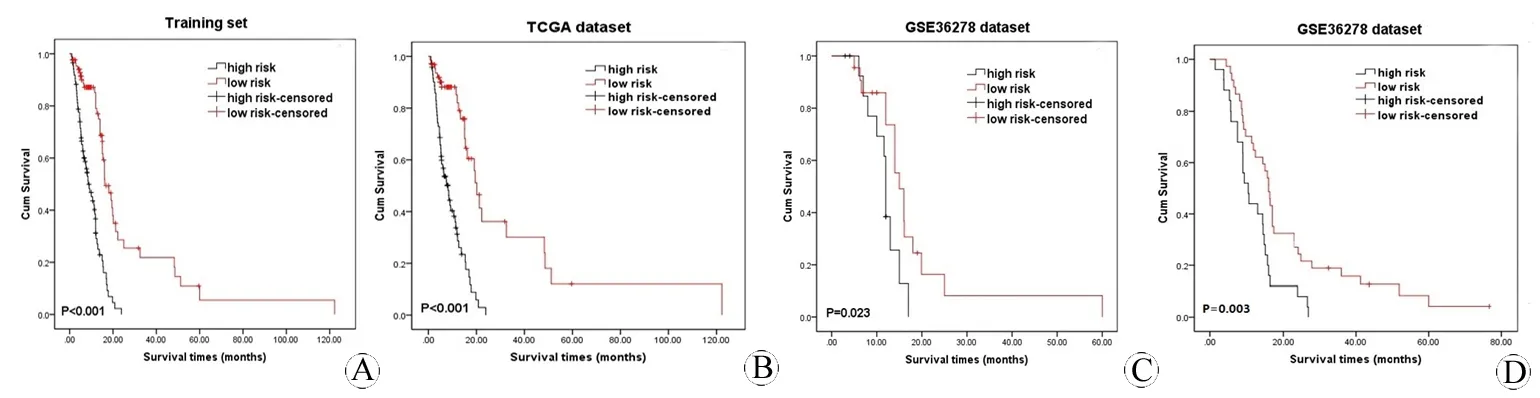
图1 Kaplan-Meier检验7-GCI shores甲基化风险评分与各病例组患者生存的关系
Fig 1 The relationship between 7-GCI shores methylation signature and the cumulative survival of patients of each group was detected by using Kaplan-Meier
A: Training-set (combined TCGA and GSE36278 group,n=173): high risk (n=87)vslow risk (n=86), χ2=41.365,P<0.001; B: TCGA group (n=136): high risk (n=72)vslow risk (n=64), χ2=35.685,P<0.001; C: GSE36278 group (n=37): high risk (n=15)vslow risk (n=22), χ2=5.193,P=0.023; D: Testing set (GSE60274,n=62): high risk (n=25)vslow risk (n=37), χ2=8.665,P=0.003.
The high-risk and low-risk group of patients are determined on the basis of the median risk score from the training set patients.
四、7-GCI shores风险评分与TCGA病例组、GSE36278组和验证组预后的关系
为减少假阳性结果,进一步验证风险评分方程对GBM患者预后的预测作用,我们分别使用风险评分方程计算了各病例的风险评分并用实验组中位风险值将TCGA病例组、GS36278病例组和实验组GSE60274分成了高风险组和低风险组。与实验组结果相似,TCGA病例组中高风险组(n=72)中位OS(8.1个月,95% CI:5.8~10.5个月)短于低风险组(n=64)中位OS(20.1个月,95% CI:17.4~22.8个月),χ2=35.685,P<0.001;GSE36278病例组中高风险组(n=15)的中位OS(12个月,95% CI:11.5~12.5个月)短于低风险组(n=22)的中位OS(15个月,95% CI:12.4~17.6个月),χ2=5.193,P=0.023;验证组GSE60274病例组中高风险组(n=25)的中位OS(10.5个月,95% CI:7.8~13.1个月)短于低风险组(n=37)的中位OS(16.0个月,95% CI:14.3~17.7个月),χ2=8.665,P=0.003。
五、7-GCI shores风险评分与患者年龄,MGMT启动子甲基化状态在预后评价中的关系
我们对所有使用替莫唑胺(temozolomide, TMZ)治疗的病例(n=125)的年龄、MGMT启动子甲基化状态同7-GCI shores风险评分一起进行多因素Cox回归模型分析。结果表明7-GCI shores风险评分是独立于年龄和MGMT启动子甲基化状态的预后因素(表3)。而在对所有病例进行Cox回归模型分析的结果也显示7-GCI shores风险评分是独立于年龄的预后因素(P<0.001, HR: 1.93, 95% CI: 1.59~2.33)。
表3 多因素Cox回归分析7-GCI shores风险评分与年龄、MGMT启动子甲基化状态对生存的影响
Tab 3 Effect of 7-GCI shores signature, age and MGMT promoter methylation status on survival by multivariate Cox analysis
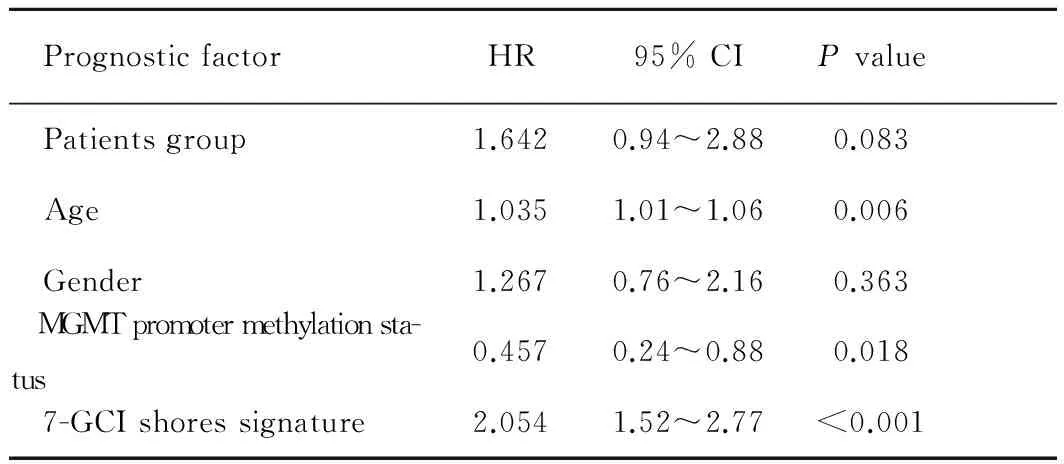
PrognosticfactorHR95%CIPvalue Patientsgroup1.6420.94~2.880.083 Age1.0351.01~1.060.006 Gender1.2670.76~2.160.363 MGMTpromotermethylationsta-tus0.4570.24~0.880.018 7-GCIshoressignature2.0541.52~2.77<0.001
HR: hazard ratio; CI: confidence interval.
讨 论
全基因组甲基化水平的高通量检测平台已经为建立大样本病例甲基化组奠定了较好的基础。使用模式分析TCGA等数据库已发现胶质瘤CGI甲基化表型(G-CIMP),随后,G-CIMP阳性和阴性表型所具有的分子基础和染色体改变特点也被偶联确认[3]。然而,G-CIMP并不能完全解释胶质瘤的所有类型,其应用具有一定的局限性。因此,研究非CGI甲基化改变模式对肿瘤的影响显得甚为重要。CGI shores是CpG密度仅次于CGI的区域,然而其甲基化改变与GBM之间的关系目前尚无研究报道。
本研究选取了包含启动子的GCI shores进行分析,最终得到了7个与GBM患者生存密切相关GCI shores。这7个GCI shores关联的基因分别为衔接蛋白-3的β3A亚基(β1 subunit of adaptor protein-3, AP3B1)基因、G蛋白信号调节蛋白16(regulator of G protein signaling 16, RGS16)基因、Mg2+/Mn2+依赖的蛋白磷酸酶1M(protein phosphatase, Mg2+/Mn2+dependent 1M, PPM1M)基因、核苷酸内焦磷酸酶/磷酸二酯酶4(ectonucleotide pyrophosphatase/phosphodiesterase 4, ENPP4)基因、高尔基体蛋白B1(golgin B1, GOLGB1)基因、钙离子电压门控通道辅助蛋白亚基α2δ2(calcium voltage-gated channel auxiliary subunit α2δ2, CACNA2D2)基因、穿膜蛋白16(transmembrane protein 16, TMEM16)基因。其中,TMEM16基因编码穿膜蛋白16(transmembrane protein16),也被称为Anoctamin,是一种钙激活氯通道(calcium-activatedchloridechannels, CaCCs)蛋白,细胞内钙离子增加可以激活其家族的Ano1(anoctamin1)从而调节唾液腺、气管、胰腺、肠道、乳腺上皮细胞的分泌及细胞体积[4]。Ano1在不同级别胶质瘤中均有表达增加,将其敲除可以抑制肿瘤细胞的增殖、迁移与侵袭[5]。RGS16基因编码GTP酶激活蛋白(GTPase-activating protein, GAP),是GTP酶激活α亚族的G蛋白偶联受体。在正常或肿瘤细胞中GAP负向调节丝裂原活化蛋白激酶(mitogen-activated protein kinase, MAPK),磷脂酰肌醇3激酶/蛋白激酶B(phosphatidylinositol 3-kinase/protein kinase B, PI3K/AKT),Rho家族蛋白A和基质细胞衍生因子1/趋化因子受体4 (stromal cell derived factor 1/chemokine receptor type 4, SDF-1/CXCR4)等癌基因通路,这些通路在多种恶性肿瘤细胞中发挥重要作用。乳腺癌患者中约有半数都会出现RGS16序列的不稳定,敲除RGS16基因可增加乳腺癌细胞表皮生长因子表达并促进肿瘤细胞增殖[6]。另外,RGS16基因表达上调可能受到p53和pRb基因的调节从而增加胰腺癌的淋巴结转移,降低患者的生存期[7]。CACNA2D2基因编码α2δ2蛋白,该基因位于肿瘤抑制基因簇的染色体3p21.3区域,该区域超甲基化或缺失在肺癌和胰腺癌中均有发生[8]。但是CACNA2D2基因对肿瘤的作用存在争议,Carboni等研究发现CACNA2D2基因高表达可促进裸鼠的非小细胞肺癌细胞凋亡,抑制肿瘤生长[9]。而在对前列腺癌的研究中,CACNA2D2基因却表现出促癌作用,其表达增加可以激活前列腺癌细胞增殖,并增加血管内皮生长因子(vascular endothelial growth factor, VEGF)分泌[10]。AP3B1基因编码衔接蛋白3 (adaptor related protein complex 3, AP-3)复合体的β3A亚基。AP-3复合体是异四聚体的复合体,系细胞质中无所不在的蛋白,为生黑色素细胞、血小板、中性粒细胞、细胞毒性T细胞和自然杀伤细胞分泌溶酶体所必须成分,神经元缺乏AP-3复合体会导致突触小泡分泌异常[11]。在乳腺癌和肝癌中,AP3B1基因表达受到抑癌miRNA mir-9的直接调控,不过其对癌细胞的影响并不清楚[12]。ENPP4基因编码核苷酸内焦磷酸酶/磷酸二酯酶4(ecto-nucleotide pyrophosphatase/phosphodiesterase 4, ENPP4),属于ENPP家族。ENPP家族可以启动核苷二磷酸和核苷三磷酸的水解作用进而影响骨和软骨软化、核苷酸循环、细胞运动以及各种嘌呤和嘌呤受体相关通路[13]。其在小鼠骨肉瘤肺转移模型中表达异常[14]。GOLGB1基因位于染色体3q13,编码高尔基体b1蛋白,是最大的高尔基复合体相关蛋白[15]。高尔基体蛋白在蛋白质糖基化和胚胎发育中起作用,于骨髓增殖性肿瘤患者中GOLGB1基因可以和血小板转化生长因子受体B形成融合基因[16]。以上基因中,部分已经证实对某些肿瘤具有影响,但在它们GBM中的研究较少,仍需进一步的离体和在体研究来深入我们对其功能的理解。
在本研究中,我们建立并验证了可以预测GBM患者生存的基于7个GCI shores区甲基化状态的风险评分方程。同样,本风险评分是独立于年龄和MGMT启动子甲基化状态的GBM患者预后因素。使用该风险评分方程可更为准确的评估GBM患者的预后,并指导GBM患者个体化治疗。本研究是第一个探索GCI shores甲基化状态与GBM预后关系的报道,加深GCI shores甲基化模式的理解将有助于完善G-CIMP以外的GBM表型,为今后进一步完善GBM甲基化图谱表明了新的方向。
参考文献
1OIKE T, SUZUKI Y, SUGAWARA K, et al. Radiotherapy plus concomitant adjuvant temozolomide for glioblastoma: Japanese mono-institutional results [J]. PLoS One, 2013, 8(11): e78943.
2RAO X, EVANS J, CHAE H, et al. CpG island shore methylation regulates caveolin-1 expression in breast cancer [J]. Oncogene, 2013, 32(38): 4519-4528.
3康恩铭, 章薇, 章翔. 胶质母细胞瘤DNA甲基化研究进展 [J]. 中华神经外科疾病研究杂志, 2017, 16(3): 311-4.
4LEE Y S, BAE Y, PARK N, et al. Surface expression of the Anoctamin-1 (ANO1) channel is suppressed by protein-protein interactions with beta-COP [J]. Biochem Biophys Res Commun, 2016, 475(2): 216-222.
5LIU J, LIU Y, REN Y, et al. Transmembrane protein with unknown function 16A overexpression promotes glioma formation through the nuclear factor-kappaB signaling pathway [J]. Mol Med Rep, 2014, 9(3): 1068-1074.
6CARPER M B, DENVIR J, BOSKOVIC G, et al. RGS16, a novel p53 and pRb cross-talk candidate inhibits migration and invasion of pancreatic cancer cells [J]. Genes Cancer, 2014, 5(11-12): 420-435.
7KIM J H, LEE J Y, LEE K T, et al. RGS16 and FosB underexpressed in pancreatic cancer with lymph node metastasis promote tumor progression [J]. Tumour Biol, 2010, 31(5): 541-548.
8LERMAN M I, MINNA J D. The 630-kb lung cancer homozygous deletion region on human chromosome 3p21.3: identification and evaluation of the resident candidate tumor suppressor genes. The International Lung Cancer Chromosome 3p21.3 Tumor Suppressor Gene Consortium [J]. Cancer Res, 2000, 60(21): 6116-6133.
9CARBONI G L, GAO B, NISHIZAKI M, et al. CACNA2D2-mediated apoptosis in NSCLC cells is associated with alterations of the intracellular calcium signaling and disruption of mitochondria membrane integrity [J]. Oncogene, 2003, 22(4): 615-626.
10WARNIER M, ROUDBARAKI M, DEROUICHE S, et al. CACNA2D2 promotes tumorigenesis by stimulating cell proliferation and angiogenesis [J]. Oncogene, 2015, 34(42): 5383-5394.
11SEONG E, WAINER B H, HUGHES E D, et al. Genetic analysis of the neuronal and ubiquitous AP-3 adaptor complexes reveals divergent functions in brain [J]. Mol Biol Cell, 2005, 16(1): 128-140.
12ZHANG J, CHENG J, ZENG Z, et al. Comprehensive profiling of novel microRNA-9 targets and a tumor suppressor role of microRNA-9 via targeting IGF2BP1 in hepatocellular carcinoma [J]. Oncotarget, 2015, 6(39): 42040-42052.
13GODING J W, GROBBEN B, SLEGERS H. Physiological and pathophysiological functions of the ecto-nucleotide pyrophosphatase/phosphodiesterase family [J]. Biochim Biophys Acta, 2003, 1638(1): 1-19.
14MIRETTI S, ROATO I, TAULLI R, et al. A mouse model of pulmonary metastasis from spontaneous osteosarcoma monitored in vivo by Luciferase imaging [J]. PLoS One, 2008, 3(3): e1828.
15SOHDA M, MISUMI Y, FUJIWARA T, et al. Molecular cloning and sequence analysis of a human 372-kDA protein localized in the Golgi complex [J]. Biochem Biophys Res Commun, 1994, 205(2): 1399-1408.
16LAN Y, ZHANG N, LIU H, et al. Golgb1 regulates protein glycosylation and is crucial for mammalian palate development [J]. Development, 2016, 143(13): 2344-2355.
