弗尼斯弧菌PCR检测方法的建立及应用❋
2018-05-10王殿夫卢福荣冯华炜麻丽丹
王殿夫, 何 平, 王 青, 于 洋, 卢福荣, 冯华炜, 麻丽丹❋❋
(1. 辽东学院农学院,辽宁 丹东 118000;2. 中国合格评定国家认可中心,北京 100062;3. 东港出入境检验检疫局,辽宁 丹东 118000;4. 丹东出入境检验检疫局,辽宁 丹东 118000)
弗尼斯弧菌(Vibriofurnissii)原名为产气河弧菌(Vibriofluvialis),1983年被Brenner重新划分为弧菌属(Vibrio)的一个新种,为革兰阴性稍弯曲或短杆菌,有长于菌体数倍的鞘生极端鞭毛和较细的侧生波状鞭毛,有动力[1]。弗尼斯弧菌是近年来国内外学者公认的新食源性致病菌,对全球公共卫生和食品安全造成潜在威胁,呈暴发流行或散发性急性胃肠炎。此菌存在于海洋,产生肠毒素,为旅游者腹泻的病原菌之一,且会引起浅表创伤感染、菌血症等疾病[2-4]。此外,该菌通过黏附和侵入宿主组织,从宿主中摄取大量营养,分泌多种毒素,从而引起欧洲鳗鱼(Anguillaanguilla)、对虾(Penaeuschinensi)、文蛤(Ruditapesphilippinarum)等水产品的弧菌病[5-12],给水产养殖带来巨大的经济损失。其中弗尼斯弧菌会导致文蛤细胞及组织病变,鳃丝溃烂、肠胃内膜上皮细胞萎缩,甚至引发其暴发性死亡,也是引起对虾发光病、红腿病、败血症等疾病的主要病原之一[12-14]。因此,亟待建立灵敏、快捷、特异的检测技术用于弗尼斯弧菌快速检测。
目前,弗尼斯弧菌的检测以传统的分离培养、生化鉴定为主,但是该方法检测周期较长、费时费力,且需要检测人员具有专业的知识背景,远不能满足疾病的快速诊断和及时治疗。以核酸为检测靶标的分子检测技术具有检测快速、灵敏度高等优点,在病原微生物的诊断和鉴定等领域得到了广泛应用,但应用PCR检测技术进行弗尼斯弧菌的鉴定检测鲜有报道。toxS基因是首次发现于霍乱弧菌(Vibriocholerae)中的毒力基因,位于toxR基因(作为霍乱弧菌毒素操纵子的转率激活子)的下游以增强toxR基因的调节转录活性[15]。toxS基因除了存在于霍乱弧菌中外,还存在于副溶血性弧菌(Vibrioparahaemolyticus)、费歇尔弧菌(Vibriofischeri)、创伤弧菌(Vibriovulcificus)和弗尼斯弧菌等弧菌中[16-21]。目前,toxS基因作为一个靶基因进行弧菌属的PCR检测已得到广泛应用[21],但是针对弗尼斯弧菌的PCR检测尚未进行研究。因此本研究以toxS基因为靶基因,建立高效、特异的PCR检测方法,用于水产品中弗尼斯弧菌的快速检测。
1 材料与方法
1.1 菌株
用于本研究的菌株共82株(见表1),采用的标准菌株购买于ATCC、CICC、IQCC,其余菌株来自于丹东出入境检验检疫局在实测样品中分离和收集的菌株,以及来自于北京、上海、广东、辽宁等出入境检验检疫局的样品分离株及疾病预防控制中心的临床分离菌株。所有菌株都经生理生化实验和测序进行验证。

表1 菌株信息表Table 1 Information of bacterial strains
续表1

编号Code菌株名称Strain来源Source地区/国别Region/Country分离时间Yearofisolation39副溶血性弧菌(Vibrioparahaemolyticus)冻墨鱼山东2010年40副溶血性弧菌(Vibrioparahaemolyticus)病人北京2011年41副溶血性弧菌(Vibrioparahaemolyticus)病人北京2011年42副溶血性弧菌(Vibrioparahaemolyticus)病人北京2011年43副溶血性弧菌(Vibrioparahaemolyticus)病人北京2011年44副溶血性弧菌(Vibrioparahaemolyticus)肛拭子上海2009年45副溶血性弧菌(Vibrioparahaemolyticus)肛拭子上海2009年46副溶血性弧菌(Vibrioparahaemolyticus)肛拭子上海2009年47副溶血性弧菌(Vibrioparahaemolyticus)肛拭子上海2009年48副溶血性弧菌(Vibrioparahaemolyticus)肛拭子上海2009年49副溶血性弧菌(Vibrioparahaemolyticus)肛拭子上海2009年50副溶血性弧菌(Vibrioparahaemolyticus)ATCC17802美国2013年51副溶血性弧菌(Vibrioparahaemolyticus)马面鱼丹东2014年52副溶血性弧菌(Vibrioparahaemolyticus)腌虾丹东2015年53霍乱弧菌(Vibriocholera)鸭绿江水丹东2004年54霍乱弧菌(Vibriocholera)鸭绿江水丹东2005年55霍乱弧菌(Vibriocholera)鸭绿江水丹东2006年56霍乱弧菌(Vibriocholera)鸭绿江水丹东2007年57霍乱弧菌(Vibriocholera)鸭绿江蟹子丹东2008年58霍乱弧菌(Vibriocholera)鸭绿江水丹东2009年59霍乱弧菌(Vibriocholera)鸭绿江水丹东2009年60霍乱弧菌(Vibriocholera)沙白鱼珠海2010年61霍乱弧菌(Vibriocholera)鲩鱼珠海2010年62溶藻弧菌(Vibrioalginolyticus)鸭绿江水丹东2007年63溶藻弧菌(Vibrioalginolyticus)冻煮玉螺肉丹东2008年64溶藻弧菌(Vibrioalginolyticus)冻小鱿鱼丹东2008年65溶藻弧菌(Vibrioalginolyticus)冻小鱿鱼丹东2008年66溶藻弧菌(Vibrioalginolyticus)盐渍毛虾丹东2011年67溶藻弧菌(Vibrioalginolyticus)冻煮河螺肉丹东2011年68溶藻弧菌(Vibrioalginolyticus)冷冻花虾丹东2011年69溶藻弧菌(Vibrioalginolyticus)病人山东2012年70溶藻弧菌(Vibrioalginolyticus)病人山东2012年71溶藻弧菌(Vibrioalginolyticus)病人山西2012年72溶藻弧菌(Vibrioalginolyticus)去马面鱼片丹东2014年73溶藻弧菌(Vibrioalginolyticus)清洗去皮马面鱼丹东2014年74溶藻弧菌(Vibrioalginolyticus)干调味马面鱼片丹东2014年75溶藻弧菌(Vibrioalginolyticus)ATCC17749美国2013年76坎氏弧菌(Vibriocampbellii)中科院微生物研究所中国2009年77大肠埃希氏菌(Escherichiacoli)ATCC25922美国2010年78大肠埃希氏菌(Escherichiacoli)苏子叶及调料丹东2014年79大肠埃希氏菌(Escherichiacoli)辣制苏子叶丹东2014年80大肠埃希氏菌(Escherichiacoli)酱汁黑豆丹东2014年81嗜水气单孢菌(Aeromonashydrophila)江鱼和江水丹东2007年82嗜水气单孢菌(Aeromonashydrophila)毛蚶大连2012年83志贺氏菌(Shigellaspp)ATCC12022美国2012年
1.2 材料
江瑶贝、蚬子肉、冻鸡肉及冻花菜等共384份样品为辽宁省丹东出入境检验检疫局进出口样品及丹东市市场监测样品。
1.3 方法
1.3.1 DNA提取 按照宝生物MiniBEST Bacteria Genomic DNA Extraction Kit Ver.3.0试剂盒操作说明进行DNA提取。
1.3.2 引物及探针 弗尼斯弧菌toxS基因的普通PCR引物及实时荧光PCR引物和探针序列见表2,由Invitrogen公司合成。
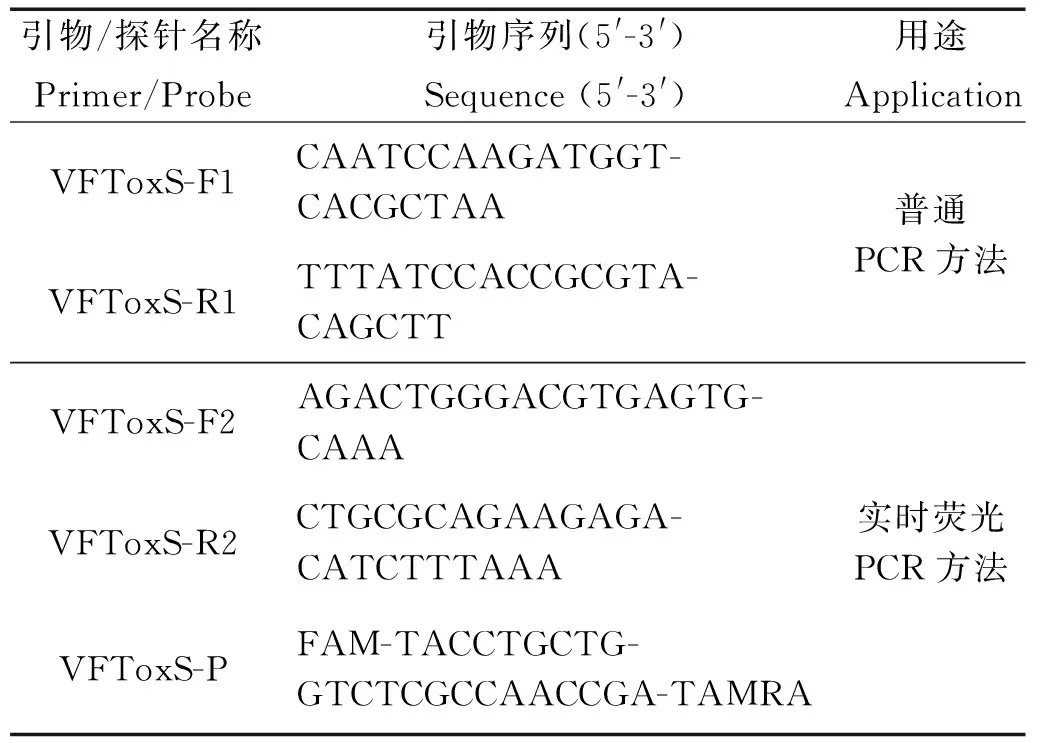
表2 弗尼斯弧菌普通PCR引物及实时荧光PCR引物和探针序列Table 2 Primers and probe used in conventional PCR and real-time fluorescence PCR assays
1.3.3 普通PCR方法及优化 使用TaKaRa Ex Taq试剂盒,确定普通PCR方法反应体系:模板液2 μL、10×PCR缓冲液2.5 μL、dNTP 0.25 μL、Taq酶0.125 μL、正反向引物(10 μmol·L-1)各0.5 μL、补充灭菌双蒸水至总体积为25 μL;以上各组分加入至0.2 mL PCR反应管中,混匀,5 000 r/min 离心10s。
反应条件:94 ℃预变性4 min,然后94 ℃ 1 min、53~61℃ 1 min、72 ℃ 1 min,35个循环,最后72 ℃延伸10 min,4 ℃保存。并以弗尼斯弧菌标准菌株ATCC 35016的基因组DNA为模板进行退火温度的优化,将PCR产物进行琼脂糖凝胶电泳,以条带最亮,特异性最强的PCR产物所对应的温度为最适退火温度。
1.3.4 实时荧光PCR方法 使用ABI的Path-IDTMqPCR Master mix Kit试剂盒,确定荧光定量PCR方法反应体系:模板液2 μL、qPCR Maser mix 12.5 μL、正向引物(10 μmol·L-1) 1 μL、反向引物(10 μmol·L-1)1 μL、探针(10 μmol·L-1) 0.3 μL,补充灭菌双蒸水至总体积为25 μL;以上各组分加入至0.2 mL PCR反应管中,混匀,5 000 r/min 离心10 s。
反应条件:95 ℃预变性10 min,然后95 ℃ 15 s、52~62 ℃ 1 min,35个循环,每次循环的退火时收集荧光。并以弗尼斯弧菌标准菌株ATCC 35016的基因组DNA(24 CFU/mL)为模板进行退火温度的优化。
1.3.5 特异性实验 选取弗尼斯弧菌菌株6株、溶藻弧菌、霍乱弧菌、副溶血弧菌、麦氏弧菌等弧菌属和肠杆菌科的菌株(共76株),分别按照普通PCR方法和实时荧光PCR方法进行扩增,以检验2种方法的特异性。
1.3.6 灵敏度实验 将弗尼斯弧菌标准菌株于3%氯化钠碱性蛋白胨水中培养18 h左右后,测其麦氏浊度,估计其菌数,然后按10倍递增进行梯度稀释到10-7,取最后4个稀释液的菌液进行平板计数,每个稀释度重复3个,按照相应的方法培养后进行菌落计数取平均值确定其真实的菌密度。将已准确计数的弗尼斯弧菌菌液进行10 倍稀,分别采用传统培养方法 、普通PCR方法和实时荧光PCR方法进行检测,以确定3种方法的检测灵敏度。
1.3.7实际样品检测 以传统的检测方法(ISO/TS 21872-2[20])为对照方法,对江瑶贝、蚬子肉、冻鸡肉及冻花菜等共384份样品分别进行普通PCR和实时荧光PCR检测,并使用ABI 3500/3500xL基因测序仪对阳性结果的PCR产物进行测序及比对,以检验本方法的效果。
2 结果
2.1 优化结果
以弗尼斯弧菌标准菌株ATCC 35016的基因组DNA为模板进行退火温度的优化。结果显示,普通PCR方法的退火温度为59 ℃(见图1),荧光定量PCR方法的退火温度为60 ℃(见图2)。
2.2 特异性试验结果
特异性实验结果显示,普通PCR方法(标准菌株特异性试验结果见图3,其他分离株特异性试验结果图略)中的6株弗尼斯弧菌均可扩增出150 bp大小的特异性片断,实时荧光PCR方法(见图4)中的6株弗尼斯弧菌均可扩增出典型的扩增曲线,而采用2种PCR方法对其余70株弧菌及肠杆菌的扩增都呈阴性,表明2种PCR方法的特异性均为100%。
2.3 灵敏度试验结果
弗尼斯弧菌的传统培养方法检测灵敏度可达2.4×104CFU/mL,而DNA梯度稀释检测结果(见图5和6)显示,普通PCR方法和实时荧光PCR检测方法对于弗尼斯弧菌的检测灵敏度分别为2 400和24 CFU/mL,表明实时荧光PCR检测方法的灵敏度是普通PCR检测方法的100倍,而普通PCR方法和实时荧光PCR检测方法的灵敏度分别是传统培养的方法的10和1 000倍。

(A:1、17:DNA分子量标准;2~4: 55 ℃;5~6:空白对照(55 ℃);7~9:57 ℃;10~11:空白对照(57 ℃);12~14:53 ℃退火;15~16:空白对照(53 ℃)。B: 1:Marker DL2000;2~3:59 ℃;4~5: 61 ℃;6:空白对照(59 ℃);7:空白对照(:61℃)。A:1、17:Marker DL2000;2-4: 55 ℃;5-6:Negative control(55 ℃);7-9:57 ℃;10-11:Negative control(57 ℃);12-14:53 ℃退火;15-16:Negative control(53 ℃).B: 1:Marker DL2000;2-3:59 ℃;4-5: 61 ℃;6:Negative control(59 ℃);7:Negative control(:61℃).)
图1 普通PCR退火温度的优化
Fig.1 Optimization of PCR annealing temperature

(1:60 ℃;2:58 ℃;3:62 ℃;4:56 ℃)图2 实时荧光PCR退火温度的优化Fig.2 Optimization of real-time fluorescence PCR annealing temperature

(1、20: DNA分子量标准;2:弗尼斯弧菌标准菌株 (ATCC 35016);3:弗尼斯标准菌株(IQCC 12301);4:弗尼斯标准菌株(IQCC 12309);5:弗尼斯弧菌分离株(病人);6:弗尼斯弧菌分离株(蚬子肉)7:弗尼斯弧菌分离株(病人);8:麦氏弧菌标准菌株(ATCC 700040);9:河弧菌标准菌株(CICC 21612);10:拟态弧菌标准菌株(CICC 21613);11:拟态弧菌标准菌株(IQCC 12304);12:创伤弧菌标准菌株(ATCC27652);13:哈氏弧菌标准菌株(ATCC35084);14:霍利斯弧菌标准菌株(ATCC33564);15:坎氏弧菌分离株(毛蚶);16:大肠埃希氏菌标准菌株(ATCC 25922);17:副溶血性弧菌标准菌株(ATCC 17802);18:志贺氏菌标准菌株(ATCC 12022);19阴性对照。1、20:Marker DL2000;2:Vibriofurnissiireference strain (ATCC 35016);3:Vibriofurnissiireference strain(IQCC 12301);4:Vibriofurnissiireference strain(IQCC 12309);5:Vibriofurnissiiisolated strain(patient);6:Vibriofurnissiiisolated strain(clam meat)7:Vibriofurnissiiisolated strain(patient);8:Vibriometschnikoviireference strain(ATCC 700040);9:Vibriofluvialisreference strain(CICC 21612);10:Vibriominicusreference strain(CICC 21613);11:Vibriominicusreference strain(IQCC 12304);12:Vibriovulcificusreference strain(ATCC27652);13:Vibrioharveyireference strain(ATCC35084);14:Vibriohollisaereference strain(ATCC33564);15:Vibriocampbelliiisolated strain(Anadarasubcrenata);16:Escherichiacolireference strain(ATCC 25922);17:Vibrioparahaemolyticusreference strain(ATCC 17802);18:Shigellasppreference strain(ATCC 12022);19:Negative control.)
图3 普通PCR检测方法特异性试验结果
Fig.3 Specificity test of conventional PCR

(1: 弗尼斯弧菌标准菌株 (ATCC 35016);2:弗尼斯标准菌株(IQCC 12301);3:弗尼斯标准菌株(IQCC 12309):4:弗尼斯弧菌分离株(病人);5:弗尼斯弧菌分离株(蚬子肉);6:弗尼斯弧菌分离株(病人)。1:Vibriofurnissiireference strain (ATCC 35016);2:Vibriofurnissiireference strain(IQCC 12301);3:Vibriofurnissiireference strain(IQCC 12309):4:Vibriofurnissiiisolated strain(patient);5:Vibriofurnissiiisolated strain(clam meat);6:Vibriofurnissiiisolated strain(patient).)
图4 实时荧光PCR检测方法特异性试验结果
Fig.4 Specificity test of real-time fluorescence PCR
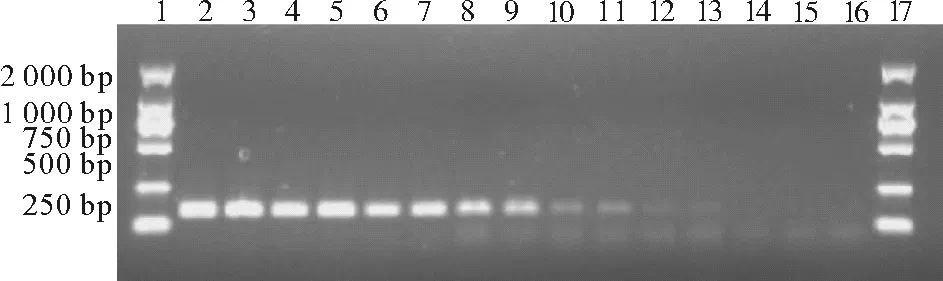
(1、17:DNA分子量标准;2、3:2.4×108CFU/mL;4、5:2.4×107CFU/mL;6、7:2.4×106CFU/mL;8、9:2.4×105CFU/mL;10、11:2.4×104CFU/mL;12、13:2.4×103CFU/mL;14、15:2.4×102CFU/mL;16:阴性对照Negative control.)
图5 普通PCR灵敏度试验结果
Fig.5 Sensitivity test of conventional PCR
2.4 食品样品检测结果
对384份样品的检测结果如表3所示:在258份水产品中,检出弗尼斯弧菌阳性6份样品,这6份样品分别来自为:牡蛎(1例)、杂色蛤(2例)、虾(1例)、章鱼(1例)、腌蟹子(1例)样品,其余126份动物及植物产品中均未检出弗尼斯弧菌。以上所有检测结果与传统生化培养方法检测结果一致, 6株阳性菌株的测序结果(见图7~12)与Genbank中登录号为:CP002377.1的弗尼斯弧菌一致性为99%~100%,证明本研究所建立2种PCR方法适用于食品中弗尼斯弧菌的检测。

(1:2.4×106CFU/mL;2:2.4×105CFU/mL;3:2.4×104CFU/mL ;4:2.4×103CFU/mL;5: 2.4×102CFU/mL;6: 2.4×101CFU/mL)

图6 实时荧光PCR灵敏度试验结果Fig.6 Sensitivity of real-time fluorescence PCR
Note: ①Number of positive samples;②Conventional PCR;③Real-time fluorescence PCR;④ISO method;⑤Aquatic products;⑥Animal products;⑦Plant products

图7 牡蛎样品中弗尼斯弧菌的序列比对结果Fig.7 Sequence alignment of Vibrio furnissii in oyster Samples

图8 杂色蛤样品(C1)中弗尼斯弧菌的序列比对结果Fig.8 Sequence alignment of Vibrio furnissii in ruditapes philippinarum Samples(C1)

图9 杂色蛤样品(C2)中弗尼斯弧菌的序列比对结果Fig.9 Sequence alignment of Vibrio furnissii in ruditapes philippinarum Samples(C2)

图10 虾样品中弗尼斯弧菌的序列比对结果Fig.10 Sequence alignment of Vibrio furnissii in shrimp Samples

图11 章鱼样品中弗尼斯弧菌的序列比对结果Fig.11 Sequence alignment of Vibrio furnissii in octopus Samples

图12 腌蟹子样品中弗尼斯弧菌的序列比对结Fig.12 Sequence alignment of Vibrio furnissii in pickled crab Samples
3 讨论
弗尼斯弧菌广泛存在于水体中,尤其多见于海产品中,近年来已有许多关于对虾(Penaeuschinensis)[9]、鱿鱼(Squid)[21]、牡蛎(Ostreagigasthunberg)[22]等水产品中检测出弗尼斯弧菌的报道,其已成为食品安全的一个重大隐患。目前,关于弗尼斯弧菌检测的报道研究中,对于弗尼斯弧菌的检测采均基于传统的分离培养及生化鉴定方法。如马秀芝等[23]采用分离培养法从132份水产品种分离出2株弗尼斯弧菌,梁健等[24]也采用传统方法从方斑东风螺中分离出1株引起方斑东风螺肿吻病的弗尼斯弧菌。传统分离方法不仅耗时耗力,还存在检测结果不准确等问题,不适于弗尼斯弧菌的高通量快速检测。国内外尚还未有弗尼斯弧菌的PCR检测方法的建立与应用的研究。
目前针对弧菌的特异性检测多针对其hly、tl、toxR、toxS等毒力基因。其中toxS基因是膜整合蛋白的编码基因,参与弧菌毒力基因的转运和表达,广泛存在于多种弧菌中。对toxS基因的生物信息学分析发现,toxS基因呈现出较大的种间差异性,种内保守而种间可变的区域可作为种鉴定的良好分子靶位点[25],以此建立基于弗尼斯弧菌toxs基因的特异性PCR 检测方法。
针对PCR检测方法的特异性试验的研究方面,王华丽等[26]对海产品中副溶血弧菌进行了PCR检测方法的建立与评价,采用11株副溶血弧菌和22株其他细菌进行特异性试验检测。姚东瑞等[27]建立的哈氏弧菌的PCR检测方法中,特异性试验采用1株哈氏弧菌及3株非哈氏弧菌进行检测。陈昌国等[28]建立的溶藻弧菌Taqman探针实时荧光PCR方法中采用1株溶藻弧菌的标准菌株及6株其他细菌进行检测。本研究建立的PCR方法在进行特异性试验时采用了6株弗尼斯弧菌及70株其他弧菌、细菌的标准菌株及分离株,远高于上述文献所报道的方法中采用的菌株数量,这使得菌株特异性试验验证结果更准确。
根据文献[26-29]报道的PCR检测方法,普通PCR方法检测弧菌的灵敏度可以达到104CFU/mL数量级[26-27],实时荧光PCR方法检测弧菌的灵敏度可以达到102CFU/mL数量级[28-29]。而本研究建立的普通PCR检测方法灵敏度分为2 400 CFU/mL,检测灵敏度比文献[26-27]报道的PCR检测方法和ISO分离培养方法均高出10倍;本研究建立的实时荧光PCR检测方法的灵敏度为 24 CFU/mL,检测灵敏度比ISO分离培养方法高出1 000倍,与文献[28-29]报道的实时荧光PCR检测方法的灵敏度基本一致。对258份食品样品检测结果显示,本研究建立的2种PCR方法与ISO方法的符合率达到100 %。这个结果可能是由于食品样品中弗尼斯弧菌的感染浓度较高,因而使得这3种方法都可以检出弗尼斯弧菌。
4 结语
本研究建立了基于PCR技术的弗尼斯弧菌快速检测方法,结果表明,普通PCR检测方法和实时荧光PCR检测方法的引物和探针具有良好的特异性(100 %),2种方法的灵敏度分别为2 400和24 CFU/mL,实时荧光PCR检测方法的灵敏度是普通PCR检测方法的100倍。采用本研究建立的方法对384份水产品和食品样品的检测结果显示,共有6份样品检出弗尼斯弧菌阳性,其检测结果与ISO方法检测结果及测序结果一致,表明该方法适用于食品中弗尼斯弧菌的检测。
参考文献:
[1] Brenner D J, Hickm An-Brenner F W, Lee J V, et al.Vibriofurnissii(form erly aerogenic biogroup ofVibriofluvialis), a new species isolated from human feces and the environment[J]. Journal of Clinical Microbiology, 1983, 18(4): 816-824.
[2] Daniels N A, Shafaie A. A review of pathogenicVibrioinfections for clinicians[J]. Infections in Medicine, 2000, 17(10): 665-685.
[3] Schirmeister F, Wieczorek A, Dieckmann R, et al. Evaluation of molecular methods to discriminate the closely related speciesVibriofluvialisandVibriofurnissii[J]. International Journal of Medical Microbiology, 2014, 304(7): 851-857.
[4] 徐嘉良, 惠书瑶, 卢昕, 等. 利用比较基因组学研究弗尼斯弧菌和霍乱弧菌在鸟氨酸脱羧酶试验中不同表型的分子遗传基础[J]. 食品科技, 2015, 40(8): 301-304.
XU Jia-liang, HUI Shu-yao, LU Xin, et al, Identification of genetic basis ofVibriofurnissiiandVibriocholeraedifferent phenotypes in iornithine decarboxylase test by comparative genomics analysis[J]. Food Science and Technology, 2015, 40(8): 301-304.
[5] Sung H H, Hsu S F, Chen C K, et al. Relationships between disease outbreak in cultured tiger shrimp, Penaeus monodon. and the composition ofVibriocommunities in pond water and shrimp hepatopancreas during cultivation[J]. Aquaculture, 2001, 192(2): 101-110.
[6] Miao L, Qian P Y. Antagonistic antimicrobial activity of marine fungi and bacteria isolated from marine biofilm and seawaters of Hong Kong[J]. Aquatic Microbial Ecology, 2005, 38(3): 231-238.
[7] Esteve C, Amaro C, Biosca E G, et al. Biochemical and toxigenic properties ofVibriofurnissiiisolated from a European eel farm[J]. Aquaculture, 1995, 132(1-2): 81-90.
[8] Jayasinghe C, Ahmed S, Kariyawasam M. The isolation and identification ofVibriospecies in marine shrimps of Sri Lanka[J]. Journal of Food & Agriculture, 2008, 1(1): 36-44.
[9] Matte G R, Matte M H, Rivera I G, et al. Distribution of potentiallyVibriosin oysters from a tropical region[J]. Journal of Food Protection, 1994, 57(57): 870-873.
[10] Janda J M, Powers C, Bryant R G, et al. Current perspectives on the epidemiology and pathogenesis of clinically significantVibriospp[J]. Clinical Microbiology Reviews, 1988, 1(3): 245-267.
[11] 张彬, 黄婷, 熊建华, 等. 文蛤主要弧菌性病害研究进展[ J]. 广东农业科学, 2012, 39(17): 128-134.
ZHANG Bin, HUANG Ting, XIONG Jian-hua, et al. Research progress ofVibriosisin hard calm,MeretrixmeretrixLinnaers.[J]. Guangdong Agricultural Sciences, 2012, 39(17): 128-134
[12] 胡梦华. 海水养殖源弧菌耐药性调查与致病性弧菌毒力基因检测[D]. 上海: 上海海洋大学, 2015.
HU Meng-hua. Investigation of Antimicrobial Resistance of Mariculture SourceVibrioand Detection of Virulence Genes of PathogenicVibrio[D]. Shanghai: Shanghai Ocean University, 2015
[13] 邓传燕, 李色东, 陈贺, 等. 凡纳滨对虾发光病病原的分离鉴定及防治措施[J]. 江西水产科技, 2013(2): 6-11.
DENG Chuan-yan, LI Se-dong, CHEN He, et al, Identification and prevention measures of luminescent pathogen fromPenaeusvannamei[J]. Jiangxi Fishery Sciences and Technology, 2013(2):6-11.
[14] 刘问, 黄惟灏, 钱冬, 等. 南美白对虾苗种淡化期间异养细菌和弧菌的数量动态[J]. 浙江海洋学院学报(自然科学版), 2004, 23(1): 24-27.
LIU Wen, HUANG Wei-hao, QIAN Dong, et al. Quantity Variation of Heterobacteria andVibriosduringPenaeusvannameiLarvae Desalting Culture[J]. Journal of Zhejiang Ocean University(Natural Science), 2004, 23(1): 24-27.
[15] Iller V L, Dirita V J, Mekalanos J J. Identification oftoxS, a regulatory gene whose product enhancestoxR-mediated activation of the cholera toxin promoter[J]. Journal of Bacteriology, 1989, 171(3): 1288-1293.
[16] Li X J, Zhao H, Su J, et al. A method for screening and detection of virulence genes ofVibrioisolates from freshwater crayfish[J]. Food Science, 2011, 32(18): 264-267.
[17] Reich K A, Schoolnik G K. The light organ symbiontVibriofischeripossesses a homolog of theVibriocholeraetransmembrane transcriptional activatorToxR[J]. Journal of Bacteriology, 1994, 176(10): 3085-3088.
[18] Lee S E, Shin S H, Kim S Y, et al.Vibriovulnificushas the transmembrane transcription activatorToxRSstimulating the expression of the hemolysin gene vvhA[J]. Journal of Bacteriology, 2000, 182(12): 3405-3415.
[19] Lux T M, Lee R, Love J. Complete genome sequence of a free-livingVibriofurnissiisp nov strain, NCTC 11218[J]. Journal of Bacteriology, 2011, 193(6): 1487-1488.
[20] Fethi B A, Kamel C, Hela K, et al. RT-PCR assays for in vivo expression ofVibrioalginolyticusvirulence genes in cultured giltheadDicentrarchuslabraxandSparusaurata[J]. Annals of Microbiology, 2009, 59(1): 63-67.
[21] 李丹丹, 徐义刚, 王昱, 等. 副溶血弧菌实时荧光定量PCR快速检测方法的建立[J]. 中国畜牧兽医, 2016, 43(1): 58-62. .
LI Dan-dan, XU Yi-gang, WANG Yu, et al. Development of real-time PCR method for rapid detection ofVibrioparahaemolyticus[J]. China Animal Husbandry & Veterinary Medicine, 2016, 43(1): 58-62. .
[22] 胡兴娟, 沈飚, 余辉, 等. 多重荧光定量PCR法检测水产品中副溶血性弧菌、沙门菌和单增李斯特菌[J]. 中国食品卫生杂志, 2016, 28(4): 440-444.
HU Xing-juan, SHEN Biao, YU Hui, et al. Detection ofVibrioparahaemolyticus, salmonella and listeria monocytogenes in seafoods using multiplex real-time quantitative PCR[J]. Chinese Journal of Food Hygiene, 2016, 28(4): 440-444.
[23] 马秀芝, 田超, 孙媛媛. 薛城区水产品致病性弧菌调查[J]. 中华现代中西医杂志, 2005, 10(1): 1.
MA Xiu-zhi, TIAN Chao, SUN Yuan-yuan. Investigation on the pathogenicity of aquatic products in xucheng district[J]. Chinese Modern Chinese and Western Medicine Magazine, 2005, 10(1): 1.
[24] 梁健, 高山, 李永仁, 等. 方斑东风螺肿吻病致病菌分离及中草药对其的预防效果[J]. 江苏农业科学, 2016, 44(11): 267-269.
GAO Jian, GAO Shan, LI Yong-ren, et al. The Pathogens was isolated from disease of Babylonia areolate and the prevention of Chinese herbal medicine on pathogens[J]. Jiangsu Agricultural Sciences, 2016, 44(11): 267-269.
[25] 慕艳娟, 许崛琼, 潘子豪, 等. 以toxR-S为靶基因的副溶血弧菌PCR检测方法的建立[J]. 中国兽医科学, 2015, 45(10): 991-999.
MU Yan-juan, XU Jue-qiong, PAN Zi-hao, et al. Development of a PCR for detection ofVibrioparahaemolyticusbased on toxR-S gene sequence[J]. Veterinary Science in China, 2015, 45(10): 991-999.
[26] 王华丽, 史贤明, 杨官品. 海产品中副溶血弧菌PCR检测方法的建立与评价[J]. 农业生物技术学报, 2006, 14(4): 627-628.
WANG Hua-li, SHI Xian-ming, YANG Guan-pin Development and evaluation of PCR method for the detection ofVibrioparahaemolyticusin seafoods[J]. Journal of Agricultural Biotechnology, 2006, 14(4): 627-628.
[27] 姚东瑞, 朱明, 陈丽, 等. 基于toxR基因病原哈氏弧菌PCR检测方法的建立[J]. 水产科学, 2011, 30(9): 559-562.
YAO Rui-dong, ZHU Ming, CHEN Li, et al. Development of PCR method for detection of pathogenic bateriumVibrioharveyi based on toxR gene[J]. Fishing Science, 2011, 30(9): 559-562.
[28] 陈昌国, 侯兵兵, 陈秋圆, 等. Taqman-探针荧光定量PCR鉴定溶藻弧菌方法的建立[J]. 实用检验医师杂志, 2017, 9(1): 1-4.
CHEN Chang-guo, HOU Bingbing, CHEN Qiu-yuan, et al. Establishment of a method for the identification ofVibrioalginolyticusby Taqman—probe fluorescence quantitative PCR analysis[J]. Chinese Journal of Laboratory Pathologist, 2017, 9(1): 1-4.
[29] 曹润冬, 王睿, 林立超. 副溶血性弧菌国家标准检测方法与快速检测方法的对比研究[J]. 中国卫生检验杂志, 2017(6): 811-813.
CAO Run-dong, WANG Rui, LIN Li-chao. Comparison between the national standard test and rapid test forVibrioparahaemolyticus[J]. Chinese Journal of Health Laboratory Technology, 2017(6): 811-813.
